外显子组测序技术对疑似鱼鳞病患儿进行分子诊断的方法学研究
2015-11-29余蕾张蕾顾峻嫣程明亮黄林夏曙华程树强简单王荔平孙朝琴王焱杨国珍
余蕾 张蕾 顾峻嫣 程明亮 黄林 夏曙华 程树强 简单 王荔平 孙朝琴 王焱 杨国珍
·技术与方法·
外显子组测序技术对疑似鱼鳞病患儿进行分子诊断的方法学研究
余蕾 张蕾 顾峻嫣 程明亮 黄林 夏曙华 程树强 简单 王荔平 孙朝琴 王焱 杨国珍
外显子组测序是利用序列捕获技术将全基因组外显子区域DNA捕捉并富集后进行高通量测序的基因组分析方法,由于其具有对常见和罕见变异的高灵敏度,能发现外显子区绝大部分疾病相关变异以及仅需对约1%的基因组进行测序等优点,使外显子组测序成为鉴定孟德尔遗传病致病基因最有效的策略,也被运用于复杂易感基因的研究和临床诊断中[1]。本研究针对1例临床上疑似鱼鳞病的患儿,通过外显子组测序实验,检测该患儿在遗传水平上携带的突变,为临床诊断和治疗提供相关信息,并探讨利用外显子组测序作为临床疑难杂症辅助治疗手段的可能性。
一、对象
患儿男,出生于2013年8月1日(剖宫产出生,其母产前5 d入院检查持续尿蛋白+++,血清白蛋白较低,输血浆不能纠正),体重2.4 kg,发育基本正常,全身皮肤硬,无弹性,火红焦化状脱皮;头发呈卷曲碾碎感。显微镜下见头发呈竹节状,临床诊断为火棉胶婴儿(图1)。除此症状之外,无其他异常,从外观上看怀疑为鱼鳞病。染色体和染色体微缺失检查正常。父母体健,否认近亲结婚,家族中未见类似患儿。
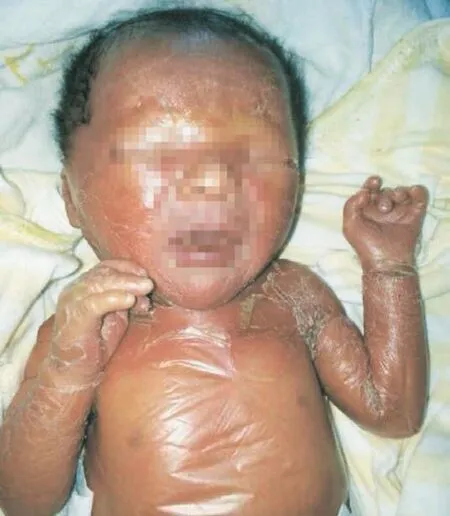
图1 疑似鱼鳞病患儿全身皮肤硬,无弹性,火红焦化状脱皮;头发呈卷曲碾碎感
二、方法
1.试剂与仪器:QIAamp DNA Blood Mini Kit(德国Qiagen公司;货号:51104),Gnomegen DNA-Seq Library Preparation Kit(美国Gnomegen公司;货号:K02422),Gnome size selector(美国 Gnomegen公司;货号:R02424L),PCR 仪(美国 ABI公司;型号:2720),Concentrator plus真空离心浓缩仪(德国eppendorf公司),小型高速离心机(德国eppendorf公司,型号:5418),实时定量PCR仪(德国eppendorf公司,型号:4376600),通用电泳仪(杭州汇尔仪器,型号:JY300C),涡旋仪(北京京辉凯业科技有限公司;型号:JHX24H),金属浴(杭州瑞诚仪器有限公司;型号:DH100-1)。
2.血液提取及基因组DNA的纯化、定量:取患儿EDTA抗凝全血2~3ml,提取DNA,DNA的纯化采用QIAamp DNA Blood Mini Kit,按试剂盒说明书操作。电泳检测基因组DNA的完整性。
3.外显子捕获:液相杂交法进行目标基因筛选:
(1)文库制备:文库构建采用Gnomegen DNA-Seq Library Preparation Kit,首先利用Covaris专利的自适应聚焦声波(Adaptive Focused Acoustics,AFA)技术将样品基因组DNA进行随机打断,获得片段化DNA,断裂要求:300~500 bp片段,以400 bp为中心峰值;然后对片段化的DNA进行末端修复和加dA尾,经过磁珠纯化之后,在纯化的DNA样品中加入连接接头,再次进行磁珠纯化,然后对带接头的纯化DNA样品进行PCR扩增,对PCR产物进行磁珠纯化后,制成300~500 bp片段文库,-20℃保存备用。
(2)液相杂交、捕获与洗脱:人工合成探针(图2B),首先将扩增的DNA文库样品与制备好的探针Nimblegen SeqCap EZ Human Exome Library v3.0(带生物素)加入适当的液相杂交体系中,经过47℃,72 h温浴,杂交完成。然后用链霉亲和素磁珠对杂交产物进行序列捕获,将目标DNA(带生物素)绑定到磁珠上。之后用3种洗脱缓冲液对磁珠进行洗脱,洗脱后的目标DNA溶液-20℃保存。
(3)筛选后文库的扩增及纯化:以洗脱后的目标DNA溶液为模板进行连接介导的PCR(Ligation.Mediated PCR,简称LM-PCR)扩增,对捕获的目标基因进行富集。PCR产物经过Gnome size selector磁珠纯化后,-20℃保存。
(4)qPCR法鉴定捕获效率:经过捕获的DNA,一般绝对数量较低,需要经过另一轮扩增才能产生用于高通量测序的样品。将捕获前后的DNA文库通过内部参照序列的qPCR分析,可以判断捕获的效率及专一性。以PCR水为空白对照模板,扩增的DNA文库为阳性对照模板,以扩增后的捕获DNA为实验组模板,选用4对引物进行qPCR SYBR Green实验,通过比较捕获组(CAP)和未捕获组(NON)的Ct值来鉴定捕获效率。
4.高通量测序:将捕获富集并验证的DNA文库采用Illumina HiSeq 2000平台测序,读长为2×100 bp双向测序,测序深度为>100×。
5.生物信息学分析:对于测序原始数据,首先采用fastqc软件对数据质量做质控,然后采用bwa软件与参考基因组hg19作比对,将比对结果去除重复片段并排序,接着采用GATK Indelrealigner对结果做重比对,然后采用GATK UnifiedGenotyper做变异检测,得到snp和indel的信息,并过滤snp和indel得到可信的snp和indel。可信的snp和indel被用来做后续的annovar、sift和polyphen注释,最后搜索OMIM数据库,查看样本疾病携带信息,并采用在线版HGMD 数据库做了注释[2-6]。
6.PCR扩增和sanger测序:再次以该患儿的基因组DNA为模板,针对检测到的突变位点,采用PCR扩增的方法,对位点附近序列进行扩增,引物序列为正向引物:5′-AGCTCAATGTAGCCTTCATGA-3′,反向引物:5′-TGGCTAAC TCCCACATAATGAA-3′。反应条件为:94 ℃ 5min,94 ℃ 30 s,58℃1min,72℃30 s,30个循环,再72℃7min。对扩增所得PCR产物回收、纯化之后,进行sanger测序。

图2 液相杂交两种探针的设计图 2A:普通探针设计使得目标序列每个位点的覆盖度为1~2×,且探针密度不够高,导致有些区域的突变无法捕获;2B:本研究设计的探针使得目标区域的每个位点都被多重覆盖,且探针密度非常高,不同位置的突变都可以被捕获
三、结果
对不明病因患儿的外显子组测序的流程包括样品收集及DNA提取,文库制备和目标基因筛选,测序及数据分析3个模块。
1.基因组DNA纯化:针对血液样本进行基因组DNA纯化,电泳检测结果显示,DNA的完整性良好。Qubit定量结果显示,该基因组DNA的质量浓度为130 mg/L,符合文库构建的标准。
2.文库构建和目标基因筛选:对纯化好的基因组DNA进行文库构建和目标基因筛选,对目标基因筛选后文库进行qRT-PCR质检,通常认为捕获前后参照序列的富集倍数(2△Ct)超过30倍,即△Ct大于5时,便可以进入下一步高通量测序。结果(表1)显示,血液样品4对引物的△Ct值均大于7,表示目标序列均富集了135倍以上。
3.高通量测序及数据分析:对捕获后的文库进行上机测序和数据分析,结果显示,该DNA样品捕获的目标区域大小为64 Mbp,符合人基因组外显子区的大小。目标区域的测序深度(即测序得到的碱基总量与目标区域大小的比值)为106×,通常认为当测序深度在10~15×以上时,目标区域覆盖度和测序错误率控制均得以保证。序列匹配时,发现有超过99.3%的高质量读长被匹配到人类基因组参考序列(hg19)上,总共有超过98.6%的外显子区域序列被覆盖了4倍以上,超过97.4%的外显子区域序列被覆盖了10倍以上,超过95.0%的外显子区域序列被覆盖了20倍以上,超过90.5%的外显子区域序列被覆盖了30倍以上,超过83.8%的外显子区域序列被覆盖了40倍以上,超过75.9%的外显子区域序列被覆盖了50倍以上。
对该患儿的外显子组测序共发现了172 639个SNP位点,20 434个插入/缺失位点,其中新的SNP位点为18 825个,新的Indels为6 890个。通过对这些突变位点进行致病性分析以及可能导致疾病的推测,共发现19个可能的致病突变(表2),其中位于第5条染色体上的SPINK5基因的第5外显子保守区序列位点发生了非同义突变c.318G>A,导致了氨基酸的改变Asp-Asn,从而可能引起内瑟顿综合征(Netherton syndrome)的发生。
内瑟顿综合征的临床特征为鱼鳞病样红皮病、发干异常和特应性素质,出生时即出现红斑,这些症状与本研究的患儿临床症状类似。将临床症状结合基因检测结果,初步确定该患儿为携带SPINK5基因杂合突变G318A的内瑟顿综合征患者。
4.序列验证:经过PCR扩增,得到了380 bp的目的片段,电泳结果见图3。对目的片段进行回收、纯化、一代测序,结果显示,该患儿的SPINK5基因确实存在G318A的杂合突变(图4)。
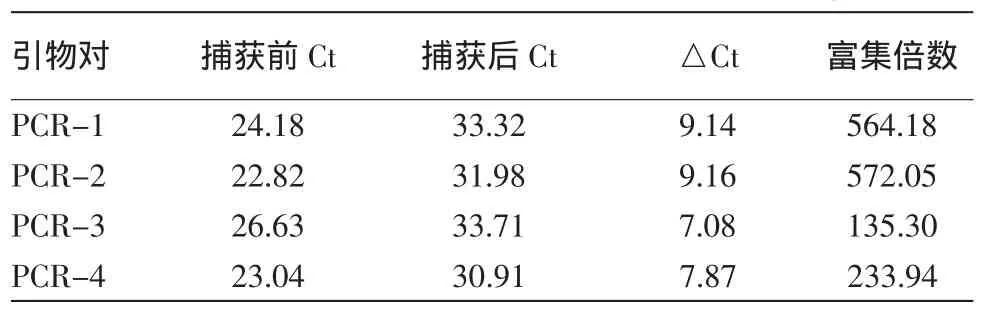
表1 捕获后血液基因组DNA文库的qRT-PCR QC结果
四、讨论
2010年,Bilgüvar等[7]运用 NimbleGen 2.1M 芯片捕获外显子,并用Genome AnalyzerⅡ测序系统对1例患者测序,发现WDR62基因与脑皮质发育异常疾病相关,从而能解释一系列的严重皮质畸形的症状,该研究提示,外显子测序对具有遗传异质性和诊断亚型困难的疾病有一定帮助。2011年,Worthey等[8]对1例临床上难以诊断疾病的患儿进行全外显子组测序,发现XIAP(X染色体的细胞凋亡抑制剂)基因上存在非同义突变,对基因功能和涉及代谢通路进行分析,结合患儿的临床症状,最终确定该患儿所得疾病为难治性炎性肠病。
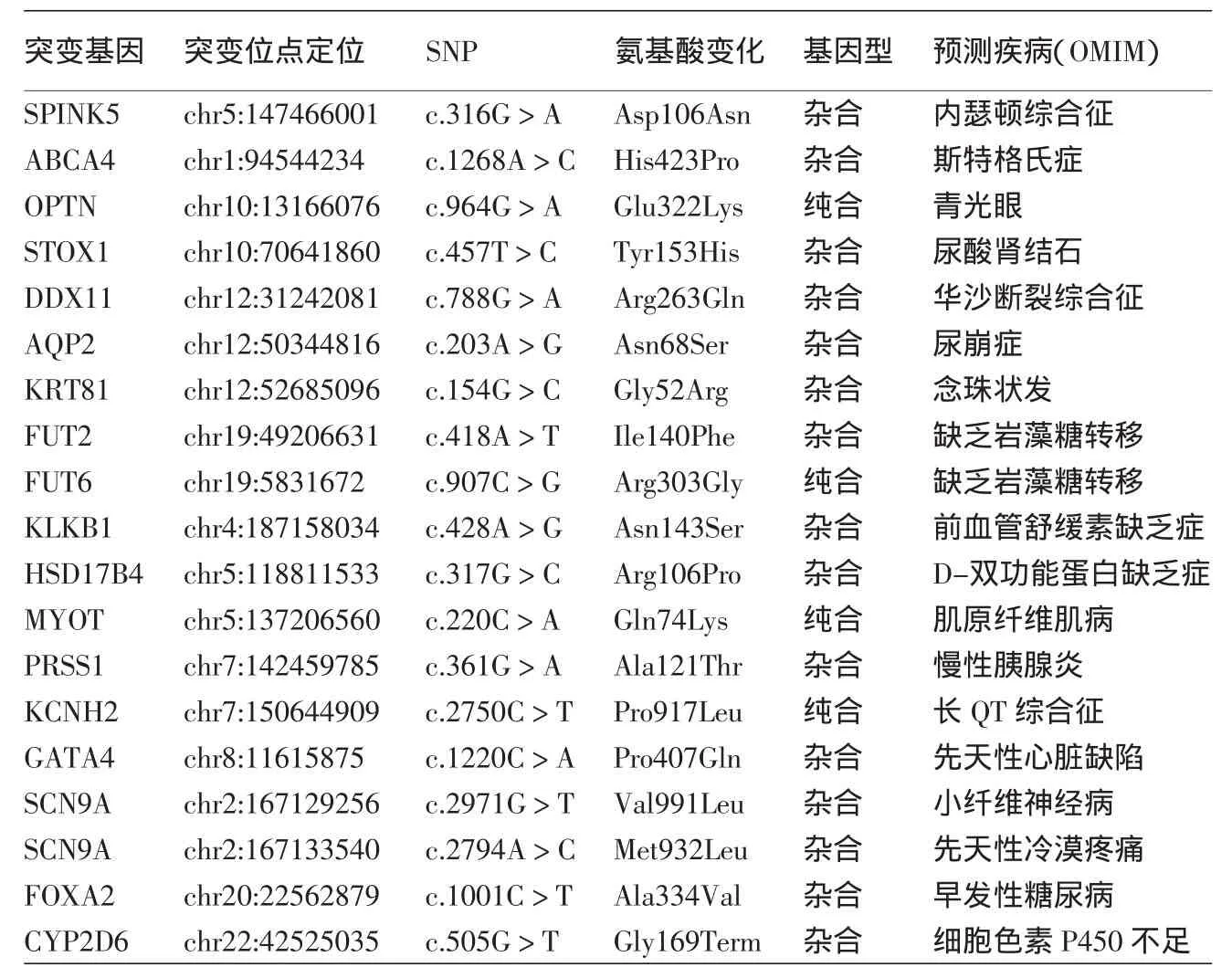
表2 疑似鱼鳞病患儿的致病突变检测结果
本研究针对1例临床上无法准确判断其病症的疑似鱼鳞病患儿,采用外显子组测序技术,对患儿的血液样本进行目标基因筛选、高通量测序及数据分析。外显子组测序技术的难点在于探针的设计,要求探针设计在维持专一性的同时,能有效捕获携带突变的片段。我们采用的是下面这种很多探针位置交错,大量起始点不同的探针去杂交捕获(由于探针是根据野生型正常序列设计,有突变序列的杂交效率低于正常序列,这种探针设计可以有效弥补这一缺陷)(图2B),可以高效地捕获发生变异的目标基因片段,定制过程中我们充分考虑了探针的密度和数量,并对杂交条件进行优化,最大可能发现更多的基因变异信息。
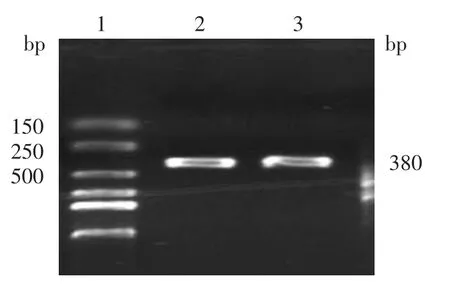
图3 SPINK5(G318A)突变位点序列PCR扩增电泳结果 目的片段为380 bp

图4 SPINK5(G318A)突变位点序列的sanger测序图谱 对目的片段进行一代测序,发现SPINK5(G318A)确实存在G318A的杂合突变
外显子测序的结果显示,64 Mbp的目标基因长度(符合人外显子的大小)、106×的测序深度、99.3%的目标区域覆盖度以及有超过90.5%的目标区域被覆盖了30倍以上,已有研究表明,30倍以上的覆盖度就足够发现低频的SNP、插入/缺失等位点[9-16],因此这样的测序深度和覆盖度足够发现发生频率很低的突变位点,可以达到预期的目的。通过对测序所得的SNP位点和Indels进行基因功能注释和致病性分析,发现一个已被确定的可以导致内瑟顿综合征的致病突变:SPINK5(G318A)。
内瑟顿综合征是由SPINK5(LEKTI)(皮肤激肽释放酶级联的一种主要抑制剂)中的突变造成的罕有常染色体隐性遗传性皮肤病,丝氨酸蛋白酶激肽释放酶(KLK)活性增加已经被证明是其临床症状的成因[17]。有文献证明,患有内瑟顿综合征的患者在编码LEKTI的SPINK5基因中出现了阅读框偏移和无义突变[18-20],LEKTI为具有对抗多种 KLK(包括KLK5、6、7、13 和 14)的活性丝氨酸蛋白酶抑制剂[21-22],这类基因缺陷会导致抑制结构域的缺失,使在正常的皮肤生理学中起到关键作用的激肽释放酶KLK蛋白活性增加,从而引起内瑟顿综合征的发生。Chavanas等[23]总结了导致内瑟顿综合征发生的SPINK5基因突变有13个。2012年,Zhang等[24]在1例中国成年内瑟顿综合征的患者中发现了SPINK5基因的杂合突变G318A,并通过免疫组化技术检测到拥有该突变的个体LEKTI的表达量明显降低,从而确定SPINK5基因的杂合突变G318A为一新的导致内瑟顿综合征发生的致病突变。从该疾病的致病机理可以推断出,给内瑟顿综合征患者补充丝氨酸蛋白酶抑制剂,理论上可以有效控制病症,具体应用需要进一步的功能验证和临床试验。
利用目标基因筛选技术,将全基因组外显子区域的序列作为靶向基因,从基因组DNA中筛选出来并进行测序和分析,既可以大幅度降低测序成本,又能增加测序深度和检测灵敏度,更重要的是,目前一致认为大部分功能变异都潜藏在外显子中[25],有机会发现更多有价值的突变,因此全外显子组测序的技术已成为现阶段基因测序工作的重心。Biesecker等[26]在 Nature Genetics 发表评论:外显子组测序使得医学基因组学成为现实。我们的研究进一步证实了外显子组测序技术作为临床难以诊断疾病的辅助诊疗手段的可能性。相信随着技术的发展、测序成本的降低以及后续分析手段的丰富,预期在不久的将来,外显子测序会广泛被运用到疾病的分子诊断,为临床一些难以诊断的疾病提供一种新的诊断思路。
[1]张鑫,李敏,张学军.全基因组外显子测序及其应用[J].遗传,2011,33(8):847-856.
[2]Li H,Durbin R.Fast and accurate long-read alignment with Burrows-Wheeler transform[J].Bioinformatics,2010,26(5):589-595.
[3]McKenna A,Hanna M,Banks E,et al.The Genome Analysis Toolkit:a MapReduce framework for analyzing next-generation DNA sequencing data[J].Genome Res,2010,20(9):1297-1303.
[4]DePristo MA,Banks E,Poplin R,et al.A framework for variation discovery and genotyping using next-generation DNA sequencing data[J].Nat Genet,2011,43(5):491-498.
[5]Wang K,Li M,Hakonarson H.ANNOVAR:functional annotation of genetic variants from high-throughput sequencing data[J].Nucleic Acids Res,2010,38(16):e164.
[6]Li H,Handsaker B,Wysoker A,et al.The Sequence Alignment/Map format and SAMtools[J].Bioinformatics,2009,25(16):2078-2079.
[7] Bilgüvar K,Oztürk AK,Louvi A,et al.Whole-exome sequencing identifies recessive WDR62 mutations in severe brain malformations[J].Nature,2010,467(7312):207-210.
[8]Worthey EA,Mayer AN,Syverson GD,et al.Making a definitive diagnosis:successfulclinicalapplication ofwhole exome sequencing in a child with intractable inflammatory bowel disease[J].Genet Med,2011,13(3):255-262.
[9]Gnirke A,Melnikov A,Maguire J,et al.Solution hybrid selection with ultra-long oligonucleotides for massively parallel targeted sequencing[J].Nat Biotechnol,2009,27(2):182-189.
[10]Ng SB,Turner EH,Robertson PD,et al.Targeted capture and massively parallel sequencing of 12 human exomes [J].Nature,2009,461(7261):272-276.
[11]Hedges DJ,Burges D,Powell E,et al.Exome sequencing of a multigenerational human pedigree [J].PLoS One,2009,4(12):e8232.
[12]Ng SB,Buckingham KJ,Lee C,et al.Exome sequencing identifies the cause of a mendelian disorder[J].Nat Genet,2010,42(1):30-35.
[13]Kim JI,Ju YS,Park H,et al.A highly annotated whole-genome sequence of a Korean individual[J].Nature,2009,460(7258):1011-1015.
[14]Baranzini SE,Mudge J,van Velkinburgh JC,et al.Genome,epigenome and RNA sequences of monozygotic twins discordant for multiple sclerosis[J].Nature,2010,464(7293):1351-1356.
[15]Roach JC,Glusman G,Smit AF,et al.Analysis of genetic inheritance in a family quartet by whole-genome sequencing[J].Science,2010,328(5978):636-639.
[16]Tewhey R,Warner JB,Nakano M,et al.Microdroplet-based PCR enrichment for large-scale targeted sequencing[J].Nat Biotechnol,2009,27(11):1025-1031.
[17]Geyer AS,Ratajczak P,Pol-Rodriguez M,et al.由SPINK5中的一个纯合子的移码突变产生的具有广泛脱屑及发育停滞的内瑟顿综合征[J].世界核心医学期刊文摘(皮肤病学分册),2005,(10):55.
[18]Chavanas S,Bodemer C,Rochat A,et al.Mutations in SPINK5,encoding a serine protease inhibitor,cause Netherton syndrome[J].Nat Genet,2000,25(2):141-142.
[19]Komatsu N,Takata M,Otsuki N,et al.Elevated stratum corneum hydrolytic activity in Netherton syndrome suggests an inhibitory regulation of desquamation by SPINK5-derived peptides [J].J Invest Dermatol,2002,118(3):436-443.
[20]Sprecher E,Chavanas S,DiGiovanna JJ,et al.The spectrum of pathogenic mutations in SPINK5 in 19 families with Netherton syndrome:implications for mutation detection and first case of prenatal diagnosis[J].J Invest Dermatol,2001,117(2):179-187.
[21] Borgoño CA,Michael IP,Komatsu N,et al.A potential role for multiple tissue kallikrein serine proteases in epidermal desquamation[J].J Biol Chem,2007,282(6):3640-3652.
[22]Egelrud T,Brattsand M,Kreutzmann P,et al.hK5 and hK7,two serine proteinases abundant in human skin,are inhibited by LEKTI domain 6[J].Br J Dermatol,2005,153(6):1200-1203.
[23]Chavanas S,Bodemer C,Rochat A,et al.Mutations in SPINK5,encoding a serine protease inhibitor,cause Netherton syndrome[J].Nat Genet,2000,25(2):141-142.
[24]Xi-Bao Z,San-Quan Z,Yu-Qing H,et al.Netherton syndrome in one Chinese adult with a novel mutation in the SPINK5 gene and immunohistochemical studies of LEKTI [J].Indian J Dermatol,2012,57(4):265-268.
[25]Botstein D,Risch N.Discovering genotypes underlying human phenotypes:pastsuccesses formendelian disease,future approaches for complex disease [J].Nat Genet,2003,33 Suppl:228-237.
[26]Biesecker LG.Exome sequencing makes medical genomics a reality[J].Nat Genet,2010,42(1):13-14.
2014-03-27)
(本文编辑:周良佳 颜艳)
10.3760/cma.j.issn.0412-4030.2015.03.019
550004贵阳医学院附属医院临检科遗传室(余蕾、程明亮、黄林、夏曙华、程树强、简单、王荔平、孙朝琴、杨国珍);吴江汇杰生物科技有限公司(张蕾、顾峻嫣);美国Gnomegen公司(王焱)
杨国珍,Email:myy12004@163.com;王焱,Email:yan.wang@huijiebio.com
