链霉菌Streptomyces olivaceus FXJ7.023多功能葡糖淀粉酶SoGA的克隆、表达及鉴定
2015-10-26王苗李园园岳昌武邵美云吕玉红保玉心黄英
王苗李园园岳昌武邵美云吕玉红保玉心黄英
(1.遵义医学院医学与生物学研究中心 贵州省普通高校微生物资源及药物开发特色重点实验室,遵义 563003;2.中国科学院微生物研究所,北京 100101)
链霉菌Streptomyces olivaceus FXJ7.023多功能葡糖淀粉酶SoGA的克隆、表达及鉴定
王苗1李园园1岳昌武1邵美云1吕玉红1保玉心1黄英2
(1.遵义医学院医学与生物学研究中心 贵州省普通高校微生物资源及药物开发特色重点实验室,遵义 563003;2.中国科学院微生物研究所,北京 100101)
根据海洋来源链霉菌Streptomyces olivaceus FXJ7.023基因组测序结果设计特异引物,通过PCR扩增获得1条全长为1 788 bp的糖苷水解酶15家族蛋白新成员完全编码区DNA片段,该片段编码1个595个氨基酸残基、分子量为66.2 kD的预测蛋白。利用基因工程技术将该片段重组入原核表达质粒pET32a并转化宿主菌BL21(DE3)plySs,IPTG诱导融合蛋白表达,表达的包涵体融合蛋白经纯化、复性后利用DNS法测定其在不同温度、pH条件下催化不同底物产生还原糖的活性。结果表明,该酶能够水解纤维素、淀粉等多种底物产生还原糖活性,且对不同底物表现不同的最适pH和最适反应温度。
链霉菌;葡糖淀粉酶;基因克隆;蛋白表达;酶活
葡糖淀粉酶(Glucoamylase,GA)是一大类能够从淀粉等底物分子的非还原端开始,逐步水解底物中的α-1,4糖苷键和α-1,6糖苷键产生葡萄糖的糖苷水解酶第15超家族(http://www.cazy. org/GH15.html)的酶类(EC 3.2.1.3)。葡糖淀粉酶的天然底物主要是各种来源的淀粉,包括直链淀粉、支链淀粉和各种糊精等,葡糖淀粉酶底物专一性较低,既能从淀粉链的非还原性末端切开α-1,4糖苷键,也能缓慢切开α-1,6糖苷键,将直链淀粉从非还原性末端依次切下葡萄糖单位,当底物为支链淀粉,遇到α-1,6糖苷键分支时,先切割α-1,6糖苷键,再切割α-1,4糖苷键,从而使支链淀粉水解成葡萄糖。在工业上由于葡糖淀粉酶主要用于水解淀粉来生产葡萄糖,因而广泛应用于酒精、淀粉糖、味精、柠檬酸、啤酒、白酒、黄酒等食品工业生产[1-8]、农产品加工[9-11]以及抗菌素医药工业等[12,13]。早期工业上应用的葡糖淀粉酶主要来自于根霉、黑曲霉、红曲霉等霉菌[1,14-20],随着生物技术的发展以及工业需求的变化,当前对于葡糖淀粉酶的研究主要集中在新型酶制剂的开发、高耐受性酶筛选及优良基因工程菌株的构建等方面,研究者对多种微生物来源的淀粉水解酶进行了筛选研究,以期获得具有良好工业利用潜力的葡糖淀粉酶[21]。迄今,碳水化合物活性酶数据库(carbohydrate-active enzymes,http://www.cazy.org)已收录1 491种不同来源的葡糖淀粉酶数据信息,其中古菌来源202种,细菌来源1 090种,真核生物来源186种,未知来源13种。在这1 000多种葡糖淀粉酶中,有57种酶的功能已得到验证,其中包括1种链霉菌Streptomyces hygroscopicus subsp. limoneus来源的葡糖淀粉酶VLdI[22]。
本研究拟利用基因工程技术克隆并原核表达链霉菌Streptomyces olivaceus FXJ7.023基因组中预测的1条葡糖淀粉酶基因,通过DNS法检测该重组蛋白在不同条件下催化不同底物的生物活性,初步评估其应用潜力,旨在为进一步开发利用该酶提供试验依据,给农产品加工、食品及医药工业生产等提供新的水解碳水化合物产生葡萄糖的葡糖淀粉酶来源。
1 材料与方法
1.1 材料
链霉菌Streptomyces olivaceus FXJ7.023(CGMCC 4.7054)由中科院微生物所黄英课题组分离自1 180 m深的南海海泥,大肠杆菌基因工程菌JM109、BL21(DE3)pLysS感受态细胞及高纯质粒提取试剂盒等购自博迈德(北京)生物科技公司;基因克隆载体pMD18-T simple 购自宝生物(大连)公司;rTaq等PCR反应试剂、限制性内切酶、T4 DNA连接酶、去磷酸化酶等分子克隆所用酶类购自富酶泰斯(深圳)生物科技公司;培养基、蛋白质表达纯化等生化药品如无特殊说明均购自碧迪医疗(上海)有限公司;DNA凝胶回收试剂盒、PCR产物回收试剂盒等购自爱思进(杭州)生物技术有限科技公司;引物合成及测序由英潍捷基(广州)有限公司完成。葡萄糖、3,5-二硝基水杨酸、 氢氧化钠、甲基纤维素钠、直链淀粉、支链淀粉及可溶性淀粉等酶活测定试剂或标准品购自阿拉丁(上海)试剂公司。
1.2 方法
1.2.1 目的基因克隆及原核表达质粒载体构建 利用primer5.0等生物软件,根据课题组前期链霉菌Streptomyces olivaceus FXJ7.023基因组测序结果(GenBank登录号APIV00000000.1)设计SoGA编码基因的完全编码区扩增引物:上游引物sogaEF:5'-CCGGAATTCATGCACCCCCGTATCGAGGA-3',下游引物sogaER:5'-CCCAAGCTTCTATCCTGCATCCTCTTCCCCG-3'(下划线分别为限制酶EcoR I和Hind III识别位点)。利用热酚法提取Streptomyces olivaceus FXJ7.023基因组DNA,加入无DNase污染的RNaseA去除RNA后,经1%琼脂糖凝胶电泳和分光光度法检测DNA质量合格后用于PCR模板进行目的基因PCR扩增,PCR体系如下:rTaq 2 U,sogaEF 20 pmol,sogaER 20 pmol,DMSO 3.0 μL,dNTPmix(10 nmol/L each)1.5 μL,模板DNA 100 ng,10×PCR buffer 5.0 μL,去离子水补足至50 μL。PCR扩增程序:95℃预变性3 min;95℃ 30 s,56℃ 30 s,72℃ 90 s,共30个循环;72℃总延伸5 min。PCR扩增产物经1%琼脂糖凝胶电泳检测,利用PCR产物纯化试剂盒纯化目的片段。取300 ng纯化的PCR产物和50 ng的pMD18-T simple 室温(约20℃)连接3 h,转化大肠杆菌JM109感受态细胞。37℃,200 r/min振荡培养1 h,取200 μL转化产物涂布于含终浓度为100 ng/μL氨苄西林的LB固体平板,37℃倒置培养过夜后,无菌牙签挑取转化子置于600 μL含终浓度为100 ng/μL氨苄西林的LB液体培养基中220 r/min,37℃培养3 h,取1 μL培养物与20 μL PCR体系中进行菌液PCR扩增,筛选重组子。菌液PCR体系如下:rTaq 0.8 U,正向引物M13-47F(5'-CGCCAGGGTTTTCCCAGTCACGAC-3')10 pmol,反向引物M13-48R(5'-AGCGGATAACAATTTCACACAGGA-3')10 pmol,DMSO 1.0 μL,dNT-Pmix(10 mmol/L each)0.75 μL,菌液1 μL,10×PCR bu-ffer 2.0 μL,去离子水补足至20 μL。菌液PCR扩增程序:95℃预变性3 min;95℃ 30 s,56℃ 30 s,72℃ 90 s,共30个循环;72℃总延伸5 min。PCR扩增产物经1%琼脂糖凝胶电泳检测,根据长度初步判断是否为阳性重组子。取500 μL经菌液PCR筛选、鉴定后的重组子菌液,高纯质粒提取试剂盒法提取质粒DNA并利用EcoR I/Hind III限制酶双酶切鉴定。经酶切进一步鉴定的重组子由英潍捷基有限公司完成测序并利用BLAST软件比对以确定克隆得到DNA片段是否为目的基因,鉴定后的阳性重组子命名为pMD-soga。利用EcoR I/Hind III限制酶双酶切2 μg的 pMD-soga质粒DNA,DNA凝胶回收试剂盒回收目的DNA片段。取500 ng回收的双酶切目的DNA片段和100 ng经EcoR I/Hind III限制酶双酶切并去磷酸化的pET32a质粒DNA,T4 DNA连接酶室温连接3 h,连接产物转化大肠杆菌JM109感受态细胞。转化子筛选、重组子鉴定流程除菌液PCR引物使用基因特异引物外与前述pMD-soga质粒鉴定完全一致,经测序验证后的原核表达质粒载体命名为pET-soga。
1.2.2 目的蛋白诱导表达 分别取100 ng pET-soga质粒和pET32a质粒,转化大肠杆菌BL21(DE3)pLysS感受态细胞,转化子经过夜培养、T7启动子引物(5'-TAATACGACTCACTATAGGG-3')/T7终止子引物(5'-TGCTAGTTATTGCTCAGCGG-3')菌液PCR鉴定后,将含重组质粒的基因工程菌分别命名为pET-soga BL21和pET32aBL21。取前述2种基因工程菌各50 μL,分别接种于5 mL含终浓度为100 ng/μL氨苄西林的LB液体培养基中220 r/min,37℃培养3 h后分别接种100 mL含终浓度为100 ng/μL氨苄西林的LB液体培养基中220 r/min,37℃培养至OD600=0.3时,取5 mL上述培养物分装至50 mL无菌离心管,加入终浓度为0、0.5、1.0和2.0 mmol/L的IPTG,分别置于18℃、28℃和37℃诱导6 h。取1 mL诱导后培养物,4 000 r/min离心5 min,菌体沉淀加入100 μL的5×SDS-PAGE电泳上样缓冲液,置于99℃金属浴中裂解10 min,12 000 r/min,室温离心10 min,上清即为菌体总蛋白裂解液。分别取10 μL总蛋白裂解液上样,10%的SDS-PAGE凝胶电泳。考马斯亮蓝R250染色6 h后,甲醇-冰醋酸脱色液脱色6 h,判断蛋白质表达情况。根据目的蛋白表达情况,选取250 mL三角瓶中加入100 mL含终浓度为100 ng/μL氨苄西林的LB液体培养基,OD600=0.3时加入1.0 mmol/L IPTG于37℃ 220 r/min振荡培养6 h为表达条件,大量诱导表达目的蛋白。
经SDS-PAGE鉴定表达目的蛋白的发酵物,4 000 r/min,4℃离心10 min,收集菌体,PBS(pH 7.6)漂洗3次后加入10 mL含终浓度为25 ng/mL溶菌酶、0.1%脱氧胆酸钠的PBS(pH7.6)重悬菌体,于超声裂解仪冰浴条件下超声破碎细菌。取100 μL裂解液,12 000 r/min,4℃离心10 min,上清加入500 μL冰冷丙酮后置冰上10 min后12 000 r/min,4℃离心10 min。菌体超声裂解物沉淀加入50 μL的5×SDS-PAGE电泳上样缓冲液,置于99℃金属浴中裂解10 min,12 000 r/min,室温离心10 min,分别取10 μL总蛋白裂解液上样,10%的SDS-PAGE凝胶电泳判断蛋白质表达状态。
1.2.3 包涵体蛋白纯化与复性 取超声裂解后的总蛋白沉淀,加入15 mL预冷PBS(pH7.6)漂洗3次去除可溶蛋白后,每1.0 g湿蛋白加入100 mL冰冷的8 mol/L尿素4℃放置过夜重溶包涵体。待无肉眼可见悬浮物后,12 000 r/min,4℃离心10 min。取上清,置冰上缓慢加入冰冷的包涵体蛋白稀释缓冲液(含0.5 mol/L精氨酸,10%甘油的PBS缓冲液,pH7.6)至尿素终浓度为0.5 mol/L。将稀释后的包涵体蛋白转移至透析袋(截留分子量300 000),冰浴条件下用包涵体蛋白稀释缓冲液每4 h换取1次透析液,50 r/min连续振荡透析24 h去除尿素。最后以不含精氨酸的包涵体蛋白稀释缓冲液透析去除精氨酸。
1.2.4 酶活测定 根据葡糖淀粉酶催化活性,以单位时间内每克酶蛋白催化产生还原性糖的摩尔数作为该酶的活力单位(U)。采用DNS法[23]测定反应产生还原性糖量。具体操作为在350 μL反应体系中加入0.1 mg酶蛋白和相应的底物,pH7.0条件下,分别在40、45、50、55、60和65℃反应30 min后,加入150 μL DNS试剂沸水煮5 min,立即冰浴冷却至室温,分别检测其波长540 nm的光密度值,根据标准曲线计算相应的温度下酶活。该酶催化最适pH测定采取梯度pH法,即各自底物最适催化温度下,分别在pH4.5、5.0、5.5、6.0、6.5、7.0、7.5、8.0、8.5和9.0条件下反应30 min后,加入150 μL DNS试剂沸水煮5 min,立即冰浴冷却至室温,分别检测其在波长540 nm的光密度值,根据标准曲线计算相应的pH下酶活。
2 结果
2.1 soga基因克隆及序列分析
通过特异引物PCR扩增得到1条全长为1 788 bp目的片段,菌液PCR筛选结合EcoR I/Hind III限制酶双酶切鉴定该片段T/A克隆的阳性重组子测序NCBI在线BLAST结果表明,成功从海洋链霉菌Streptomyces oLivaceus FXJ7.023克隆到1条编码595个氨基酸残基、分子量为66.2 kD编码糖苷水解酶15家族蛋白新成员,命名为soga。预测的SoGA氨基酸序列与已鉴定功能的微生物来源的糖苷水解酶15家族葡糖淀粉酶进行系统进化分析,结果(图1)表明,与SoGA氨基酸序列相似性最高的葡糖淀粉酶是Streptomyces hygroscopicus subsp. limoneus来源的VLdI(相似性为58%),与真核微生物Blastobotrys adeninivorans来源的GaA的氨基酸序列相似性24%,与古菌Thermoplasma acidophilum来源的TaGA的氨基酸序列相似性仅为19%,表明该蛋白是一个较新的编码糖苷水解酶15家族蛋白。

图1 微生物来源已知功能葡糖淀粉酶系统进化分析
2.2 Trx-SoGA融合表达及纯化
SDS-PAGE结果(图2)表明,与未加IPTG诱导的基因工程菌pET-soga BL21总蛋白相比,加入1.0 mmol/L IPTG诱导的基因工程菌pET-sogaBL21总蛋白中出现1条明显的分子量约为80 kD新蛋白条带,与预期重组蛋白分子量大小一致,表明Trx-SoGA成功被诱导表达。进一步分析表明诱导表达菌裂解上清液中无该目的蛋白存在,表明该蛋白是以包涵体的形式表达。诱导表达的菌体沉淀超声裂解物经PBS漂洗多次后,可见乳白色沉淀且能完全溶解入8 mol/L尿素。包涵体尿素溶液经包涵体稀释液缓慢稀释到尿素终浓度为0.5 mol/L时,未见蛋白沉淀析出,表明包涵体通过稀释成功复性。将复性成功的Trx-SoGA透析去除残留尿素,SDS-PAGE复性效果表明,Trx-SoGA以可溶形式存在于透析后上清液中。
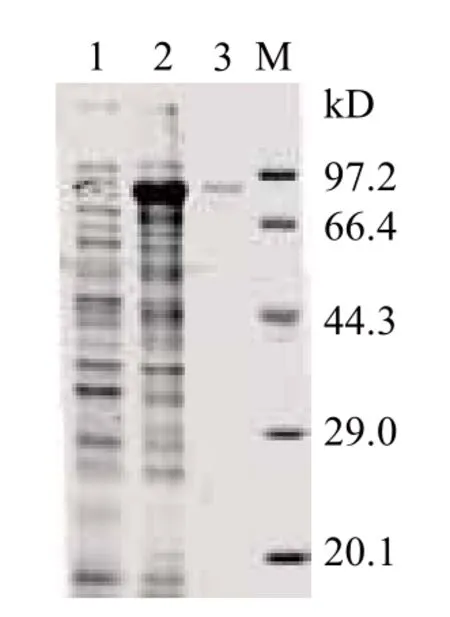
图2 重组蛋白Trx-SoGA表达及纯化效果分析
2.3 温度对Trx-SoGA酶活的影响
利用DNS法在pH7.0条件下,分别在不同温度下测定Trx-SoGA催化不同底物产生还原性糖的酶活,结果(图3)表明,Trx-SoGA催化底物羧甲基纤维素的酶活在40℃时达到最大值(1.36 mmol/g);以可溶性淀粉为底物时,Trx-SoGA酶活在40℃时达到最大值(1.95 mmol/g);而分别以直链淀粉和支链淀粉为底物时,该重组蛋白的最适反应温度则分别为55℃(酶活为2.03 mmol/g)和45℃(酶活为1.90 mmol/g)。
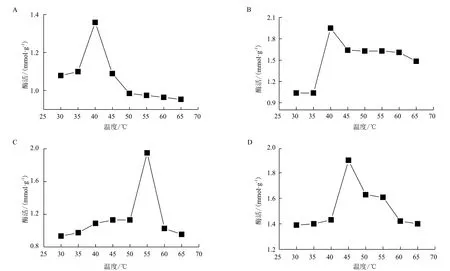
图3 温度对Trx-SoGA催化底物产生还原性糖酶活的影响
2.4 pH对Trx-SoGA酶活的影响
Trx-SoGA水解不同底物产生还原性糖酶活的结果(图4)表明,该蛋白水解羧甲基纤维素的最适pH为6.0(酶活为1.34 mmol/g),水解可溶性淀粉为最适pH为6.5(酶活为3.39 mmol/g),水解直链淀粉的最适pH为8.5(酶活为1.04 mmol/g),而催化支链淀粉的最适pH是8.0(酶活为1.59 mmol/g)。
3 讨论
工业上用于水解生产糖类的淀粉原料主要有小麦淀粉、玉米淀粉、大米淀粉及薯类淀粉等。利用淀粉水解产生葡萄糖的方法主要有酸水解法[24,25]、酶水解法[26,27]和酸酶结合水解法[28,29]。酸水解法以酸(无机酸或有机酸)为催化剂,在高温高压下将淀粉水解转化为葡萄糖的方法,虽然具有过程简单、产能大、时间短、技术含量低等优点,但存在要求设备耐腐蚀、耐高温、高压,淀粉利用率低以及淀粉原料要求严格(颗粒大小均匀等)、淀粉浓度不宜过高,且在酸水解过程中有副反应的发生,特别是产物直接用于食品和药品原料时存在安全隐患等缺点。而酶法则能很好地克服酸水解法的这些缺点,特别是条件温和、转化率高和产品安全,尽管存在耗时较长、后期加工流程长和设备多等缺点,但只要随着新型酶制剂的发现及酶解工艺的改进,酶法将会取代酸水解法成为工业制糖的主要方法。
农产品加工、食品及医药工业生产上应用的葡糖淀粉酶都是利用其热稳定性,目前研究者主要是通过霉菌、酵母等真菌中筛选热稳定的糖化酶生产菌株方法获得热稳定性好的葡糖淀粉酶[15,30],由于还没有理想的常温水解生淀粉的葡糖淀粉酶产品,真正得到应用的基因工程葡糖淀粉酶较少。一般情况下,糖化酶都具有较宽的pH适应范围,但最适pH多为4.5-6.0,不同来源的葡萄糖淀粉酶的糖化最适温度和pH值可能存在一定的差异。例如,黑曲霉为55-60℃,pH值3.5-5.0;根霉50-55℃,pH值4.5-5.5;拟内孢霉为50℃,pH值4.8-5.0。葡糖淀粉酶底物专一性较低,可水解直链淀粉、支链淀粉及糊精等产生还原性糖,但目前尚未见同时具有水解纤维素活性的葡糖淀粉酶的报道。虽然本研究的结果离工业应用可能还有一定的距离,但如果结合现代基因工程技术对该酶进行相应的突变等相关操作,进一步提高其催化活力和改善催化条件,可望在农产品加工、食品及医药等工业生产得以应用。
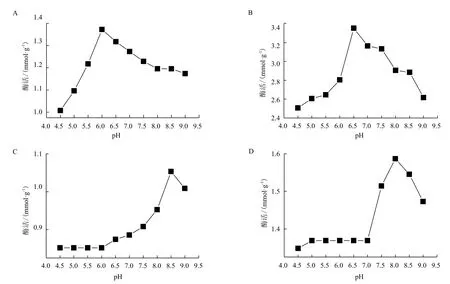
图4 pH对催化底物产生还原性糖酶活的影响
4 结论
本研究利用PCR技术从海洋来源的链霉菌Streptomyces olivaceus FXJ7.023基因组DNA中成功克隆1个预测编码葡糖淀粉酶基因soga,并在大肠杆菌中成功表达出分子量约为80 kD的重组葡糖淀粉酶Trx-SoGA,DNS法检测结果表明该酶可水解直链淀粉、支链淀粉及可溶性淀粉等不同来源淀粉及羧甲基纤维素产生还原性糖,且该酶对不同底物表现出较广的最适pH范围(pH6.0-8.5)和较低最适反应温度范围(40-55℃)。
[1] Maelida Y, Hassan SN, Radu SZ, et al. Purification and characterization of sago starch degrading glucoamylase from Acremonium sp. endophytic fungus [J]. Food Chemistry, 2000, 71(2):221-227.
[2] 贾可华, 马晓军.高温淀粉酶法制备大麦饮料的工艺研究[J].食品工业科技, 2013, 34(22):183-187, 191.
[3] 郭春玉.酶解米乳饮品的研制及其稳定性研究[J].食品工业,2013, 34(10):90-93.
[4] 赵红岩, 王娜.应用α-淀粉酶和糖化酶的协同作用提高葛根饮料稳定性和口感[J].食品与发酵工业, 2013, 39:125-127.
[5] 杨婧曦, 于海峰.酶法制备白果甜玉米澄清饮料的研究[J].食品科技, 2013, 8(9):76-78.
[6] 付艳丽, 周霞, 苏畅.荞麦黑米酒液化及糖化工艺的研究[J].食品工业, 2013, 34(5):100-103.
[7]Yang S, Li B, Zhang L, et al. Study on process conditions ofpreparation of microporous potatoes starch by complex enzyme method [J]. Chinese Food Science, 2012, 1(1):4-16, 19.
[8]Wang R, Wang D, Gao X, et al. Direct fermentation of raw starch using a Kluyveromyces marxianus strain that expresses glucoamylase and alpha-amylase to produce ethanol [J]. Biotechnol Prog, 2014,30(2):338-347.
[9]Liu K, Han J. Enzymatic method for measuring starch gelatinization in dry products in situ [J]. J Agric Food Chem, 2012, 60(17):4212-4221.
[10]Lee J, Seo E, Kweon DH, et al. Fermentation of rice bran and defatted rice bran for butanol 5 production using clostridium beijerinckii NCIMB 8052[J]. J Microbiol Biotechnol, 2009, 19(5):482-490.
[11] Attia RM, Ali SA. Utilization of agricultural wastes of Aspergillus awamori for the production of glucoamylase [J]. Zentralbl Bakteriol Parasitenkd Infektionskr Hyg, 1977, 132:322-325.
[12]谭瑶瑶, 吴亨, 古碧, 等.不同酶制备木薯抗性淀粉的性质比较[J].食品工业科技, 2013, 34(11):80-91.
[13]Ventura L, Ramón D. Transformation of Aspergillus terreus with the hygromycin B resistance marker from Escherichia coli [J]. FEMS MicrobioL Lett, 1991, 66(2):189-193.
[14]彭微, 杨雪娟, 张军.类食品乳杆菌412发酵酸面团中风味物质分析[J].食品工业科技, 2013, 34(14):126-130.
[15]Bagheri A, Khodarahmi R, Mostafaie A. Purification and biochemical characterisation of glucoamylase from a newly isolated Aspergillus niger:Relation to starch processing [J]. Food Chem,2014, 161:270-278.
[16]Duran RM, Gregersen S, Smith TD, et al. The role of Aspergillus flavus veA in the production of extracellular proteins during growth on starch substrates [J]. Appl Microbiol Biotechnol, 2014, 98(11):5081-5094.
[17]刘波, 邬应龙, 张霞, 等.红曲霉产生淀粉酶固态发酵条件的优化及部分酶学性质[J].食品与发酵工业, 2013, 39(9):106-110, 115.
[18]Sakaguchi M, Matsushima Y, Nankumo T, et al. Glucoamylase of Caulobacter crescentus CB15:cloning and expression in Escherichia coli and functional identification [J]. AMB Express, 2014, 4(1):5.
[19]Stamford TL, Stamford NP, Coelho LC, et al. Production and characterization of a thermostable glucoamylase from Streptosporangium sp. endophyte of maize leaves [J]. Bioresour Technol, 2002, 83(2):105-109.
[20]Sauer J, SigurskjoLd BW, Christensen U, et al. Glucoamylase:structure/function relationships, and protein engineering [J]. Biochim Biophys Acta, 2000, 1543(2):275-293.
[21] Pandy A, Nigam P, Soccol CR, et al. Advances in microbial amylases [J]. Biotechnol Appl Biochem, 2000, 3(1):35-52.
[22]Singh D, Seo MJ, Kwon HJ, et al. Genetic localization and heterologous expression of validamycin biosynthetic gene cluster isolated from Streptomyces hygroscopicus var. limoneus KCCM 11405(IFO 12704) [J]. Gene, 2006, 376(1):13-23.
[23]Miller GL. Use of dinitrosalicylic acid reagent for determination of reducing sugar [J]. Anal Chem, 1959, 31(3):426-428.
[24]Tirpanalan O, Reisinger M, Huber F, et al. Wheat bran biorefinery:An investigation on the starch derived glucose extraction accompanied by pre- and post-treatment steps [J]. Bioresour Technol, 2014, 163:295-299.
[25]Fouladi E, Mohammadi Nafchi A. Effects of acid-hydrolysis and hydroxypropylation on functional properties of sago starch [J]. Int J Biol Macromol, 2014, 68:251-257.
[26]Luana N, Rossana C, Curiel JA. Manufacture and characterization of a yogurt-like beverage made with oat flakes fermented by selected lactic acid bacteria [J]. Int J Food Microbiol, 2014,185C:17-26.
[27]Hong Y, Liu G, Gu Z. Recent advances of starch-based excipients used in extended-release tablets:a review [J]. Drug Deliv, 2014,15:1-9.
[28] Li H, Zhu Y, Jiao A, et al. Impact of α-amylase combined with hydrochloric acid hydrolysis on structure and digestion of waxy rice starch [J]. Int J Biol Macromol, 2013, 55:276-281.
[29] Srichuwong S, Isono N, Mishima T, et al. Structure of lintnerized starch is related to X-ray diffraction pattern and susceptibility to acid and enzymehydrolysis of starch granules [J]. Int J Biol Macromol, 2005, 37(3):115-121.
[30]Zaroog MS, Abdul Kadir H, Tayyab S. Stabilizing effect of various polyols on the native and the denatured states of glucoamylase [J]. Scientific World Journal, 2013:570859.
(责任编辑 马鑫)
Isolation,Expression and Identification of Multifunctional Glucoamylase from Marine Streptomyces olivaceus FXJ7.023
Wang Miao1Li Yuanyuan1Yue Changwu1Shao Meiyun1Lü Yuhong1Bao Yuxin1Huang Ying2
(1. Guizhou Key Laboratory of Microbial Resources & Drug Development,Zunyi Medical College,Zunyi 563003;2. Institute of Microbiology,Chinese Academy of Sciences,Beijing 100101)
SoGA, a predicted glucoamylase encoded by a full-length 1 788 bp gene consists of 595 amino acids was cloned from Streptomyces olivaceus strain FXJ7.023 genomic DNA by PCR. Heterologous expression of SoGA in Escherischia coli strains BL21(DE3) plysS was performed and a fusion protein with molecular weight of 83.22 kD was obtained. The novel recombinant glucoamylase showed multifunctional catalytic activity of hydrolyze carboxymethylcellulose and starch under different temperature and pH.
streptomycetes;glucoamylase;gene clone;protein expression;enzyme activity
10.13560/j.cnki.biotech.bull.1985.2015.02.024
2014-06-23
国家自然科学基金项目(31160004),国家重点基础科学研究计划(2011CB808800),贵州省教育厅特色重点实验室建设项目(黔教合KY字[2012]011号),贵州省科技学技术基金项目(黔科合J字[2010]2156号,黔科合J字[2012]2348号,黔科合SY字[2013]3013号)
王苗,女,硕士,研究方向:微生物天然产物生物合成;E-mail:wmhappy@nwsuaf.edu.cn
岳昌武,男,博士,副研究员,副教授,硕士生导师,研究方向:微生物天然产物生物合成;E-mail:changwuyue@126.com黄英,研究员,博士生导师,研究方向:放线菌系统学与资源;E-mail:huangy@im.ac.cn.
