羧酶体结构及其CO2浓缩机制研究进展
2014-06-24张宝燕田平芳
张宝燕,田平芳
北京化工大学生命科学与技术学院 北京市生物加工过程重点实验室,北京 100029
羧酶体结构及其CO2浓缩机制研究进展
张宝燕,田平芳
北京化工大学生命科学与技术学院 北京市生物加工过程重点实验室,北京 100029
羧酶体 (Carboxysome) 是高效的固碳微体,在CO2浓缩机制 (CO2-concentrating mechanism,CCM) 中发挥重要作用。在蓝藻及某些化能自养菌中,羧酶体作为类细胞器包裹1,5-二磷酸核酮糖羧化酶/加氧酶(RubisCO) 和碳酸酐酶 (Carbonic anhydrase,CA),它与无机碳转运蛋白共同在胞质中积累HCO3–,通过增加RubisCO周围的CO2浓度来提高固碳效率。随着羧酶体结构和功能的阐明,异源表达羧酶体已成功实现,并且已鉴定出编码羧酶体壳蛋白及内部组分的基因。首先简要介绍羧酶体的发现和种类,然后系统分析其结构及在CCM机制中的作用,并对其在代谢工程上的广阔应用前景进行了展望。
蓝藻,微体,碳酸酐酶,1,5-二磷酸核酮糖羧化酶/加氧酶,CO2浓缩机制
羧酶体 (Carboxysome) 作为原核细胞的“细胞器”,因参与固定CO2及其进化生物学意义而引起了广泛关注。亚细胞组分分化是真核细胞和原核细胞的主要区别之一。真核细胞中的结构区域化 (Compartmentalization) 既实现了代谢途径的高保真性,提高了酶催化反应效率,又降低了代谢物对细胞的毒性。原核细胞只含有一种结构相对完整的细胞器,即核糖体。羧酶体标志着原核细胞的功能区域化,它提高了原核细胞代谢反应的效率,淡化了原核细胞和真核细胞之间的严格界限。
无机碳进入生物圈主要通过卡尔文循环(Calvin-Benson-Bassham cycle)。该循环中,1,5-二磷酸核酮糖羧化酶/加氧酶 (RubisCO) 作为限速酶,将CO2固定在核酮糖-1,5-二磷酸(RuBP) 上,生成3-磷酸甘油酸 (3-PGA)。因为CO2和O2具有化学相似性[1],所以通过RubisCO催化的固碳反应对O2高度敏感。然而,大气中O2含量远远超过CO2。为适应大气环境,自然界进化出CO2浓缩机制 (CO2-concentrating mechanism,CCM) (图1A和B),即通过在细胞内高浓度积累CO2来提高RubisCO的固碳效率。在CCM中,羧酶体将RubisCO和碳酸酐酶(Carbonic anhydrase,CA) 包裹在半透性外壳中,既阻止了O2进入酶的活性位点,又提高了RubisCO周围CO2的浓度。
1 羧酶体的发现
1956年首次在钩状席藻Phormidium uncinatum中发现原核细胞中的羧酶体 (当时称蛋白复合体)[2]。1973年Shively等[3]通过差速离心和蔗糖梯度离心,从那不勒斯盐生硫杆菌Halothiobacillus neapolitanus中分离出这种蛋白复合体。该复合体外围是蛋白质外壳,内部包含丰富的RubisCO。为纪念该发现,Shively将该复合体命名为羧酶体。随后,科学家发现很多细菌都有羧酶体。迄今为止,携带羧酶体的细菌包括全部蓝藻及部分化能自养菌,如蓝藻中的集胞藻属Synechocystis、原绿球藻属Prochlorococcus、念珠藻Nostoc punctiforme、聚球藻属Synechococcus和鱼腥藻Anabaena cylindrica等;化能自养菌中的H. neapolitanus、嗜酸性氧化亚铁硫杆菌Acidithiobacillus ferrooxidans和维氏硝化杆菌Nitrobacter winogradskyi等[4]。研究最多的是蓝藻中Synechocystis sp. PCC 6803和细长聚球藻Synechococcus elongates PCC 7942及化能自养菌H. neapolitanus。
2 羧酶体的种类
根据所含RubisCO的不同,可将羧酶体分为两种:α-羧酶体,包含1A型RubisCO;β-羧酶体,包含1B型RubisCO[5-6]。基因突变和基因组分析显示[7],编码羧酶体结构蛋白的基因主要聚集在基因组的同一基因座上,涉及两个启动子:cso和ccm (图1C)。迄今发现,由cso启动子编码的α-羧酶体存在于α, β和γ-变形菌及单细胞的Synechococcus等海洋蓝藻中[8],以H. neapolitanus为模式生物。由ccm启动子编码的β-羧酶体则只存在于蓝藻 (包括纤维状的蓝藻、含异型胞群的蓝藻及其分支类型和组合体,但藻青菌UCYN-A除外[10]) 中[9],以Synechocystis sp. PCC 6803为模式生物。
3 羧酶体的结构
Schmid等[11]通过低温电子断层扫描(Cryo-ET) 和扫描透射电镜 (STEM) 技术观察到羧酶体呈二十面体对称结构 (图2)。此外,Iancu等[12-13]通过电镜观察了3种化能自养菌的羧酶体,发现除了典型的二十面体形状外,羧酶体还有其他形状 (如不规则型及细长型等)。羧酶体内部包裹胞内大部分RubisCO,外围是一层选择渗透性蛋白外壳。选择渗透性是由外壳上带电荷的孔隙形成的。这种外壳既能阻止O2进入,又能阻止CO2逸出,即在最小化光呼吸的前提下提高了RubisCO周围CO2浓度。另外,外壳必须对HCO3−具有渗透性,并能透过RubisCO的底物 (RuBP) 和产物 (3-PGA)。
除RubisCO外,羧酶体中还有少量CA,CA催化HCO3−转变为CO2。目前已报道多种CA[14-15],如CsoSCA、CcaA和CcmM等。CA是羧酶体内以HCO3−为光合可利用CO2的最基本酶。
3.1 羧酶体外壳

图2 电镜观察聚球藻属Synechococcus sp. WH8102羧酶体[7]Fig. 2 Electron microscopy of carboxysomes in Synechococcus sp. WH8102[7].
羧酶体外壳由几千个小蛋白亚基组成,直径约800−1 400 Å (埃,光谱线波长单位)[6]。这些蛋白亚基主要含有两种结构域:BMC (Bacterialmicrocompartment;Pfam00936) 结构域和构域的CcmL/EutN (Pfam03319) 结构域。含有BMC结蛋白聚集成六聚体,六聚体相互连接构成二十面体的平面;而含有CcmL/EutN结构域的蛋白聚集成五聚体,五聚体构成二十面体的顶点[15-18]。此外,在六聚体对称轴心处有不同大小及带电类型的孔隙[19]。研究发现,孔隙处带有正电荷,而孔隙周围呈疏水性。据推测,这些孔隙可作为HCO3−、RuBP及3-PGA等小分子物质的代谢通道。HCO3−等阴离子物质能轻易通过带正电荷的孔隙,而O2和CO2等不带电分子则不能穿过羧酶体 (图3)。
3.1.1 BMC六聚体面蛋白

图3 羧酶体外壳蛋白高清结构[26]Fig. 3 High-resolution structures of carboxysome shell proteins[26].
BMC蛋白 (具有共同BMC结构域的蛋白质) 是羧酶体最主要的结构成分,由此聚集成的六聚体呈盘装结构,相互连接构成羧酶体的平面。在β-羧酶体中,拥有单一BMC结构域的蛋白为CcmK1-4,而CcmO蛋白由两个BMC结构串联而成[20]。Kerfeld等[16]首次从Synechocystis sp. PCC 6803中确定了β-羧酶体外壳蛋白CcmK2和CcmK4的结构。CcmK2蛋白六聚体面同向排列,构成均匀的一层;而由CcmK4蛋白构成的分子层,其六聚体的凹凸面交替排列。随后,Cai等[21]发现β-羧酶体中的假定蛋白——CcmP,认为CcmP是β-羧酶体外壳上的串联BMC结构域蛋白,形成双层蛋白结构,能促进大分子代谢物通过羧酶体。其他细菌的β-羧酶体中可能有更多BMC蛋白。
在α-羧酶体中,CsoS1A-C蛋白具有单独BMC结构域。从H. neapolitanus中分离的羧酶体外壳蛋白CsoS1A[17]和CsoS1C[22]与β-羧酶体中的CcmK2结构类似。序列分析显示,CsoS1A和CsoS1C蛋白仅相差两个氨基酸,而CsoS1B蛋白在C端额外延伸出12个氨基酸。此外,Roberts等[23]从原绿球藻Prochlorococcus marinus MED4中分离出一种新型外壳蛋白CsoS1D。研究显示,CsoS1D蛋白不由cso操纵子编码,且具有串联的BMC结构域,可能作用于羧酶体外壳的结构或功能。
3.1.2五聚体顶点蛋白
羧酶体外壳看似由六聚体组成,但晶体结构显示,其外壳上还存在一个椎形结构域构成羧酶体的顶点,即CcmL/EutN结构域。羧酶体中含有CcmL/EutN结构域的五聚体蛋白非常保守,α-羧酶体中有CsoS4A和CsoS4B,而β-羧酶体中只有一种,即CcmL。Tanaka等[6]指出:CsoS4A和CcmL蛋白与六聚体蛋白一样,具有明显的凹凸面。CcmL蛋白用其突起的C端与相邻亚基紧密连接,留下一个狭窄孔隙,CO2通过该孔隙穿过羧酶体。随后,Cai等[18]发现,在缺乏CsoS4顶点蛋白的H. neapolitanus突变株中,羧酶体外观结构看似正常,但不再限制CO2的渗透,也不再具有RubisCO催化优势。因此,顶点蛋白可能有助于羧酶体行使CO2渗透屏障功能。
由此得知,羧酶体外壳平面由大量嵌合的BMC蛋白组成,顶点由少量特异性五聚体蛋白填充。另外,Samborska和Kimber[24]最近发现,β-羧酶体外壳可能由双层BMC蛋白组成。该观点与羧酶体的厚度一致[25]。而且,随着α-羧酶体外壳蛋白CsoS1D双层结构的报道[23],Cai等[21]也提出,β-羧酶体中的CcmP蛋白具有双层结构,这加深了我们对羧酶体外壳结构的认识。
3.2 羧酶体内部结构
α-羧酶体外壳下方有一个结合CA CsoSCA的区域。1989年Zarzycki和Iancu发现,细胞质在正常环境下不含CA,CA的异常表达会破坏胞质中的HCO3−池,而使CCM机制短路,所以CA是羧酶体功能模型中的重要因素。另有报道称RubisCO与外壳紧密相连[9,12],但连接机制尚不确定。Menon等[26]提出:RubisCO通过大亚基与外壳连接,但也有人认为是小亚基在发挥作用[27]。除RubisCO和CsoSCA外,α-羧酶体中还有大量外壳连接蛋白,如假定的结构蛋白CsoS2等。在某些物种中,CsoS2蛋白以一种多肽存在;而在H. neapolitanus中[23],CsoS2蛋白包含两种多肽:CsoS2A和CsoS2B。有人猜想[10],CsoS2蛋白可能是组织蛋白,因CsoS2蛋白和β-羧酶体CcmM蛋白 (如下) 一样,拥有两个功能未知的重复氨基酸序列,但没有证据证明CsoS2蛋白以相似的机制组织α-羧酶体。
β-羧酶体也有一个富含CA CcaA的内壳层[25,28-31],CA负责将碳酸氢盐脱水,转变为CO2。β-羧酶体基因簇编码两种蛋白:CcmM和CcmN,它们参与羧酶体内部的催化或组装。CcmM蛋白通过转录形成2种多肽:C端包含3−5个RubisCO蛋白小亚基的重复单位,转录形成短链 (35 kDa,M35),连接RubisCO;N端是γ-CA的同源物,转录形成长链 (58 kDa,M58),连接羧酶体CA CcaA。CcmM与RubisCO的大亚基 (RbcL)、外壳蛋白及CcmN都可相互作用。另外,许多β-蓝藻中的CcmM-58都有CA活性[32]。CcmN蛋白功能未知,但基因簇序列高度保守,其N端主要与羧酶体CcmM (即RubisCO和CA CcaA) 相互作用;C端与羧酶体外壳连接。有报道称:CcmN蛋白C端多肽对研究羧酶体的起源具有重要意义[33]。羧酶体蛋白组成如表1所示。
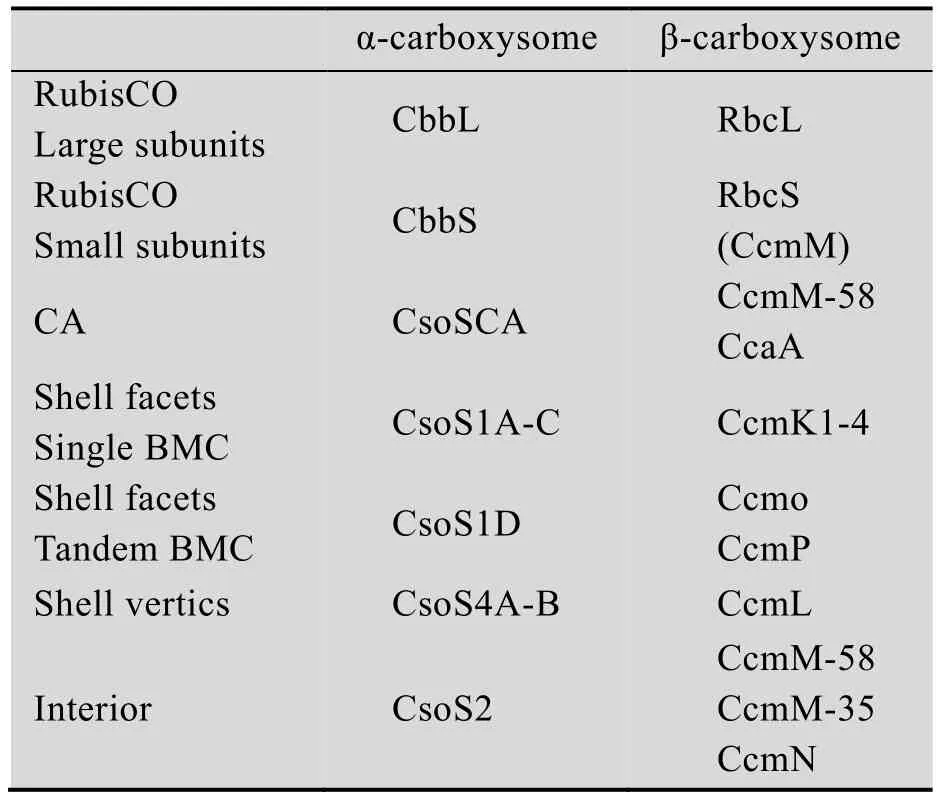
表1 α和β-羧酶体蛋白组成Table 1 Protein components of α- and β-carboxysome
4 CCM机制与羧酶体
CCM机制由两步构成[34]:第一步由跨膜的碳泵将胞外无机碳泵入胞内,在胞质中积累HCO3−;第二步通过羧酶体中的CA将HCO3−转化为CO2。羧酶体中的CO2在RubisCO催化下生成3-PGA,3-PGA逸出羧酶体后参与其他代谢。因RubisCO不仅对CO2亲和力低,而且对O2敏感度高,所以通过CCM增加RubisCO周围的CO2浓度可有效提高固碳效率。其他现存的CCM机制包括真核生物体中的淀粉核[35],及C4光合代谢机制和景天酸代谢机制[36]等。
除羧酶体外,在肠道沙门氏菌Salmonella enterica中还发现两种类羧酶体微体:包含1,2-丙二醇代谢途径的Pdu微体[37]和包含乙醇胺代谢途径的Eut微体[38]。PduU蛋白是报道的首个类羧酶体外壳蛋白。EutG蛋白将乙醛转化为乙醇,并能促进羧酶体中NADH的再循环。大肠杆菌Escherichia coli中也含有Eut微体[39]。这些类羧酶体器官能保存挥发性代谢物或活性中间产物,如CO2等。BMC蛋白存在于1/5的细菌中,可以想象自然界中尚有多少不为人知的微体。
5 羧酶体的功能拓展
为研究RubisCO在羧酶体中的作用,Menon等[26]构建了一系列RubisCO突变株。研究发现,缺乏RubisCO的羧酶体能组装成形状和组分都正常的空壳。Rae等[20]通过敲出外壳蛋白CcmK2或CcmO发现,突变株产生不含外壳的结构。因此,Menon等[26]认为羧酶体的外壳特征并不取决于其所包含的酶,并首次指出羧酶体的内含物是可以被操纵的。为探究羧酶体结构的可塑性,辛鑫等[40]首先分析其他固碳酶的理化性质,将鲍曼不动杆菌Acinetobacter baumannii的磷酸烯醇式丙酮酸羧化酶(Phosphoenolpyruvate carboxylase,PEPC) 转化原核宿主。结果显示,PEPC可以促进细胞生长,且不会带来生长负担。因此,在羧酶体内引入PEPC,可望构建全新羧酶体。
更令人振奋的是哈佛大学Silver课题组[41]近期发现,由10个基因编码的细长聚球藻Synechococcus elongates PCC 7942羧酶体能在大肠中异源表达。异源表达的羧酶体与原始宿主中的很相似,纯化后发现其结构良好,不仅包含预期的分子成分,而且可在体内固定CO2。基于此,Silver研究小组建立了一种基因系统,该系统能够在不同宿主中产生固碳模块。这不仅使我们更好地理解了羧酶体的复杂分子结构,还为构建人工羧酶体奠定了基础。将人工羧酶体作为合成“细胞器”植入异养生物中,有望赋予其固定CO2的性能,从而将其转变为自养生物。
随着对羧酶体结构和功能的阐明,很多学者认为羧酶体在本质上类似于噬菌体,加之其结构的可塑性,认为它可作为载体用于表达外源基因。此外,增加胞内羧酶体的拷贝数可望提高固定CO2能力。Price等[42]将蓝藻CCM机制中HCO3−转运蛋白:BicA和SbtA分别转入C3植物中,两种转基因植物的光合作用均提高约28%。Synechocystis sp. PCC 6803是研究光合作用的模式生物,但生长缓慢、发酵周期长的缺点严重影响了在生物能源方面的应用价值。可以设想,在Synechocystis sp. PCC 6803中构建羧酶体表达载体,增加其拷贝数,有望增强固定CO2能力,从而提高其生长速度。
综上所述,作为原核细胞的“细胞器”,羧酶体是当今研究热点,其发现不仅具有进化生物学和细胞生物学理论意义,而且在代谢工程(Metabolic engineering) 和合成生物学 (Synthetic biology) 特别是光合分子育种领域具有非常广阔的应用前景。
REFERENCES
[1] Tcherkez GGB, Farquhar GD, Andrews TJ. Despite slow catalysis and confused substrate specificity, all ribulosebisphosphate carboxylases may be nearly perfectly optimized. Proc Natl Acad Sci USA, 2006, 103(19): 7246–7251.
[2] Drews G, Niklowitz W. Cytology of cyanophycea. II. Centroplasm and granular inclusions of Phormidium uncinatum. Arch Microbiol, 1956, 24(2): 147–162.
[3] Shively JM, Ball F, Brown DH, et al. Functional organelles in prokaryotes: polyhedral inclusions (carboxysomes) of Thiobacillus neapolitanus. Science, 1973, 182(4112): 584–586.
[4] Shively JM. Inclusion bodies of prokaryotes. Annu Rev Microbiol, 1974, 28, 167–188.
[5] Badger MR, Bek EJ. Multiple Rubisco forms in proteobacteria: their functional significance in relation to CO2acquisition by the CBB cycle. J Exp Bot, 2008, 59(7): 1525–1541.
[6] Tanaka S, Kerfeld CA, Sawaya MR, et al. Atomic-level models of the bacterial carboxysome shell. Science, 2008, 319(5866): 1083–1086.
[7] Rae BD, Long BM, Whitehead LF, et al. Cyanobacterial carboxysomes: microcompartments that facilitate CO2fixation. J Mol Microb Biotech, 2013, 23(4/5): 300–307.
[8] Rae BD, Forster B, Badger MR, et al. The CO2-concentrating mechanism of Synechococcus WH5701 is composed of native and horizontally-acquired components. Photosynth Res, 2011, 109(1/3): 59–72.
[9] Zarzycki J, Axen SD, Kinney JN, et al. Cyanobacterial-based approaches to improving photosynthesis in plants. J Exp Bot, 2013, 64(3): 787–798.
[10] Tripp HJ, Bench SR, Turk KA, et al. Metabolic streamlining in an open-ocean nitrogen-fixing cyanobacterium. Nature, 2010, 464(7285): 90–94.
[11] Schmid MF, Paredes AM, Khant HA, et al. Structure of Halothiobacillus neapolitanus carboxysomes by cryo-electron tomography. J Mol Biol, 2006, 364(3): 526–535.
[12] Iancu CV, Ding HJ, Morris DM, et al. The structure of isolated Synechococcus strain WH8102 carboxysomes as revealed by electron cryotomography. J Mol Biol, 2007, 372(3): 764–773.
[13] Iancu CV, Morris DM, Dou Z, et al. Organization, structure, and assembly of α-carboxysomes determined by electron cryotomography of intact cells. J Mol Biol, 2010, 396(1): 105–117.
[14] Cannon GC, Heinhorst S, Kerfeld CA. Carboxysomal carbonic anhydrases: Structure and role in microbial CO2fixation. BBA-Proteins Proteom, 2010, 1804(2): 382–392.
[15] Pena KL, Castel SE, Araujo C de, et al. Structural basis of the oxidative activation of the carboxysomal γ-carbonic anhydrase, CcmM. Proc Natl Acad Sci USA, 2010, 107(6): 2455–2460.
[16] Kerfeld CA, Sawaya MR, Tanaka S, et al. Protein structures forming the shell of primitive bacterial organelles. Science, 2005, 309(5736): 936–938.
[17] Tsai Y, Sawaya MR, Cannon GC, et al. Structural analysis of CsoS1A and the protein shell of the Halothiobacillus neapolitanus carboxysome. PLoS Biol, 2007, 5(6): e144.
[18] Cai F, Menon BB, Cannon GC, et al. The pentameric vertex proteins are necessary for the icosahedral carboxysome shell to function as a CO2leakage barrier. PLoS ONE, 2009, 4(10): e7521.
[19] Kinney JN, Axen SD, Kerfeld CA. Comparative analysis of carboxysome shell proteins. Photosynth Res, 2011, 109(1/3): 21–32.
[20] Rae BD, Long BM, Badger MR, et al. Structural determinants of the outer shell of β-carboxysomes in Synechococcus elongates PCC 7942: roles for CcmK2, K3-K4, CcmO, and CcmL. PLoS ONE, 2012, 7(8): e43871.
[21] Cai F, Sutter M, Cameron JC, et al. The structure of CcmP, a tandem bacterial microcompartment domain protein from the β-carboxysome, forms a subcompartment within a microcompartment. J Biol Chem, 2013, 288(22): 16055–16063.
[22] Tsai Y, Sawaya MR, Yeates TO. Analysis of lattice-translocation disorder in the layered hexagonal structure of carboxysome shell protein CsoS1C. Acta Crystallogr D Biol Crystallogr,2009, 65(9): 980–988.
[23] Roberts EW, Cai F, Kerfeld CA, et al. Isolation and characterization of the Prochlorococcus carboxysome reveal the presence of the novel shell protein CsoS1D. J Bacteriol, 2012, 194(4): 787–795.
[24] Samborska B, Kimber MS. A dodecameric CcmK2 structure suggests β-carboxysomal shell facets have a double-layered organization. Structure, 2012, 20(8): 1353–1362.
[25] Long BM, Rae BD, Badger MR, et al. Over-expression of the β-carboxysomal CcmM protein in Synechococcus PCC 7942 reveals a tight co-regulation of carboxysomal carbonic anhydrase (CcaA) and M58 content. Photosynth Res, 2011, 109(1/3): 33–45.
[26] Menon BB, Dou Z, Heinhorst S, et al. Halothiobacillus neapolitanus carboxysomes sequester heterologous and chimeric RubisCO species. PLoS ONE, 2008, 3(10): e3570.
[27] Holthuijzen YA, van Breemen JFL, Kuenen JG, et al. Protein composition of the carboxysomes of Thiobacillus neapolitanus. Arch Microbiol, 1986, 144(4): 398–404.
[28] Ludwig M, Sultemeyer D, Price GD. Isolation of ccmKLMN genes from the marine cyanobacterium, Synechococcus sp. PCC 7002 (Cyanophyceae), and evidence that CcmM is essential for carboxysome assembly. J Phycol, 2000, 36(6): 1109–1119.
[29] Cot SSW, So AKC, Espie GS. A multiprotein bicarbonate dehydration complex essential to carboxysome function in cyanobacteria. J Bacteriol, 2008, 190(3): 936–945.
[30] Long BM, Badger MR, Whitney SM, et al. Analysis of carboxysomes from Synechococcus PCC 7942 reveals multiple Rubisco complexes with carboxysomal proteins CcmM and CcaA. J Biol Chem, 2007, 282(40): 29323–29335.
[31] Long BM, Tucker L, Badger MR, et al. Functional cyanobacterial β-carboxysomes have an absolute requirement for both long and short forms of the CcmM protein. Plant Physiol, 2010, 153(1): 285–293.
[32] Espie GS, Kimber MS. Carboxysomes: cyanobacterial RubisCO comes in small packages. Photosynth Res, 2011, 109(1/3): 7–20.
[33] Kinney JN, Salmeen A, Cai F, et al. Elucidating essential role of conserved carboxysomal protein CcmN reveals common feature of bacterial microcompartment assembly. J Biol Chem, 2012, 287(21): 17729–17736.
[34] Yeates TO, Kerfeld CA, Heinhorst S, et al. Protein-based organelles in bacteria: carboxysomes and related microcompartments. Nat Rev Microbiol, 2008, 6(9): 681–691.
[35] Meyer M, Griffiths H. Origins and diversity of eukaryotic CO2-concentrating mechanisms: lessons for the future. J Exp Bot, 2013, 64(3): 769–786.
[36] Sage RF. The evolution of C4 photosynthesis. New Phytol, 2004, 161(2): 341–370.
[37] Crowley CS, Sawaya MR, Bobik TA, et al. Structure of the PduU shell protein from the Pdu microcompartment of Salmonella. Structure, 2008, 16(9): 1324–1332.
[38] Penrod JT, Roth JR. Conserving a volatile metabolite: a role for carboxysome-like organelles in Salmonella enterica. J Bacteriol, 2006, 188(8): 2865–2874.
[39] Tanaka S, Sawaya MR, Yeates TO. Structure and mechanisms of a protein-based organelle in Escherichia coli. Science, 2010, 327(5961): 81–84.
[40] Xin X, Wang ZX, Ge XZ, et al. Cloning and prokaryotic expressin of phosphoenolpyruvate carboxylase. J Beijing Univ Chem Technol: Nat Sci Ed., 2012, 39(5): 98–101 (in Chinese).
辛鑫, 王朝绚, 葛喜珍, 等. 磷酸烯醇式丙酮酸羧化酶基因的克隆与原核表达. 北京化工大学学报: 自然科学版,2012, 39(5): 98–101.
[41] Bonacci W, Teng PK, Afonso B, et al. Modularity of a carbon-fixing protein organelle. Proc Natl Acad Sci USA, 2012, 109(2): 478–483.
[42] Price GD, Pengelly JJL, Forster B, et al. The cyanobacterial CCM as a source of genes for improving photosynthetic CO2fixation in crop species. J Exp Bot, 2013, 64(3): 753–768.
(本文责编 陈宏宇)
Progress in structure and CO2-concentrating mechanism of carboxysomes
Baoyan Zhang, and Pingfang Tian
Beijing Key Laboratory of Bioprocess, College of Life Science and Technology, Beijing University of Chemical Technology, Beijing 100029, China
Carboxysomes are extremely efficient microcompartments committed to CO2fixation due to tailored CO2-concentrating mechanism (CCM). In cyanobacteria and some chemoautotrophs, carboxysomes as organelle-like microbodies encapsulate ribulose-1,5-bisphosphate carboxylase/oxygenase (RuBisCO) and carbonic anhydrase (CA). Together with active inorganic carbon uptake transporters, carboxysomes accumulate HCO3(-) in the cytoplasm, leading to high efficiency of carbon fixation. Based on the elucidation of structures and functionalities, heterologous production ofcarboxysomes has been achieved so far. In fact, the genes encoding either vacant carboxysome shell or only interior components have been characterized. This review summarizes the discovery along with types, showcases molecular structures and roles of carboxysomes in CCM, and presents their broad applications in metabolic engineering.
cyanobacteria, microcompartments, carbonic anhydrase, RubisCO, CO2-concentrating mechanism
November 12, 2013; Accepted: December 10, 2013
Pingfang Tian. Tel: +86-10-89136369; E-mail: tianpf@mail.buct.edu.cn
张宝燕, 田平芳. 羧酶体结构及其CO2浓缩机制研究进展. 生物工程学报, 2014, 30(8): 1164−1171.
Zhang BY, Tian PF. Progress in structure and CO2-concentrating mechanism of carboxysomes. Chin J Biotech, 2014, 30(8):1164−1171.
Supported by: National Natural Science Foundation of China (Nos. 21076013, 21276014), National Basic Research Program of China (973 Program) (No. 2012CB725200).
国家自然科学基金 (Nos. 21076013, 21276014),国家重点基础研究发展计划 (973计划) (No. 2012CB725200) 资助。
