一种基于核酸外切酶Ⅲ的PCR产物克隆方法
2014-06-24王艳艳张春雨王兴春刘斌
王艳艳,张春雨,王兴春,刘斌
1 山西农业大学生命科学学院,山西 太谷 030801
2 中国农业科学院作物科学研究所,北京 100081
3 山西农业大学研究生院,山西 太谷 030801
一种基于核酸外切酶Ⅲ的PCR产物克隆方法
王艳艳1,2,3*,张春雨2*,王兴春1,刘斌2
1 山西农业大学生命科学学院,山西 太谷 030801
2 中国农业科学院作物科学研究所,北京 100081
3 山西农业大学研究生院,山西 太谷 030801
基因克隆作为一种常规技术被广泛应用于DNA及蛋白质的研究。在传统的基因克隆中,一般利用限制性内切酶对DNA片段进行消化,然后使用DNA连接酶完成连接,因此这种方法受到插入片段和载体酶切位点的限制。一些ligase-free 克隆技术虽然克服了酶切位点的限制,但费时费力,成本较高。为了弥补其他ligase-free技术的不足,文中介绍了一种新的利用核酸外切酶Ⅲ进行ligase-free克隆的方法,反应时间仅需要30 min,提高了克隆效率并有效降低经济成本,有利于大规模基因克隆的应用。
核酸外切酶Ⅲ,LIC系统,PCR产物克隆
为了提高克隆效率,近年来一些ligase-free技术已经被开发用于高效克隆PCR产物,包括GATEWAY系统[1-3]、In-fusion系统[4-6]、尿嘧啶DNA糖基化酶 (Uracil-DNA glycosylase,UDG)[7-9]、序列和连接反应的高效克隆基因方法(Sequence-ligation independent cloning,SLIC)[10],以及基于T4 DNA 聚合酶的连接反应克隆(Ligation-Independent cloning,LIC)[11-15]等。但它们依然存在一些缺陷,例如GATEWAY系统操作步骤繁琐,引物的两端需加入较长特定序列,反应所需的酶类价格昂贵,并需要特定的载体[10]。In-fusion虽然反应条件简单,效率较高,但昂贵的酶让人望而却步,在大规模的克隆中消耗太大。LIC作为一种快速高效的方法被应用于大规模的基因克隆[16]及载体构建[17-19]。这种方法利用T4 DNA聚合酶的外切酶活性作用于载体与PCR产物的两端,从而产生互补的5′粘性末端,在退火条件下不需要连接酶的作用,而直接通过5′突出的粘性末端发生重组,重新环化成新的质粒[20]。但LIC方法需要在重叠区段缺少某个特定核苷酸[11-15],因此需要特定的载体或通过PCR的方法将载体线性化。另外,如果这段粘性末端在编码区内,就会在重组蛋白的N端出现多余的肽段,这些都在一定
程度上限制了该方法的广泛应用[11]。
核酸外切酶是从分子链的末端顺次水解磷酸二酯键而生成单核苷酸作用的酶,核酸外切酶Ⅲ作用于双链DNA,沿3′→5′方向逐步催化去除单核苷酸[21-22]从而产生类似于限制性内切酶消化所产生的粘性末端[23]。已有报道利用核酸外切酶Ⅲ进行亚克隆方法的研究[22-24],但在这些方法中,依然存在操作步骤繁琐、消耗时间、工作效率低等问题。文中介绍了一种快速、高效的核酸外切酶Ⅲ亚克隆,很好地解决了上述问题。
1 材料与方法
1.1 材料与试剂
1.1.1材料
大肠杆菌DH5α感受态 (北京康为世纪生物科技有限公司),pUC19克隆载体,gus质粒。
1.1.2主要试剂
核酸外切酶Ⅲ,10×NE Buffer 1,BamHⅠ(NEB公司),10×NE Buffer 3,琼脂糖凝胶回收试剂盒,高保真的KOD酶 (Toyobo公司),50×TAE,1×TAE,SYBR GreenⅠ染料 (北京普博新生物科技有限公司),BM5000 DNA Marker (Biomed公司)。
1.2 方法
1.2.1引物设计
gus-BamHⅠ-F (pUC19):5′-CGGTACCCGGGG ATCTTTCGATGCGGTCACTCA-3′;gus-BamHⅠ-R (pUC19):5′-CGACTCTAGAGGATCCACTT GCGGACGGGTATC-3′。
1.2.2 gus片段的扩增和回收
PCR反应体系:10 μmol/L F/R引物,2.5 mmol/L dNTPs 5 μL,10×Buffer 5 μL,KOD酶 1 μL,25 mmol/L MgSO43 μL,模板 (质粒) 1 μL,加灭菌的超纯水至总体积50 μL;PCR反应程序:95 ℃ 2 min;98 ℃ 10 s,58 ℃ 30 s,68 ℃ 25 s,32个循环。
将PCR反应得到的目的产物,按照AXYGEN的Axyprep DNA凝胶回收试剂盒进行纯化回收。
1.2.3载体线性化
利用限制性内切酶BamHⅠ (参考NEB公司的限制性内切酶BamHⅠ的反应体系) 线性化pUC19克隆载体,按照PCR片段与载体摩尔数为5∶1、3∶1、2∶1、1∶1、1∶2、1∶3和1∶5的比例将两者在冰上混合;加入无菌水使反应体系达到8 μL;在冰上加入1 μL的10×NE Buffer 1;将100个单位的核酸外切酶Ⅲ用水稀释10倍,吸取1 μL加入体系,冰上孵育30 s至3 min。
PCR程序为:70 ℃ 20 min,42 ℃ 10 min,待PCR仪加热至70 ℃后,立即将反应体系放到PCR仪上进行反应;反应结束后,吸取2 μL反应液用于转化。
1.2.4大肠杆菌(DH5α)的热转化
DH5α感受态细胞放置在冰上溶解;在感受态细胞中加入2 μL反应混合液,轻轻混匀后,冰浴30 min;42 ℃热激45 s;冰上放置2 min;加入800 μL LB液体培养基,37 ℃、220 r/min振荡预培养60 min;将200 μL培养液涂布到含50 mg/L卡那霉素的LB平板上,37 ℃培养12–16 h。
随机挑取96个单克隆,置于96孔板进行菌落PCR,来估计总克隆数和阳性克隆数,找出最佳片段与载体摩尔比。在此基础上继续探索最佳片段与载体的最佳浓度、冰上孵育的最佳时长、最佳退火温度,具体内容参考结果与分析。
2 结果与分析
2.1 片段与载体的最佳摩尔比
通过保持载体浓度、改变PCR片段的浓度,调整反应体系中PCR片段与载体摩尔数的比值。如图1所示,随着片段与载体摩尔比的降低,总的克隆数逐渐上升,并在比值为2∶1时达到最大值。当片段与载体的摩尔比在1∶1时,总的克隆数依然保持较高值,而当片段浓度低于载体时,总克隆数出现了较大幅度的下降,并随着摩尔比的降低呈急剧下降的趋势。阳性克隆率一直保持较高水平,说明在片段的摩尔数等于或大于载体的摩尔数时,克隆效率较高,并当PCR片段与载体的摩尔数比为2∶1时,效率最高。
2.2 片段与载体的最佳浓度
在保持片段与载体摩尔比为2∶1的条件下,设置7个PCR片段浓度。如图2所示,随着片段浓度的增加,总的克隆数首先呈现出直线上升的趋势,当反应体系中片段浓度达到50 ng时即有较高的克隆数,并在150 ng时总克隆数达到最大值,且阳性克隆比率也到达到最高。说明该浓度为反应的最佳浓度。当体系中PCR片段浓度大于150 ng时,总的克隆数则表现出下降的趋势,这可能是因为DNA片段过多而反应体系小,或DNA片段过多而外切酶切割不充分造成的。
2.3 冰上孵育最佳时长
冰上孵育时间对总克隆数及阳性克隆率的影响如图3A所示,随着冰上孵育时间的增加,总的克隆数表现出了逐渐下降的趋势,孵育时间越长,下降的幅度则越大。当孵育时长为30 s时,克隆数最多,转化效率最高。但考虑到操作时间问题,在实际的操作中,冰上孵育时间至多不应超过3 min。

图2 片段浓度对总克隆数和阳性克隆数的影响Fig. 2 Effect of the fragment concentration on the total number of clones and the number of positive clones.
冰上孵育的过程中,应将PCR仪预先升温至70 ℃。孵育结束后立即将反应体系放在70 ℃的PCR仪上加热20 min,使酶失去活性。如果将反应体系放到PCR仪后升温,则会严重影响DNA片段的连接效率 (图3B)。这可能因为核酸外切酶Ⅲ在温度逐渐上升至灭火温度的过程中,对DNA片段过度切割造成的。
2.4 最佳退火温度
如图4所示,70 ℃灭活20 min后,将PCR仪温度下调进行退火。在设置的6个不同梯度的退火温度中,以42 ℃时总的克隆数最多,说明此时为最佳的退火温度。较低的退火温度并未对总的克隆数及阳性克隆率造成明显影响,相反,当退火温度高于42 ℃时,总的克隆数随着温度的上升而逐渐下降,说明过高的温度不利于PCR片段与载体重组。因此,在设置退火温度时应避免温度过高而导致效率的降低。
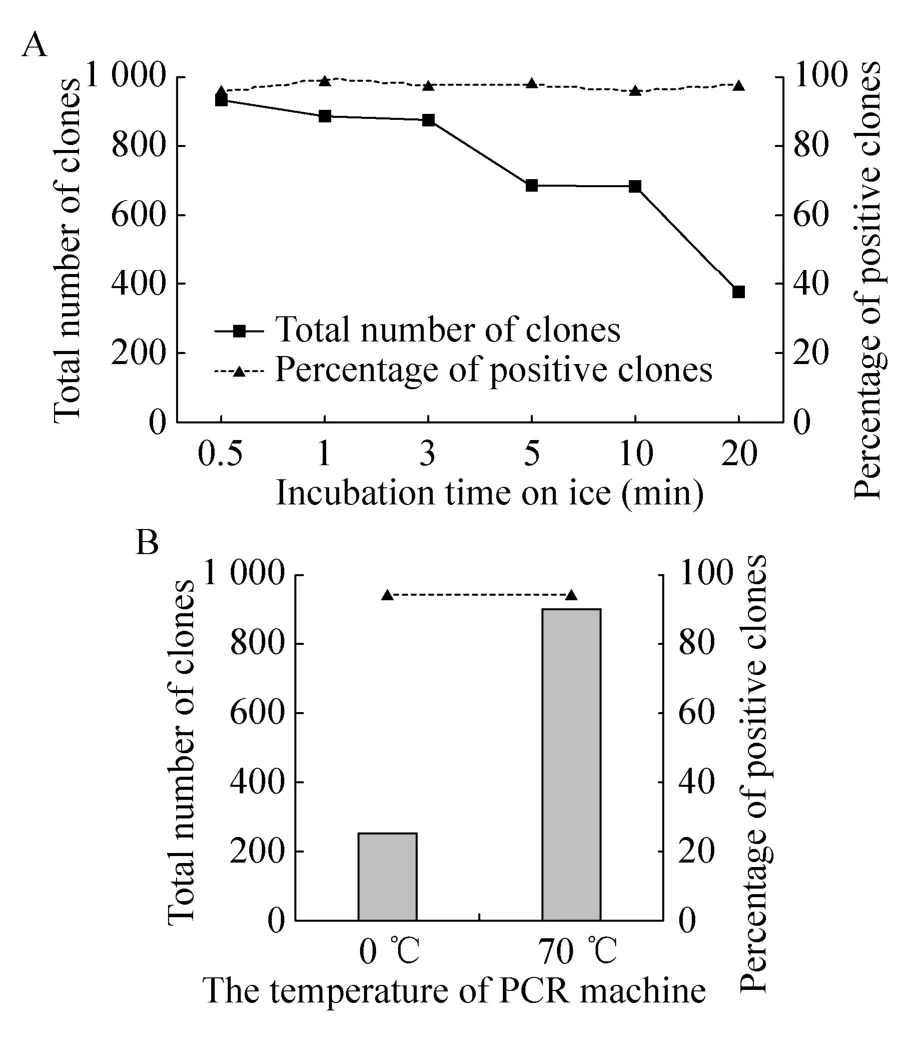
图3 冰浴时间以及PCR仪预热70 ℃对总克隆数和阳性克隆数的影响Fig. 3 Effect of the incubation time on ice (A) and the temperature of PCR machine (B) on the total number of clones and the number of positive clones.
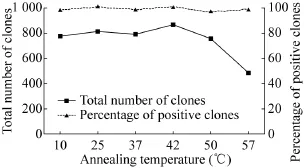
图4 退火温度对总克隆数和阳性克隆数的影响Fig. 4 Effect of the annealing temperature on the total number of clones and the number of positive clones.
2.5 与其他ligase-free技术的比较
将文中新方法分别与基于T4的ligase-free技术和In-fusion系统进行比较 (Clontech In-Fusion HD克隆试剂盒是一种基于痘病毒DNA聚合酶3′-5′外切酶活性的ligase-free系统,该酶同样能沿着载体及PCR片段两端3′羟基方向消化产生互补单链,从而完成重组)[17]。T4的方法参考Li等[10]的方法并略有修改,In-fusion参照产品说明书进行操作。文中的方法转化效率介于T4和In-fusion之间,其中以In-fusion的转化效率最高,三者阳性率都处于较高的水平 (表1),但在价格方面E3则占据最大的优势,低于T4且远远低于In-fusion (图5A)。虽然In-fusion的转化效率稍高,但从总克隆数与每个反应价格的比值可以看出,E3的效率要远远高于其他两种方法 (表2和图5B)。

表1 三种亚克隆方法的克隆效率比较Table 1 Comparative analysis of cloning efficienty of the three different cloning methods

图5 三种酶的效价比Fig. 5 Comparative analysis of the potency ratio of the three kinds of enzymes. (A) The price per reaction of the three different enzymes. (B) The total number of clones of the three different enzymes.
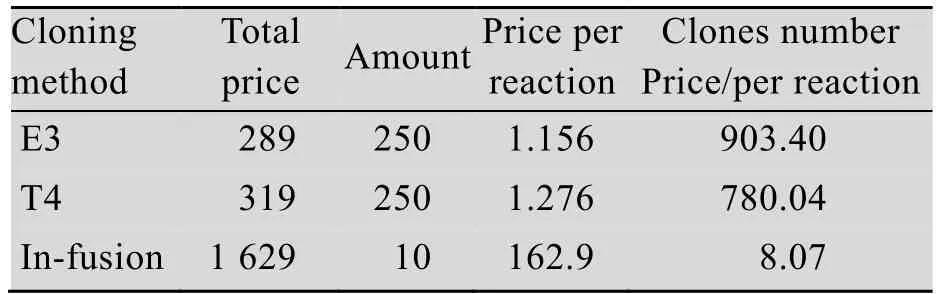
表2 三种亚克隆方法的价格效价比Table 2 Comparative analysis of the price of the three methods of the clone potency ratio
3 讨论
本文介绍了一种新的利用核酸外切酶Ⅲ进行PCR片段亚克隆的方法,实验流程如图6所示。
核酸外切酶Ⅲ主要在冰上完成对DNA片段的切割,形成较长的粘性末端,从而使重组变得更稳定并且不需要其他的连接反应[23]。在其他ligase-free技术中,人们往往通过在重组序列中缺失某一特定碱基[11-15],或利用尿嘧啶基化酶[7-9]等方法经过外切酶活性消化产生固定长度的单链末端,但这在一定程度上限制了此类方法的使用。本方法利用核酸外切酶Ⅲ在冰上消化PCR产物及载体产生单链末端,未采用特殊方法控制切割长度,这并没有对转化效率及阳性率造成影响。有报道表明,在4 ℃条件下,核酸外切酶Ⅲ每分钟能够切割25个碱基,因此仍需要控制反应体系在冰上的孵育时间。时间过长可能会破坏DNA片段的结构,或导致细菌对重组后的质粒无法完成修复。控制冰上孵育的时间在较短范围内,并在孵育后立即到70 ℃灭活,对于提高克隆效率至关重要。

图6 基于核酸外切酶Ⅲ的克隆图解Fig. 6 Schematic diagram of exonuclease Ⅲ based cloning.
有研究表明,在LIC系统中,被T4 DNA聚合酶处理的载体经凝胶纯化后,克隆的重组率能够达到100%[25],即使不经过纯化,克隆的重组率依然很高,能够达到96%以上[15]。本方法在各种处理下,阳性克隆率均大于95%,且变化幅度不明显,表明该方法同样具有其他ligase-free技术高阳性率的优势。但当载体经酶切后,一般会产生几个碱基互补的粘性末端,如果将退火温度设置过低,它们可能会发生重组环化成不含有插入片段的空质粒。因此,建议退火温度不宜设置过低而导致阳性率降低。
载体与PCR片段重组序列的长度决定着LIC系统的连接效率。有研究表明,12个碱基是连接的最小长度,当长度为10个碱基时,转化效率下降到25%,而当长度缩短为8个碱基时,则没有克隆出现[15]。而本研究结果表明,转化效率可能与退火温度存在密切的关系。本方法中载体两端与PCR片段同源的15个碱基的Tm值分别为52 ℃和46 ℃ (A-T为2 ℃,G-C为4 ℃),以此可以推算其退火温度约在40–50 ℃之间,这与结果中42 ℃为最佳退火温度相符。因此,在设定退火温度时,应参考互补序列的Tm值,这可以提高载体与插入片段的连接效率。
T4 DNA聚合酶能够利用其3′-5′外切酶活性消化DNA片段3′末端序列,并用某种dNTP终止反应,从而产生5′突出的单链末端[25]。因此,T4 DNA聚合酶被广泛应用于LIC系统[10-11,15,20,25-26]。本文参照Li等的方法[10],利用T4 DNA聚合酶消化DNA片段产生5′突出的单链末端,然后在75 ℃条件下灭活20 min,最后退火环化成新的质粒。该方法的转化效率虽然低于E3,但仍然处于较高水平。因此,采用热灭火终止反应适用于此类利用外切酶活性克隆PCR产物的方法。
与其他ligase-free方法相比该方法有下列优点:1) 在PCR引物上仅添加了与载体上同源的15个碱基,重组后不会编码多余蛋白;2) 几乎适用于所有的载体,不需要带有特定接头的商业化载体或通过PCR扩增载体;3) 阳性率高,DNA片段的过度切割未影响到转化效率及克隆的阳性率;4) 操作步骤简洁,反应仅约30 min;5) 价格低廉,远远低于in-fusion等其他ligase-free技术。因此适用于大规模基因克隆中,具有良好的应用前景。
REFERENCES
[1] Walhout AJ, Temple GF, Brasch MA, et al. GATEWAY recombinational cloning: application to the cloning of large numbers of open reading frames or ORFeomes. Methods Enzymol, 2000, 328: 579–592.
[2] Atanassov II, Atanassov II, Etchells JP, et al. A simple, flexible and efficient PCR-fusion/Gateway cloning procedure for gene fusion, site-directed mutagenesis, short sequence insertion and domain deletions and swaps. Plant Methods, 2009, 5: 14.
[3] Esposito D, Garvey LA, Chakiath CS. Gateway cloning for protein expression. Methods Mol Biol, 2009, 498: 31–54.
[4] Berrow NS, Alderton D, Owens RJ. The precise engineering of expression vectors using high-throughput in-Fusion PCR cloning. Methods Mol Biol, 2009, 498: 75–90.
[5] Zhu B, Cai G, Hall EO, et al. In-fusion assembly: seamless engineering of multidomain fusion proteins, modular vectors, and mutations. Biotechniques, 2007, 43(3): 354–359.
[6] Berrow NS, Alderton D, Sainsbury S, et al. A versatile ligation-independent cloning method suitable for high-throughput expression screening applications. Nucleic Acids Res, 2007, 35(6): e45.
[7] Bitinaite J, Rubino M, Varma KH, et al. USER friendly DNA engineering and cloning method by uracil excision. Nucleic Acids Res, 2007, 35(6): 1992–2002.
[8] Geu-Flores F, Nour-Eldin HH, Nielsen MT, et al. USER fusion: a rapid and efficient method for simultaneous fusion and cloning of multiple PCR products. Nucleic Acids Res, 2007, 35(7): e55.
[9] Nour-Eldin HH, Hansen BG, Nørholm MH, et al. Advancing uracil-excision based cloning towards an ideal technique for cloning PCR fragments. Nucleic Acids Res, 2006, 34(18): e122.
[10] Li MZ, Elledge SJ. Harnessing homologous recombination in vitro to generate recombinant DNA via SLIC. Nat Methods, 2007, 4(3): 251–256.
[11] Tachibana A, Tohiguchi K, Ueno T, et al. Preparation of long sticky ends for universal ligation-independent cloning: sequential T4DNA polymerase treatments. J Biosci Bioeng, 2009, 107(6): 668–669.
[12] Weeks SD, Drinker M, Loll PJ. Ligation independent cloning vectors for expression of SUMO fusions. Protein Expr Purif, 2007, 53(1): 40–50.
[13] Eschenfeldt WH, lucy S, Millard CS, et al. A family of LIC vectors for high-throughput cloning and purification of proteins. Methods Mol Biol, 2009, 498: 105–115.
[14] Bardóczy V, Géczi V, Sawasaki T, et al. A set of ligation independent in vitro translation vectorsfor eukaryotic protein production. BMC Biotechnol, 2008, 8: 32.
[15] Aslanidis C, de Jong PJ, Schmitz G. Minimal length requirement of the single-stranded tails for ligation-in dependent cloning (LIC) of PCR products. PCR Methods Appl, 1994, 4(3): 172–177.
[16] Vernet E, Sauer J, Andersen A, et al. Predictive mutagenesis of ligation-independent cloning (LIC) vectors for protein expression and site-specific chemical conjugation. Anal Biochem, 2011, 414(2): 312–314.
[17] Dortay H, Akula UM, Westphal C, et al. High-throughput protein expression using a combination of ligation-independent cloning (LIC) and infrared fluorescent protein (IFP) detection. PLoS ONE, 2011, 6(4): e18900.
[18] Xu G, Sui N, Tang Y, et al. One-step, zero-background ligation-independent cloning intron-containing hairpin RNA constructs for RNAi in plants. New Phytol, 2010, 187(1): 240–250.
[19] Balhana R, Stoker NG, Sikder MH, et al. Kendall SLRapid construction of mycobacterial mutagenesis vectors using ligation-independent cloning. J Microbiol, 2010, 83(1): 34–41.
[20] Yang YS, Watson WJ, Tucker PW, et al. Construction of recombinant DNA by exonuclease recession. Nucleic Acids Res, 1993, 21(8): 1889–1893.
[21] Rogers SG, Weiss B. Exonuclease Ⅲ of Escherichia coli K-12, an AP endonuclease. Methods Enzymol, 1980, 65(1): 201–211.
[22] Rogers SG, Weiss B. Cloning of the exonucleaseⅢ gene of Escherichia coli. Gene, 1980, 11(3/4): 187–195.
[23] Kaluz S, Kolble K, Reid KB. Directional cloning of PCR products using exonuclease Ⅲ. Nucleic Acids Res, 1992, 20(16): 4369–4370.
[24] Hsiao K. Exonuclease Ⅲ induced ligase-free directional subcloning of PCR products. Nucleic Acids Res, 1993, 21(23): 5528–5529.
[25] Aslanidis C, de Jong PJ. Ligation-independent cloning of PCR products (LIC-PCR). Nucleic Acids Research, 1990, 18(20): 6069–6074.
[26] Yang J, Zhang Z, Zhang XA, et al. A ligation-independent cloning method using nicking DNA endonuclease. Biotechniques, 2010, 49(5): 817–821.
(本文责编 郝丽芳)
A method for PCR product cloning based on exonuclease Ⅲ
Yanyan Wang1,2,3*, Chunyu Zhang2*, Xingchun Wang1, and Bin Liu2
1 College of Life Sciences, Shanxi Agricultural University, Taigu 030801, Shanxi, China
2 Crop Research Institute, Chinese Academy of Agricultural Sciences, Beijing 100081, China
3 Graduate School, Shanxi Agricultural University, Taigu 030801, Shanxi, China
Gene cloning is one of the most important and widely used technologies in molecular biology research.Generally, DNA fragment is cut with restriction enzyme, and then the product is ligated to a linearized vector with complementary sticky end or blunt end by DNA-ligase. This traditional DNA cloning method requires compatible enzyme recognition sites existing in both PCR fragment and targeted vector. Several ligase-free methods have been established to avoid the using of restriction enzyme. However, those methods are time-consuming, labor-intensive and expensive. To overcome these shortcomings, we developed an Exonuclease Ⅲ based DNA cloning method that takes only 30 minutes with high cloning efficiency and significant economic advantage. Therefore, this method is suitable for large-scale gene cloning.
exonuclease Ⅲ, LIC system, PCR cloning product
Junuary 19, 2014; Accepted: March 14, 2014
Xingchun Wang. Tel: +86-354-6287191-306; E-mail: wxingchun@163.com
王艳艳, 张春雨, 王兴春, 等. 一种基于核酸外切酶Ⅲ的PCR产物克隆方法. 生物工程学报, 2014, 30(8): 1266−1273.
Wang YY, Zhang CY, Wang XC, et al. A method for PCR product cloning based on exonuclease Ⅲ. Chin J Biotech, 2014,30(8): 1266−1273.
Supported by: National Natural Science Foundation of China (Nos. 31171352, 31100235), Natural Science Foundation of Shanxi Province, China (No. 2013011028-1).
Bin Liu. Tel: +86-10-82108435; E-mail: liubin05@caas.cn
* These authors contributed equally to this study.
国家自然科学基金 (Nos. 31171352, 31100235),山西省自然科学基金 (No. 2013011028-1) 资助。
时间:2014-04-02 网络出版地址:http://www.cnki.net/kcms/doi/10.13345/j.cjb.140042.html
