门多萨假单胞菌Pseudomonas mendocina P10脂肪酶基因的克隆及其在酵母中的表达
2013-12-06王建荣陈丽芝罗长财钟开新聂金梅李阳源
王建荣,陈丽芝,罗长财,钟开新,聂金梅,李阳源
(广东溢多利生物科技股份有限公司,广东珠海519060)
脂肪酶(Lipase,E.C.3.1.1.3),全称为三酰基甘油酰水解酶(triacylglycerol acylhydrolase),它属于α/β折叠酶家族,是一种丝氨酸水解酶。脂肪酶催化三酰甘油的酯键水解,释放更少酯键的甘油酯或甘油及脂肪酸[1]。脂肪酶主要来源于动植物和微生物中,相对于动植物,微生物分泌的脂肪酶具有更广的底物专一性、作用温度以及作用pH范围。脂肪酶在微生物界分布很广,目前已发现曲霉[2]、根霉[3]、假单胞菌[4]等多种微生物可产脂肪酶。在众多来源的脂肪酶中,来源于假单胞菌属的脂肪酶广泛应用于各个工业领域,包括食品工业、洗涤工业、手性拆分和药物合成等领域中。如门多萨假单胞菌的脂肪酶用于洗涤剂添加剂、荧光假单胞菌和铜绿假单胞菌的脂肪酶则被广泛用于有机合成等[5]。由于来源于假单胞菌的脂肪酶具有良好的应用特性,为了进一步提高假单胞菌分泌脂肪酶的能力,许多学者开始应用分子生物学的方法研究其脂肪酶基因的克隆、表达、调控等。到目前为止已有多种假单胞菌的脂肪酶基因被克隆并且实现了异源表达,如荧光假单胞菌[6]、铜绿假单胞菌[7]、洋葱勃克霍尔德菌[8]等。本研究采用同源克隆法得到了门多萨假单胞菌的脂肪酶基因并对该基因进行了分析,同时将该基因转入毕赤酵母X33中,进行了初步的发酵实验,为进一步研究该基因奠定了基础。
1 材料与方法
1.1 材料与仪器
表达载体pPICZαA、Pichia pastoris X-33 购自Invitrogen公司;门多萨假单胞菌Pseudomonas mendocina P10 购自中国工业微生物菌种保藏管理中心;大肠杆菌Top10、高保真Pfu酶、T4 DNA连接酶、限制性内切酶EcoRI、NotI、质粒提取试剂盒、细菌基因组提取试剂盒、PCR纯化试剂盒 均购自上海生工公司;PCR引物合成以及DNA测序 均由深圳华大基因公司完成;其他生化试剂 均为国产分析纯。
Biometra热密闭PCR仪T-1 德国Biometra公司;Bio-Rad凝胶成像系统 美国Bio-Rad公司;Nano Drop分光光度计ND-1000 美国Nano Drop公司;电泳仪DYY-6C 北京市六一仪器厂。
1.2 实验方法
1.2.1 门多萨假单胞菌基因组DNA的提取 挑取单菌落接入50mL LB液体培养基中,30℃,150r/min培养过夜,离心取菌体,参照细菌基因组DNA提取试剂盒说明书提取基因组DNA。用Nano Drop分光光度计(ND-1000)测定DNA的浓度,选取A260/A280值在1.8~2.0的样品,置于冰箱中-20℃保存备用。
1.2.2 脂肪酶基因的克隆 经过分析NCBI上已报道的Pseudomonas mendocina NK-01的全基因组DNA序列(Genebank登录号为:CP002620.1),设计了一对引物,上游引物:5’-ATCGGAATTCATGGCCAGTCTTCC GCTGTAT-3’和下游引物:5’-ATATGCGGCCGCCTA GCCCTTCATCGGCGCATC-3’(分别含有EcoRI和NotI酶切位点)。以提取的门多萨假单胞菌基因组DNA为模板进行PCR扩增。PCR反应体系:DNA模板1μL(约50ng),上、下游引物各2μL,2×Pfu Master Mix 50μL,用ddH2O补充到总体积为100μL。PCR反应条件:94℃预变性5min后,分别于94℃变性50s,58℃退火50s和72℃延伸1min,共33个循环,最后在72℃延伸10min。PCR产物经凝胶电泳鉴定正确后切胶纯化存于冰箱中-20℃备用。
1.2.3 表达载体pPICZαA-pml的构建 pPICZαA质粒和目的基因PCR产物都用EcoRI和NotI进行双酶切,切胶纯化后,用T4DNA连接酶16℃过夜连接酶切产物,然后用CaCl2转化法转入E.coli Top10,在含25μg/mL Zeocin的LB培养基平板上涂板,过夜培养。挑取阳性转化子过夜培养后提取质粒,进行双酶切鉴定。将阳性质粒提交华大基因公司进行序列测定。
1.2.4 序列分析 核酸和氨基酸序列的比对在NCBI BLAST(http://www.ncbi.nlm.nih.gov)上进行,核酸序列分析用DNAman 5.0完成,蛋白结构与功能的预测在Swissprot(http://www..expasy.ch/sprot)网站上提供的链接的平台上进行。
1.2.5 重组毕赤酵母的构建、诱导表达以及酶活检测 重组质粒用限制性内切酶SacI进行线性化后,使用电转化法转化到毕赤酵母X-33,取转化产物涂布于含有100μg/mL的Zeocin YPD平板上,30℃培养2~3d,进行菌落PCR鉴定。挑取经菌落PCR鉴定的转化子,接种于含有50mL BMGY培养基的250mL三角瓶中,30℃,250r/min振荡培养16~20h至OD600达到2~6。离心收集菌体,再将其重悬浮于BMMY培养基中,稀释至OD600为0.8~1.0,继续振荡培养,每隔24h向BMMY培养基中补加甲醇至终浓度为1.0%进行诱导表达,同时测定重组菌的菌体浓度和脂肪酶酶活。脂肪酶活性定量测定采用碱滴定法,具体步骤参照文献[9]中报道的方法。
2 结果与分析
2.1 门多萨假单胞菌脂肪酶基因pml的克隆及序列比对
以提取的门多萨假单胞菌基因组DNA为模板,用设计的引物进行PCR扩增,经琼脂糖凝胶电泳可见PCR产物大小约为820bp,大小与理论值相符(见图1)。将该目的片段回收并克隆入表达载体pPICZαA,提取重组质粒进行EcoRI和NotI双酶切鉴定,片段大小正确(见图2),测序结果表明,pml基因全长为801bp,起始和终止密码子分别为ATG和TAG,推测其完整的ORF编码267个氨基酸。利用NCBI网站上的BLAST软件分析表明,推测的门多萨假单胞菌脂肪酶的氨基酸序列同其他已知细菌的脂肪酶氨基酸序列有较高的相似性。其中最高的为Pseudomonas mendocina NK01(相似性为97%),其次为Pseudomonas mendocina DLHK(相似性为82%)。采用DNAman 5.0进行同源比对分析(见图3),发现门多萨假单胞菌脂肪酶基因pml的氨基酸序列中含有高度保守的“Gly-X-Ser-X-Gly”五肽功能元件。研究表明,该功能元件在脂肪酶识别和结合底物方面发挥着重要的作用[10]。同时在该脂肪酶氨基酸序列中还发现了构成脂肪酶活性中心催化活性位点的三个氨基酸残基(Ser-Asp-His),它们分别位于156、212、247位。通过以上分析表明从门多萨假单胞菌中克隆得到了完整的脂肪酶基因。

图1 PCR扩增电泳图Fig.1 Electrophoretogram of PCR

图2 重组质粒的酶切鉴定Fig.2 Digestion and identification of recombinant plasmid
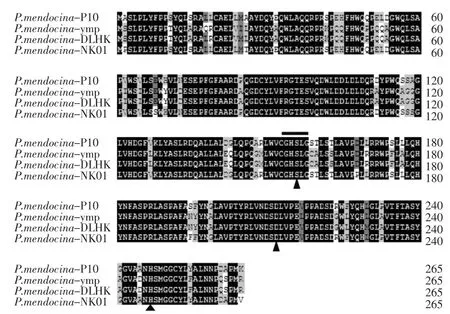
图3 脂肪酶氨基酸序列多重比对图Fig.3 The multiplex alignment of the lipase
2.2 门多萨假单胞菌脂肪酶基因pml生物信息学分析
门多萨假单胞菌脂肪酶基因pml编码297个氨基酸,利用在线软件Protparam预测表明,该脂肪酶的等电点和相对分子量大小分别为4.82、29.95ku,半衰期理论值为30h,不稳定参数为61.80,属于不稳定蛋白质。利用在线网站SinglelP4.0预测信号肽定位,结果显示该氨基酸序列不含有信号肽。采用在线软件ProtScale分析此蛋白的疏水性和亲水性,发现第21位的丙氨酸和22位的谷氨酸的疏水性最强(分值为2.189),位于第41位的精氨酸亲水性最强(分值为-2.967)。用SOPMA对此蛋白质的二级结构进行了预测,在该蛋白质的二级结构中无规则卷曲的含量最大为47.74%,其次为α螺旋含量为34.59%,延伸链的含量为15.41%,而β转角的含量最少为2.26%。通过NCBI网站上的conserve domain对该氨基酸序列进行保守结构域分析,结果显示该序列属于脂肪酶超家族中的第三类脂肪酶。
2.3 重组毕赤酵母的构建以及筛选
Zeocin抗性平板上长出数百个转化子(见图4),随机挑取单菌落进行菌落PCR,阴性对照和阳性对照分别以毕赤酵母X-33的基因组DNA和构建好的脂肪酶表达载体为模板进行PCR扩增,结果如图5所示。重组菌和阳性对照都能扩增出一条820bp大小的条带,与预期大小相符,表明重组质粒已成功整合到了毕赤酵母基因组中。

图4 拟转化子Fig.4 Putative transformants

图5 拟转化子PCR鉴定Fig.5 PCR detection of putative transformants
2.4 重组转化子脂肪酶基因的诱导表达及酶活测定
按照方法“1.2.5”对筛选的重组毕赤酵母进行诱导培养,每隔24h取样测量发酵液的菌体浓度和上清发酵液的脂肪酶酶活。结果如图6所示,诱导到168h时,菌体浓度达到最高,菌体浓度为432g/L;诱导至192h时,酶活力达到最大为8U/mL。

图6 重组毕赤酵母生长曲线和产酶曲线Fig.6 Cell growth and lipase production of recombinant yeast
3 结论
采用同源克隆法从门多萨假单胞菌中克隆得到脂肪酶基因,该基因全长801bp,编码267个氨基酸。通过对该脂肪酶基因的序列及其编码的氨基酸进行分析发现:该脂肪酶基因与已报道的脂肪酶基因具有很高的相似性,相似性最高可达97%;同时在该脂肪酶氨基酸序列中还存在“Gly-X-Ser-X-Gly”五肽功能元件以及脂肪酶活性中心催化活性位点的三个氨基酸残基(Ser-Asp-His),通过保守区域分析,该脂肪酶适于脂肪酶超家族中的第三类脂肪酶。将该脂肪酶基因连接到pPICZαA表达载体上转化Pichia pastoris X-33,脂肪酶基因在Pichia pastoris X-33中实现了分泌性表达。当诱导培养时间诱导到168h时,菌体浓度达到最高,菌体浓度为432g/L;诱导至192h时,酶活力达到最大为8U/mL。
[1]Jaeger K E,Eggert T.Lipases for biotechnology[J].Curr Opin Biotechnol,2002,13(4):390-397.
[2]Shi B,Zeng L,Song H,et al.Cloning and Expression of Aspergillus tamarii FS132 Lipase Gene in Pichia pastoris[J].Int J Mol Sci,2010,11(6):2373-2382.
[3]颜兴和,王栋,徐岩.根霉脂肪酶的研究进展[J].工业微生物,2005,35(3):45-49.
[4]张博,杨江科,闫云君.假单胞菌脂肪酶的研究进展[J].生物技术通报,2007,2:52-57.
[5]Gupta R,Gupta N,Rathi P.Bacterial lipases:an overview of production,purification and biochemical properties[J].Appl Microbiol Biotechnol,2004,64(6):763-781.
[6]Chung GH,Lee YP,Jeohn GH,et al.Cloning and nucleotide sequence ofthermostable lipase gene from Pseudomonas fluorescens SIK W1[J].Agric Biol Chem,1991,55(9):2359-2365.
[7]Ogino H,Hiroshima S,Hirose S,et al.Cloning,expression and characterization of a lipase gene(lip3) from Pseudomonas aeruginosa LST-03[J].Mol Genet Genomics,2004,271(2):189-196.
[8]Jorgensen S,Skov KW,Diderichsen B.Cloning,sequence,and expression of a lipase gene from Pseudomonas cepacia:lipase production in heterologous hosts requires two Pseudomonas genes[J].J Bacteriol,1991,173(2):559-567.
[9]Saxena R K,Davidson W S,Sheoran A,et al.Purification and characterization of an Alkaline thermostable lipase from Aspergillus carneus[J].Process Biochem,2003,39(2):239-247.
[10]Tan Y,Miller K J.Cloning,expression,and nucleotide sequence of a lipase gene from Pseudomonas fluorescens B52[J].Appl Environ Microbiol,1992,58(4):1402-1407.
