黄海冷水团可培养细菌的多样性研究
2011-09-13刘吉文张晓华
王 红,刘吉文,张晓华
(中国海洋大学海洋生命学院海洋生物遗传育种教育部重点实验室,山东青岛266003)
黄海冷水团可培养细菌的多样性研究
王 红,刘吉文,张晓华**
(中国海洋大学海洋生命学院海洋生物遗传育种教育部重点实验室,山东青岛266003)
本文研究了2008年7月黄海冷水团海域可培养细菌的多样性。AODC和DVC计数总菌数和活菌数分别为(1.3~4.8)×105/mL和(0.6~1.6)×105/mL;2216E平板以菌落计数法可培养细菌浓度为(0.56~2.2)×103cfu/mL。从分离到的475株中选取163株进行16S rDNA扩增,并用HhaⅠ酶进行ARDRA(扩增性rDNA限制酶切片段分析)多态性分析,取114株不同带型测序。结果显示,冷水团细菌归为4个细菌类群:变形菌门(Proteobacteria)、放线菌门(Actinobacteria)、厚壁菌门(Firmicutes)和拟杆菌门(Bacteroidetes),共24个属,其中包括α-,β-,γ-变形菌纲;非冷水团细菌也归为这4个门类,共15个属,但未分离到β-变形菌纲。γ-变形菌纲在冷水团和非冷水团区域的不同深度都是优势菌群,不同的是冷水团区域α-变形菌纲比例较高(26.8%),而非冷水团区域α-变形菌纲比例相对较低(15.6%)。这表明黄海冷水团和非冷水团区域细菌多样性都很丰富,但其群落组成和优势菌群有所不同。此外,16S rDNA测序结果表明,6株细菌可能为海洋细菌新种。
可培养细菌;多样性;16S rDNA分析;黄海冷水团
黄海冷水团(Yellow Sea Cold Water Mass,YSCWM)是我国陆架浅海上1个重要的海洋现象,多年来一直为我国海洋学家所关注[1-2]。7~8月,冷水团达鼎盛期,9月以后,随垂直混合逐渐加深而消失。其主要特征是温度低且温差大,盐差小,温度变化范围通常约为5~12℃,盐度变化范围通常约为31.5~32.5。黄海冷水团是由冬季进入黄海的外海水与沿岸水混合而成,并因海面的冷却作用而下沉到深底层,冬季呈垂直均匀状态[3-5]。
ARDRA(Amplified rDNA restriction analysis,扩增性rDNA限制酶切片段分析)是基于不同种群16S rDNA序列的差异,用限制性内切酶对PCR扩增产物进行酶切,然后进行凝胶电泳分离,每种微生物可得到特异酶切指纹图谱,以此来区分是否属于同一种微生物[6]。16S rDNA序列分析主要是基于已建立的基因序列数据库,用于确定细菌的系统发育关系及未知菌的鉴定,目前已被广泛应用于海洋微生物多样性的研究[7]。本研究利用ARDRA对平板分离得到的黄海冷水团和非冷水团细菌进行聚类,然后对不同带型的细菌用16S rDNA序列分析进行鉴定,分析比较了冷水团和非冷水团可培养微生物的群落组成和优势菌群,一方面有助于了解冷水团的存在对微生物多样性产生的影响,另一方面可以丰富我国海洋微生物菌种库的菌种资源。
1 材料与方法
1.1 样品采集
2008年7月21~2008年8月2日随中国科学院海洋研究所和青岛市科技局联合资助的“2008年中国近海海洋科学考察开放航次”出海采集海水样品,该航次由中国科学院海洋研究所的“科学三号”考察船执行。采样站如图1所示。采样点的物理、化学信息如表1。分别在3500-5,3600-6,3600-8,3400-6和3800-2站点(冷水团区域)以及3200-3和3850-5站点(非冷水团区域),取0,10,30 m深度的海水,同时在3500-5、3600-6和3400-6站点也取了60 m深度海水,用CTD采水器采集海水样品。取原样、1/10和1/100 3个稀释度各100μL在2 h之内涂布2216E平板。
1.2 培养基与试剂
2216E培养基:蛋白胨5 g,酵母膏1 g,FePO4微量,琼脂20 g,海水1 000 mL,p H=7.6~7.8,121℃灭菌20 min。
试剂均为分析纯产品,限制性内切酶、PCR试剂等均购自TaKaRa公司。
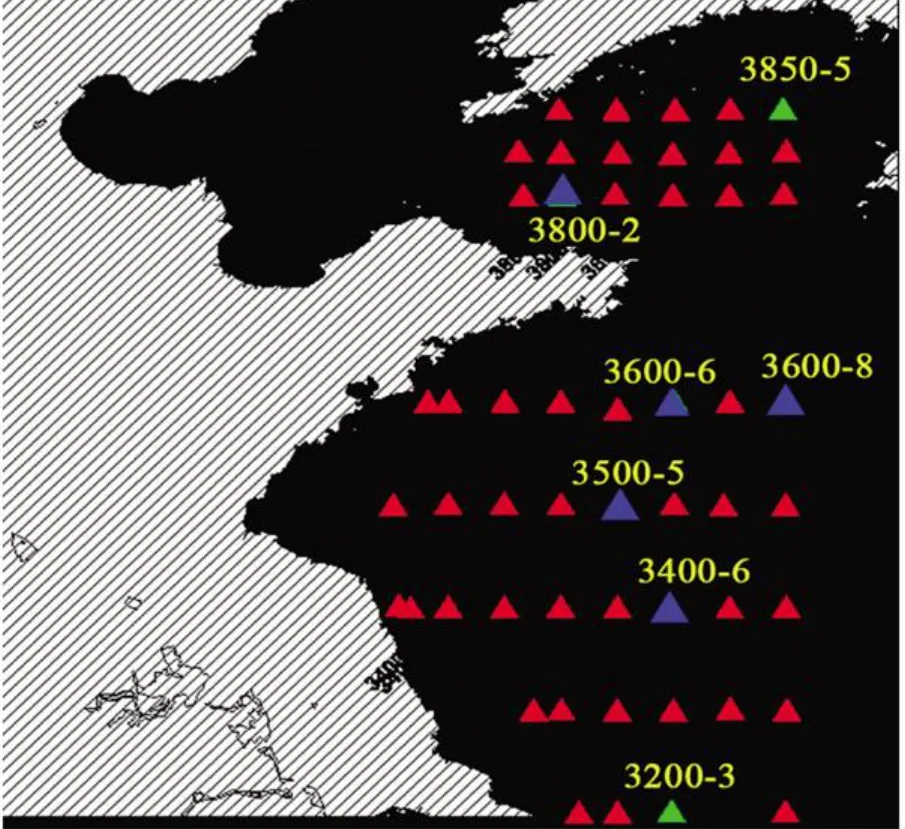
图1 采样站位Fig.1 Map of sampling stations

表1 采样站位的物理、化学信息Table 1 Physical and chemical information of sampling stations
1.3 实验方法
1.3.1 细菌计数及可培养细菌的分离纯化
(1)海水中细菌总数的测定用吖啶橙染色直接计数法(AODC)[8]测定。取一定量水样(1 mL),用吖啶橙(4~7滴)染色(3~5 min),用伊拉克黑预染的孔径为0.25μL的滤膜抽滤,将滤膜置于荧光显微镜下检测,随机计数至少10个视野,换算出样品中所含的细菌数量。
(2)海水中活菌数测定用活菌直接镜检计数法(DVC)[9]测定。吸取1 mL海水样品到1.5 mL的EP管中,向管中加入0.01 mL 0.2%萘啶酮酸和0.01 mL 2.5%酵母膏溶液,混合均匀后,将EP管放入28℃温箱中培养6 h之后,取出EP管加入0.05 mL甲醛固定水样。然后按AODC法用吖啶橙染色,荧光显微镜计数。视野中那些长大或变粗的发橙红色荧光的是活菌。
(3)平板计数及分离培养。用无菌海水对样品进行10倍系列稀释,涂布2216E琼脂平板,每个样品涂布3个平板,每个平板涂布0.1 mL稀释液,在28℃培养箱中培养4 d进行计数。根据菌落形态等特征挑取不同菌落,在2216E平板上划线纯化,每个菌落纯化至少2次。用4.5 mL灭菌保种液(85%生理盐水+15%甘油)将分离纯化的菌株保存于-80℃的超低温冷冻箱中。
1.3.2 DNA提取 根据菌落的大小、颜色、透明度、边缘是否规则、湿润或干燥、菌落突起、凹陷或平坦等指标选取特征不同的菌株用酚-氯仿法进行基因组DNA的提取。方法参考Ausubel等[10]。
1.3.3 16S rDNA扩增 扩增引物:上游引物27F: 5’-AGAGTTTGATCCTGGCTCAG-3’;下游引物1492R:5’-GGTTACCTTGTTACGAC TT-3’。引物由上海生工生物工程有限公司合成。PCR反应条件为: 94℃预变性5 min;94℃变性1 min;55℃退火1 min 30 s; 72℃延伸1 min,30个循环;72℃延伸10 min。PCR产物用1.0%琼脂糖凝胶电泳检测,点样量为5μL。
1.3.4 ARDRA分析 将PCR产物用HhaⅠ进行酶切。酶切反应体系如下:10×Buffer 1μL、HhaⅠ1.5 μL、16S rDNA扩增产物5μL、双蒸水7.5μL,使反应体系为15μL。37℃水浴2 h。酶切产物用2.0%琼脂糖凝胶50V电泳1 h检测。
1.3.5 16S rDNA序列测定及构建系统树 根据ARDRA结果将所有的菌株进行归类,将各大类菌株的16S rDNA PCR产物送上海博尚生物公司进行测序,将测得的序列与GenBank数据库中的基因序列进行相似性比较,用MEGA软件构建系统进化树。将获得的16S rDNA序列提交GenBank,并获得序列号。
2 结果与分析
2.1 海水样品细菌计数
冷水团3个站点不同深度海水样品中,每毫升海水中的总菌数、活菌数及可培养细菌的数目如表2所示。总菌数为(1.3~4.8)×105/mL,活菌数为(0.6~1.6)×105/mL,可培养细菌数为(0.56~2.2)×103cfu/mL,活菌的比例为33.3%~46.1%。
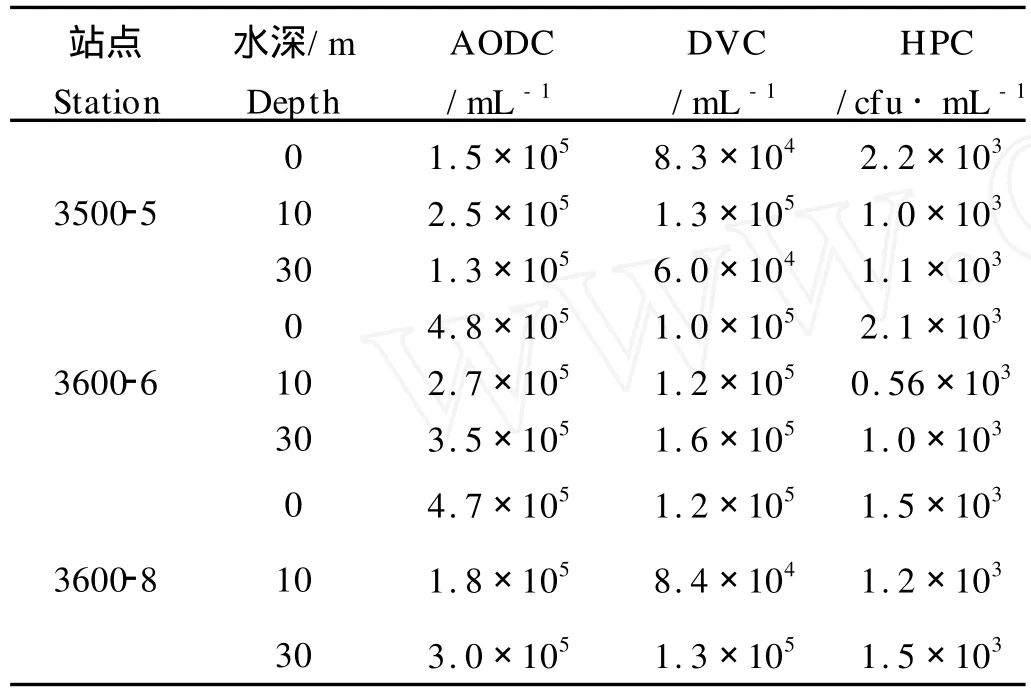
表2 黄海冷水团3个站点海水样品细菌计数Table 2 Enumeration of bacteria in seawater of 3 stations inter of YSCWM
2.2 细菌菌株DNA的提取和PCR扩增
分别从冷水团3500-5站点挑选51株菌,3600-6站点19株菌,3600-8站点18株菌,3400-6站点19株菌, 3800-2站点9株菌,非冷水团3200-3站点27株菌,3850-5站点20株菌提取的DNA电泳可见清晰的总DNA条带,获得的提取物经PCR扩增后电泳均能得到约1.5 kb的单一条带(见图2),表明扩增产物无明显非特异性扩增现象。下图为部分菌株16S rDNA扩增图谱。

图2 部分菌株16S rDNA PCR条带图谱Fig.2 16S rDNA PCR electrophoresis for part of strains
2.3 细菌菌株ARDRA分析
将这163株菌的16S rDNA扩增产物分别经HhaⅠ酶切,不同站点分离的菌株所获得的带型数见表3。部分菌株的酶切图谱见图3,可以看出每个菌株的带型都在7种以上,说明HhaⅠ酶酶切条带较多,有较高的分辨率。菌株WH120和WH113 2种酶切带型在酶切条带图中非常常见(见图3),通过鉴定它们分别是γ -变形菌纲的交替单胞菌属(A lteromonas)和假交替单胞菌属(Pseudoalteromonas),从而证明了海水中γ-变形菌纲占优势。

表3 不同站点水样分离的菌株数和ARDRA带型数Table 3 Numbers of strains isolated from different stations and numbers of ARDRA patterns
2.4 冷水团和非冷水团区域菌株的多样性比较
7个站点每个带型各取1-2株代表菌株共114株进行测序,将获得的序列运用Blast程序与GenBank数据库中已有的细菌16S rDNA序列进行相似性比较,获得菌株的分类地位。冷水团和非冷水团海水分离菌株所属类群见表4。114株细菌的序列均已提交GenBank,获得的序列号为FJ866642-FJ866748和FJ847830-FJ847836。
分别选取冷水团和非冷水团区域的部分菌株,与GenBank数据库中相似性较高的序列建立系统发育进化树,见图4、图5。
根据比对结果,黄海冷水团区域5个站点的82个测序菌株分布在4个细菌类群:变形菌门(Proteobacteria)、放线菌门(Actinobacteria)、硬壁菌门(Firmicutes)和拟杆菌门(Bacteroidetes)(见表4和图4),共24个属,其中变形菌门的菌数最多,其中γ-变形菌纲47株,9个属;α-变形菌纲22株,8个属;β-变形菌纲3株,3个属;硬壁菌门6株,2个属;放线菌门2株,1个属;拟杆菌门1株,1属。非冷水团32株测序的菌株也有4个细菌类群:变形菌门、放线菌门、硬壁菌门和拟杆菌门(见表4和图5),同样主要由变形菌门的细菌构成,共15个属,其中γ-变形菌纲21株,8个属;α-变形菌纲5株,4个属;硬壁菌门占有4株,1个属;放线菌门和拟杆菌门各1株,1属。细菌种类比非冷水团区域多。拟杆菌门在冷水团分离到的1株菌为黄杆菌纲(Flavobacteria),非冷水团分离到的为鞘脂杆菌纲(Sphingobacteria)。以上均说明冷水团区域和非冷水团区域细菌多样性都很丰富,但其群落组成有所不同。

图3 部分菌株16S rDNA PCR产物HhaⅠ限制性酶切条带Fig.3 Parts of RFLP patterns derived from the digestion of 16S rDNA PCR products byHhaⅠ

表4 冷水团和非冷水团海水分离菌株所属类群Table 4 Taxonomy position of strains isolated from inter and exterior of YSCWM
2.5 不同深度海水中细菌多样性比较
7个站点不同深度海水分离菌株所属类群见表5, γ-变形菌纲在不同深度海水中都是优势菌群,然而不同深度海水中其他菌群的分布有所差异:表层海水(0 m)中α-变形菌纲的种类数量较多;中层海水(10 m),厚壁菌门有较高的丰度;深层海水(60 m)中除了γ-变形菌纲外,优势菌群不明显,α-变形菌纲、β-变形菌纲,硬壁菌门和拟杆菌门均有分布。
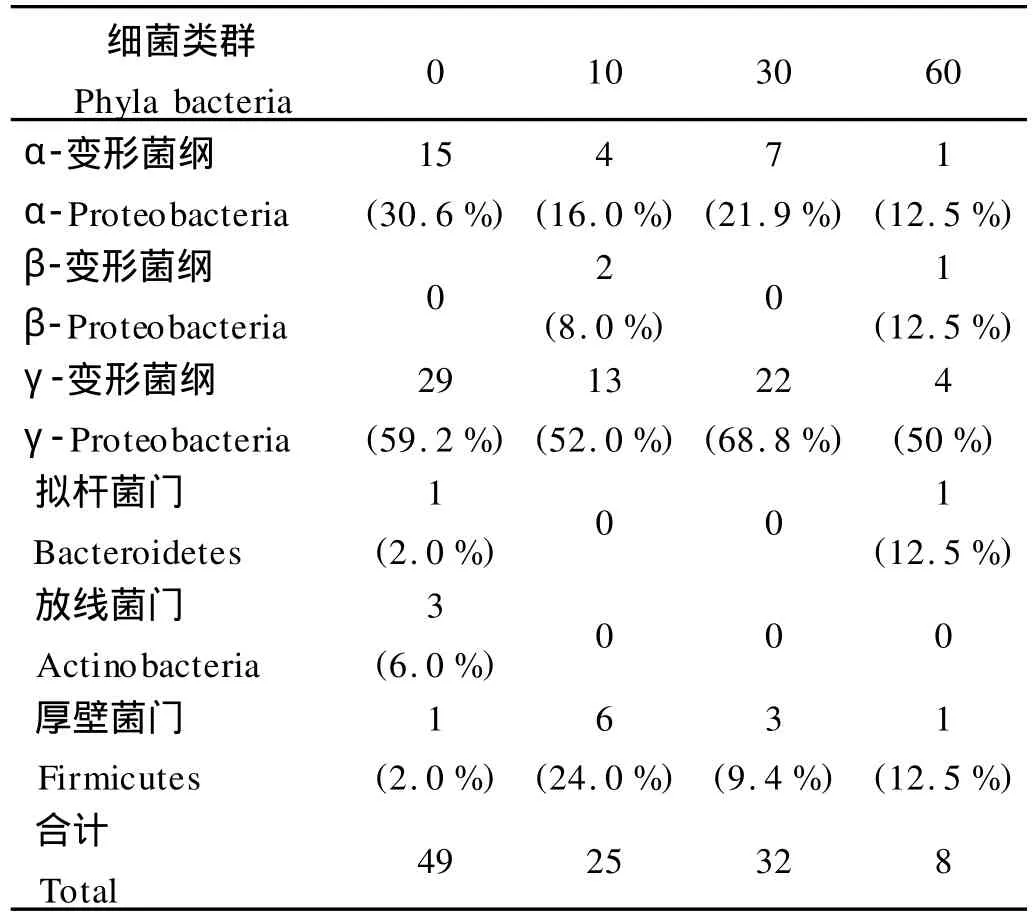
表5 7个站点不同深度海水分离菌株所属类群Table 5 Taxonomy position of strains isolated from different depths of seven stations/m
冷水团变形菌门包括α-,β-,γ-变形菌纲(见表4),其中β-变形菌纲分离自3500-5站点10,60 m深处。β-变形菌纲包括3个属Achromobacter(无色菌属), Delf tia(戴尔福特氏菌属)和L imnobacter(湖沼杆菌属)。而非冷水团区域没有分离到β-变形菌纲。冷水团区域特有的3个属为Stenotrophomonas(寡养单胞菌属),A lcanivorax(食烷菌属)和Oceanobacter(大洋杆菌属),而非冷水团区域特有的2个属为A garivorans(食琼脂菌属)和Melitea。α-变形菌纲在冷水团区域特有的4个属为Paracoccus(副球菌属),Ruegeria(鲁杰氏菌属),Oceanicaulis(大洋柄菌属)和Phaeobacter(褐杆菌属),说明冷水团区域α-变形菌纲

图4 冷水团菌基于16S rDNA用Mega 4.0建的Neighbor-joining进化树Fig.4 Neighbor-joining tree showing the relationships between strains isolated from the inter of YSCWM and various reference taxa

图5 非冷水团菌基于16S rDNA用Mega 4.0建的Neighbor-joining进化树Fig.5 Neighbor-joining tree showing the relationships between strains isolated from the exterior of YSCWM and various reference taxa
3 讨论
分离海洋微生物常用的培养基有2216E培养基, TCBS培养基(弧菌培养基),海水琼脂培养基,R2A培养基等。由于不同的微生物所需要的营养不同,任何1种培养基都不能分离培养出所有的海洋细菌。2216E培养基主要用于分离和培养海洋异养好氧细菌。本实验用AODC、DVC和2216E平板计数法计数黄海冷水团海水中的总菌数、活菌数和可培养细菌数,实验结果显示活菌的比例为33.3%~46.1%,说明活菌的比例较高,但可培养细菌的比例较低,因此作者所做的可培养细菌多样性与真正的细菌多样性仍有差别。
使用ARDRA分析细菌群落系统发育,多选取2~5种限制性内切酶[11]。使用1种限制性内切酶得到的相同带型菌株,可能会被另一种酶切出不同的带型。本文使用HhaⅠ酶切,是1种识别四碱基对的限制性内切酶,虽然只有1种限制酶切,但此酶分辨率较高,对冷水团区域和非冷水团区域的菌株都有较多的酶切带型,能显示出两者均有较高的微生物多样性。
1997年Pinhassi等[12]用培养方法发现波罗的海北部沿岸海域浮游细菌的优势菌群是α-变形菌纲,γ-变形菌纲,β-变形菌纲和拟杆菌门;1999年,Lasse等[13]用PCR-DGGE分析阿拉伯海域的细菌群落,发现主要为δ-变形菌纲和放线菌门,并没有发现γ-变形菌纲和拟杆菌门;同一年,Christoph等[14]利用同样的方法研究地中海西部海水,发现细菌群落组成包括α-变形菌纲,β-变形菌纲和拟杆菌门。可以看出不同海域的细菌群落组成不同,可能与各自的环境特征有关。黄海冷水团的微生物群落组成,囊括了α-变形菌纲,γ-变形菌纲,β-变形菌纲,放线菌门和拟杆菌门群落,但是放线菌门,拟杆菌门和β-变形菌纲种类数量很少,γ-变形菌纲为优势种群。这些差异可能既与海区有关,也与冷水团的生态特殊性相关。
刘敏等[15]直接提取黄海冷水团水样混合细菌的总基因组,利用PCR-DGGE技术分析了黄海冷水团非培养细菌的群落组成,而本研究采用的是纯培养方法,所得到的结果与他们的结果有一定的差异。刘敏等[15]没发现黄海冷水团水样有α-变形菌纲、放线菌门和厚壁菌门的存在,但发现有δ-变形菌纲存在,而且刘敏等[15]发现冷水团不同深度水域细菌群落构成与本实验得到的结果也有一定的差异。这些结果说明,虽然非培养方法在某些方面优于纯培养方法,从理论上来讲其得到的细菌多样性应远远高于纯培养方法,但是由于多种原因使其结果具有一定的局限性。比如对混合细菌的总基因组进行PCR扩增时,16S rDNA的通用引物经常对不同类群的细菌基因组有不同的亲和性,使其不能够反应真正的多样性。如果需要深入地研究某一特殊区域的微生物群落多样性,需要纯培养方法与非培养方法相结合,才能得到更加准确的结果。
菌株WH020-1,WH063,WH82,WH134, WH169和WH278在使用BLAST进行比对时,与最相近菌种的相似性分别为97.6%,97.9%,97.0%, 96.6%,96.2%和96.8%可能为未被鉴定的新种。其中与WH20-1最相近菌株的分类地位为变形菌门,β-变形菌纲,产碱杆菌科(Alcaligenaceae),无色杆菌属(Achromobacter);与WH63,WH82,WH169和WH278最相近的菌株为同一个菌种,其分类地位为变形菌门,γ-变形菌纲,交替单胞菌科(Alteromonadaceae),海滩杆菌属(Aestuariibacter);与WH134最相近的菌株为变形菌门,γ-变形菌纲,弧菌科(Vibrionaceae),弧菌属(Vibrio)。相似性低仅为新菌的一个鉴定标尺,为了确定这些菌株是否为新种,还需要做进一步的生理生化反应鉴定。目前菌株WH169已被鉴定为海滩杆菌属新种——凝集海滩杆菌(Aestuariibacter aggregatussp.nov.)[16],而WH134已被鉴定为弧菌属新种——冷水团弧菌(Vibriomarisf lavisp. nov.)[17]。
[1] 刁焕祥,沈志良.黄海冷水域水化要素的垂直分布特征[J].海洋科学集刊,1985,25:41-51.
[2] 王保栋.黄海冷水域生源要素的变化特征及相互关系[J].海洋学报,2000,22(6):47-54.
[3] Su J L,Huang D J.On the current field associated with the Yellow Sea Cold Water Mass[J].Oceanologr Limnol Sin supplement,1995,26(5):1-7.
[4] 翁学传,张以恳,王从敏,等.黄海冷水团的变化特征[J].青岛海洋大学学报,1989,19(1)Ⅱ:119-131.
[5] Zhao B.The fronts of the Huanghai Sea cold water mass induced by tidal mixing[J].Oceanologr Limnol Sin,1985,16(6):451-459.
[6] 陈晓蕾,张忠泽.微生物的ARDRA检测[J].微生物学杂志, 1999,19(4):40-43.
[7] 薛超波,王国良,金珊,等.海洋微生物多样性研究进展[J].海洋科学进展,2004,22(3):377-384.
[8] Hobbie J E,Daley,R J,Jasper S.Use of nucleopore filters for counting bacteria by fluorescence microscopy[J].Appl Environ Microbiol,1977,33:1225-1228.
[9] Könneke M,Bernhard A E,Torre J R,et al.Isolation of an autotrophic ammonia-oxidizing marine archaeon[J].Nature,2005, 437:543-546.
[10] Ausubel F M,Brent R,Kingston R E,et al.Short protocols in molecular biology:a compendium of methods from current protocols in molecular biology[M],3rd edn.New York:Wiley. 1995.
[11] 柴丽红,王涛,崔晓龙,等.青海柯柯盐湖16株细菌的ARDRA筛选及系统发育初步分析[J].云南大学学报:自然科学版, 2003,25(6):541-544.
[12] Pinhassi J,Zweifel U L,Hagstrom A.Dominant marine bacterioplankton species found among colony-forming bacteria[J].Applied and Environmental Microbiology,1997,63(9):3359-3366.
[13] Lasse R,Grieg F S,Laura B F,et al.Bacterial community composition during two consecutive NE Monsoon periods in the Arabian Seastudiedbydenaturinggradientgelelectrophoresis (DGGE)of rRNA genes[J].Deep-Sea ResearchⅡ,1999,46 (6):1791-1811.
[14] Christoph MH,Sabine R,Dietmar V,et al.Investigation of 0.2 μm filterable bacteria from the Western Mediterranean Sea using a molecular approach:dominance of potential starvation forms [J].FEMS Microbiology Ecology,1999,31:153-161.
[15] 刘敏,朱开玲,李洪波,等.应用PCR-DGGE技术分析黄海冷水团海域的细菌群落组成[J].环境科学,2008,29(4):1082-1090.
[16] Wang Y,Wang H,Liu J,et al.Aestuariibacter aggregatussp. nov.,a moderately halophilic bacterium isolated from seawater of the Yellow Sea[J].FEMS Microbiology Letters doi:10.1111/j. 1574-6968.2010.02011.x.
[17] Wang H,Liu J,Wang Y,et al.Vibrio marisf lavisp.nov.,a novel marine bacterium isolated from seawater near the Yellow Sea Cold Water Mass,China[J].The International Journal of Systematic and Evolutionary Microbiology doi:10.1099/ijs.0. 022285-0.
Abstract: Phylogenetic diversity of cultivable bacteria from the inter of the Yellow Sea Cold Water Mass (YSCWM)area was studied in July 2008,and was compared with that from the exterior of YSCWM area. Sea water samples were collected in five stations 3500-5,3600-6,3600-8,3400-6 and 3800-2 from the inter of YSCWM,and two stations 3200-3 and 3850-5 from the exterior of YSCWM.According to the AODC and DVC results,the total bacterial number and the total number of viable cells of the marine water samples were(1.3~4.8)×105/mL and(0.6~1.6)×105/mL,respectively;from the colony count method with 2216E plates,the abundance of the heterotrophic bacteria of sea water was(0.56~2.2)× 103cfu/mL.475 bacterial strains were isolated from 2216E plates,and the PCR-ARDRA technique was performed on these strains for cluster analysis,with fragments of 16S rDNA digested by restriction enzymeHhaⅠ.The 16S rDNA of all major RFLP patterns were sequenced.Phylogenetic analysis of 114 bacterial strains showed that bacterial phylotypes from the inter of YSCWM were made up of four phyla bacteria,i.e.Proteobacteria,Actinobacteria,FirmicutesandFlavobacteria,including 24 genera,and from the exterior were made up of the same four taxon,including 15 genera.α-,β-andγ-Proteobacteria subdivisions of theProteobacteriawere discovered from the inter of YSCWM.Howeverβ-Proteobacteria was not discovered from the exterior of YSCWM.γ-Proteobacteriarepresented the vast majority of bacterial isolated at every depth.By comparing,α-Proteobacteriarepresented higher proportion with percentage of 26.8%inter of YSCWM and lower proportion with percentage of 15.6%exterior of YSCWM. These indicated that samples from the inter as well as the exterior of YSCWM showed high bacterial diversity,but bacterial community composition,as well as the predominant bacterial groups,from the inter of YSCWM was different from the exterior of YSCWM.In addition,according to the 16S rDNA gene sequences analysis,6 strains might be novel species.
Key words: cultivable bacteria;diversity;16S rDNA analysis;YSCWM
责任编辑 于 卫
Diversity of Cultivable Bacteria in the Yellow Sea Cold Water Mass
WANG Hong,LIU Ji-Wen,ZHANG Xiao-Hua
(Key Laboratory of Marine Genetics and Breeding,Ministry of Education,College of Marine Life Sciences,Ocean University of China,Qingdao 266003,China)
Q938.8
A
1672-5174(2011)06-061-07
国家高技术研究发展计划项目(2007AA09Z434)资助
2010-05-17;
2010-11-22
王 红(1984-),女,硕士生,研究方向:海洋微生物学。
**通讯作者:E-mail:xhzhang@ouc.edu.cn
