菊粉酶基因在酿酒酵母中的表达及乙醇发酵
2011-02-09李楠楠袁文杰王娜辛程勋葛旭萌白凤武
李楠楠,袁文杰,王娜,辛程勋,葛旭萌,白凤武
1 大连理工大学 生命科学与技术学院,大连 116024
2 大庆九环生物能源有限公司,大庆 163511
目前国内外燃料乙醇主要使用糖质和淀粉质原料生产,随着陈化粮的消耗殆尽,燃料乙醇需求的增长,寻找非粮原料生产燃料乙醇已成为我国燃料乙醇产业发展的基本方向。
菊芋Jerusalem artichoke耐瘠薄、耐盐碱、抗干旱、生物质产量高,其主要成分是菊粉,占块茎干重的60%~70%,由果糖残基经β-2,1糖苷键脱水聚合而成,末端为一个葡萄糖分子[1],可以通过酸解和酶解等途径转化为易于发酵的果糖[2]。与目前国内外普遍关注的秸秆类生物质相比,不需要复杂的预处理,且水解产物也不含难以利用的五碳糖[3-4],是我国燃料乙醇及其他生物基化学品生产的良好粮食替代原料。
国内外对利用菊芋块茎生产乙醇进行了大量的文献报道[5-11],其中效果最好的当属克鲁维酵母的一步法发酵工艺,菊芋的糖化和发酵均由克鲁维酵母自身完成,原料不需预先糖化,节约了能耗和额外添加酶的费用[12],但其缺点是克鲁维酵母属于非常规酵母,其乙醇产生机制尚不明了,乙醇耐性较酿酒酵母差,发酵过程乙醇收率仅为 80%左右,终点乙醇浓度 6%~7%,远远低于淀粉质原料乙醇发酵收率达到理论值 90%以上和发酵终点乙醇浓度10%~12%的技术指标,并且发酵速率慢,时间很长,导致发酵罐设备生产强度很低。因此,通过诱变育种或采用基因工程的方法选育能够高效发酵菊芋生产乙醇的菌种是菊芋原料生产燃料乙醇的发展方向,其中最有效的方法就是以乙醇耐性较高的酿酒酵母为受体菌,构建能够高效分泌菊粉酶的基因工程菌株。
在酵母基因组中,ho单拷贝基因编码核酸内切酶,负责完成酵母a型和α型之间的转换,普遍存在于出芽繁殖的酵母中[13],并且该基因是生长非必需的,破坏 ho基因不会影响酵母生长[14]。Warren等[15]构建了以ho起始密码−2720~−1814为左边界,+1 199~+1 699为右边界的整合载体,实现了外源基因在酿酒酵母中的表达。
本研究以工业酿酒酵母作为受体菌株,以ho位点为整合位点,使菊粉酶基因在酿酒酵母中得到表达,进而使酿酒酵母在自身优良的发酵性能基础上利用菊芋粉一步发酵得到乙醇,为菊粉发酵燃料乙醇的工业生产奠定基础。
1 材料与方法
1.1 材料
1.1.1 菌株和质粒
本实验过程中涉及的菌株及质粒列于表1中。
其中酿酒酵母Saccharomyces cerevisiae 6525为本实验用的出发菌株,具有良好的乙醇发酵性能;酿酒酵母Saccharomyces cerevisiae 288c、马克斯克鲁维酵母Kluyveromyces marxianus及大肠杆菌E. coli DH5α为本实验室保存菌种;HI6/6为本实验构建的含有带自身启动子的菊粉酶基因的基因工程菌;HPI6/3为本实验构建的带pgk启动子的菊粉酶基因的基因工程菌;HO-2 (6 062 bp) 质粒为本实验室保存;质粒HO/inu (7 729 bp) 是不带启动子的菊粉酶整合表达载体,为本实验室构建并保存;HO/p-inu (8 349 bp) 是带自身启动子的菊粉酶基因的整合表达载体,为本研究构建;HO/pgk-inu (8 547 bp) 是带pgk启动子的菊粉酶基因的整合表达载体,为本研究构建。
1.1.2 工具酶和化学试剂
引物合成和序列测定委托TaKaRa (大连) 公司完成。Taq DNA聚合酶、限制内切酶、T4 DNA连接酶、DNA marker等购自TaKaRa (大连) 公司;菊粉购自内蒙古亿利生物技术有限公司;菊芋购自山东济宁,用前进行烘干粉碎并过60目筛,经测定,100 g菊芋粗粉中含有 63 g的菊糖。G418购自Sigma公司;DNA纯化试剂盒、质粒提取试剂盒与凝胶回收试剂盒均购自Solarbio公司。

表1 实验菌株和质粒Table 1 Strains and plasmids used in this study
1.1.3 培养基
YPD培养基 (g/L):无水葡萄糖20,酵母浸粉10,蛋白胨20,121 ℃灭菌15 min,用于酵母菌的培养。
YPD选择培养基 (g/L):无水葡萄糖 20,酵母浸粉10,蛋白胨20,121 ℃灭菌15 min,冷却后加入G418,使其终浓度为300 mg/L,用于酵母菌转化子的培养。
LB培养基 (g/L):氯化钠 10,酵母浸粉5,蛋白胨10,pH 7.0,121 ℃灭菌15 min,用于E. coli菌株的培养。
LB选择培养基 (g/L):氯化钠10,酵母浸粉5,蛋白胨10,pH 7.0,121 ℃灭菌15 min,冷却后加入Amp,使其终浓度为100 mg/L,用于E. coli转化子的培养。
菊粉培养基 (g/L):菊粉40,酵母粉4,蛋白胨4,121 ℃灭菌15 min,用于酵母菌转化子的产酶培养。
菊芋粗粉培养基:60目以下的菊芋粗粉加自来水配成的培养基,料水比为1∶4,菊糖浓度为126 g/L,无其他成分添加,未灭菌,用于酵母菌转化子的乙醇发酵。
1.2 方法
1.2.1 菊粉酶基因整合表达载体的构建
根据菊粉酶 (GenBank Accession No. X57202)序列,设计引物p-inu forward primer和p-inu reverse primer,以 K. marxianus基因组为模板扩增 p-inu基因 (引物序列见表2)。PCR程序:94 ℃预变性5 min;94 ℃ 1 min,63.6 ℃ 1 min ,72 ℃ 3 min,30个循环;72 ℃延伸10 min。根据pgk启动子序列(GenBank Accession No. BK006937.1),设计引物pgk forward primer和pgk reverse primer,以酿酒酵母S. cerevisiae 288c基因组为模板扩增pgk启动子基因。PCR程序:94 ℃预变性5 min;94 ℃ 1 min,61 ℃ 1 min ,72 ℃ 1 min,30个循环;72 ℃延伸10 min。PCR产物经胶回收纯化后,分别与载体pMD19-T连接。阳性克隆提取质粒后测序,分别命名为pMD19T/p-inu和pMD19T/pgk。pMD19T/p-inu经 BamHⅠ、BssHⅡ双酶切后与经过同样处理的HO-2进行连接;pMD19T/pgk经BsiWⅠ、BamHⅠ双酶切后与经过同样处理的HO/inu进行连接。连接产物转化E. coli DH5α感受态细胞,在LB选择培养基上培养,挑取单菌落,小量提取质粒酶切鉴定并进行 DNA序列测定,得到整合载体 HO/p-inu和HO/pgk-inu。引物设计利用Pimer Premier 5.0软件,质粒的提取、酶切、连接和E. coli转化均按Sambrook等[16]的方法进行。

表2 本实验所用的引物序列Table 2 Primer sequences used in this study
1.2.2 S. cerevisiae 6525的转化
整合载体HO/p-inu、HO/pgk-inu,经NotⅠ酶切后,进行琼脂糖电泳,回收目的DNA片段,采用电击法转化S. cerevisiae 6525感受态细胞,在含有300 mg/L G418的YPD平板上筛选转化子。详细操作参考BIORAD说明书。
1.2.3 转化子的验证
分别提取 HI6/6及 HPI6/3转化子的基因组DNA。采用PCR法进行验证。HI6/6的验证所使用的引物序列为HI6/6 forward primer和HI6/6 reverse primer。HPI6/3的验证所使用的引物序列为 HPI6/3 forward primer和HPI6/3 reverse primer。
1.2.4 转化子产酶情况考察
取 S. cerevisiae 6525和整合子 HI6/6及HPI6/3各100 μL,接至50 mL的YPD液体培养基中,30 ℃培养24 h后,按10%接种到菊粉培养基,100 mL/250 mL摇瓶,每24 h取样测定生物量、菊粉酶活性。
1.2.5 转化子发酵性能考察
取S. cerevisiae 6525和转化子HI6/6及HPI6/3各100 μL,接至50 mL的YPD液体培养基中,30 ℃培养24 h后,按10%接种到菊芋粗粉发酵培养基中,100 mL/250 mL摇瓶,厌氧发酵,每12 h取样测定菊粉酶的活性、还原糖、总糖及乙醇含量。乙醇对理论值的收率=乙醇/(总糖−剩余总糖)/0.511。
1.2.6 分析方法
还原糖及总糖测定方法:还原糖采用DNS法测定,总糖经酸水解后用 DNS 法测定。
菊粉酶活的测定方法:取适量发酵上清液,用0.1 mol/LHAc-NaAc缓冲液 (pH 4.6) 稀释,取50 μL稀释液加入450 μL 5%菊粉溶液,混匀,60 ℃水浴反应10 min (精确计时),立即取出沸水浴5 min灭活,从反应液中取出50 μL,加入1.5 mL DNS试剂+1.95 mL水,混匀,沸水浴中5 min,冷水冷却,用容量瓶定容至25 mL,测OD540。对应果糖标准曲线计算得样品反应液中的含糖量(mg)。空白对照:取发酵上清稀释液沸水5 min灭活,同上反应,作为对照。菊粉酶活性单位定义:每分钟产生1 μmol果糖所需酶量。
乙醇测定方法:乙醇浓度利用SBA生物传感分析仪 (山东省科学院生物研究所) 测定。
生物量测定采用干重法[17]:取一定体积的样品置于预先烘干称重的离心管中,离心后的沉淀用蒸馏水洗涤2次,然后置85 ℃的恒温烘箱中烘至恒重后称重。
2 结果与分析
2.1 菊粉酶基因整合表达载体的构建
构建菊粉酶基因整合表达载体的图谱如图 1所示。
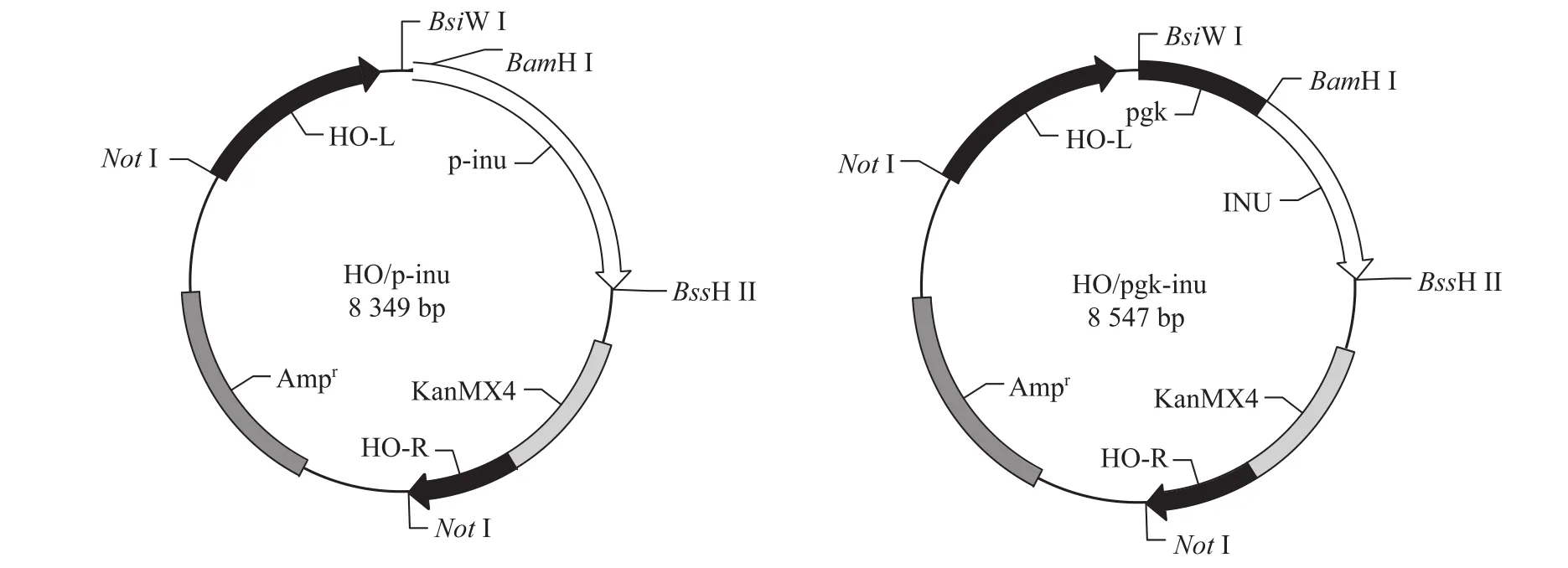
图1 菊粉酶基因整合载体的示意图Fig. 1 Map of the inulinase integrating expression system.
首先以K. marxianus基因组DNA 为模板,采用PCR法扩增p-inu基因,扩增出特异性谱带,大小约为2.4 kb,与预期一致;以S. cerevisiae288c基因组DNA为模板,采用PCR法扩增pgk基因,扩增出特异性谱带,大小约为800 bp,结果如图2所示,与预期一致。经Blast比对,与模板的相似性达100%。HO/p-inu经NotⅠ酶切出现3.1 kb和5.3 kb两条谱带,HO/pgk-inu经NotⅠ酶切出现3.1 kb和5.4 kb两条谱带,结果如图3所示,证明整合载体的连接成功。

图2 PCR法扩增马克斯克鲁维酵母的菊粉酶基因和酿酒酵母288c的pgk启动子基因Fig. 2 Amplification of the p-inu gene from K. marxianus and the pgk gene from S. cerevisiae 288c by PCR. M1: DL2000 DNA marker; 1−3: pgk promoter; 4: p-inu gene; M2: 1 kb marker.

图3 重组质粒的NotⅠ酶切鉴定Fig. 3 Recombinant plasmids digested with Not I. M: 1 kb marker; 1−5: HO/pgk-inu digested with Not I; 6: HO/p-inu digested with Not I.
2.2 S. cerevisiae 6525的转化及转化子验证
NotⅠ酶切载体HO/p-inu、HO/pgk-inu,分别回收5.3 kb和5.4 kb的线性目的片段,电击法转化,涂布含有300 μg/mL G418的YPD培养基。挑取选择平板上的抗性菌落进行培养。对生长出的菌体依次进行基因组的提取和PCR扩增,HO/p-inu转化子扩增的片段包括HO的左边界,菊粉酶基因和kam基因,预期的目标序列的长度为4.8 kb;HO/pgk-inu转化子扩增的片段包括 pgk的后半部分和菊粉酶基因的前半部分,预期的目标序列的长度为1 kb,而出发菌株无整合片段,PCR结果应为阴性。PCR的电泳结果如图4所示。HI6/6和HPI6/3的产物大小与预期相符,对PCR产物进行测序证明外源基因随HO片段与染色体同源区域发生重组而整合到染色体上。基因工程菌在无筛选压力下连续培养20代以上,仍然保持G418抗性,说明整合的基因片段是稳定的。

图4 转化子HI6/6和HPI6/3的PCR验证Fig. 4 Confirmation of the recombinants HI6/6 and HPI6/3 by PCR. M: 1 kb marker; 1: plasmid HO/p-inu; 2, 6: S. cerevisiae 6525; 3: HI6/11; 4: HI6/6; 5: plasmid HO/pgk-inu; 7: HPI6/3; 8: HPI6/4.
2.3 转化子的产酶情况
为了考察转化子由于 ho基因的破坏是否对酵母的生长产生不利的影响及转化子产酶的情况,按照前述的实验方法在摇瓶中对转化子的生长和产酶能力进行测定,结果如图5所示。可见HPI6/3的生物量在24 h达到了6.4 g/L,在168 h时达到9 g/L,几乎与出发菌株生长一致,而 HI6/6的生物量虽然略低于出发菌株与HPI6/3,但终点的生物量仍然达到8.2 g/L,说明转化子的生长状况并没有受到影响。至于产酶情况,转化子 HI6/6的酶活明显高于出发菌株,48 h前酶活没有明显提高,之后酶活迅速提高,在144 h达到最高酶活86 U/mL,是出发菌株的4.5倍,而HPI6/3的酶活略高于出发菌株,在120 h达到最高,为23.8 U/mL。由此可以看出,该菊粉酶基因利用自身的启动子比pgk启动子表达的菊粉酶活要高。

图5 宿主菌和转化子HI6/6及HPI6/3的生长情况及菊粉酶活力的变化Fig. 5 Yeast cell growth and inulinase production of the host and recombinants HI6/6 and HPI6/3 at aerobic conditions. Biomass and inulinase activity: S. cerevisiae 6525 (◆ and ◇ ); HI6/6 (▲ and △ ); HPI6/3 (■ and □).
2.4 转化子的发酵情况

图6 转化子HI6/6及HPI6/3的菊芋粉乙醇发酵性能Fig. 6 Ethanol fermentation performance of HI6/6 and HPI6/3 in the batch fermentations from Jerusalem artichoke. (A) Total sugar and ethanol concentration. S. cerevisiae 6525 (◆ and ◇ ) , HI6/6 (▲ and △ ) and HPI6/3 (■ and □). (B) Reducing sugar concentration and inulinase activity. S. cerevisiae 6525 (◆ and ◇ ); HI6/6 (▲ and △ ); HPI6/3 (■ and □).
在摇瓶中对转化子HI6/6及HPI6/3的乙醇发酵性能进行了测定,结果如图6所示。由图可以看出,转化子在前24 h,乙醇浓度迅速提高,总糖含量迅速下降,还原糖浓度降低到3 g/L,是发酵的主要阶段;24 h后,乙醇浓度增加缓慢,到48 h基本上达到发酵终点,乙醇浓度达到最高,总糖和还原糖浓度基本上降到最低水平。HI6/6与HPI6/3的最终乙醇浓度分别为55 g/L和52 g/L,均高于出发菌株49 g/L,较出发菌株最终乙醇浓度分别提高了12.24%和6.12%,糖醇转化率分别为0.495和0.453,为理论值的 96.9%和 88.6%,虽然 HPI6/3不如 HI6/6的终点乙醇浓度高,但是其发酵时间缩短了12 h。在发酵过程中,3株菌的酶活逐渐增高,到60 h基本上达到最高点,其中HI6/6的酶活力最高,达到26 U/mL,明显高于出发菌株。总之,HI6/6和HPI6/3的发酵性能均高于出发菌株,说明菊粉酶基因在酿酒酵母中得到了很好的表达,而 HI6/6的发酵性能要优于HPI6/3。
3 讨论
酿酒酵母是生产燃料乙醇的最成熟的菌种,具有生长速度快、乙醇得率高、遗传背景清楚、生物安全等优点,广泛应用于国内外的燃料乙醇生产中。菊芋因其具有抗旱、抗盐、抗虫等优点成为极具开发价值的非粮燃料乙醇生产原料。一般认为,普通的酿酒酵母因不产生菊粉酶而不能直接发酵菊粉生产乙醇[18-19],但酿酒酵母具有分泌蔗糖转化酶的能力,该酶 (SUC2) 的氨基酸序列与K. marxianus的菊粉酶的氨基酸序列有67%的相似性[20],表现出部分菊粉酶活力,所以,酿酒酵母能部分地水解菊芋生成乙醇,但残余总糖较高,不能满足工业化生产的需要。因此,本文通过采用菊粉酶启动子和酿酒酵母3-磷酸甘油酸激酶基因 (pgk) 启动子实现来源于K. marxianus的菊粉酶在酿酒酵母中的高表达。实验结果表明,两种启动子及目的基因在ho基因位点整合后,不影响菌体的生长,且都能够启动菊粉酶基因的表达。其中菊粉酶自身启动子效率较高,最高菊粉酶酶活达到 86 U/mL,是出发菌株的 4.6倍;而 pgk启动子的效率略高于出发菌株,最高菊粉酶酶活为23.8 U/mL,是出发菌株的1.5倍。浓度为200 g/L的粗菊芋粉的生料发酵结果表明,重组表达菊粉酶的酿酒酵母的乙醇发酵速度较快,终点乙醇浓度分别为55 g/L和52 g/L,均高于出发菌株 49 g/L,较出发菌株最终乙醇浓度分别提高了12.24%和6.12%,糖醇转化率分别为0.495和0.453,为理论值的96.9%和88.6%。为提高终点乙醇浓度,我们尝试了补料发酵,当菊芋粉浓度提高到280 g/L时,乙醇的浓度可达到10%以上,发酵可在60 h完成 (数据另文发表),表现出潜在的工业化应用价值。
REFERENCES
[1] Szambelan K, Nowak J, Czarnecki Z. Use of Zymomonas mobilis and Saccharomyces cerevisiae mixed with Kluyveromyces fragilis for improved ethanol production from Jerusalem artichoke tubers. Biotechnol Lett, 2004, 26(10): 845−848.
[2] Vandamme EJ, Derycke DG. Microbial inulinases: fermentation process, properties, and applications. Adv Appl Microbiol, 1983, 29(2): 139−176.
[3] Wymen CE, Dale BE, Elander RT, et al. Coordinated development of leading biomass pretreatment technologies. Bioresour Technol, 2005, 96(18): 1959−1966.
[4] Szambelan K, Nowak J, Jelen H. The composition of Jerusalem artichoke (Helianthus tuberosus L.) spirits obtained from fermentation with bacteria and yeasts. Eng Life Sci, 2005, 5(1): 68−71.
[5] Favala-Torres E, Allais JJ, Baratti J. Kinetics of batch fermentations for ethanol production with Zymomonas mobilis growing on Jerusalem artichoke juice. Biotechnol Bioeng, 1986, 18(8): 850−856.
[6] Allais JJ, Torres EF, Baratti J. Continus production of ethanol with Zymomonas mobilis growing on Jerusalem artichoke juice. Biotechnol Bioeng, 1987, 29(6): 778−782.
[7] Ohta K, Hamada S, Nakamura T. Production of high concentrations of ethanol from inulin by simultaneous saccharification and fermentation using Aspergillus niger and Saccharomyces cerevisiae. Appl Environ Microbiol, 1993, 59(3): 729−733.
[8] Ge XY, Zhang WG. A shortcut to the production of high ethanol concentration from Jerusalm artichoke tubers. Food Technol Biotechnol, 2005, 43(3): 241−246.
[9] Margaritis A, Bajpai P. Ethanol production from Jerusalem artichoke tubers (Helianthus tuberosus) using Kluyveromyces marxianus and Saccharomyces rosei. Biotechnol Bioeng, 1982, 24(4): 941−953.
[10] Kim YH, Nam SW, Chung BH. Simultaneous saccharification of inulin and ethanol fermentation by recombinant Saccharomyces cerevisiae secreting inulinase. Biotechnol Bioproc Eng, 1998, 3(2): 55−60.
[11] Bajpai P, Margaritis A. Kinetics of ethanol production by immobilized Kluyveromyces marxianus cells at varing sugar concentrations of Jerusalem artichoke juice. Appl Microbiol Biotechnol, 1987, 26(5): 447−449.
[12] Eksteen JM, van Rensburg P, Cordero Otero RR, et al. Starch fermentation by recombinant Saccharomyces cerevisiae strains expressing the α-amylase and glucoamylase genes from Lipomyces kononenkoae and Saccharomycopsis fibuligera. Biotechnol Bioeng, 2003, 84(6): 639−646.
[13] Tamai Y, Tanaka K, Umemoto N, et al. Diverisity of the HO gene encoding an endonuclease for mating-type conversion in the bottom fermenting yeast Saccharomyces pastorianus. Yeast, 2000, 16(14): 1335−1343.
[14] Baganz F, Hayes A, Marren D, et al. Suitability of replacement markers for functional analysis studies in Saccharomyces cerevisiae. Yeast, 1997, 13(16): 1563−1573.
[15] Voth WP, Richards JD, Shaw JM, et al. Yeast vectors for integration at the HO locus. Nucl Acids Res, 2001, 29(12): e59.
[16] Sambrook J, Fritsch EF, Maniatis T. Molecular Cloning: A Laboratory Manual. 2nd ed. New York: Cold Spring Harbor Laboratory Press, 1989.
[17] Chang BL, Yuan WJ, Zhao XQ, et al. Ethanol Fermentation from Jerusalem artichoke juice with self-flocculating yeast Saccharomyces cerevisiae flo. The Chin Proc Engin, 2009, 9(4): 796−800.常宝磊, 袁文杰, 赵心清, 等. 自絮凝颗粒酵母发酵菊芋汁生产乙醇. 过程工程学报. 2009, 9(4): 796−800.
[18] Yang LB, Zhao WL, Zhang G, et al. Mutation screening of yeast strains with higher inulinase activity. China Brewing, 2010(5): 139−142.杨利博, 赵卫利, 张桂, 等. 高菊粉酶活酿酒酵母的诱变选育. 中国酿造, 2010(5): 139−142.
[19] Zhang T, Chi Z, Zhao CH, et al. Bioethanol production from hydrolysates of inulin and the tuber meal of Jerusalem artichoke by Saccharomyces sp. W0. Bioresour Technol, 2010, 101(21): 8166−8170.
[20] Chen XM, Chen HQ, Chen TX, et al. Enzymatic properties and molecular biology of inulinase. J Food Sci Biotechnol, 2009, 28(5): 577−588.陈晓明, 陈寒青, 陈天祥, 等. 菊粉酶的酶学特性与分子生物学. 食品与生物技术学报, 2009, 28(5): 577−588.
