基于图像与文本特征的在线生物文献MRI图像库构建
2010-09-18赵鹏飞钱沄涛郑文斌李吉明
赵鹏飞 钱沄涛 郑文斌 李吉明 居 斌
(浙江大学计算机学院,杭州 310027)
基于图像与文本特征的在线生物文献MRI图像库构建
赵鹏飞 钱沄涛*郑文斌 李吉明 居 斌
(浙江大学计算机学院,杭州 310027)
近年来,利用在线文献构建生物数据库引起越来越多的关注,包括论文中生物数据的自动收集、组织和分析。在线文献中图像形式表示的数据具有特别重要的意义。利用在线生物文献中的图像和图像标注,构建在线生物文献核磁共振(MRI)图像库,其中识别文献中的MRI图像是重要的一步。作为从在线文献中构建MRI图像库的必要部分,综合利用图像信息和图像文本标注信息,采用后验相乘、特征拼接和协同学习3种方法来识别文献中的MRI图像。实验表明,综合利用图像和文本两类信息训练得到的分类器,比基于单种信息训练得到的分类器具有更高的识别精度,为构建能解释在线文献中MRI图像的知识系统这一长期目标提供了基础支持。
MRI;在线文献;图像分类
引言
随着信息技术的快速发展,信息的来源呈多元化趋势,而信息的数量呈爆炸性增长趋势。目前,专业科学和技术工作者更依赖于共同的研究平台和资源(大型专业数据库),以及海量信息的高效检索和组织能力(搜索引擎)。通过对非结构化文献数据的信息抽取和整理,建立具有一定标准结构的专业数据库,为信息提取、处理和组织提供了有效的手段。在文献中,图表包含了关键的实验结果和性能指标,工程技术和实验科学文献尤为明显。从文献构建专业图像数据库,是收集整理文献提供的各种不同条件、对象、时间、方法下取得的专业图像,并对这些图像进行分类、标注、存储、索引、链接。
近年来,国外出现了一些利用文献中的图表(figure)、图表标题(caption)、摘要(abstract)、文本(text)等信息构建的专业数据库。Carnegie MellonUniversity的Murphy实验室开发了SLIF(Subcellular Location Image Finder)系统,该系统利用在线文献中抽取的图表和图表标题,建立荧光显微图像数据库,采用图像结合文本特征对荧光图和非荧光图进行分类,其分类精度为 88.6%[1-3];EBSCO 数据库能提供期刊文献订购、出版及检索等服务,能根据期刊文献的关键词、主题、出版物、索引、图像和参考文献等信息进行检索,在学术界有很强的影响力[4];Shatkay把文献的图表数据整合到基于文献的文本信息的分类方法中,对生物医学文献进行分类[5];Yu和Lee建立文献中摘要和图表的联系,通过摘要信息去检索包含感兴趣图表的文献[6];Rafkind利用文献中的图表信息和文本信息,自动把文献中的图表分成凝胶图(gel images)、图表、实物图(image-of-thing)、混合图(mixtures)和模型图(models)等多类图表[7];Xu等开发了一套基于关键词检索生物医学图片和文献的搜索引擎(YIF)[8]。上述工作尽管比较初步,但已经显示,通过在线文献这一巨大和规范的数据源,可以建立为专门研究和商业应用服务的专业数据库,前景广阔。
本研究的背景是从在线生物文献中构建MRI图像库,MRI在临床医学和脑科学研究中有着重要的作用[9-10],因此建立 MRI图像库的工作有着重要的意义。此前,笔者已完成了在线生物文献中图表和图表标题信息的提取,图表-图表标题对(figure-caption,如图1所示)分割为嵌图(panel)和嵌图标注(annotation),并以嵌图-嵌图标注对(panel-annotation)的形式存储在图像库中[11-12],下一步的重点在于如何识别在线文献中的MRI图像和提高识别精度。在传统方法中,一般仅利用图像特征来做图像识别。本研究提出综合利用图像库中的图像信息(SIFT特征)和文本信息(图像对应文本标注的词频特征),训练较高分类精度的支持向量机(support vector machine,SVM)分类器。采用3种图像特征结合文本特征的方法:一是分别基于图像特征和文本特征训练SVM分类器对数据做分类,输出后验概率值,然后相乘其后验概率值作为最终分类结果;二是直接拼接图像和文本两类特征,用拼接后的特征训练SVM分类器对数据做分类;三是根据协同学习的思想,利用一些未标记样本,并综合图像特征和文本特征训练SVM分类器对数据做分类。

图1 在线生物文献中的图表-图表标题对[13]Fig.1 The figure-caption in online biological literature[13]
1 系统简介
从在线生物文献构造MRI图像库是一个复杂的过程,图2是MRI图像库的基本功能模块,主要包括:获取在线生物文献,并从文献内捕获图表和对应的图表标题;图表中嵌图的分割,一般文献中一张图表内往往包含多个嵌图,需要对图表进行划分以保证每个嵌图作为独立的图像;嵌图序号识别,如果图表包含多个嵌图,那么一般每个嵌图都有一个序列号,用来对应嵌图和图表标题中的文本;图表标题的处理,得到图像指针(image points)和每个图像指针在图表标题中的所辖范围,图像指针和嵌图序号一起为嵌图和标题提供一一对应关系;每个图像指针在图表标题中的所辖范围是对图像指针所对应嵌图的说明;嵌图识别、分类/聚类、语义标注。通过这些步骤,最终构建一个在线生物文献MRI图像库。
在图2中,虚线表示部分是本研究所关注和解决的问题,即使用图像和文本特征来识别图像库中的MRI图像。
2 基于图像和文本信息融合的MRI图像识别
2.1 图像特征提取
MRI是断层成像的一种,它利用磁共振现象从人体中获得电磁信号,并重建出人体信息。通常,MRI图像的背景为黑色,图像中间为较明亮影像物体,影像物体局部的形态和特征与其他图像相比具有较明显的区别。通过构造SIFT特征词汇表(bagof-features)[14]作为图像特征来识别 MRI图像。SIFT是David G.Lowe提出的一种基于尺度空间的、对图像缩放、旋转甚至仿射变换保持不变性的图像局部特征描述算子[15],因此,其特征适合区分MRI图像和其他图像。一幅图像SIFT特征向量的生成算法包含以下3个主要步骤。

图2 MRI图像库构造流程Fig.2 The construction process of MRI Database
步骤1:尺度空间极值点检测,以确定关键点位置和所在尺度。在图像二维平面空间和DoG(difference-of-gaussian)尺度空间中,同时检测局部极值作为关键点。DoG算子定义为两个不同尺度的高斯核的差分,有

式中,G(x,y,σ)为二维高斯函数,σ为高斯正态分布的方差,(x,y)为图像的像素位置,L为图像的尺度空间。
步骤2:利用关键点领域像素的梯度方向分布特征,为每个关键点指定方向参数,使算子具备旋转不变性。点(x,y)处梯度的模值和方向为

步骤3:生成特征描述子。首先将坐标轴旋转为关键点的方向,以关键点为中心取8像素×8像素的窗口,然后在每2像素×2像素的小块上计算8个方向的梯度方向直方图。因此,一个关键点由4×4共16个小块组成,每个小块有8个方向向量信息,从而获得16×8=128维的特征描述子作为SIFT特征。
在生成图像库中所有图像的SIFT特征词汇表主要包括以下4个步骤。
步骤1:把所有样本图像放缩到一样的大小,用Canny算子对图像做边缘检测。在边缘检测后得到的灰度图中,找出灰度值最大100个像素作为关键点。由此,可以确定这些关键点在图像中的坐标。
步骤2:根据上述SIFT特征的生成算法,计算样本图像中所有关键点的SIFT特征向量。相当于每个样本图像中提取100个SIFT特征向量,来表示该图像。
步骤3:收集所有样本中的SIFT特征向量作为一个数据集,用k-means算法[15]对数据集做聚类,产生300个聚类中心。从而构成了样本图像的SIFT特征词汇表。
步骤4:针对每个样本图像中100个SIFT特征向量,分别计算每个特征向量到这300个聚类中心的相似度,选取相似度最大的聚类中心来表示该特征向量,从而生成一个300维的频率分布直方图来表示一个样本图像。
2.2 图像特征提取
在线生物文献图像库中,不仅存储了大量嵌图,还包含对应嵌图的标注。这些标注能一定程度地解释嵌图所要表达的内容,所以可以根据嵌图标注等文本信息来识别MRI图像。同时,也可以利用嵌图标注信息,结合图像信息的方法来识别MRI图像。
文本特征提取主要包括3个步骤:文本预处理、索引统计、特征选择。文本预处理涉及统一文本编码格式,去除停用词。索引统计包括提取文本中的词项,并与词典进行匹配和计数,建立样本文本特征空间。特征选择是文本分类的重要问题,因为通常文本特征空间的维数很高,有几千上万维,标准的分类算法很难处理如此大的特征集,而且分类结果不可靠。因此,需要进行特征选择,既可以大大降低向量空间的维数,也可以减少“过拟合”的问题。
在特征选择中,若不考虑特征项之间的相关性,常见的算法有:词频特征(TermFrequency,TF)、文档频率(documentfrequency,DF)、信息增益值(information gain,IG)、互信息 (multi-information,MI)[16]。采用计算信息增益值的方法来做特征选择,信息增益的计算为

式中:P(ci)表示类别ci出现的概率,等于属于类别ci的样本数量除以总的样本数;P(tk)表示特征(tk)出现的概率,等于出现过特征(tk)样本数除以总样本数;P(ci|tk)表示出现特征tk时,样本属于类别ci的概率,等于出现特征tk并且属于类别ci的样本数除以出现了特征tk的样本数。
在选择文本特征时,先计算所有特征词的信息增益值,然后从中选取300个拥有最大信息增益值的特征词作为文本特征,最终构成300维的文本特征空间。
2.3 图像结合文本特征的分类
在获取图像特征和文本特征后,可以单独利用图像特征或文本体征来识别MRI图像,也可以用图像和文本信息融合的方法来识别MRI图像。本研究选择基于线性核函数的SVM方法作为识别算法[17],采用3种图像和文本信息融合的方法。
1)提取图像库中每个嵌图-嵌图标注对的图像特征、文本特征,用SVM方法,训练分别基于图像特征(x1)的分类器和基于文本特征(x2)的分类器,对测试样本做分类,分别输出后验概率值p(ci|x1)和p(ci|x2)。假设样本的图像特征和文本特征是相互独立的,则满足式(4),根据式(5)可求得基于图像和文本特征的后验概率p(ci|x1,x2),即

2)直接拼接图像和文本特征。对图像库中的每个嵌图-嵌图标注对提取300维图像特征(x1)和300维文本特征(x2),把两类特征向量拼接为一个600维的特征向量x=(x1,x2)。根据拼接后的特征,训练SVM分类器来识别MRI图像。
3)根据协同学习的思想[18],利用一些非标记样本,并综合图像特征、文本特征,训练分类器来识别MRI图像。因为在图像库中,除了一些标记好类别的样本外,还存在大量未标记的样本。协同学习是一种半监督方法,它的理论假设是:数据集可以被自然地分成两个独立的特征子集,并要求这两个子集满足一致性和独立性。前者要求对大多数样本在每个特征子集上预测的类别是相同的。后者要求对指定类别的任意样本在两个特征子集中的描述是独立的。在图像库中,对每个嵌图-嵌图标注对提取300维图像特征(x1)和300维文本特征(x2),该数据集的图像特征子集和文本特征子集满足协同学习要求的一致性和独立性。对协同学习算法进行描述:
输入:L表示已经标记好的样本数据,U表示未标记的样本数据,C表示用于缓存一小部分U的缓冲器。
输出:增加了未标记样本后的训练样本数据L
伪代码:
Step 1:将U分解为C和U′,满足U=CUU′
Step 2:循环m次:
{
Step 3:用L中的图像特征(x1)训练一个SVM分类器h1;
Step 4:用L中的文本特征(x2)训练一个SVM分类器h2;
Step 5:用分类器h1对C进行分类;
Step 6:依据分类结果和一定原则,从C中选择一部分数据Ch1(包括p个MRI样本和n个非MRI样本),加入到 L,使得,C=C-Chi,L=LUChi;
Step 7:用分类器h2对C进行分类;
Step 8:依据分类结果和一定原则,从C中选择一部分数据Ch2(包括p个MRI样本和n个非MRI样本),加入到 L,使得 C=C-Ch2,L=LUCh2;
Step 9:从U′中随机选择2p+2n个样本,记为P,加入到C中,使得C=CUP;
}
在协同学习算法中,设定未标记样本缓存器C=300,循环次数m=30,并根据图像库中MRI图像和非MRI图像的比例,设定每次循环加入的MRI样本的数量p=1,非MRI样本的数量n=3。
3 分类器的评价方法
从自建的图像库中,选出892个图表-图表标题对,并把这些图表-图表标题对分割为嵌图-嵌图标注对,其中MRI图像和非MRI图像的比例大约为1:3。在选取实验数据时,为了避免同个图表-图表标题对中的不同嵌图-嵌图标注对的相似性,在每个图表-图表标题对中只选取一个嵌图-嵌图标注对。同时,选取1 000个未标记嵌图-嵌图标注对,用于协同学习算法。
在实验中,随机选取90%带标记样本数据作为待选取的训练样本,剩余10%样本数据作为测试样本。设定训练样本占待选取训练样本的比例为p,p从1/9递增到1,每次递增1/9,然后用基于图像特征、基于文本特征、后验相乘、特征拼接、协同学习的5种分类方法识别测试样本,分别计算每种分类方法在不同训练样本数量下的识别精度。这个过程在实验中重复20次,最终求得每种方法在不同数量训练样本下的平均识别精度。
Kappa统计值经常作为衡量新方法的一个评测指标[19]。在实验中,随机选取90%的样本数据作为训练样本,剩余10%样本数据作为测试样本,用5种分类方法识别测试样本,然后分别计算每种分类结果与金标准的Kappa统计值。这个过程在实验中重复20次,然后求每种方法平均的 Kappa值。Kappa统计值的范围在0~1.0之间,值越大说明该方法越稳定、性能越好。
ROC 曲线是以“灵敏度”为纵坐标′[20],“1-特异度”为横坐标,根据ROC曲线空间中曲线的分布来分析一个分类器是否可靠。在实验中,随机选取90%的样本数据作为训练样本,剩余10%的样本数据作为测试样本,用5种分类方法识别测试样本,设定分界点k从0.01递增到0.99,每次递增0.01,分别计算每种分类方法的结果与金标准在分界点为k时的ROC曲线坐标值。这个过程在实验中重复20次,然后求每种方法平均的ROC曲线坐标值。在ROC曲线空间,如果曲线沿着左边线,然后沿着上边线越紧密,则实验准确度越高,该曲线对应的分类方法的识别效果越好。
4 实验结果
识别精度的实验结果如图3所示;从整体上看,5种方法随着训练样本数量的增加,分类的准确率基本上稳步提高。当训练样本数量大于总样本数量20%的时候,基于文本特征比基于图像特征的识别精度略高(高1%左右),说明文本特征适合做MRI图像的识别。从总体上看,3种图像结合文本的方法均能有效提高MRI图像的识别精度,尤其是协同学习方法,当训练样本比较少的情况下(比如10%样本作为训练样本时),其分类准确率依然比较高,其效果优于其他两种图像文本特征结合方法。这说明协同学习方法能有效利用大量的未标记数据,提高识别MRI图像的准确率。随着训练样本数量的增加(比如70%以上样本作为训练样本时),特征拼接的方法能更有效地识别MRI图像。

图3 在不同数量训练样本下5种识别方法的分类情况Fig.3 The classification performance of five different algorithms on different training sample sets
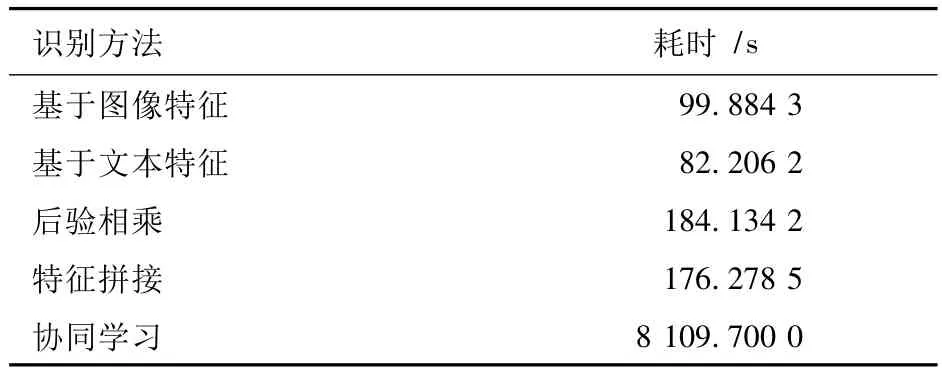
表1 5种识别方法所消耗的时间Tab.1 The consuming time of five different algorithms
表1是实验中每种识别方法在不同数量训练样本下完成分类所消耗的总时间。图像和文本特征融合的方法在提高MRI图像识别精度的同时,也不同程度上增加了训练时间。但是,特征拼接法和后验相乘法增加的训练时间是有限的,而协同学习的训练计算量相对比较大。由于训练是离线进行的,因此计算消耗不是太大的问题。
表2为5种识别方法的Kappa值,可知后验相乘、特征拼接和协同学习3种方法的Kappa值更大,说明这3种方法的稳定性和准确率都优于基于单种特征的方法。

表2 5种识别方法的Kappa值Tab.2 Kappa Statistics of five different algorithms
图4为5种识别方法的ROC曲线,可知后验相乘、特征拼接和协同学习3种方法的ROC曲线分布更接近左边线和上边线,说明这3种方法的稳定性和准确度都优于基于单种特征的方法。

图4 5种识别方法的ROC曲线Fig.4 ROC curve analysis of five different algorithms
5 讨论和结论
当前,随着大量学术文献和信息的数字化,从中检索和重组符合个性化和专业化要求的信息资源,并构造更方便使用的结构化数据库,已经成为数字信息资源深度挖掘的重要方向。笔者以如何从在线生物文献构造MRI图像数据库为研究背景,重点研究在线文献中MRI图像的识别方法。由于在线文献中的图像存在分辨率较低、形态复杂、分布随机、无统一标准等问题,对MRI图像实现高识别低漏检是一个富有挑战的问题。而文献中图像的突出特点是一定会有对应的图像标注对图像进行一定的说明,由此提供了一个很好的识别MRI图像的信息来源。因此,笔者提出了综合利用图像和文本特征,采用后验相乘、特征拼接和协同学习3种方法来识别MRI图像的方法,通过图像本身特征和标注特征的互补性及互证性,提高了识别精度,降低了漏检率。实验表明,综合利用图像、文本两类特征训练得到的分类器,比单种特征训练得到的分类器的分类精度要高。研究为建立在线生物文献MRI图像库和后续的知识挖掘等工作打下扎实的基础。
在提出的3种特征融合分类方法中,后验相乘最简单,可以分别对不同种类的特征进行训练,但性能提高上比其他两种方法略低;特征拼接需要注意的是异构特征的归一化问题;而协同学习尽管时间比较长,但特别适合小训练样本集的场合。另外,其他特征融合方法和分类器融合方法是下一阶段的研究课题之一。
本研究提出的方法对于从在线文献构造任意其他图像数据库是具有普遍借鉴意义的。不同图像库构建的主要区别在于需要针对不同图像的特点构造不同的图像和文本特征,由于荧光显微图像就更适合用纹理特征。而且每一类图像都有自己特有的专业术语,因此选择特征词典可以结合所需构造图像库的特点进行。构造更具区分性的图像和文本特征,以及更具实际需求从在线文献建立其他图像数据库,也是进一步的主要工作之一。
[1] Qian Yuntao,Murphy RF.Improved recognition offigures containing fluorescence microscope images in online journal articles using graphical models [J].Bioinformatics,2008,24(4):569-576.
[2 ] Murphy RF,Velliste M,Yao J,et al.Searching online journals for fluorescence microscope images depicting protein subcellular location patterns[A].In:Bourbakis NG,eds.IEEE International Symposium on Bioinformatics and Bioengineering [C].Washington DC:IEEE Computer Society,2001.119 –128.
[3] Murphy RF,Velliste M,Porreca G.Robust Numerical features for description and classification of subcellular location patterns in fluorescence microscope Images[J].The Journal of VLSI Signal Processing,2003,35(3):311 -321.
[4] StephensJT.EBSCO Databases[DB/OL].http://web.ebscohost.com,2010-03-06/2010-05-25.
[5] Shatkay H,Chen N,Blostein D.Integrating image data into biomedical text categorization [J].Bioinformatics,2006,22(14):446-453.
[6] Yu H,LeeM.Accessingbioscienceimagesfrom abstract sentences[J].Bioinformatics,2006,22(14):547 -556.
[7] Rafkind B,Lee M,Chang SF,et al.Exploring text and image features to classify images in bioscience literature[A].In:Verspoor K,Cohen KB,Ben Goertzel,eds.Proceedings of the HLT-NAACL BloNLP Workshop on Linking Natural Language Processing and Biology[C].New Jersey: Association for Computational Linguistics,2006.73 -80.
[8] Xu Songhua,McCusher J,Krauthammer M.Yale Image Finder(YIF):a new search engine for retrieving biomedical images[J].Bioinformatics,2008,24(17):1969 -1970.
[9] 过哲,张晶,梁伟,等.骨巨细胞瘤的动态增强MRI与^1H-MR波谱研究[J].中国医学影像技术,2008,24(10):1490-1492.
[10] 杨贵昌,李文进,周平,等.MRI诊断非出血性脑弥漫性轴索损伤的临床价值[J].中国医学影像学杂志,2003,11(4):273 -274,311.
[11] 赵鹏飞,钱沄涛,郑文斌.基于在线生物学文献的MRI图像获取[J].中国体视学与图像分析,2009,14(2):216 -211.
[12] 赵鹏飞,钱沄涛,郑文斌.在线文献的嵌图序号检测与识别[A].见:刘国权,编.第八届全国信号与信息处理联合学术会议[C].北京:中国体视学学会,2009.151-155.
[13] Tripathi M,Singh M,Padma MV,et al.Surgical outcome of cortical dysplasias presenting with chronic intractable epilepsy:a 10-year experience[J].Neurology India,2008,56(2):138 -143.
[14] Li Feifei,Perona P.A bayesian hierarchical model for learning natural scene categories[A].In:Schmid C,Soatto S,Tomasi C,eds.Proceedings of the 2005 IEEE Computer Society Conference on Computer Vision and Pattern Recognition[C].Washington DC:IEEE Computer Society,2005.524 -531.
[15] Lowe DG.Distinctive image features from scale-invariant keypoints[J].International Journal of Computer Vision,2004,60(2):91-110.
[16] Sebastiani F.Machine learning in automated text categorization[J].ACM Computing Surveys,2002,34(1):1 -47.
[17] Cortes C,Vapnik V.Support vector networks[J].Machine learning,1995,20(3):273 -297.
[18] Blum A,Mitchell T.Combining labeled and unlabeled data with co-training[A].In:Bartlett P,Mansour Y,eds.Proceedings of the Eleventh Annual Conference on Computational Learning Theory[C].New York:Association for Computer Machinery,1998.92-100.
[19] 郑卓肇,孙忠强,范家栋,等.Kappa统计量评价半月板MRI诊断[J].中国医学影像技术,2002,18(6):587 -588.
[20] Fawcett T.An introduction to ROC analysis[J].Pattern Recognition Letters,2006,27(8):816 -874.
MRI Database Construction Using Image and Text Features Extracted from Online Biological Literature
ZHAO Peng-FeiQIAN Yun-Tao*ZHENG Wen-Bin LI Ji-Ming JU Bin
(College of Computer Science,Zhejiang University,Hangzhou 310027,China)
Recently,building biological databases from online literature has attracted more attentions,which includes automating the collection,organization and analysis of biological data in the research literature.Images,as an important type of data in online literature,present great significance.It is necessary to build a magnetic resonance imaging(MRI)database that extracts information regarding images and texts in online biological literature.In this paper,MRI image recognition was studied.For better comprehensive utilization of image and text features,we propose three fusion approaches,including merging both features,multiplying both posterior probabilities and Co-training algorithm.Experimental results show a significant improvement in the average accuracy of the three fusion classifiers as compared with classifiers only based on image or text features.
MRI;online literature;image classification
R318.08
A
0258-8021(2010)05-0697-07
10.3969/j.issn.0258-8021.2010.05.010
2010-06-12,
2010-08-23
国家自然科学基金资助项目(60872071)
*通讯作者。 E-mail:ytqian@zju.edu.cn
