陆地棉TRM基因家族的鉴定及纤维品质相关优异单倍型分析
2024-04-28王凯迪高晨旭裴文锋杨书贤张文庆宋吉坤马建江王莉于霁雯陈全家
王凯迪 高晨旭 裴文锋 杨书贤 张文庆 宋吉坤 马建江 王莉 于霁雯 陈全家


doi:10.6048/j.issn.1001-4330.2024.03.001
摘 要:【目的】鉴定陆地棉TRM基因家族序列及理化性质,分析与棉纤维品质相关的优异单倍型基因差异。
【方法】利用生物信息学分析陆地棉TRM基因家族进化关系、理化性质和聚类表达;利用基因单倍型效应分析筛选调控纤维品质性状(纤维长度、比强度、马克隆值)的候选基因。
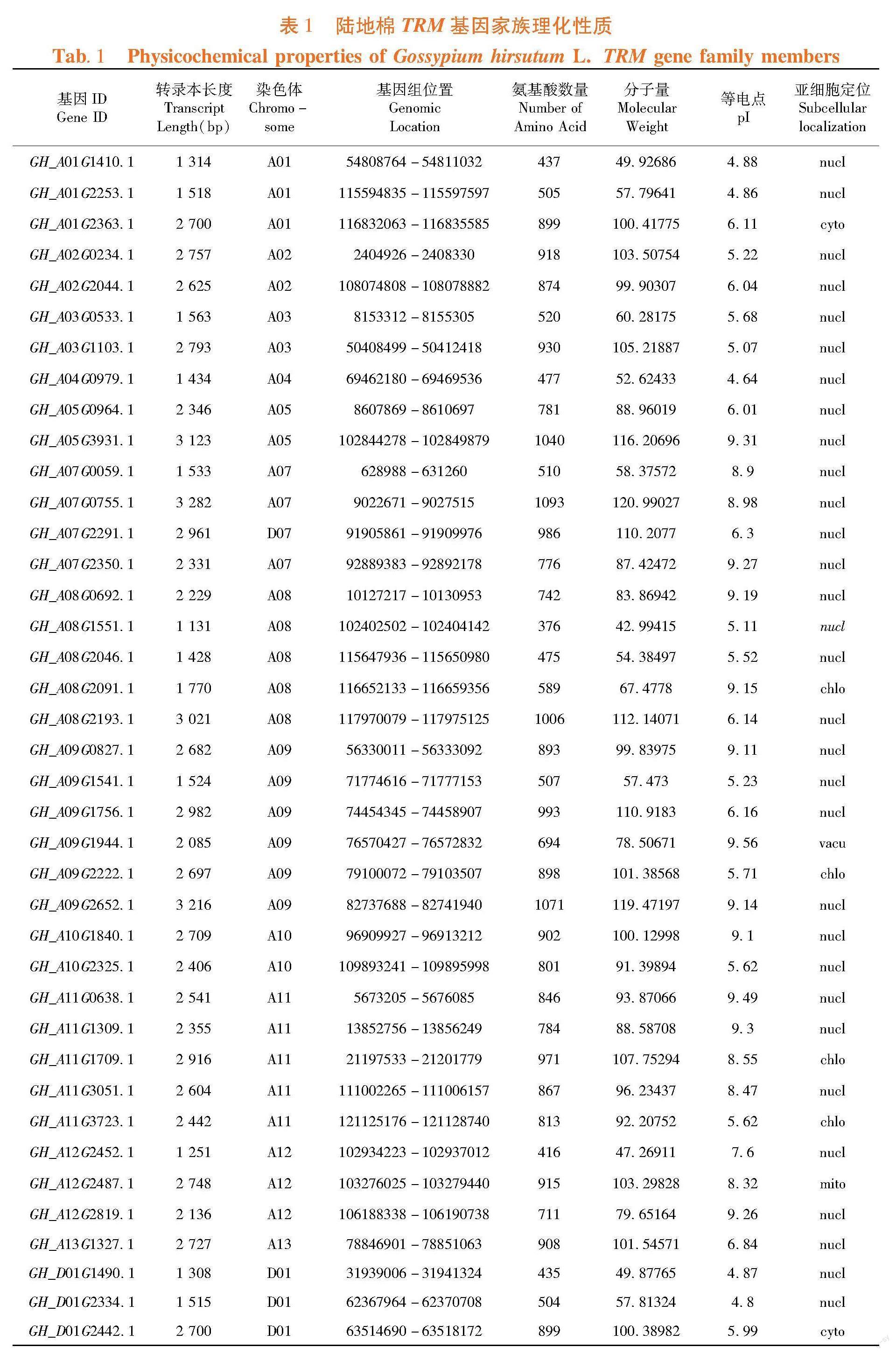
【结果】陆地棉TRM基因家族编码的氨基酸为376~1 093,等电点为4.64~9.56。亚细胞定位预测发现多达60个陆地棉TRM基因家族定位于细胞核中。TRM基因家族含有较多的光响应、激素响应、胁迫响应和生长发育相关的元件。60个TRM基因家族在纤维发育时期优势表达,调控棉花纤维发育。每个基因的单倍型个数为1~8,并筛选到纤维长度、强度以及马克隆值相关的优异单倍型TRM基因分别有14、18和15个,其中有11个基因同时具有长度、强度、马克隆值3种改良纤维品质的优异单倍型。GH_D09G0775的Hap_4单倍型和GH_D03G1434的Hap_3单倍型在增加纤维长度、强度的同时降低了马克隆值。
【结论】在陆地棉(Gossypium hirsutum L.)中鉴定出75个TRM基因家族成员分布在24条染色体上,系统进化将其分为Cluster Ⅰ~Ⅲ 3个亚族。
关键词:陆地棉;基因家族;基因表达;纤维品质;单倍型
中图分类号:S562 文献标志码:A 文章编号:1001-4330(2024)03-0521-16
收稿日期(Received):
2023-07-11
基金项目:
河南省农业良种联合攻关项目(2022010301);新疆维吾尔自治区自然科学基金项目(2021D01B113);中央级公益性科研院所基本科研业务费专项(1610162022013)
作者简介:
王凯迪(1998-),男,陕西延安人,硕士研究生,研究方向为棉花分子育种,(E-mail)424416709@qq.com
通讯作者:
于霁雯(1978-),女,河南安阳人,研究员,博士,硕士生/博士生导师,研究方向为棉花分子育种,(E-mail)yujw666@hotmail.com
陈全家(1972-),男,新疆乌鲁木齐人,教授,博士,硕士生/博士生导师,研究方向为棉花遗传育种,(E-mail)chqjia@126.com
0 引 言
【研究意义】棉纤维是纺织工业的主要原料[1]。棉纤维品质的优劣关系到棉纺织品的质量,因此改良棉纤维品质是棉花育种的主要目标。常见的异源四倍体棉种陆地棉由A和D两个二倍体亚组组成[2],其基因组已经公布[3-4]。GRAND(http://grand.cricaas.com.cn/home)数据库公布的棉属表达数据为开展基因家族的全基因组鉴定与生物信息学分析提供了基础信息[5]。【前人研究进展】TRM(TON1 Recruiting Motif)基因家族对于植物生长发育起着重要作用,其基因家族分析在多种植物中已完成[6]。在拟南芥中鉴定到34个TRM蛋白,所有TRM中拥有6个短保守基序,其中一个是与TON1相互作用的基序。TRM1能够与微管蛋白结合,并通过与TON1的相互作用将其招募到微管蛋白[7]。TRM5是TRM 基因家族的一个重要成员,AtTRM5对拟南芥的生长发育起着重要作用,AtTRM5突变体具有明显的发育缺陷表型如叶片生长变慢、花发育遲缓、根变短等[8]。TRM4与拟南芥种子粘液质的分泌相关[9]。TRM7与TRM8突变体损害有丝分裂前期的早前期带(preprophase band,PPB)的形成,但不影响间期微管[10]。Tang 等[11]的研究将拟南芥中AtTRM6突变后会导致胚胎致死和种子败育。【本研究切入点】生物信息学方法在全基因组分析中发挥着重要作用,棉花的多个重要基因家族已经通过生物信息学的全基因组分析取得了相应成果[12-13]。作物种质资源中蕴藏着丰富遗传优异等位基因,可以鉴别与表型性状相关的候选基因核苷酸变异,还可以评估新的单倍型等[14]。TRM基因在植物的生长发育中起着重要作用,而陆地棉TRM基因家族鉴定及纤维品质相关优异单倍型分析仍未见报道。需研究鉴定陆地棉TRM基因家族的序列及分析纤维品质相关优异单倍型。【拟解决的关键问题】采集已公布的陆地棉基因组数据,系统分析棉花TRM基因家族成员的进化关系、基因结构变异﹑共线性关系和基因表达模式以及单体型,为研究TRM基因在陆地棉中的相关功能提供数据支撑。
1 材料与方法
1.1 材 料
陆地棉TRM基因家族下载陆地棉(http://www.cottonfgd.org/)[15]的基因组序列与注释数据。根据拟南芥TRM家族蛋白序列,依据BLASTP[16](E值设为1e-5)及TRM家族的隐马尔科夫模型文件PF14383、PF14309,HMMER程序[17]陆地棉TRM基因家族同源蛋白序列进行比对。通过在线工具NCBICD-Search(https://www.ncbi.nlm.nih.gov/cdd/)和Search Pfam(http://pfam.xfam.org/search)进行验证,过滤掉保守结构域不完整的序列,得到TRM家族成员序列。借助TBtools[18](Protein Paramter Calc)计算棉花TRM家族成员的分子量和等电点。利用亚细胞定位工具 WoLF PSORT(https:/l/wolfpsort.hgc.jp/)预测陆地棉TRM基因家族的亚细胞定位。
1.2 方 法
1.2.1 陆地棉TRM基因家族的进化与基因结构与保守结构域
利用MEGA11[19]内置的Clustal W[20]对陆地棉TRM蛋白序列进行比对,采用邻接法(Neibour-joining method),Bootstrap重复1 000次构建进化树。利用TBtools[18]分析陆地棉TRM基因家族成员的基因结构。利用在线工具 MEME(http://meme-suite.org/)分析TRM蛋白序列的保守结构域,最大基序设置为10,其他参数为默认值,同时利用NCBI Batch CD-Search(https://www.ncbi.nlm.nih.gov/)分析TRM蛋白序列的保守结构域,用TBtools[18]进行可视化。
1.2.2 陆地棉TRM基因家族的染色体定位与顺式作用元件
利用TBtools陆地棉TRM基因家族成员在染色体上的位置图。TBtools[18]从基因组序列中提取TRM基因家族上游2 000 bp作为启动子序列,运用在线工具 PlantCARE[21]分析启动子顺式作用元件。
1.2.3 陆地棉TRM基因家族成员的共线性
利用TBtools[18]软件分析TRM家族在陆地棉中成员的共线性关系,最小模块设置为5个基因。
1.2.4 陆地棉TRM基因家族的表达
从三代转录组数据中得到TRM基因家族在不同组织中的转录数据[22],使用TBtools[18]软件将数据制作热图进行可视化。
1.2.5 陆地棉TRM基因家族成员单倍型
使用cottonMD(http://yanglab.hzau.edu.cn/cott/PublicFun/total_jump target=vir/search&gene)所提供的基因型和表型数据[23],以及基因内snp和Indel作为分型依据,进行基因分型,结合群体纤维品质表型数据,对各单倍型的效应进行分析。
2 结果与分析
2.1 陆地棉TRM基因家族数量及其理化性质
研究表明,陆地棉TRM基因家族 CDS 长度在1 131(GH_A08G1551.1与GH_D08G1569.1)~3 282(GH_A07G0755.1)bp,氨基酸数在376~1 093,分子量在42.82(GH_D08G1569.1)~120.99(GH_A07G0755.1)kD,理论等电点为4.64~9.56,其中40个基因编码蛋白呈酸性,其余为碱性。有2个蛋白定位在细胞质中,分别为GH_A01G2363.1、GH_D01G2442.1,2个蛋白定位在液泡中,分别为GH_A09G1944.1、GH_D09G1898.1,8个蛋白定位在叶绿体中,分别为GH_A08G2091.1、GH_A09G2222.1、GH_A11G1709.1、GH_A11G3723.1、GH_D08G2107.1、GH_D10G2421.1、GH_D11G1747.1、GH_D11G3750.1,3个蛋白定位在线粒体中,分别为GH_A12G2487.1、GH_D02G0253.1、GH_D12G2500.1,其60个蛋白全部定位在细胞核内。表1
2.2 陆地棉TRM基因家族的进化与结构
研究表明,拟南芥中34个TRM基因家族与陆地棉中的75个TRM基因家族能聚类为3个亚组。其中Cluster Ш中成员最多,TRM基因家族数量最多,包含52个(36个陆地棉TRM基因和16个拟南芥TRM基因),家族成员占总数的48%。图1
TRM基因家族的外显子个数在2~7个,并
从中共鉴定出10个保守基序依次命名为Motif 1 ~ Motif 10。所有TRM基因家族成员均含有相同的保守基序Motif 4,另外有47个具有N端的Motif 5 ,其余28个缺失 N端的Motif 5。保守结构域除了GH_D12G2464.1、GH_A12G2452.1、GH_D01G1490.1以外,其余都含有DUF4378结构域。图2,图3
2.3 陆地棉TRM基因家族的啟动子顺式作用元件
研究表明,TRM基因家族含有较多的光响应相关元件、激素反应相关元件、胁迫响应相关元件和生长发育相关元件。其中光反应元件的数量较多且种类丰富。在激素响应元件中,生长素响应元件含量较多。图4
2.4 陆地棉TRM基因家族的染色体分布与共线性
研究表明,陆地棉TRM基因家族在染色体上的分布比较广泛,且数量有较大差异。其中A08、A09、A11、D07、D08、D09和D11共7条染色体均有多达5个基因,在A07染色体分布有4个基因,在A01、A12、D01、D02、D12 5条染色体分布有3个基因,在A02、A03、A05、A10、D03、D04、D05、D10 8条染色体上分布有2个基因,在A04、A13、D13三条染色体上分布有1个基因。图5
160个共线性基因对其中A亚族有29对,D亚族有30对,AD亚族有101对,陆地棉TRM基因家族为种间协同进化。图6
2.5 陆地棉TRM基因家族的表达
研究表明,75个陆地棉TRM基因家族成员分为3个亚家族,其中第Ⅰ亚家族共有17个成员,普遍在纤维中优势表达,第Ⅱ亚家族有35个成员,普遍在0 d的胚珠与早期纤维中优势表达。第Ⅲ共有共23个成员,在植物的各个组织中都有表达。图7
2.6 陆地棉TRM基因家族的单倍型
研究表明,全部陆地棉TRM基因家族成员在1 245陆地棉材料中基因的单倍型个数在1~8个。其中在46个基因中鉴定出与棉花纤维的长度、强度、马克隆值相关的优异单倍型。表2
与纤维长度相关单倍型的基因有41个,其中有14个基因携带显著增长纤维长度的优异单倍型。与纤维强度相关单倍型的基因有38个,其中18个基因携带显著增强纤维强度的优异单倍型。与马克隆值相关单倍型的基因有35个,其中15个基因携带显著降低马克隆值的优异单倍型。由于其中一些优异单倍型存在一因多效,29个基因同时拥有与纤维长度、强度、马克隆值均具有显著差异的单倍型,其中11个基因的携带一因多效的优异单倍型。其中GH_D09G0775的Hap_4单倍型显著增长纤维长度,增强纤维强度,显著降马克隆值;GH_D03G1434的Hap_3单倍型同样显著增长纤维长度,增强纤维强度,显著降低马克隆值。图8
3 讨 论
有报道显示TRM1能够与微管蛋白结合,并通过与TON1的相互作用将其招募到微管蛋白[7]。AtTRM5对拟南芥的生长发育起着重要作用,AtTRM5突变体具有明显的发育缺陷表型如叶片生长变慢、花发育迟缓、根变短等[8]。AtTRM4参与纤维素的合成从而调控拟南芥种子粘液质的沉积[9]。
基因组质量的完整性是全基因组水平分析基因家族的基础。随着基因组测序技术的不断深入,棉花基因组从二倍体棉到四倍体陆地棉不断更新[24,25,26]。研究通过生物信息学方法,以拟南芥中的34个TRM家族蛋白为参考从陆地棉全基因组中鉴定出75个TRM基因家族成员,依据进化可以将其与75个陆地棉TRM基因家族成员分为3个亚族,所编码的氨基酸数量在376~1 093,都含有相同的保守结构域MOTIF4(是TON1的互作元件)。陆地棉TRM基因家族成员在除了A06与D06两条染色体以外的24条染色体上均有分布,共线性分析结果发现其家族为种间协同进化。Ma等在群体中定位到的mqFL-lg15-3区间中出现TRM基因家族[27],TRM基因家族成员普遍在纤维发育时期优势表达,其可能与纤维发育相关。
4 结 论
陆地棉TRM基因家族具有丰富的序列及理化性质变异,该基因家族在棉花中可能参与不同发育过程。陆地棉TRM家族成员可能与棉花纤维发育相关。共有50个陆地棉TRM基因在纤维中较高表达,陆地棉TRM基因潜在调控棉花纤维发育。
46个基因的不同单倍型在纤维品质性状有表型差异,均在纤维中优势表达,同时存在一因多效,与纤维长度相关拥有优异单倍型的基因是14个;与纤维强度相关拥有优异单倍型的基因是18个;与纤维马克隆值相关拥有优异单倍型的基因是15个。同时拥有与纤维长度、强度、马克隆值相关优异单倍型的基因是11个。
参考文献(References)
[1]
赵坤宇,陈甜甜.棉花转录因子SRS基因家族的鉴定与分析[J/OL].分子植物育种,2022:1-11.(2022-08-04).https://kns.cnki.net/kcms/detail/46.1068.S.20220803.1704.014.html.
ZHAO Kunyu,CHEN Tiantian.Genome-wide identification and analysis of SRS gene family in cotton [J/OL].Molecular Plant Breeding:2022:1-11.(2022-08-04).https://kns.cnki.net/kcms/detail/46.1068.S.20220803.1704.014.html.
[2]王坤波,刘旭.棉属多倍化研究进展[J].中国农业科技导报,2013,15(2):20-27.
WANG Kunbo,LIU Xu.Study progress of polyploidization [J].Journal of Agricultural Science and Technology,2013,15(2):20-27.
[3]Hu Y,Chen J D,Fang L,et al.Gossypium barbadense and Gossypium hirsutum genomes provide insights into the origin and evolution of allotetraploid cotton[J].Nature Genetics,2019,51(4):739-748.
[4]Wang M J,Tu L L,Yuan D J,et al.Reference genome sequences of two cultivated allotetraploid cottons,Gossypium hirsutum L. and Gossypium barbadense[J].Nature Genetics,2019,51(2):224-229.
[5]李雨哲,邢淋雪,刘梦洁,等.棉花苯丙氨酸解氨酶基因家族的生物信息学分析[J].棉花学报,2021,33(1):66-74.
LI Yuzhe,XING Linxue,LIU Mengjie,et al.Bioinformatics analysis of the phenylalanine ammonia lyase(PAL) gene family in cotton[J].Cotton Science,2021,33(1):66-74.
[6]李豪,虎亞静,张清霞,等.黄瓜TRM基因家族鉴定及分析[J].山东农业大学学报(自然科学版),2021,52(3):358-363.
LI Hao,HU Yajing,ZHANG Qingxia,et al.Identification and analysis on TRM family in cucumber[J].Journal of Shandong Agricultural University(Natural Science Edition),2021,52(3):358-363.
[7]Drevensek S,Goussot M,Duroc Y,et al.The Arabidopsis TRM1-TON1 interaction reveals a recruitment network common to plant cortical microtubule arrays and eukaryotic centrosomes[J].The Plant Cell,2012,24(1):178-191.
[8]Guo Q Q,Ng P Q,Shi S S,et al.Arabidopsis TRM5 encodes a nuclear-localised bifunctional tRNA guanine and inosine-N1-methyltransferase that is important for growth[J].PLoS One,2019,14(11):e0225064.
[9]Yang B,Voiniciuc C,Fu L B,et al.TRM4 is essential for cellulose deposition in Arabidopsis seed mucilage by maintaining cortical microtubule organization and interacting with CESA3[J].The New Phytologist,2019,221(2):881-895.
[10]Schaefer E,Belcram K,Uyttewaal M,et al.The preprophase band of microtubules controls the robustness of division orientation in plants[J].Science,2017,356(6334):186-189.
[11]Tang J,Jia P F,Xin P Y,et al.The Arabidopsis TRM61/TRM6 complex is a bona fide tRNA N1-methyladenosine methyltransferase[J].Journal of Experi-mental Botany,2020,71(10):3024-3036.
[12]Qanmber G,YU D Q,LI J,et al.Genome-wide identification and expression analysis of Gossypium RING-H2 finger E3 ligase genes revealed their roles in fiber development,and phytohormone and abiotic stress responses[J].Journal of Cotton Research,2018,1(1):1.
[13]YANG X,XU Y C,YANG F F,et al.Genome-wide identification of OSCA gene family and their potential function in the regulation of dehydration and salt stress in Gossypium hirsutum[J].Journal of Cotton Research,2019,2(1):11.
[14]柳思思.玉米耐旱功能標记开发及优异单倍型应用[D].乌鲁木齐:新疆农业大学,2012.
LIU Sisi.The Development of Functional Market and the Application of Superior Haplotype Associated with Drought Tolerance in Maize(zea Mays L.)[D].Urumqi:Xinjiang Agricultural University,2012.
[15]Zhu T,Liang C,Meng Z G,et al.CottonFGD:an integrated functional genomics database for cotton[J].BMC Plant Biology,2017,17(1):101.
[16]Mount D W.Using the basic local alignment search tool(BLAST)[J].Cold Spring Harbor Protocols,2007,2007(7):pdb.top17.
[17]Finn R D,Clements J,Eddy S R.HMMER web server:interactive sequence similarity searching[J].Nucleic Acids Research,2011,39:W29-W37
[18]Chen C J,Chen H,Zhang Y,et al.TBtools:an integrative toolkit developed for interactive analyses of big biological data[J].Molecular Plant,2020,13(8):1194-1202.
[19]Hall B G.Building phylogenetic trees from molecular data with MEGA[J].Molecular Biology and Evolution,2013,30(5):1229-1235.
[20]Larkin M A,Blackshields G,Brown N P,et al.Clustal W and Clustal X version 2.0.[J].Bioinformatics,2007,23(21):2947-2948.
[21]Lescot M.PlantCARE,a database of plant cis-acting regulatory elements and a portal to tools for in silico analysis of promoter sequences[J].Nucleic Acids Research,2002,30(1):325-327.
[22]Ma Z Y,He S P,Wang X F,et al.Resequencing a core collection of upland cotton identifies genomic variation and loci influencing fiber quality and yield[J].Nature Genetics, 2018,50(6):803-813.
[23]He S P,Sun G F,Geng X L,et al.The genomic basis of geographic differentiation and fiber improvement in cultivated cotton[J].Nature Genetics,2021,53(6):916-924.
[24]Li F,Fan G,Lu C,et al.Genome sequence of cultivated Upland cotton(Gossypium hirsutum L. TM-1) provides insights into genome evolution[J].Nat Biotechnol,2015,33(5):524-530.
[25]Zhang T Z,Hu Y,Jiang W K,et al.Sequencing of allotetraploid cotton(Gossypium hirsutum L.acc.TM-1) provides a resource for fiber improvement[J].Nature Biotechnology,2015,33(5):531-537.
[26]Li F,Fan G,Wang K,et al.Genome sequence of the cultivated cotton Gossypium arboreum[J].Nat Genet,2014,46(6):567-72.
[27]Ma J J,Jiang Y F,Pei W F,et al.Expressed genes and their new alleles identification during fibre elongation reveal the genetic factors underlying improvements of fibre length in cotton[J].Plant Biotechnology Journal,2022,20(10):1940-1955.
Identification of TRM gene family and fiber quality related excellent haplotype analysis in Gossypium hirsutum L.
WANG Kaidi1,GAO Chenxu2,PEI Wenfeng1,2,YANG Shuxian2,ZHANG Wenqing2,SONG Jikun1,2,MA Jianjiang2,WANG Li2,YU Jiwen1,2,CHEN Quanjia1
(1.Cotton Engineering Research Center,Ministry of Education / College of Agronomy,Xinjiang Agricultural University,Urumqi 830052,China;2.Institute of Cotton Research of Chinese Academy of Agricultural Sciences/State Key Laboratory of Cotton Biology/Key Laboratory of Cotton Genetic Improvement,Ministry of Agriculture,Anyang Henan 455000,China)
Abstract:【Objective】 Identification of TRM gene family and fiber quality related excellent haplotype analysis in Gossypium hirsutum L.
【Methods】 Bioinformatics was used to analyze the evolutionary relationship,physicochemical properties and cluster expression of TRM gene families.Candidate genes regulating fiber quality traits(fiber length,strength,and Macron value) were screened by gene haplotype analysis.
【Results】 The analysis of physicochemical properties showed that the amino acids encoded by this family gene ranged from 376-1,093,with isoelectric points ranging from 4.64-9.56.Subcellular localization predicted that up to 60 TRM family genes were localized in the nucleus.The results of cis-acting element analysis showed that TRM gene family contained more elements related to light response,hormone response,stress response and growth and development.Transcriptome data showed that 60 TRM family genes were expressed preferentially during fiber development,which might regulate cotton fiber development.Furthermore,1,245 upland cotton materials were used to genotype TRM,and the number of haplotypes of each gene was found to be 1-8,and 14,18 and 15 excellent haplotype TRM genes related to fiber length,strength and Macron value were screened,respectively.Among them,11 genes had excellent haplotypes of length,strength and micronotype.The Hap_4 haplotype of GH_D09G0775 and the Hap_3 haplotype of GH_D03G1434 increased fiber length and strength while decreasing the Macron value.
【Conclusion】 In this study,75 TRM family members were identified on 24 chromosomes in upland cotton(Gossypium hirsutum L.),which were systematically divided into cluster evolution Ⅰ-Ⅲ3 subtribe.
Key words:Gossypium hirsutum L.; gene family; gene expression; fiber quality; haploidgenotype
Fund projects:Henan Province Agricultural Improved Seed Joint Research Project(2022010301); Natural Science Foundation of Xinjiang Uygur Autonomous Region(2021D01B113); Basic Research Funds for Public Welfare Research Institutes of the Central Government(1610162022013)
Correspondence author:YU Jiwen(1978-),female,Anyang,from Henan,researcher,doctors,research direction: cotton molecular breeding,(E-mail) yujw666@hotmail.com
CHEN Quanjia(1972-),male,from Urumqi, Xinjiang,professor,doctors,research direction: cotton genetic breeding,(E-mail)chqjia@126.com
