集胞蓝细菌PCC 6803中非生物胁迫响应的核心基因分析
2023-12-19程亚蕊黄世鹏张天缘张建猛王锋尖
程亚蕊,黄世鹏,张天缘,张建猛,王锋尖
集胞蓝细菌PCC 6803中非生物胁迫响应的核心基因分析
程亚蕊1,黄世鹏1,张天缘2,张建猛1,王锋尖1
(1. 汉江师范学院 化学与环境工程学院,湖北 十堰 442000;2. 华中农业大学 生命科学技术学院,湖北 武汉 430070)
以集胞蓝细菌PCC 6803的多组转录组数据为对象,鉴定蓝细菌在非生物胁迫下共同表达的核心差异基因(CDGs)).结果表明,9个对氮、铁、碳、磷缺乏响应的CDGs被鉴定.京都基因和基因组百科全书(KEGG)分析表明,氮代谢、ABC转运蛋白和氨基酸代谢与营养缺乏有关.研究还鉴定了热激处理下共同表达的CDGs,并且代谢通路ABC转运蛋白和碳固定也与热胁迫相关.同时构建了CDGs的蛋白质-蛋白质相互作用网络,以预测基因在应对胁迫可能的响应机制.这些候选基因库将有助于加速理解蓝细菌在应激中的调控模式.
集胞蓝细菌PCC 6803;非生物胁迫;核心差异基因;KEGG;蛋白互作
蓝细菌属于革兰阴性的光合生物,通过捕获太阳能来增强其高效的光自养代谢,进而固定无机碳和氮[1].蓝细菌的光系统与高等植物相似,可在各种环境压力下成功生存,因此其可以作为高等植物研究非生物胁迫下光合作用和信号转导机制的优秀模型[2].由于集胞蓝细菌PCC 6803(sp. PCC 6803)生长速度相对较快、基因组较小且易于修饰的特点,已成为研究各种应激响应的模式生物[3]71.组学测序技术已成功应用于研究蓝细菌对胁迫的响应.前期研究通过整合转录组学、蛋白质组学和代谢组学数据,鉴定了集胞蓝细菌对盐胁迫响应的基因以及激活平衡渗透压的基因[4].盐浓度过高会破坏细胞内离子稳态,并导致活性氧的积累,进而导致光合系统的破坏和膜脂质的过氧化[5].蓝细菌对3种不同光照(蓝、橙、红)适应过程的相关基因也通过转录组测序进行了鉴定[6].集胞蓝细菌PCC 6803在缺铁和氧化胁迫下的转录组数据也确定了该菌株在各种应激下的调控机制[7].
不同类型的胁迫不仅会导致蓝细菌发生类似的生理变化,还可能诱导一系列相同基因的表达,并激活相关蛋白来响应非生物应激.基于不同的组学数据可以比较分析响应压力源的共同基因和代谢途径.集胞蓝细菌应激的多转录组比较分析确定了其对己醇、苯甲醇和丁醇等响应的共同表达基因[8].多组氮缺乏的转录组数据的分析揭示了可能参与异形胞分化的新候选基因[9]2545.然而,尚未有多组转录组数据的比较分析来确定蓝细菌对营养缺乏响应的共同基因和代谢途径.
本文重新分析集胞蓝细菌PCC 6803的3种不同营养限制和2种热激处理转录组数据集,并确定细胞对应激因素的响应.通过生物信息学分析,确定胁迫响应的共同表达基因和代谢途径,构建共同表达基因的蛋白质-蛋白质相互作用(protein-protein interaction,PPI)网络,确定参与蓝细菌抗胁迫的重要基因.这些候选基因库将有助于加速理解蓝细菌在应激中的调控机制,以期为蓝细菌和植物对胁迫响应机制提供理论基础.
1 材料与方法
1.1 转录组数据采集及分析
集胞蓝细菌PCC 6803不同应激处理的转录组数据集从国家生物技术信息中心(NCBI,https://www.ncbi.nlm.nih.gov/sra)下载.本文共筛选4个不同的数据集(SRP102282,缺氮4 h,每个处理2个重复;SRP301698,缺铁4 h,每个处理3个重复;SRP032228,缺碳20 h,缺磷12 h,42℃ 30 min,对照2个重复,其他无重复;SRP3328835,39℃ 1 h,每个处理3个重复),共21个样本组成.通过FastQC处理原始数据获得高质量的数据.Bowtie2用于将高质量数据与集胞蓝细菌PCC 6803参考基因组进行比对[10].通过比较应激和对照样品中的基因表达水平来鉴定共同表达的核心差异基因(core differential genes,CDGs).
1.2 CDGs的富集分析
通过比较不同数据集中的差异表达基因(倍数变化≥2.00和<0.05),并基于维恩图分析应激响应的CDGs,然后对这些基因进行基因本体论(Gene Ontology,GO)、京都基因和基因组百科全书(Kyoto Encyclopedia of Genes and Genomes,KEGG)富集分析,以确定与营养缺乏和热激有关的代谢途径.使用clusterProfiler 3.8版进行GO和KEGG富集分析.
1.3 PPI网络分析
基于STRING数据库(https://string-db.org/cgi/input.pl)可以对已知和未知的蛋白质-蛋白质关联数据进行收集和整合[11].可能的相互关系参考的标准是置信度得分为0.4,交互因素的最大数量为0.交互预测主要来自文本挖掘、实验、数据库、共表达、邻域、基因融合和共出现.
2 结果与分析
2.1 与营养缺乏相关的CDGs鉴定
首先分析在氮、铁、碳、磷缺乏下的转录组数据集.在营养缺乏的情况下,不同数据集的总体基因表达谱(见图1a)是相似的,表明集胞蓝细菌对胁迫的反应是相容的.由维恩图(见图1b)可见,在不同的样品中共鉴定出1 132个差异表达基因,但不同数据集仅鉴定了9个在不同营养限制中保守的CDGs.这9个基因分别编码缺铁诱导的叶绿素结合蛋白、Fe2+转运蛋白、50S核糖体蛋白L4、异天冬氨酰肽酶和未知蛋白等(见表1).每个数据集特有的数百个基因可能是由菌株对不同应激处理和生长条件的耐受水平差异引起的.

图1 与营养缺乏相关CDGs的鉴定
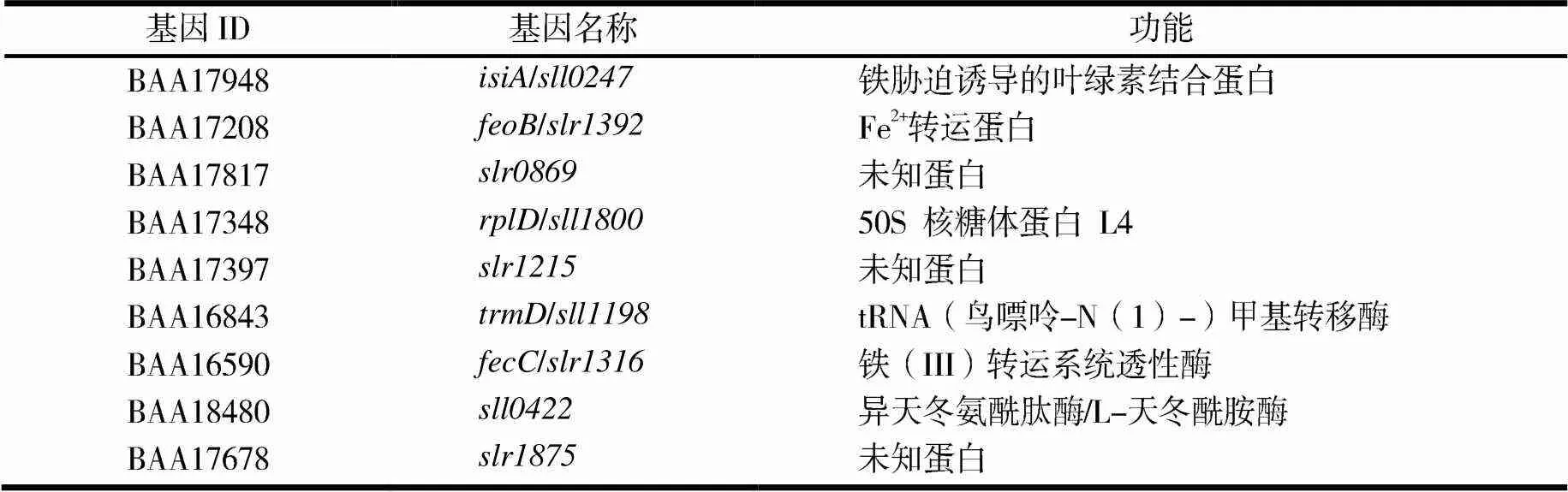
表1 不同数据集中响应营养限制的CDGs
对在不同数据集中检测共同表达的CDGs进行了GO和KEGG富集分析(见图2).在铁和氮缺乏的情况下,对46个CDGs的GO分析(见图2a)表明,所富集的生物过程与膜的组成、类囊体膜和质膜有关.KEGG分析(见图2b)表明,ABC转运蛋白是最显著富集的代谢通路,其次是氮代谢,分别有35%,18%的CDGs参与这些途径,表明转运蛋白在营养缺乏的情况下发挥着关键作用.在氮、碳、磷缺乏的情况下,对48个CDGs的KEGG富集分析(见图2c)表明,核糖体是最显著的生物过程,其次是氨基酸代谢.在铁、碳、磷缺乏的情况下,CDGs主要富集在ABC转运蛋白和丙氨酸、天冬氨酸和谷氨酸代谢(见图2d).这些结果表明,氨基酸代谢可能与营养限制有关,因为菌株需要更多的氨基酸来合成蛋白质以应对营养缺乏.
2.2 响应温度的CDGs鉴定
对转录组数据集SRP332228(42℃,30 min)和SRP3328835(39℃,1 h)进行比较分析,以探究与温度相关的基因.35个共同表达的CDGs被检测(见图3a).由缺氮、缺铁和热激下的CDGs(见图3b)可见,该结果仅检测到3个共同表达的CDGs,包括BAA17923(,未知蛋白)、BAA16628(,ABC转运蛋白)和BAA16842(,铁摄取蛋白A1),其在碳和磷缺乏下并未差异表达.该结果表明,在不同胁迫处理下,共有基因较少,可能是由于蓝细菌本身的基因数量较少及不同数据集中菌株的生长状态和胁迫处理不同.GO分析(见图3c)主要富集在细胞质、ATP结合和蛋白水解.KEGG分析(见图3d)显示,ABC转运蛋白是最富集的代谢途径,其次是碳固定,该结果与营养缺乏下的分析一致,表明ABC转运蛋白可能在各种胁迫下均发挥重要作用,这可能是由于菌株在应激时会增强营养物质的吸收和运输.

图3 与热激相关CDGs的鉴定
2.3 CDGs的PPI网络分析
为了探究在营养缺乏下9个共有表达CDGs的功能,基于STRING数据库中寻找这些基因的PPI信息,每个PPI网络由11个节点组成.部分CDGs的蛋白互作网络见图4.与IsiA相关的蛋白质是光系统I的亚基,与RplD相关性较高的蛋白质是核糖体亚基;与FecC相关性较高的蛋白主要与铁转运有关;与未知蛋白质(Sll0422,Slr0869,Slr1726,Slr1113)相互作用的蛋白质主要是未知蛋白质.这说明应对胁迫的基因并不是独立发挥作用,而是与其具有类似功能的基因共同作用来抵抗不利条件.

图4 CDGs的PPI网络分析
3 讨论
本文通过比较不同胁迫下的转录组数据集,以确定蓝细菌对应激响应的共有基因和代谢途径.与植物相比,集胞蓝细菌的基因数量相对较少,只有3 000多个[3]1173.因此,本文通过比较转录组分析确定的共有表达的CDGs较少.念珠藻PCC 7120中通过将2组转录组数据进行比较分析来筛选与异形胞分化相关的基因,但该研究并未使用系统生物信息学的分析方法[9]2555.前期在集胞蓝细菌中也未有相关的比较分析来确定其在非生物胁迫下的共有核心基因.
在营养缺乏的情况下,CDGs富集在ABC转运蛋白、氮代谢和氨基酸代谢中.集胞蓝细菌PCC 6803基因组中有50多个基因编码ABC转运蛋白,其功能主要涉及各种化合物和营养物质的运输[12].植物在营养限制下会诱导蛋白质降解,激活呼吸途径,并利用氨基酸作为替代底物来应对能量不足.氨基酸也可以作为信号分子来协调植物生长和胁迫响应[13],蓝细菌也可能具有与植物类似的响应机制.在氮和铁缺乏下,CDGs也富集到氮代谢.铁摄取调节因子FurA参与念珠藻PCC 7120中的异形胞分化[14].总氮调节因子NtcA可以控制基因的表达以调节铁的获取,从而保护蓝细菌免受缺铁引起的氧化应激的影响[15].这表明蓝细菌中的铁和氮调节网络存在相互交叉.响应温度的CDGs的KEGG分析主要富集在ABC转运蛋白和碳固定途径.为了应对低温胁迫导致的光合速率降低,香蕉会上调磷酸烯醇丙酮酸羧化酶,以提高固碳效率[16].因此,在不利条件下生长的植物可能会导致光合效率下降,并通过调节碳固定来应对能量不足.
PPI网络显示与IsiA高度相关的蛋白质均为PS I亚单位.基因与光系统II的具有序列相似性,前者参与铁缺乏、氧化胁迫、热激或盐胁迫下的调节[17-19].这表明蓝细菌中IsiA在胁迫下的调控发挥重要作用.大多数FeoB相关蛋白属于ABC转运蛋白,其在N-末端具有典型的ATP结合基序,其基因的缺失导致细胞利用亚铁的能力显著降低.Ssr2333是一种未知的蛋白质,但与FeoB的相关性最高,这表明Ssr2333可能也属于ABC转运蛋白.未知蛋白Sll0422和CphB(藻青素酶)具有最高的相关性,CphB可以水解-Asp-Arg键,其也与细菌的碳利用有关[20].这一系列基因在集胞蓝细菌PCC 6803应对胁迫时的调控机制还有待进一步的验证.因此,应构建并筛选基因的缺失和过表达菌株,观察其在不同胁迫下的生长,进而判断基因对蓝细菌抗逆性的影响.若突变体的表型有明显变化,则可通过转录组测序来进一步确定该基因在各胁迫下的调控机制.
综上所述,本文从集胞蓝细菌PCC 6803的不同转录组数据集中鉴定了响应不同非生物胁迫的CDGs.KEGG分析显示,ABC转运蛋白、氮代谢、氨基酸代谢和碳固定等代谢途径与非生物胁迫相关.本文鉴定了9个在营养缺乏下共同表达的CDGs,并基于PPI网络分析揭示了可能在蓝细菌应激反应作为重要调节因子的假定蛋白质.此研究将为了解蓝细菌对非生物应激反应的通用信号奠定坚实的基础.
[1] Veaudor T,Blanc-garin V,Chenebault C,et al.Recent advances in the photoautotrophic metabolism of cyanobacteria:biotechnological implications[J].Life,2020,10(5):71.
[2] Jensen P E,Leister D.Cyanobacteria as an experimental platform for modifying bacterial and plant photosynthesis[J].Frontiers in Bioengineering and Biotechnology,2014(2):7.
[3] Kaneko T,Tabata S.Complete genome structure of the unicellular cyanobacteriumsp. PCC 6803[J].Plant and Cell Physiology,1997,38(11):1171-1176.
[4] Klähn S,Mikkat S,Riediger M,et al.Integrative analysis of the salt stress response in cyanobacteria[J].Biology Direct,2021,16(1):26.
[5] YANG Z,LI J L,LIU L N,et al.Photosynthetic regulation under salt stress and salt-tolerance mechanism of sweet sorghum[J].Frontiers in Plant Science,2020,10:1722.
[6] Luimstra V M,Schuurmans J M,Hellingwerf K J,et al.Blue light induces major changes in the gene expression profile of the cyanobacteriumsp. PCC 6803[J].Physiologia Plantarum,2020,170(1):10-26.
[7] CHENG Y,ZHANG T,WANG L,et al.Transcriptome analysis reveals IsiA-regulatory mechanisms underlying iron depletion and oxidative-stress acclimation insp. strain PCC 6803[J].Applied and Environmental Microbiology,2020,86(13):e00517-e00520.
[8] Mironov K S,Kupriyanova E V,Shumskaya M,et al.Alcohol stress on cyanobacterial membranes:New insights revealed by transcriptomics[J].Gene,2021,764:145055.
[9] Brenes-álvarez M,Mitschke J,Olmedo-verd E,et al.Elements of the heterocyst-specific transcriptome unravelled by co-expression analysis insp. PCC 7120[J].Environmental Microbiology,2019,21(7):2544-2558.
[10] Langmead B,Salzberg S L.Fast gapped-read alignment with Bowtie 2[J].Nature Methods,2012,9(4):357-359.
[11] Szklarczyk D,Franceschini A,Wyder S,et al.STRING v10:protein-protein interaction networks,integrated over the tree of life[J].Nucleic Acids Research,2015,43:D447-D452.
[12] Shvarev D,Maldener I.ATP-binding cassette transporters of the multicellular cyanobacteriumsp. PCC 7120:a wide variety for a complex lifestyle[J].FEMS Microbiology Letters,2018,365(4):fny012.
[13] Heinemann B,Hildebrandt T M.The role of amino acid metabolism in signaling and metabolic adaptation to stress-induced energy deficiency in plants[J].Journal of Experimental Botany,2021,72(13):4634-4645.
[14] González A,Bes M T,Peleato M L,et al.Expanding the role of FurA as essential global regulator in cyanobacteria[J].PLoS One,2016,11(3):e0151384.
[15] Kaushik M S,Srivastava M,Singh A,et al.Impairment ofgene revealed its role in regulating iron homeostasis,ROS production and cellular phenotype under iron deficiency in cyanobacteriumsp. PCC 7120[J].World Journal of Microbiology & Biotechnology,2017,33(8):158.
[16] LIU J,Takáč T,YI G,et al.Acceleration of carbon fixation in chilling-sensitive banana under mild and moderate chilling stresses[J].International Journal of Molecular Sciences,2020,21(23):9326.
[17] Kojima K,Suzuki-Maenaka T,Kikuchi T,et al.Roles of the cyanobacterial isiABC operon in protection from oxidative and heat stresses[J].Plant Physiology,2006,128(3):507-519.
[18] MA F,ZHANG X,ZHU X,et al.Dynamic changes of IsiA-containing complexes during long-term iron deficiency insp. PCC 6803[J].Molecular Plant,2017,10(1):143-154.
[19] Yousef N,Pistorius E K,Michel K P.Comparative analysis of idiA and isiA transcription under iron starvation and oxidative stress inelongatus PCC 7942 wild-type and selected mutants[J].Archives of Microbiology,2003,180(6):471-483.
[20] Hejazi M,Piotukh K,Mattow J,et al.Isoaspartyldipeptidase activity of plant-type asparaginases[J].Biochemical Journal,2002,364(1):129-136.
Analysis of core genes associated with abiotic stress response insp. PCC 6803
CHENG Yarui1,HUANG Shipeng1,ZHANG Tianyuan2,ZHANG Jianmeng1,WANG Fengjian1
(1. School of Chemistry and Environmental Engineering,Hanjiang Normal University,Shiyan 442000,China;2. School of Life Science and Technology,Huazhong Agricultural University,Wuhan 430070,China)
Multiple sets of transcriptome datasets ofsp. PCC 6803 was applied to identify core differential genes(CDGs)under abiotic stress.Results showed that 9 co-expressed CDGs responsive to nitrogen,iron,carbon and phosphorus deficiency were identified.The Kyoto Encyclopedia of Genes and Genomes(KEGG)analysis showed that nitrogen metabolism,ABC transporters,and amino acid metabolism were related to nutrient deficiency.The co-expressed CDGs were also identified under heat shock,the ABC transporters and carbon fixation were also related to heat stress.At the same time,a protein-protein interaction network of CDGs was constructed to predict the possible response mechanism of genes in response to stress.These candidate gene libraries will help accelerate the understanding of the regulatory patterns of cyanobacteria under various stresses.
sp. PCC 6803;abiotic stress;core differential genes;KEGG;protein-protein interaction
1007-9831(2023)11-0050-06
Q93
A
10.3969/j.issn.1007-9831.2023.11.010
2023-07-08
汉江师范学院重点项目(XJ2022A02);湖北省大学生创新创业训练计划项目(S202210518023);汉江师范学院一般项目(XJ2022030)
程亚蕊(1994-),女,河南信阳人,讲师,博士,从事微生物多样性和生物信息学研究.E-mail:chengyarui@hjnu.edu.cn
王锋尖(1976-),男,江西高安人,教授,博士,从事微生物生态学研究.E-mail:13972500499@163.com
