帚状江蓠质体UPA和线粒体CO I 基因的序列比较及其系统进化分析
2023-09-06陈素文段亚飞张文文周传朋
徐 瑞,陈素文,段亚飞,张文文,王 珺,周传朋,王 芸
(1.上海海洋大学水产与生命学院,上海 201306;2.中国水产科学研究院南海水产研究所,农业农村部水产品加工重点实验室,广州 510300)
帚状江蓠(Gracilariaedulis),隶属于红藻门(Rhodophyta),江蓠目(Gracilariales),江蓠科(Gracilariaceae),江蓠属,是一种主要分布于海南省和广东省沿海的大型海藻[1-2]。帚状江蓠不仅具有改善水体环境、修复生态系统的功能[3-4],且富含海藻多糖、藻胶、多种氨基酸和微量元素,是生产工业琼胶和制作水产动物饵料的重要原料[5-7]。最新研究表明,帚状江蓠提取物在抗菌和抑制肿瘤细胞增殖等医学领域亦发挥作用[8-10]。目前获得帚状江蓠的途径主要依靠捕捞野生资源,因此,为了实现帚状江蓠种质资源的可持续利用,需要开展帚状江蓠种质资源的鉴定评价与遗传多样性分析。
质体基因组和线粒体基因组具有细胞质基质基因组单系遗传和克隆进化的特征,在植物群体遗传学研究中应用广泛[11]。其中,质体UPA基因(质体23S rRNA V区域)作为通用扩增子,具有通用性强、扩增率高等特点,在大型海藻的分子鉴定中常被选为候选标记[12-14]。线粒体COⅠ基因片段具有母系遗传、进化速度快的特点[15],已被公认为是有效区分不同动物物种的DNA条形码标准区域[16],作为海洋红藻的DNA条形码,可以用于区分亲缘关系相近的物种和种内群体的生物地理亚群[17-18]。本研究采集了海南省文昌和莺歌海潮间带的野生帚状江蓠,对其质体UPA基因片段和线粒体COⅠ基因片段进行了序列分析,并结合GenBank数据库中红藻门其他藻类的UPA和COⅠ序列,从分子水平上对其遗传距离与系统进化进行分析,以期为帚状江蓠种质鉴定和系统进化研究提供基础数据。
1 材料与方法
1.1 实验材料
帚状江蓠样品于2020年7月采自海南文昌和莺歌海近海潮间带,采集30株整株藻体,活体运回实验室,取藻体幼嫩茎叶组织置于95%乙醇,-20℃保存,以待后续提取DNA。
1.2 DNA的提取与PCR扩增测序
取出95%乙醇中保存的帚状江蓠茎叶组织吸干水分(约20 mg),采用液氮研磨和吸附柱法提取样品DNA,试剂盒选用广州迈宝瑞生物科技有限公司的磁珠法植物DNA提取试剂盒。提取的DNA经1%琼脂糖凝胶电泳和核酸定量仪检测后于-80℃保存。
以帚状江蓠基因组DNA为模板,进行UPA基因片段和COⅠ基因片段的扩增,引物的选择参照文献[12]和文献[18](表1)。引物p23SrV_F和p23SrV_R在红藻门中具有较好的扩增效果,目前已在红藻门和绿藻门中成功扩增多个物种[12,19-22];COⅠ基因选用的引物GazF1和GazR1是目前红藻门、褐藻门藻类最常用的引物[18-22]。引物购自上海生工生物工程股份有限公司,PCR试剂购自艾科瑞生物工程有限公司,反应体系参照试剂说明。

表1 本实验所用引物序列及反应程序Tab.1 Primer sequences and reaction procedure used in this study
将经琼脂糖凝胶电泳检验过有明亮清晰条带的产物送往上海生工生物工程股份有限公司进行纯化回收测序。为避免测序机器测量误差及确保PCR产物为目的基因扩增产物,每个样品做两个平行,送两管PCR产物进行测序。
1.3 序列分析
使用SeqMan程序对测序数据进行峰图校正和序列拼接,通过BLAST比对确定所获得序列为目的基因片段,利用MEGA 11进行序列比对并去除引物两端序列,获得完整目的基因序列。利用DNAsp 6.0计算序列核苷酸多样性指数和单倍型多样性指数,并用MEGA 11计算序列碱基组成,基于Kimura-2-parameter模型计算帚状江蓠种内和种间遗传距离,利用邻接法(neighbor joining method)构建系统进化树,Bootstrap value设置为1 000次重复[23]。
2 结果与分析
2.1 序列组成分析
帚状江蓠UPA和COⅠ基因片段PCR产物经测序拼接后,通过与NCBI数据库进行BLAST比对,确认所得序列为江蓠的UPA和COⅠ基因片段。利用MEGA 11软件进行序列比对,删去测序不准确的序列,分别得到370 bp的UPA基因片段和664 bp的COⅠ基因片段。
30个帚状江蓠样品的UPA基因片段共检测到1种单倍型,无多态性位点,碱基T、C、A、G和A+T的平均含量分别为26.2%、16.2%、30.3%、27.3%和56.5%。30个帚状江蓠样品的COⅠ基因片段共检测到2种单倍型(hap1、hap2),3个多态性位点,均为简约信息位点,其中转换位点2个,均为A/G转换,分别在第325和第379位点,颠换位点1个,为A/C颠换,位于第532位点,转换颠换比值为2.0(表2)。序列无缺失和插入位点;碱基T、C、A、G和A+T的平均含量分别为39.2%、15.1%、27.6%、18.1%和66.8%,与其他藻类的碱基组成研究结果一致[24-25]。

表2 帚状江蓠不同单倍型CO I 基因序列的变异位点分布Tab.2 Distribution of variab le sites of CO I gene sequence from different hap lotypes of G.edu lis
利用DNAsp 6.0软件对帚状江蓠UPA和COⅠ基因片段的遗传多样性参数进行统计(表3)。其中,UPA基因只有1种单倍型,各项遗传多样性参数均为0,UPA基因片段的遗传多样性参数均低于COⅠ基因片段,说明UPA基因片段比COⅠ基因片段更保守。

表3 帚状江蓠UPA和CO I 基因片段的遗传多样性参数Tab.3 Genetic diversity parameters of UPA and CO I gene fragments of G.edulis
2.2 基于UPA基因的遗传距离与系统进化分析
从GenBank中选择15种红藻的UPA序列作为外群物种的UPA序列,包含江蓠属、角叉菜属(Chondrus)、海头红属(Plocamium)、沙菜属(Hypnea)、蜈蚣藻属(Grateloupia)、石花菜属(Gelidium)和仙菜属(Ceramium)共7个属。外群物种的序列信息见表4,15个外群物种的UPA基因序列碱基含量差异不大,相同属的碱基含量则更为接近。
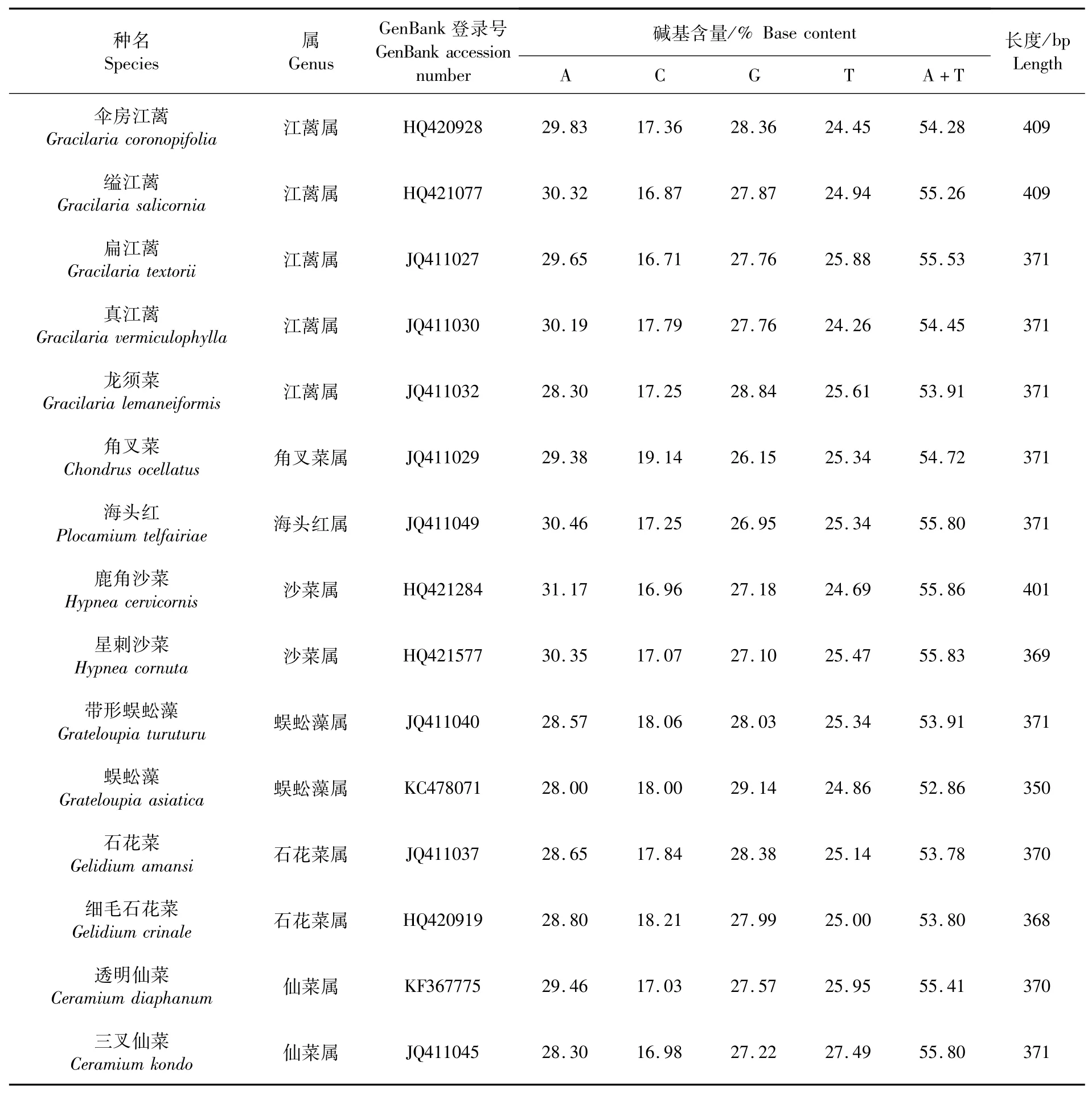
表4 外群物种UPA基因序列基本信息Tab.4 Basic data of UPA gene sequences of outgroup species
基于Kimura-2-parameter模型,计算得到PCR扩增帚状江蓠UPA基因序列的1种单倍型与15种外群物种UPA序列的遗传距离(表5)。江蓠属中,伞房江蓠(Gracilariacoronopifolia)与扁江蓠(Gracilariatextorii)遗传距离最近,为0.025;帚状江蓠与真江蓠(Gracilariavermiculophylla)遗传距离最远,为 0.089。16 种藻类中,石花菜(Gelidium amansi)和细毛石花菜(Gelidium crinale)遗传距离最近,为0.005,真江蓠与细毛石花菜遗传距离最远,为0.152。不同属间,海头红属与沙菜属间遗传距离最近,江蓠属和石花菜属间遗传距离最远。

表5 基于UPA基因片段的Kimura-2-parameter遗传距离(对角线左下)Tab.5 K imura-2-parameter genetic distances(lower left of the diagonal)based on UPA gene fragments
邻接法构建的16种红藻的UPA基因系统进化树(图1)显示,伞房江蓠与扁江蓠先汇为一支,真江蓠和缢江蓠(Gracilariasalicornia)先汇为一支,这二支汇聚后再与帚状江蓠汇聚,最后再与龙须菜(Gracilarialemaneiformis)汇聚为江蓠属。沙菜属、仙菜属和石花菜属首先分别聚为一支,随后,石花菜属与仙菜属汇为一支,沙菜属与海头红汇为一支,蜈蚣藻属和角叉菜汇为一支,最后与江蓠属汇聚。
2.3 基于CO I 基因的遗传距离与系统进化分析
从GenBank中选择15种红藻的COⅠ序列作为外群物种的COⅠ序列,包含江蓠属、角叉菜属、海头红属、沙菜属、蜈蚣藻属、石花菜属和仙菜属共7个属。外群物种的序列信息见表6,15个外群物种的COⅠ基因序列碱基含量差异不大。A+T含量显著高于C+G,相同属藻类的碱基含量更为接近。
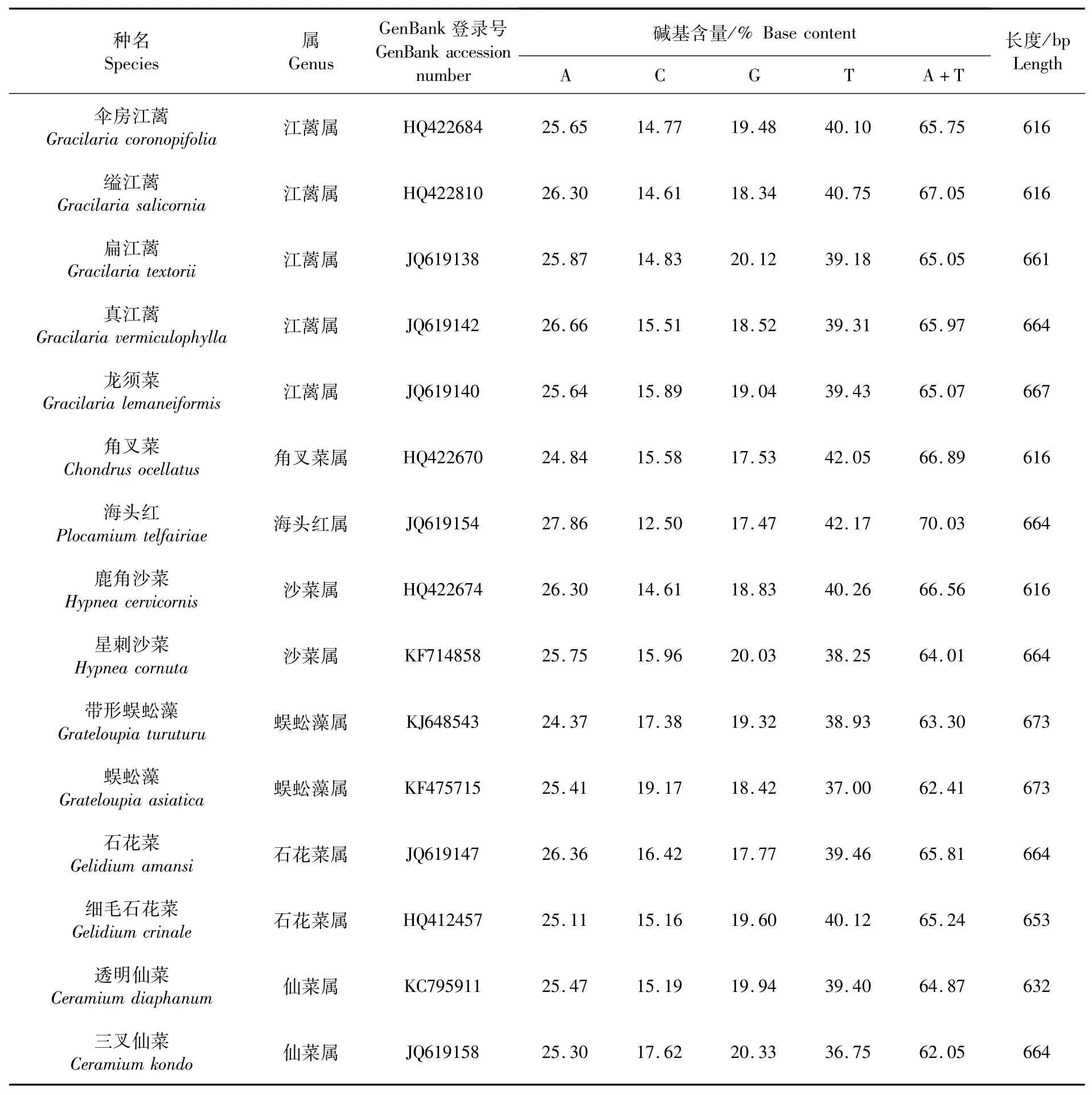
表6 外群物种CO I 基因序列信息Tab.6 Data of CO I gene sequences of outgroup species
基于Kimura-2-parameter模型,计算得到PCR扩增的帚状江蓠COⅠ基因序列的2种单倍型(hap1、hap2)与15种外群物种COⅠ序列的遗传距离(表7),帚状江蓠2种单倍型的遗传距离为0.005。江蓠属中,缢江蓠和伞房江蓠遗传距离最近,为0.127,扁江蓠和龙须菜遗传距离最远,为0.175。16种藻类中,带型蜈蚣藻和蜈蚣藻间遗传距离最近,为0.120;扁江蓠和三叉仙菜(Ceramiumkondo)遗传距离最远,为0.275。不同属间,江蓠属与仙菜属遗传距离最远,海头红属和角叉菜属遗传距离最近。

表7 基于CO I 基因片段的K imura-2-parameter遗传距离(对角线左下)Tab.7 K imura-2-parameter genetic distances(lower left of the diagonal)based on CO I gene fragments
邻接法构建的16种红藻的COⅠ基因系统进化树(图2)显示,伞房江蓠和扁江蓠先汇为一支,真江蓠和缢江蓠分别独立为一支,帚状江蓠的2种单倍型聚在一起成一支,这5种江蓠汇聚后再与龙须菜汇为江蓠属。石花菜属、蜈蚣藻属、海头红、角叉菜、沙菜属和仙菜属先各自分别汇为一支,随后,沙菜属与角叉菜和海头红汇为一支,然后再与蜈蚣藻属和江蓠属汇聚,最后与石花菜属和仙菜属汇聚。

图2 基于CO I 基因片段构建的NJ进化树Fig.2 NJ phylogenetic tree based on CO I gene fragments
3 讨论
3.1 帚状江蓠UPA和CO I 基因片段的序列比较
本研究采用PCR技术对帚状江蓠的UPA和COⅠ基因序列进行扩增,分别得到370 bp和664 bp的基因序列,序列分析结果表明,帚状江蓠UPA基因和COⅠ基因的A+T含量(分别为56.5%和66.8%)均高于G+C。SHERWOOD等[25]研究发现,红藻门藻类COⅠ基因的A+T含量大于60%,而UPA基因的碱基含量更为均衡,A+T和G+C含量通常各约占50%。相关研究表明,相对于COⅠ基因,UPA基因进化速度更慢,更为保守;COⅠ基因比UPA基因拥有更高的变异度[14]。
本研究中,帚状江蓠的野生群体30个样本中,UPA基因只检测出1个单倍型,COⅠ基因则检测出2个单倍型和3个多态性位点,COⅠ基因片段的各项遗传参数均高于UPA基因片段。结果表明,与COⅠ基因序列相比,UPA基因序列的遗传变异更小,序列更保守。本研究中,UPA片段只检测到一种单倍型,推测与UPA基因的保守性相关。SHERWOOD等[25]的研究表明,由于COⅠ基因是一个编码基因,密码子的第3个碱基位点的差异是大部分变异的来源,COⅠ基因片段显示出最大的置换饱和度,而UPA基因片段则没有显示出置换饱和度,这也验证了UPA基因更加保守。对沙菜属的研究结果表明,COⅠ基因在6个沙菜种间的差异为10.1%~16.3%,而UPA基因只有2.5%~4.4%的种间差异,也证明了UPA基因的保守性[26]。本研究中,帚状江蓠UPA基因片段与伞房江蓠、缢江蓠、扁江蓠、真江蓠和龙须菜的序列差异分别为4.5%、7.1%、5.0%、8.9%和8.6%,而帚状江蓠COⅠ基因片段与伞房江蓠、缢江蓠、扁江蓠、真江蓠和龙须菜的序列差异均大于10.0%。UPA和COⅠ基因均能够区分江蓠属内不同种间的物种,本研究表明,UPA基因不适合帚状江蓠种内的遗传多样性分析,但仍能用于属内种间的物种鉴定;与UPA基因相比较,COⅠ基因适用于帚状江蓠种内遗传多样性分析,在物种鉴定上也更具优势。但本研究中,UPA基因只检测到1种单倍型,COⅠ基因也只检测到2种单倍型,且COⅠ基因的3个变异位点中包含2个转换位点和1个颠换位点,转换颠换比值高,COⅠ基因变异尚未达到饱和,也反映了海南海域的帚状江蓠种质资源相对单一。
3.2 帚状江蓠的系统进化
本研究中,基于UPA基因的江蓠属内的遗传距离为0.025~0.089,基于COⅠ基因的江蓠属内遗传距离为0.136~0.175,帚状江蓠种内遗传距离为0.005,符合相关研究中提出的种间差异大于种内差异的10倍的标准[27]。COⅠ基因的种间差异度高,已经作为DNA条形码成功鉴定了许多红藻物种[28-29]。
对于传统形态学难以鉴定的生物,DNA序列分析提供了一个分类新工具,同时也被广泛应用于系统进化关系的研究[30]。基于COⅠ基因构建的NJ系统进化树表明,帚状江蓠与伞房江蓠、缢江蓠、扁江蓠、真江蓠、龙须菜汇聚为一支,江蓠属、沙菜属、仙菜属、石花菜属和海头红属各自汇成独立的一支,这与形态学分类结果一致[1,31]。基于UPA基因构建的NJ进化树在江蓠属内和蜈蚣藻属的划分和COⅠ基因有些不一致:UPA构建的进化树中,缢江蓠和真江蓠、伞房江蓠和扁江蓠分别汇为一支,而COⅠ构建的进化树中,缢江蓠和真江蓠被分为独立的两支,伞房江蓠和扁江蓠汇为一支;UPA构建的进化树中,蜈蚣藻被单独分为一支,带形蜈蚣藻与角叉菜汇为一支,而COⅠ构建的进化树中,蜈蚣藻属的两个种汇聚到一起,角叉菜单独一支。黄艳等[20]的研究中,同样也出现UPA基因无法区分亲缘关系较近的种的现象。相较于UPA基因,COⅠ更能区分亲缘关系更近的物种。结合形态学分类特征,COⅠ基因比UPA基因更适合构建红藻的系统进化关系;COⅠ基因适合进行属内种间的系统进化分析,UPA基因则适合在更高阶的分类单元应用。
大型海洋藻类种类繁多,形态复杂,采用单一的形态鉴定方法确定物种较为困难。DNA条形码技术多应用于获取未鉴定生物的分类学信息[32],在大型海藻的鉴定和遗传分析中也被广泛应用[33],本研究所选用的UPA和COⅠ基因片段在红藻门藻类的分子鉴定中具有较高的适用性[34]。UPA基因在分析帚状江蓠的遗传多样性时,其保守性可能会导致对南海海区帚状江蓠遗传多样性的低估,但UPA序列易于获取,通用性强,仍可作为红藻门DNA条形码的一个备选。COⅠ基因在本研究和其他学者的研究中,均表现出较强的区分物种的能力,适合作为红藻门藻类的DNA条形码,但也有研究表明,COⅠ基因在某些藻类中存在扩增和测序方面的问题[35]。
用于研究海洋藻类的DNA条形码还有许多,本研究仅选择了UPA和COⅠ基因对帚状江蓠进行评估,未来将选择更多DNA条形码进行海洋藻类种质资源及遗传多样性研究。本研究结果显示,南海海区帚状江蓠种质资源相对单一,未来应着手开展江蓠资源保护工作。
