人肝癌细胞系HepG2和Huh7中的基因组拷贝数变异分析
2023-06-05冀梦蝶卞晓翠杨玉容
冀梦蝶,苑 赞,卞晓翠,杨玉容,郭 鑫,王 琦,陈 阳*
1.中国医学科学院基础医学研究所 北京协和医学院基础学院 生物化学与分子生物学系医学分子生物学国家重点实验室,北京 100005;2.华中农业大学 信息学院 农业生物信息湖北省重点实验室,湖北 武汉 430070
肝癌是中国和世界范围内常见的恶性肿瘤之一。据国际癌研究机构(IARC)统计,2020年全球有近83万人死于肝癌,并且每年确诊肝癌以及因此死亡的人数预计将增长55%以上。中国是肝癌大国,肝癌负担占全球一半以上[1],已严重威胁到中国国民的生命健康。
拷贝数变异(copy number variation, CNV)指的是基因拷贝数目的改变,可通过基因剂量效应直接改变所在基因的表达水平,或通过染色质构象改变引起的位置效应调控远处基因表达,以及通过基因融合或断裂效应阻碍基因的表达[2],因此可导致癌基因的激活和抑癌基因的失活[3]。研究表明,CNV的重复或缺失会影响基因的表达和癌相关的生物学过程[2]。
随着近些年测序技术的快速发展,被鉴定发现的CNV数量越来越多。基于CNV和基因表达谱的关联分析,使研究者可以在分子水平上进一步了解肝癌发生发展相关的分子机制,从而有利于肝癌的分型、诊断、预后及靶向治疗药物的设计等。本文利用Bionano光学基因组图谱(optical genome mapping, OGM)技术解析肝细胞癌基因组的大片段CNV,并结合肝细胞癌转录组和临床表达数据分析肝细胞癌拷贝数变异对基因表达、肝癌患者预后的影响。
1 材料与方法
1.1 材料
1.1.1 细胞系:人肝细胞癌细胞系HepG2、Huh7(中国医学科学院基础医学研究所细胞资源中心)。
1.1.2 试剂及试剂盒:光学基因组图谱技术建库试剂盒(Bionano Prep SP DNA Isolation Kit);DNA定量试剂盒(Qubit® HS dsDNA Assay Kit);DLE-1酶RNA提取试剂(Invitrogen Trizol Reagent);RNA-seq建库试剂盒(VAHTS Universal V6 RNA seq Library Prep Kit for Illumina®)。
1.1.3 公共数据:肝细胞癌临床生存数据来源于GEPIA数据库(http://gepia.cancer-pku.cn/);HEK293T基因表达数据来源于NCBI GEO数据库(GSE203529)。
1.2 实验方法
1.2.1 光学基因组图谱DNA文库构建:将HepG2、Huh7细胞分别培养到2.5×106cell/mL后,裂解和消化细胞,通过纳米结合盘结合、洗涤和洗脱程序提取基因组DNA(genomic DNA, gDNA),用DLE-1酶对gDNA进行序列特异性标记,对标记的DNA染色以显示其主干,Qubit荧光仪对gDNA定量后上机测序。
1.2.2 RNA-seq实验检测:将HepG2、Huh7细胞分别培养到1×106cell/mL,Trizol裂解细胞,poly-T低聚吸附磁珠纯化mRNA后,分解成短片段,再合成双链cDNA,对cDNA进行末端修复、加尾、连接测序接头,最后通过片段选择和PCR富集得到最终的cDNA文库,用于上机测序。
1.3 统计学分析
1.3.1 拷贝数变异的分析:数据比对到hg38参考基因组后上传到线上分析软件(Access 1.7 Standalone),导出拷贝数变异位点。由于算法是按照500bp为一个单位来识别CNV,为了最大程度保留真实的CNV,按照confidence 0.99、size 500 bp过滤数据,confidence越高可信度越高越真实。
1.3.2 拷贝数变异基因功能的分析:DAVID[4](https://david.ncifcrf.gov/tools.jsp)数据库对拷贝数变异基因进行KEGG分析,分析结果使用Sangerbox3.0[5](http://sangerbox.com/home.html)工具绘图。
1.3.3 蛋白质相互作用网络的分析:分别取两种细胞系KEGG分析的前5条富集通路的基因,用String[6](https://string-db.org/)生成蛋白质相互作用关系(protein-protein interaction, PPI)网络,置信度为0.9,导入到cytoscape软件中,利用Network Analyser对网络中的各个节点进行分析,得到各个节点的节点度(degree)值。用节点的面积大小和颜色深浅以degree值表示,节点面积越大,颜色越深,degree值越大,并选择Attribute Circle Layout对所有的蛋白节点进行排布。

1.3.5 临床预后的分析:从蛋白质相互作用网络中选取节点度前15的基因,使用GEPIA[7](http://gepia.cancer-pku.cn/)数据库分析基因表达与肝癌患者预后的关系。
2 结果
2.1 全基因组中肝细胞癌的拷贝数变异
在HepG2细胞系中总计观测到84个功能获得型CNV和1个功能缺失型CNV(图1A,1C)。对主要CNV的染色体分布情况进行统计,包括CNV的染色体覆盖区域、平均拷贝数及拷贝数变异类型(表1)。其中2、6、11、14、16、17、20号染色体出现较多的拷贝数增加;5号染色体出现拷贝数减少,且覆盖范围不到1M。
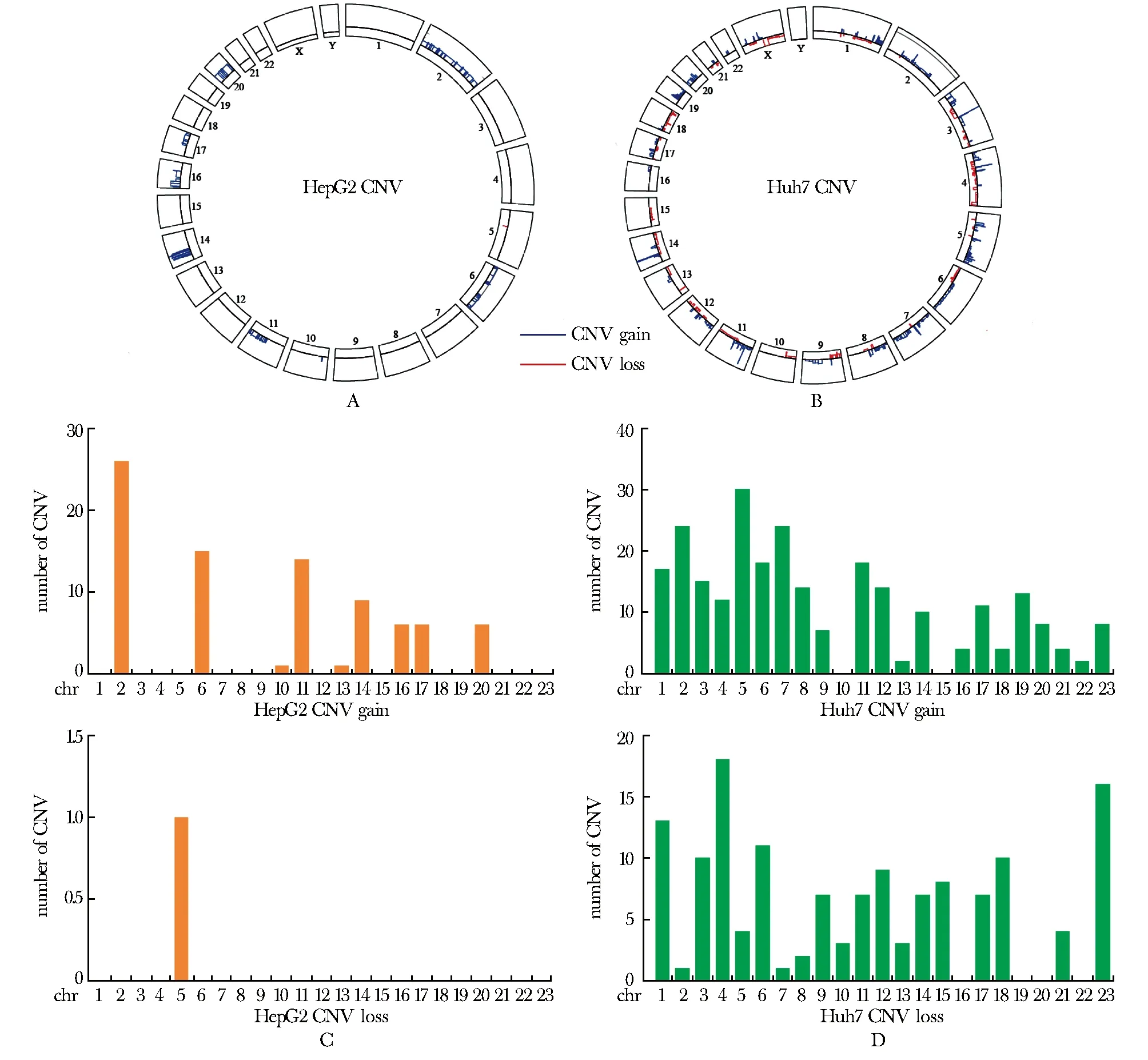
A.circos plot of CNV in HepG2 cells; B.circos plot of CNV in Huh7 cells; C.chromosome distributions of CNV numbers in HepG2 cells; D.chromosome distributions of CNV numbers in Huh7 cells.图1 肝细胞癌全基因组拷贝数变异图谱Fig 1 Genome-wide copy number variation map of hepatocellular carcinoma
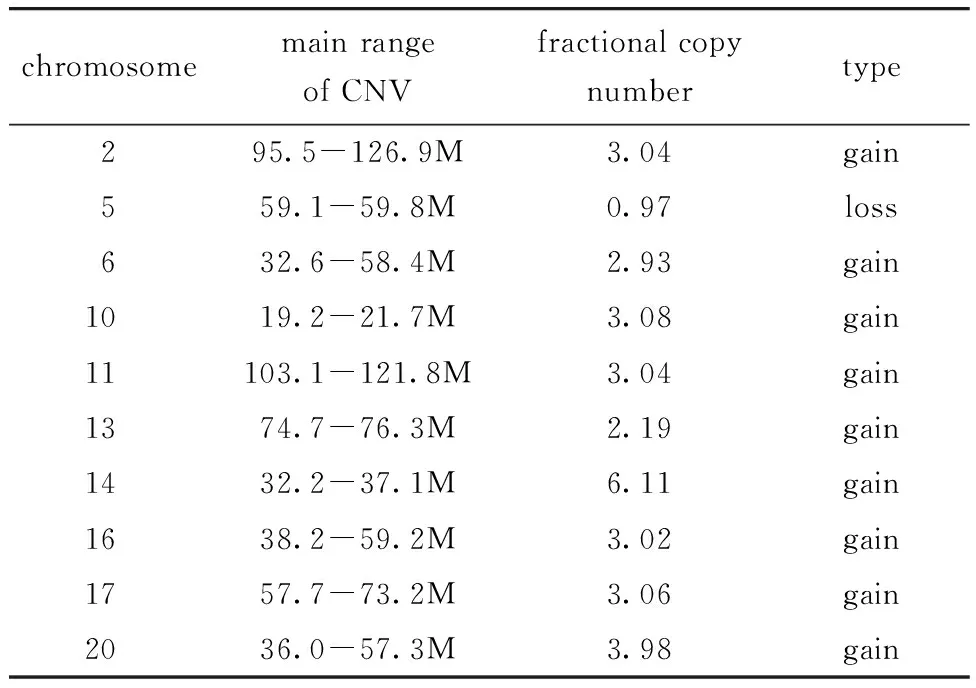
表1 HepG2细胞系主要CNV的染色体分布Table 1 Chromosome distribution of main CNVs in cell line HepG2
在Huh7细胞系染色质中观测到更多拷贝数变异情况(图1B,1D),总计259个功能获得型CNV和141个功能缺失型CNV,对每条染色体主要的CNV分布情况进行统计(表2)。该细胞系拷贝数变异的主要特征是:1、3、4、5、6、9、11、12号染色体既有拷贝数增加,又有拷贝数缺失;2、7、8、17、19、20号染色体主要出现拷贝数增加,10、13、14、15、18、23号染色体主要出现拷贝数缺失。

表2 Huh7细胞系主要CNV的染色体分布Table 2 Chromosome distribution of main CNVs in cell line Huh7
2.2 拷贝数变异基因功能分析
HepG2和Huh7细胞系共有4 711个相同的拷贝数变异基因,2 854个HepG2特异的拷贝数变异基因,15 789个Huh7特异的拷贝数变异基因(图2A)。HepG2细胞系中拷贝数变异基因呈现了5条富集的关键通路(图2B),包括雌激素信号通路、金黄色葡萄球菌感染、Th17细胞分化、抗原处理和呈递、1型糖尿病;Huh7细胞系中拷贝数变异基因呈现了5条富集的关键通路(图2C),包括嗅觉传导、细胞因子-细胞因子受体相互作用、酒精性肝病、RIG-Ⅰ样受体信号通路、丙酮酸代谢。

A.overlap of copy number variation genes in HepG2 and Huh7 cells; B.KEGG analysis of copy number variation genes in HepG2 cells;C.KEGG analysis of copy number variation genes in Huh7 cells.图2 HepG2和Huh7细胞系拷贝数变异基因的功能分析Fig 2 Functional analysis of copy number variation genes in HepG2 and Huh7 cells
2.3 关键基因代谢网络分析
在蛋白质相互作用网络中,节点表示网络中的单元,而边表示单元之间的相互作用。具有高节点度的点往往充当网络不同部分之间的桥梁, 因此可能在网络的整体组成中起重要作用。HepG2细胞中,PPI原始网络主要由92个节点和333条边组成,节点度前25个基因组成了PPI网络的核心模块(图3A),其网络由25个节点和123条边组成,均为拷贝数增加的基因。
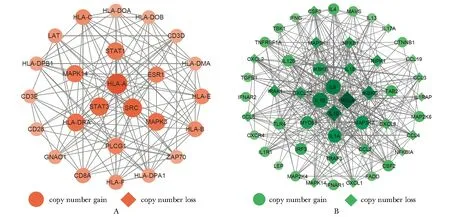
A.protein interaction network in HepG2 cells; B.protein interaction network in Huh7 cells.图3 蛋白质相互作用网络Fig 3 Protein interaction network
Huh7细胞中,PPI原始网络主要由253个节点和981条边组成。节点度前50个基因组成PPI网络的核心模块(图3B),其网络由50个节点和351条边组成,其中23个是拷贝数减少的基因,27个是拷贝数增加的基因。
本文也分别展示了两细胞系PPI网络核心基因的节点度(degree)和中介中心性(betweenness centrality)(表3,表4),显示了网络中每个基因的2种拓扑性质,可以很好地反映每个基因在网络中的重要性。度或中介中心性越高,基因在网络中的作用越重要。
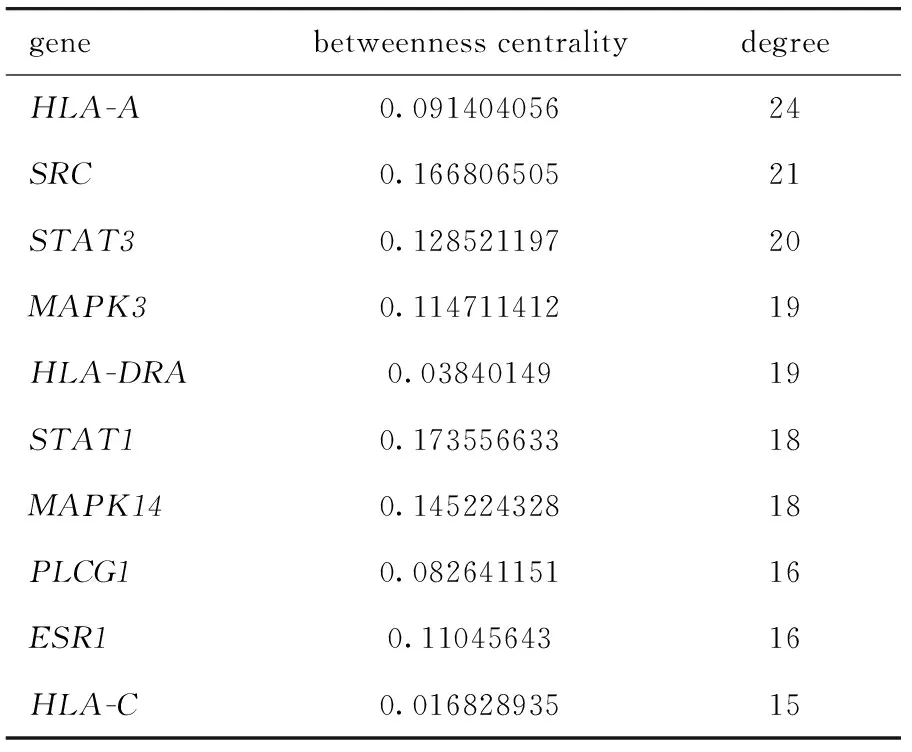
表3 HepG2细胞PPI网络基因核心的节点度Table 3 Degree of the PPI network hub gene in HepG2 cells
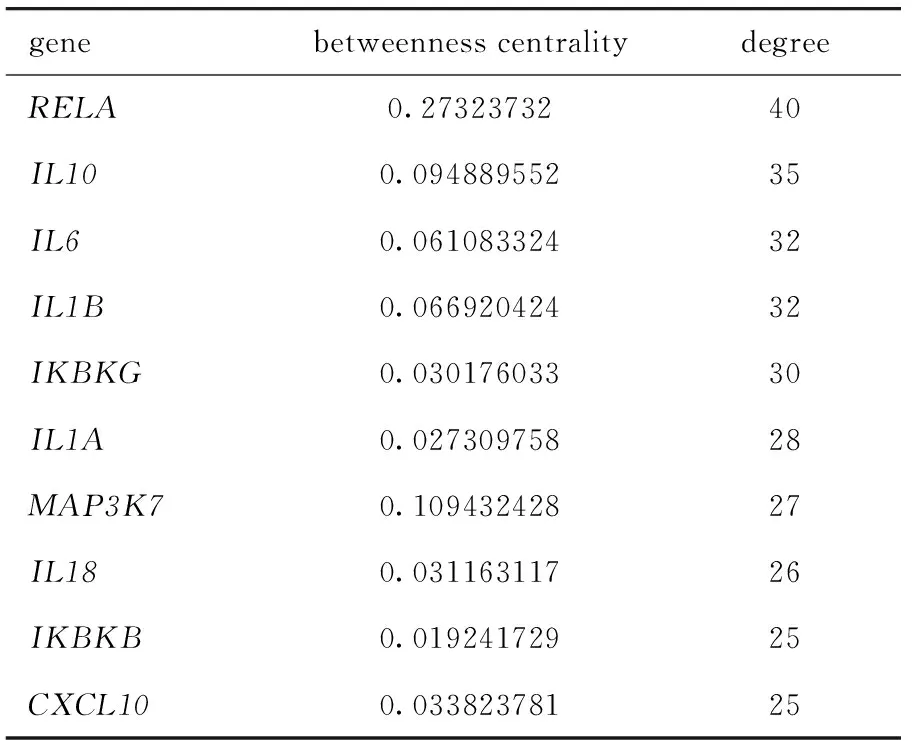
表4 Huh7细胞PPI网络基因核心的节点度Table 4 Degree of the PPI network hub gene in Huh7 cells
2.4 关键拷贝数变异基因的预后分析与验证
两个细胞的核心网络中有3个基因不仅表达量和拷贝数正相关,还与临床预后相关,包括原癌基因SRC、编码MAP激酶家族成员MAPK3、丝氨酸/苏氨酸蛋白激酶MAP3K7。
其中编码酪氨酸蛋白激酶的原癌基因SRC[8],在两个细胞系中拷贝数均增加。而MAPK3、MAP3K7分别仅在HepG2和Huh7拷贝数增加。RNA-seq实验结果显示,SRC(图4A)、MAP3K7(图4C)在HepG2和Huh7细胞系中mRNA水平均显著高于HEK293T,提示了肝细胞癌的特异性变异,MAPK3(图4B)无差异。同时,SRC(图4D)、MAPK3(图4E)、MAP3K7(图4F)这3个基因在肝癌患者的高表达均与患者生存期显著相关。

A-C.expression levels of SRC, MAPK3 and MAP3K7 mRNA in HEK293T(n=3), HepG2(n=4) and Huh7(n=4) (*P<0.001 compared with HEK293T); D-F.prognostic correlation of SRC, MAPK3 and MAP3K7 in hepatocellular carcinoma patients (P<0.05 compared with low expression).图4 关键基因表达和临床生存曲线Fig 4 Hub gene expression and clinical survival curve
3 讨论
编码酪氨酸蛋白激酶的原癌基因SRC[8],在两个细胞系中拷贝数均增加,该基因是目前研究最深入的癌基因之一,可以调控癌细胞的增殖、迁移、侵袭和分化参与肿瘤的恶性发展[9]。在HCC组织中,该基因编码的蛋白质c-Src表达显著升高,促进肝细胞癌的生长和肿瘤发生,并与患者生存成负相关[10],研究结果与GEPIA数据库中结果一致。该基因的拷贝数增加,可能提示了肝癌的一致性变异。
MAPK3、MAP3K7分别仅在HepG2和Huh7拷贝数增加。MAPK/ERK信号通路在信号级联中发挥重要作用,调节响应各种细胞外信号[11],如增殖、分化和细胞周期进程。在已确定的HCC相关信号通路中, MAPK/ERK信号通路是HCC发展中最关键的通路之一[12]。MAP3K7主要通过TGF β及TAK1结合蛋白调节炎性反应、细胞分化和凋亡[13]。它们可能揭示了肝细胞癌的细胞特异性变异。
其中,拷贝数变异基因SRC、MAP3K7的基因表达与患者的预后显著相关,不仅支持这两个基因在肝细胞癌研究中的关键作用,也揭示了拷贝数变异对于肝癌的发展和异质性的重要影响。因此,研究结果为寻找肝细胞癌生物标志物提供了实验和数据支持,对肝癌拷贝数变异的深入探索将有机会促进肝细胞癌发生发展分子机制的研究。
