鼠伤寒沙门菌sRNA SdsR 靶基因的预测及验证分析
2022-06-23张家莉令狐远凤段世宇
张家莉,令狐远凤,段世宇,潘 永,杨 琦,4
(1.贵州大学动物科学学院,贵州 贵阳 550025;2.贵州大学动物疫病研究所,贵州 贵阳 550025;3.贵州省农业科学院畜牧兽医研究所,贵州 贵阳 550025;4.贵州省动物疫病研究室,贵州 贵阳 550025)
0 引言
【研究意义】鼠伤寒沙门菌是一种常见的人畜共患病原菌,对我国的畜禽产业造成了严重的威胁。转录后水平的调控是调节细菌基因表达的重要方式,sRNA 通过分子内碱基配对与靶mRNA 相互作用,通常通过隔离靶mRNA 的核糖体结合位点(RBS)来调节翻译起始[1]。揭示sRNA 与靶基因的调控理论,找到沙门氏菌防控新方式,对保障人与畜牧业的健康发展具有重要意义。【前人研究进展】sRNA SdsR 以两种形式存在,一种是全长 100 nt sRNA,另一种是经过加工的 70 nt 分子[2]。作为一种基因转录后调节因子,SdsR 影响细菌对压力条件的适应、毒力及生物膜形成[3]。SdsR 是固定相 sigma 因子 (σS) 调节子的成员,通过有限且不完美的碱基互补配对与靶mRNA 结合[4],结合区域常为相邻的6~8 个碱基[5]。Fröhlich 等[6]确定并验证了ompDmRNA为SdsR 的直接目标。之后通过SdsR 脉冲表达与全基因组转录组学相结合,发现了 20 个以前未知的 SdsR候选靶标,其中有18 个靶标受SdsR 的依赖性调节[7]。【本研究切入点】sdsR基因几乎存在于所有肠杆菌基因组中,说明sRNA 一定存在额外的、保守的靶基因。TargetRNA2 是建立在TargetRNA[8]、IntaRNA[9]、RNApredator[10]之上的用于识别细菌中被sRNA 调控的mRNA 靶标,其特点是既准确又有效[11]。然而有关鼠伤寒沙门菌sRNA SdsR 靶基因的预测及验证的研究还有待深入进行。【拟解决的关键问题】本研究利用TargetRNA2 软件预测sRNA SdsR 的潜在靶标,结合前期对sRNA SdsR 敲除后的转录组数据,将预测到的部分基因注释到GO、KEGG 以及eggNOG数据库中进行分析,并利用RT-qPCR 对部分预测靶基因进行验证,筛选出可能受sRNA SdsR 直接调控的靶基因,为进一步明确鼠伤寒沙门菌sRNA SdsR的调控机理奠定基础。
1 材料与方法
1.1 试验材料
1.1.1 菌株及质粒 野生型鼠伤寒沙门菌标准株3409由法国国家科学研究中心(CNRS)分子遗传学Bossi实验室惠赠;SdsR 敲除株(基因型为△sdsR::cat)由本实验室构建。
1.1.2 主要试剂及仪器 试剂:RNA 提取试剂盒购自贵州卓一生物科技有限公司,反转录试剂盒[Recombinant NovoScript 1st Strand cDNA Synthesis SuperMix(gDNA Purge)]及荧光染料AceQ Universal SYBR qPCR Master Mix 均购自宏达尔生物科技有限公司,试剂均为分析纯。
仪器:荧光定量PCR 仪(CFX Connect optics Module),美国Bio-RAD 公司;微量核酸测定仪(Nano Photometer NP80),德国Implen 公司。
1.2 TargetRNA2 预测SdsR 靶标
进入TargetRNA2 靶基因预测网站(http://cs.wellesley.edu/~btjaden/TargetRNA2/),输入sRNA SdsR序列,分析对象为沙门氏菌肠道亚种,鼠伤寒沙肠杆菌血清型LT2 染色体。勾选sRNA conservation and accessibilty。目标参数为从上游80 NTs 到mRNA 翻译起始位点下游的20 NTs,sRNA 窗口大小为13,杂交种子数为7,P值阈值为0.05,过滤器尺寸为400。点击search,将预测到的结果导出并选择杂交能量最高的5 个基因进行后续的试验。
1.3 预测基因的生物学信息分析
本实验室前期已经将鼠伤寒沙门菌sRNA SdsR基因敲除后进行转录组测序。转录组测序结果已注释到GO、KEGG 以及eggNOG 数据库中。将预测到的靶标定位在测序结果中,分析其涉及的信号通路、所行使的生物学功能、蛋白质功能及其分类。
1.4 预测基因的RT-qPCR 验证
选择杂交能量较高的5 个预测到的靶基因hemA、STM0951、mreC、STM1252、dcoC,对其进行RT-qPCR验证。根据GenBank 提供的基因序列设计相应的RT-qPCR 引物(表1),送上海生工生物工程有限公司进行合成。以SdsR 敲除株为试验组、3409 标准株为对照组,提取总RNA 样本,反转录成cDNA 后进行RT-qPCR 试验,反应体系为:2×AceQ qPCR SYBR Green Master Mix 10 μL,上下游引物各0.4 μL,50×ROX Reference Dye 1 0.4 μL,cDNA 2 μL,ddH2O 7.8 μL;反应程序为:95 ℃ 0.5 min;95 ℃ 5 s,60 ℃ 0.5 min,39 个循环,每个样本重复3 次。数据采用2-ΔΔCT法进行处理。将得到的结果结合转录组数据进行分析。

表1 引物序列Table 1 Primer sequence
2 结果与分析
2.1 sRNA SdsR 靶标预测结果
Target RNA2 软件预测到29 个靶标(表2),其中hemA、STM0951、mreC、STM1252、dcoC的杂交能量较高。hemA编码谷氨酰-tRNA 还原酶(GlutamyltRNA reductase),STM0951编码细胞质蛋白(Cytoplasmic protein),mreC编码杆状决定蛋白MreC(Rod shape-determining protein MreC),STM1252 编码细胞质蛋白(Cytoplasmic protein),dcoC编码草酰乙酸脱羧酶亚基γ(Oxaloacetate decarboxylase subunit gamma)。
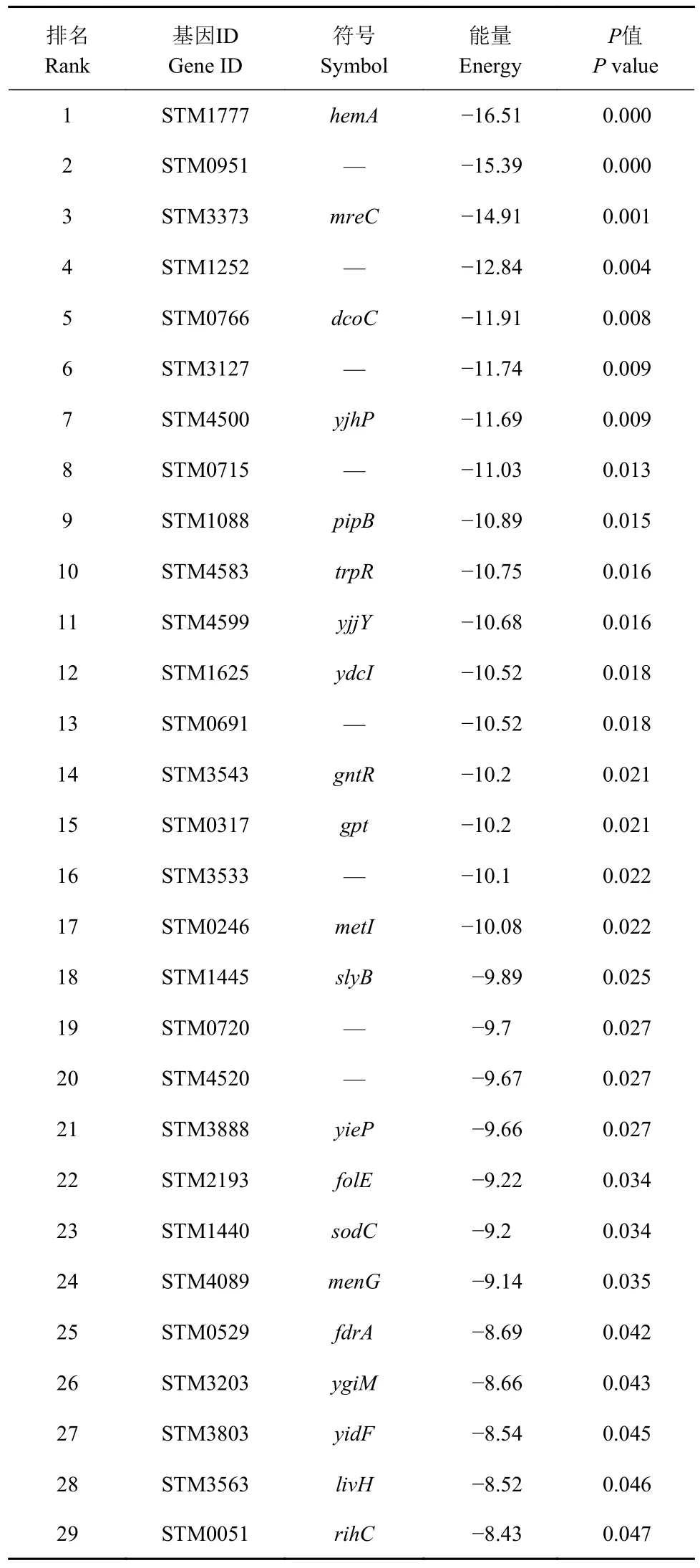
表2 TargetRNA2 软件预测结果Table 2 Prediction by TargetRNA2
杂交能量较高的5 个基因的靶位点的预测结果(表3)显示:STM0951 与dcoC在5′UTR 区域与sRNA SdsR 形成了4 个连续的碱基互补配对,而hemA、mreC、STM1252 则是在CDS 区域与sRNA SdsR 发生大于7 个碱基的连续或不连续的互补配对。该结果说明所选择的5 个基因均能与sRNA SdsR 形成连续的碱基互补配对,且可能存在其他有待发现的作用机制。

表3 靶位点预测结果Table 3 Predicted target sites
2.2 预测靶基因GO、KEGG 和eggNOG 数据库注释
将筛选到的5 个潜在靶基因注释到GO、KEGG和eggNOG 数据库中(表4)。GO 数据库注释结果显示,在生物过程方面,hemA主要参与原卟啉原IX 生物合成过程、氧化还原过程,STM0951 只参与氧化还原过程,mreC影响细胞形状的调节,dcoC参与脂质代谢过程;在细胞成分方面,merC和dcoC同时与膜的组成部分质膜相关;在分子功能方面,hemA和STM0951 同时涉及槲皮素 2,3-双加氧酶活性,而dcoC涉及钠离子跨膜转运蛋白活性、草酰乙酸脱羧酶活性、水解酶活性。STM1252 的生物学功能未被富集到。KEGG 数据库注释结果显示hemA与dcoC分别富集到卟啉和叶绿素代谢途径与丙酮酸代谢途径。eggNOG 数据库注释结果显示,筛选出的5 个基因均可注释到eggNOG 数据库中的直系同源蛋白功能分类。其中hemA、STM0951、mreC、STM1252、dcoC分别注释到辅酶转运和代谢、仅一般功能预测、细胞壁/膜/包膜生物发生、碳水化合物运输和代谢以及能源生产和转化几个分类中。表明上述基因可能参与细菌的新陈代谢以及基本的生命活动。结合转录组数据,sRNA SdsR 敲除后hemA、mreC分别下 调0.23 和0.39 倍;STM0951、STM1 252、dcoC分别上调0.51、0.35 和1.86 倍。由此可见,dcoC基因表达差异倍数最大,但表达倍数小于2 且未达到差异阈值,其差异性需要进一步验证。

表4 预测靶基因GO、KEGG 和eggNOG 数据库注释结果Table 4 Annotation of predicted target genes by GO,KEGG,and eggNOG databases
2.3 预测靶基因的RT-qPCR 验证
对预测到的杂交能量较高的5 个潜在靶基因进行RT-qPCR 验证,结果(图1)显示,hemA、mreC、STM0951、STM1 252、dcoC与野生型沙门菌LT2 相比转录水平分别下调0.70 倍、下调0.59 倍、上调1.34 倍、上调0.47 倍和上调2.46 倍。以上结果表明,hemA、mreC、STM0951、STM1252、dcoC是本研究筛选被sRNA SdsR 调控的靶基因。
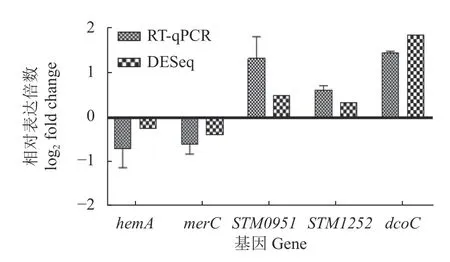
图1 RT-qPCR 验证结果Fig. 1 Results of RT-qPCR verification
3 讨论
sRNA SdsR 是伴侣蛋白Hfq 依赖性sRNA 中的一员,它在肠杆菌的保守性以及经过验证的靶标的生物学作用使得 SdsR 成为sRNA 研究领域中深入研究的主题。Choi 等[12]对sRNA SdsR 进行RNA-seq 来寻找SdsR 驱动的细胞死亡的靶基因。虽然此方法能筛选到大量的潜在靶基因,但由于筛选到的基因总数太大,验证sRNA 直接调控的靶基因有着很大程度上的不确定性,导致直接调控靶基因的确定较困难。TargetRNA2 在sRNA 目标预测系统中具有独特的能力,可以整合RNA-seq 数据以提高预测结果。本研究采用TargetRNA2 预测到29 个靶基因,选择其中的5 个基因进行RT-qPCR 验证,结果发现RT-qPCR与DESeq 的上下调趋势均相符,但差异倍数不完全符合,这可能与验证方法不同及仪器灵敏度差异有关,且RT-qPCR 检测结果整体高于DESeq 检测结果,说明荧光定量PCR 的敏感性高于DESeq 测序方法。以上结果表明这些基因可能受SdsR 直接调控。很明显,这种针对研究对象特性并结合软件预测的研究方法具有较高的筛选效率,同时结合转录组测序的数据能极大提高靶基因的筛选效率。
SdsR 是Hfq 依赖性sRNA,Hfq 相关sRNA 通常通过反式编码调节靶mRNA,导致在翻译、RNA 稳定性或两者水平上抑制或激活靶标[13]。反式编码调控小RNA 对靶mRNA 的调控作用以负调节作用最为常见[14]。在本次研究中,筛选出的5 个靶基因中hemA、merC的表达量下调,说明在沙门菌中,SdsR 对某些靶基因的表达有促进作用,其调控机制值得进一步深入研究。SdsR 与靶mRNA 互补配对的位置通常位于其5′UTR 靠近翻译的起始位点的区域,而预测结果显示SdsR 与hemA、mreC、STM1252 的结合位点为起始密码子的CDS 区,这种结合方式并不常见。Verena Pfeiffer[15]验证了与Hfq 相关的sRNA MicC 通过CDS(密码子23-26)中的r12-bp RNA 双链体沉默鼠伤寒沙门氏菌ompDmRNA,此外,ArcZ-TPX[16]、RybB-fdaL[17]、SgrS-manX[18]等都属于sRNA 与靶基因的CDS 区域相互作用。此机制已被证明是一种比以前认为的更为常见的机制。这些研究支撑了hemA、mreC、STM1 252 作为SdsR 靶标的可能性。
TargetRNA2 预测到的5 个基因中,hemA编码谷氨酰-tRNA 还原酶[19],该酶是催化血红素生物合成途径中的第一个关键步骤。血红素可作为一种主要的铁来源,铁是大多数生物包括细菌生命的必需营养元素[20]。STM0951 基因GO 富集到氧化还原过程,鼠伤寒沙门菌作为需氧及兼性厌氧型细菌,细菌的许多生命活动都会进行着复杂的氧化还原反应,如呼吸作用、固氮作用及生物体内许多代谢活动。merC与膜的组成部分质膜相关,STM1 252 编码细胞质蛋白。dcoC编码草酰乙酸脱羧酶亚基γ,草酰乙酸脱羧酶还有α、β 另外两个亚基,其具备钠离子转运功能;另外,研究还发现草酰乙酸脱羧酶对细菌的致病性及毒力起重要作用,如在军团菌中草酰乙酸脱羧酶突变导致其在宿主体内的增殖能力减弱[21]。沙门菌作为重要的人畜共患病原菌,掌握其sRNA与靶基因的互作方式及了解其靶基因的组织结构、生理功能等有利于人们应对沙门氏菌的威胁,保证人类和动物的健康。
