我国芝麻主要育成品种遗传多样性分析及优异变异位点挖掘
2022-05-27段迎辉苗红梅张海洋
李 春,段迎辉,琚 铭,苗红梅,杜 华,张海洋
(1. 河南省农业科学院 芝麻研究中心,河南 郑州 450002;2. 河南省特色油料基因组学重点实验室,河南 郑州 450002)
芝麻(Sesamum indicumL.)是世界上最古老的优质油料作物之一,也是我国重要的特色优质农产品,在食品加工中具有重要的地位。2016—2020 年世界芝麻年均种植面积1 215 万hm2,年均产量约610 万t[1],主要分布在亚洲、非洲、拉丁美洲等发展中国家,苏丹、印度、缅甸、坦桑尼亚、尼日利亚和中国是芝麻主要生产国。2016—2020 年,我国芝麻年均种植面积为26.26 万hm2,年均产量为41.51 万t,年均单产1 578 kg/hm2,在世界芝麻生产和国际贸易中占有重要地位[1]。截至目前,我国芝麻育种家通过常规选育、复合杂交、理化诱变、轮回杂交、远缘杂交等育种技术共选育出181个芝麻品种[2-3]。但受芝麻栽培种物种进化单一等因素限制,现有芝麻种质遗传基础相对狭窄,育成品种遗传多样性相对降低,进一步选育高产稳产芝麻新品种的难度逐步增大[3-4]。
近年来,为解析芝麻种质资源重要性状的遗传特征,加快芝麻育种研究进程,国内外芝麻学者相继开展了芝麻种质资源的遗传多样性和群体结构分析[4-9]。岳文娣等[4]利用42 对具明显多态性的SSR(Simple sequence repeat)标记,分析了国内外545 份芝麻品种的遗传多样性和群体结构,发现我国芝麻种质资源遗传基础较为狭窄,遗传多样性与地理分布不完全相关,而国外资源遗传多样性丰富。WU等[5]选用88 个SSR 和InDel 标记对130 份中国芝麻种质材料进行分析,通过PCA(Principal coordinate analysis)、进化树和AMOVA(Analysis of molecular variance)分析,将130 份材料分为5 个亚群。WEI等[6]通过简化基因组测序发现,705份芝麻材料大体可分为与纬度相关的2 个亚群,分别对应北方材料和南方材料。DOSSA等[7]选用33对SSR标记分析了来自22 个国家的96 份芝麻种质,结果将材料划分为5 个亚群,并指出亚群分布与材料起源地存在关系。同时,CUI 等[8]采用89 924 个SNP 标记和366 份芝麻种质材料,也获得了类似的结论,发现材料中存在3个亚群,分别为南方材料、北方材料和混合型材料。BASAK 等[9]采用5 292 个SNP 标记分析了地中海地区的芝麻核心种质,发现来自美洲和欧洲地区的材料多样性较高,且材料的亲缘关系与地区分布间无明显的关联。
在芝麻育成品种遗传多样性研究方面,WEI等[2]选用140 对分子标记开展了151 个芝麻品种的指纹图谱构建,发现芝麻品种材料间的相似度为0.51~0.99,指出我国芝麻育成品种相似度较高,遗传多样性相对较低。WEI 等[6]研究了705 份芝麻种质材料的多样性,指出我国芝麻育成品种的分子多样性显著低于地方种,且亲缘关系较为集中,同时也发现与产量、油脂含量、抗病性紧密连锁的31 个SNP位点在地方种和育成品种中的等位基因频率改变程度超过30%。但截至目前,尚未有对我国芝麻育成品种中遗传多样性显著改变的位点、数量或分布等特征进行系统分析的研究报道;同时,基因多样性如何与品种人工改良发生关联也不甚清晰。为此,以95 份我国芝麻育成品种和610 份国内外种质资源(其中国外种质资源205 份,国内地方种405份)为材料,基于基因组测序数据,比较分析了材料间的分子多样性,探明在芝麻育成品种中被富集的部分位点,为构建芝麻分子育种技术体系提供重要理论和技术参考信息。
1 材料和方法
1.1 数据来源
705 份芝麻种质资源材料基因组PE 双末端(Pair-end)测序数据(NCBI Bioproject PRJEB8078)共617.86 Gb(Giga bases),单个样品数据量为199~5 401 Mb(Million bases),平均每个样品数据量为876.39 Mb[6]。数据通过NCBI 下载。705 份芝麻种质资源材料包括95 份我国育成芝麻品种(Modern cultivar,简写为MC)、405 份国内地方种(Land race,简写为LR)以及205份国外种质[6]。
1.2 基因组变异分析
以芝麻豫芝11 号(Yuzhi 11)为参考基因组[10],对上述705份基因组测序数据进行比对分析。采用软件Trimmomatic 0.33[11]以默认参数进行碱基质量控制;通过BWA(Burrows wheeler aligner)0.7.17 程序[12]比对至参考基因组(默认参数),获得SAM(Sequence alignment/Map)文件,然后以SAM 文件为基础,通过SAMtools[13]筛选获得高质量比对序列。通过GATK 4.0 程序,依据Short variants calling best practice[14],确 定 各 样 本 的SNP(Single nucleotide polymorphism)及InDel(Insert/deletion)变异位点。以最低材料数量(Min count)和稀有等位基因频率(Minor allele frequency,MAF)为主要参数,使用TASSEL 5.2.43程序[15]过滤并筛选上述变异位点。
使用河南省农业科学院芝麻遗传育种研究团队获得的560份芝麻核心种质材料的基因组重测序数据(简称为GWAS560,未公开)和搭建的SNP/InDel变体数据库,进一步对在多个材料中普遍存在的变异位点进行筛选,以获得群体间材料多样性比较的稳定结果。
依据ZHANG 等[10]构建的芝麻超高密度SNP 遗传图谱(13条染色体),采用滑动窗口法分析变异位点在芝麻13 条染色体上的分布,窗口大小设置为10 kb(Kilo bases),步移设置为2 kb。
1.3 材料多样性分析
依据GATK 4.0 程序产生的VCF(Variant call format)文件,使用VCFtools 0.0.17 程序[16]计算芝麻育成品种和地方种的核苷酸多样性指数π[17]及群体间的遗传分化系数Fst[18]。
通过模拟置换检验(Permutation test)进行核苷酸多样性指数π和遗传分化系数Fst的显著性检验。在核苷酸多样性指数π的检验中,从500 份(95 份育成品种和405 份国内地方种)材料中随机抽取95 份样本,组成模拟群体。在遗传分化系数Fst的检验中,将500 份材料随机分为2 个群体,群体大小分别为95 和405,组成2 个模拟群体。以自主编写的PERL 脚本进行1 000 次模拟假设检验,显著性阈值设定为P=0.001。
2 结果与分析
2.1 样本变异位点的鉴定与筛选
为确定705 份芝麻样本的变异位点,以豫芝11号基因组为参考,分析了705 份芝麻种质材料的基因组PE 数据,确定了7 799 977 个原始变异位点(6 757 883个SNP和1 042 094个InDel),建立了705份样本的变异位点矩阵。在该矩阵中,69.13%的变异位置覆盖度≤2 层,41.55%的变异位置覆盖度为0,表明上述材料的测序数据量较少,对变异位点的可靠度分析有一定影响。
为进一步筛选在多个材料中普遍存在的变异位点,剔除稀有等位基因及群体特异性变异,以用于群体间材料多样性的比较,选用了560 份芝麻种质的全基因组重测序数据和变异位点数据库(GWAS560 数据库),分别以“Min count=564(总材料的80%)、493(70%)或420(60%)”和“MAF=0.05”为参数,对上述原始变异位点进行过滤。结果显示,以“Min count=564”结合“MAF=0.05”参数,获得了12 704 个位点,其中12 654 个(99.61%)位点可同时在GWAS560 数据库中被检测到。因而,确认上述12 704 个位点为多个材料中普遍存在的变异位点。
在上述12 704 个变异位点中,共有8 384 个变异位点分布于13 条染色体上,每条染色体为116(Chr8)~1 445 个标记(Chr3)(图1)。滑动窗口法(Window size=10 kb,Step=2 kb)分析结果显示,在8 384 个位点中,每窗口内变异位点数量为1~203个,平均为9.55 个,部分变异位点存在聚集现象(图1)。
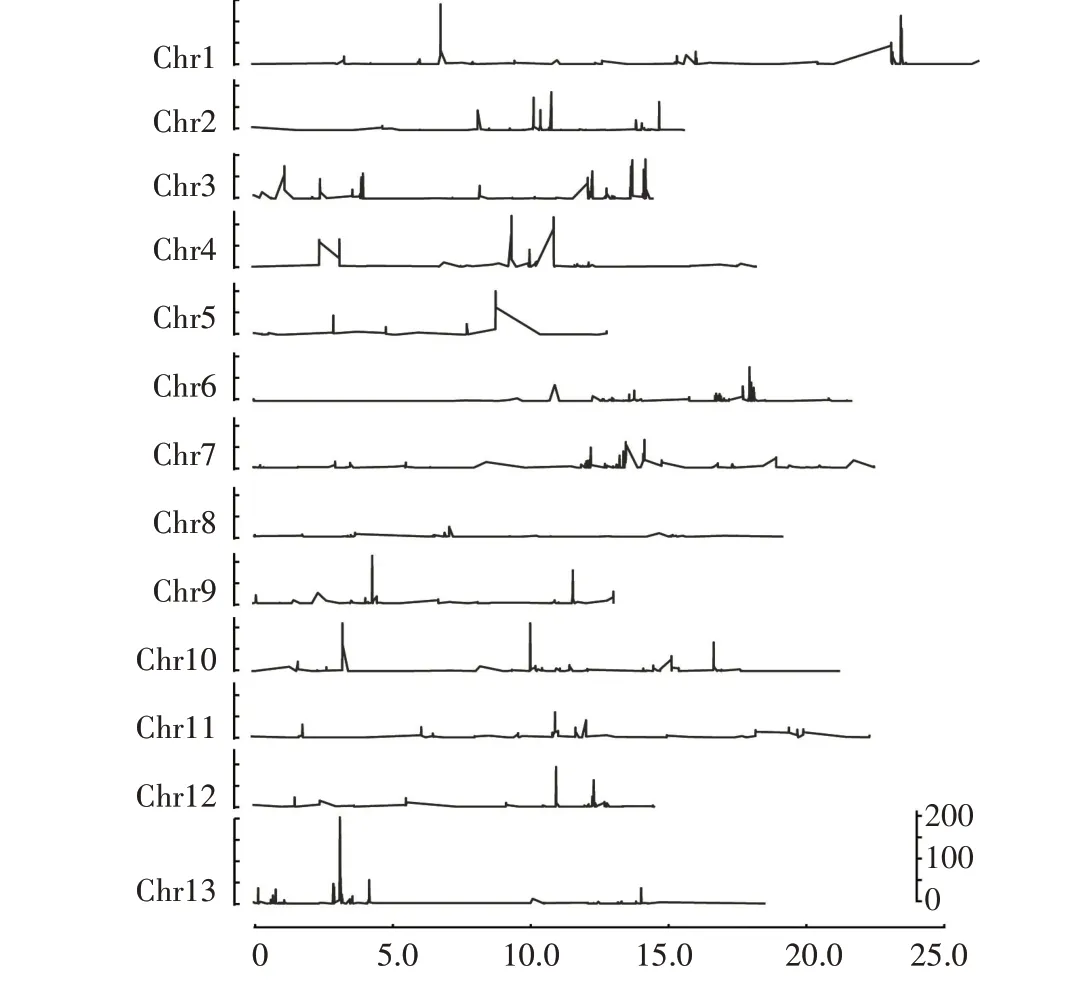
图1 8 384个变异位点在13条染色体上的分布密度曲线Fig.1 The density plot of 8 384 SNP/InDel variants on the 13 chromosomes of sesame
2.2 芝麻育成品种的多样性分析
为分析材料间的进化关系,依据上述12 704 个变异位点,对材料进行系统进化分析。结果(图2)显示,705 份材料可被划分为4 个进化亚组,分别为Ⅰ(包含79 份材料)、Ⅱ(52 份)、Ⅲ(182 份)和Ⅳ(392 份)组。其中,国外种质材料主要分布在亚组Ⅳ,共包括了160 份国外资源,占国外材料的78.05%;育成品种主要集中在亚组Ⅰ(51 份),在其余3 个亚组中,育成品种呈零散分布(Ⅱ组6 个,Ⅲ组14个,Ⅳ组24个)。

图2 芝麻育成品种、地方种和国外材料的系统进化树Fig.2 The Phylogenetic tree of the sesame modern cultivars,landraces and germplasm lines collected from the other countries
随后,以核苷酸多样性指数π为参数,比较了95 份芝麻育成品种和405 份国内地方种的遗传多样性(图3)。结果显示,在育成品种和地方种中具有多样性的变异位点共计12 670 个;育成品种材料的π值为0~0.653,平均为0.158,地方种的π值为0.006~0.622,平均0.246,地方种显著高于育成品种(One tailT-test,P<0.01)。同时,在芝麻的13条染色体中,育成品种的多样性也明显低于地方种。

图3 芝麻育成品种和地方种间核苷酸多样性小提琴图比较Fig.3 The violin plot comparison of nucleotide diversity between the sesame cultivars and landraces across the 13 chromosomes of sesame
此外,使用Permutation 置换检验对上述变异位点的多样性进行分析。结果发现,在育成品种中,有2 483 个位点(19.60%)表现出多样性显著降低(P<0.001),有115 个位点表现为多样性显著升高(P<0.001)。
2.3 芝麻育成品种与地方种间的遗传分化分析
为明确我国芝麻育成品种与地方种之间在分子多样性方面的差异,选用遗传分化系数Fst为参数,比较了2 组样本的12 704 个变异位点的分化特征(图4)。通过1 000 次模拟假设检验,共发现1 290 个位点在芝麻育成品种和地方种间表现为显著分化(P<0.001)。其中,1 125 个位点与核苷酸多样性指数π显示育成品种多样性显著降低的位点相同,80个位点与育成品种多样性显著升高的位点相同(图4)。

图4 育成品种材料(MC)中多样性显著升高、降低位点,以及育成品种-地方种(LR)间分化位点Venn图Fig.4 The venn diagram across the variants with significantly lower or higher nucleotide diversity in modern cultivars(MC),and the ones significantly divergent between cultivars and landraces(LR)
进一步分析显示,在芝麻育成品种和地方种间显著分化的1 290 个位点中,1 081 个(83.80%)变体位点呈现聚集性,相邻位点间的距离小于2 kb。如,显著分化的位点Chr1:3380935、3381182 和3381193,集中于500 bp 的区间内。使用滑动窗口法计算Fst,也获得了类似的结果。
2.4 芝麻育成品种的等位基因富集分析
为探索芝麻育成品种中富集的等位基因,进一步对上述在育成品种中多样性显著升高且在育成品种和地方种间显著分化的80 个位点进行分析。结果(表1)显示,80 个变异位点共分布于9 个染色体和2 个重叠群(Contig)上;其中,共有79 个变异位点具有2 个等位变异,仅SNP 位点Chr2:8177102 存在3 个等位变异。此外,有3 个变异位点(Chr1∶26400643、Chr13∶3548465 和Chr13∶3608441)只在育成品种中存在多样性(表1)。
在80 个变异位点中,17 个(21.25%)分布于13个基因的外显子中,11 个(13.75%)位于基因间区(Intergenic),52 个(65.00%)位 于 基 因 上 下 游(Upstream/Downstream)。位于基因外显子区的17个变异引起了基因突变,其中同义突变(Synonymous)3 个(Chr2∶8177098、Chr10∶3442162和Chr10:3442942),错义突变(Missense)12 个,移码框突变(Frame shift)1个(Chr1:27360152),以及终止密 码 子 丢 失 突 变(Stop codon lost)1 个(C22∶4704132)。上述变异位点中,错义突变、移码框突变和终止密码子丢失突变(共14 个)具有强突变效应,可能影响了U0183.30(Cytochrome P450)、C_2_2.176(Disease resistance NB-LRR protein)、C37.81(Disease resistance NB-LRR protein)和aC22.188(Retrotransposon gag protein)的基因功能(表1)。在影响上述2 个抗病相关基因(C_2_2.176和C37.81)的强突变效应位点中,位点Chr2:8176782 和Chr10∶3441593 在育成品种中的基因频率较地方种分别提高了12.93倍和3.44倍(表1)。

表1 通过核苷酸多样性和遗传分化分析选定的80个位点统计信息及变异效应预测Tab.1 The statistical information and variant effect annotation of the 80 loci selected from the intersection of the nucleotide diversity and population divergent analysis

续表1 通过核苷酸多样性和遗传分化分析选定的80个位点统计信息及变异效应预测Tab.1(Continued) The statistical information and variant effect annotation of the 80 loci selected from the intersection of the nucleotide diversity and population divergent analysis

续表1 通过核苷酸多样性和遗传分化分析选定的80个位点统计信息及变异效应预测Tab.1(Continued) The statistical information and variant effect annotation of the 80 loci selected from the intersection of the nucleotide diversity and population divergent analysis
3 结论与讨论
本研究通过705份芝麻种质材料的变异位点发掘与分布分析,系统比较了95 份育成芝麻品种和405 份国内地方种的变异位点分布特征和分化差异,揭示了80 个变异位点受人工选择后的分化差异,为后续芝麻分子育种技术体系构建提供了理论和技术支持。人工选择是改良农作物品种性状、改变遗传多样性的重要手段。人工选择可导致一些重要农艺性状相关的位点发生多样性改变,并促进个别等位基因被保留下来[19-20]。因此,群体间多样性的差异分析结果可反映一些与重要农艺性状相关的变异位点。CAVANAGH 等[21]以2 994份小麦地方种和育成品种为材料,通过比较各亚群间的Fst系数,鉴定出了32个与育成品种改良相关的基因组区域,其中就包含了在“绿色革命”中发挥了重大作用的矮秆基因Rht-B1。QIN 等[22]通过类似的方法在辣椒基因组中鉴定出包含乙烯响应元件结合因子、10 个抗病基因等在内的34 个重要位点。在芝麻种质GWAS(Genome-wide association study,全基因组关联分析)分析中,WEI 等[6]确定了调控芝麻每叶腋花数(一叶三花或一叶一花)性状的等位基因,该等位基因位点的频率从59.5%(种质资源)富集提升到98.9%(育成品种)。
研究结果表明,农作物基因组中一般有2%~7%的基因位点在育种过程中被人工选择过。如,玉米中被人工选择的基因位点占全部基因的2%~4%[23],大豆被人工选择位点占比约7%[24]。本研究依据多样性在育成品种中显著升高且在育成品种-地方种间显著分化的原则,获得了80 个位点,并认定这些位点上的某个等位变异在芝麻育成品种中被富集。在80个位点中,发现3个位点只在育成品种中芝23号(Zhongzhi 23,ID 为G564)、中芝13 号(Zhongzhi 13,G610)和皖芝3 号(Wanzhi 3,G689)等材料中发现了多样性,在地方种无多样性,推测这些品种中可能引入了材料342(5)(ID 为G456,日本)、U.C.R/82NO201NS(G478,美国)、Padathar(G513,缅甸)或/和K2(G593,几内亚)等材料的血统。
在芝麻遗传育种研究中,选育高产、抗病(抗枯萎病、茎点枯病)、抗逆(耐渍、耐旱、抗倒伏)、适于机收等优异新品种是当前国内外芝麻育种家们的共同目标[3,25]。本研究中,在80 个位点涉及的13 个基因内,发现了2 个抗病相关的基因C_2_2.176和C37.81,这2 个 基 因 均 编 码NB-LRR(Nucleotide binding site leucine rich repeat)蛋白,且位点的变异均引起了氨基酸的错义突变。同时,在2个基因内,育成品种中等位变异的基因频率较地方种分别最高提高了12.93倍和3.44倍。该结果说明,上述2个基因在育成品种中被富集,并可能在提升品种的抗病性方面发挥了重要作用。NB-LRR类基因是植物R 基因(Resistance gene)中较大的一类基因家族,且可分为2 类:TIR-NB-LRR(Toll/inte rleukin receptorNB-LRR)和CC-NB-LRR(Coiled-coilNB-LRR)[26]。C_2_2.176和C37.81均属于CC-NB-LRR(CNL)类的抗病基因,与拟南芥RPP8(抗黑胫病)、RPS4(抗斑点病)以及小麦Lr10(抗叶锈病)等基因同源[27-28]。NB-LRR相关R 基因在芝麻中的研究还较少。此外,本研究也发现了基因U0183.30发生了移码突变,其等位变异在育成品种中较地方种提升了2.70 倍。该基因编码细胞色素P450(Cytochrome P450)相关基因,也与植物的抗病性相关[29]。
本研究以核苷酸多样π为参数,比较芝麻育成品种和地方种间的差异。对于一个含有2个等位基因的位点,π的计算公式可简化为π=n*2pq/(n-1),其中p和q分别为2 个等位基因的频率,且q=1-p;n为样本量[17]。依据该公式,π在p与q相等时取最大值。文中选择的π显著升高的位点,实际上是在地方种中p和q差异较大而在育成品种群体中趋于缩小的位点。但是,该策略忽略了另一类在育种过程中发挥重要作用的位点,如经历了Selective sweep(选择性清除)的位点[30]。这类位点在种质资源中,各个等位基因的基因频率差异不大,表现为p和q差异较小;但在育成品种群体中某个等位基因被富集,获得了极高的等位基因频率(下文称之为B 类位点)。
本研究共发现了2 483个B类位点,占总位点数量的近20%,远高于其他作物中估计的2%~7%,暗示着其中应包含较多数量的假阳性位点,其原因可能是B 类位点的检测受测序深度制约较大[31]。本研究所用基因组数据量少,基因组覆盖度较低,难以有效进一步降低假阳性位点的数量。WEI 等[6]针对与产量、油脂含量、抗病性紧密连锁的31 个SNP 位点开展多样性分析,发现这些位点在芝麻地方种和育成品种中的等位基因频率改变程度>30%。该结果暗示了在育种过程中发挥重要作用的优异等位变异,其等位基因频率在育成品种和地方种间可能发生了明显的改变。而在本研究获得的育成品种中多样性显著升高且在育成品种-地方种间显著分化的80 个位点中,大多位点(78 个)的等位基因频率在育成品种和地方种中发生了显著变化,差异倍数均超过了2.0,这些位点受假阳性的影响应较有限。为揭示芝麻品种变异位点选择与进化特征,后续研究将选用测序深度更高的种质数据库GWAS560,进一步分析并阐明芝麻育成品种中的重要变异位点和选择区域,为搭建完善的芝麻分子育种技术体系奠定坚实的基础。
