重组酶RAD51和DMC1功能保守和分化研究进展
2022-05-24郭雨萱严顺平王应祥
郭雨萱,严顺平,王应祥
综 述
重组酶RAD51和DMC1功能保守和分化研究进展
郭雨萱1,严顺平2,王应祥1
1. 复旦大学生命科学学院,植物科学研究所,上海 200438 2. 华中农业大学生命科学技术学院,武汉 430070
减数分裂(meiosis)是有性生殖细胞中发生的特殊分裂方式,在这个过程中DNA复制一次,细胞核分裂两次,最终产生单倍体的配子。雌雄配子融合后基因组又恢复到二倍体水平,不仅保证了有性生殖过程中世代间基因组的稳定性,还导致后代的遗传多样性。减数分裂同源重组(homologous recombination, HR)是其前期I的核心事件之一,它不仅保证了后续同源染色体的正确分离,而且允许同源染色体之间遗传信息发生交换,增加了后代的遗传多样性。RAD51 (RADiation sensitive 51)和DMC1 (disruption Meiotic cDNA 1)是HR过程中必需的重组酶,二者有一定的共性和特性。本文从起源、进化、结构和功能等方面总结并比较了它们间的保守和分化,并对未来的研究方向提出了展望,为进一步深入研究减数分裂的重组机制提供了借鉴。
减数分裂;同源重组;RAD51;DMC1
减数分裂包括减数分裂I和减数分裂II两个时期,分别涉及同源染色体和姐妹染色单体的分离。减数分裂与有丝分裂不同之处在于减数第一次分裂存在特别的前期I,又分为细线期、偶线期、粗线期、双线期和终变期。在减数分裂I过程中同源染色体之间发生了多个重要事件,比如配对、联会、重组和分离[1]。减数分裂重组是前期I核心事件之一,为生物变异提供了重要的物质基础。研究减数分裂重组的机制对作物改良和育种有十分重要的意义[2,3]。
已有的研究表明,减数分裂重组过程在物种间相对保守(图1),其起始于DNA 双链断裂(double strand break, DSB)的形成,DSB由高度保守的拓扑异构酶SPO11 (sporulation 11)蛋白和其他关键蛋白共同作用产生。DSB形成后,末端被MRN复合体进一步加工成单链DNA (single-stranded DNA, ssDNA),不同物种中参与该过程的蛋白不完全相同。在酿酒酵母()中主要由RED50 (radiation sensitive 50)、MER2 (meiotic recombination 2)、MEI4 (meiosis defective4)、MRE11 (meiotic recombination 11)、REC102 (recombination-deficient 102)、REC104 (recombination-deficient 104)、REC114 (recombination-deficient 114)、SKI8 (superkiller 8)和XRS2 (X-ray sensitive 2)这9个蛋白形成的复合物参与[4,5]。而在拟南芥()中,主要由MRE11、RAD50(RAD50 double strand break repair protein)和NBS1(nijmegen breakage syndrome 1)蛋白参与[6,7]。复制蛋白A复合体(replication protein A, RPA)与ssDNA 3′端结合,保护末端以避免形成DNA的二级结构,促进重组酶的装载。随后,RecA/RAD51及DMC1取代RPA与ssDNA结合形成核蛋白丝,核蛋白丝在同源双螺旋链上搜索匹配序列并进行链入侵形成D环结构(D-loop)[8]。由于RPA与ssDNA具有更强的结合能力,会抑制重组酶的装载,因此需要BRCA2 (breast cancer 2)等其他RAD51同源物介导重组酶结合到ssDNA上[9]。在体外实验中,BRCA2可以自主执行这一功能,但在细胞中,该过程则需要它的“伴侣”——PALB2 (partner and localizer of BRCA2)[10]。最后,DNA聚合酶替代RAD51/DMC1启动DNA的合成[11]。减数分裂DSB修复途径有3种:一种是在ZMMs (ZIP-MSH-MER)蛋白作用下进行DNA合成、DSB的第二端捕获和连接,形成dHJ (double holliday junction)中间体,并最终产生干涉敏感型(I型)交叉[12];第二种是在MUS81 (methyl methane sulfonate and ultraviolet sensitive 81)和FANCD2 (FA complementation group D2)蛋白质作用下产生干涉不敏感型(II型)交叉,现阶段在裂殖酵母()中发现该过程中存在sHJ(single holliday junction)中间体,但在动植物中该途径的中间体还不清楚[13,14];第三种途径是由解旋酶处理D-loop,通过合成依赖链退火途径(synthesis-dependent strand annealing, SDSA)产生非交叉重组(图1)。
基因家族不仅在减数分裂同源重组过程中发挥重要作用,在DNA修复和基因组稳定方面也至关重要[15,16]。对动植物的进化分析表明,除了RAD51,还有6个RAD51同源物,分别是RAD51B、RAD51C、RAD51D、XRCC2、XRCC3和DMC1。其中,DMC1只在减数分裂过程中发挥作用,而其他RAD51蛋白在有丝分裂和减数分裂DSB修复过程中都发挥作用[8],并且倾向于以异源复合体的形式存在于细胞中,如异源四聚体BCDX2 (RAD51B/ C/D/XRCC2)、异源三聚体BDX2 (RAD51B/D/XRCC2)、CDX3 (RAD51C/D/XRCC3)和异源二聚体CX3 (RAD51C/XRCC3)等。不同的蛋白复合物参与不同的DSB修复途径。体细胞中DSB修复的两种主要途径是SDSA和单链退火(single-strand annealing, SSA)。在水稻()中,CDX3 (RAD51C/D/ XRCC3)复合物在SDSA途径中发挥作用,而BCDX2复合物在SSA途径中发挥作用[17]。在减数分裂中,拟南芥CX3复合体通过介导RAD51在染色体上的定位保证其在减数分裂重组中的正常作用,RAD51B和XRCC2可能参与了减数分裂重组的抑制[18,19]。拟南芥DMC1在着丝粒配对中起重要作用且在同源染色体间的同源重组过程中不可或缺,而RAD51、RAD51C和XRCC3在同源染色体臂配对中起重要作用,RAD51则更多地参与姐妹间的重组[20,21]。在减数分裂中同源染色体的搜索和链交换被认为是由减数分裂特异DMC1完成的,RAD51只起辅助作用,然而在减数分裂过程中缺乏RAD51会产生染色体碎片[22,23]。因此,认识减数分裂重组中RAD51和DMC1之间的保守性和分化性对于探索二者在减数分裂重组中的具体分工和减数分裂重组修复机制具有重要意义。
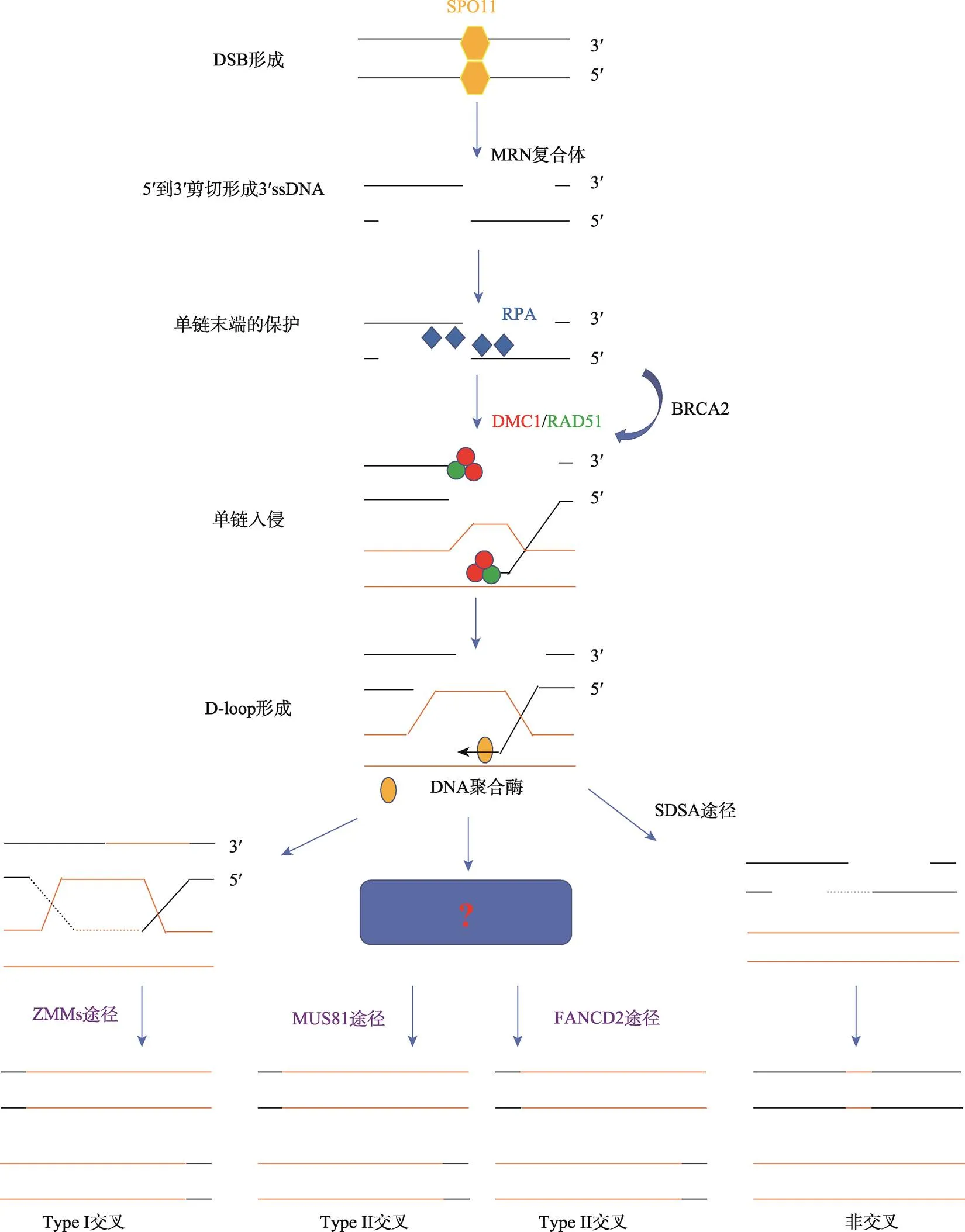
图1 减数分裂重组模型
黑色和橙色线条分别描绘了两条父母亲本双链DNA。减数分裂重组起始于SPO11及相关蛋白诱导的DSB (double strand break)形成,接着MRN复合体在DSB末端进行5'→3'剪切,形成3'ssDNA。RPA结合ssDNA保护其不被降解。RAD51和DMC1在BRCA2等蛋白作用下加载到ssDNA上,形成缠绕在ssDNA周围的DMC1/RAD51螺旋状细丝,侵入同源双链DNA形成D-loop。D-loop可以形成dHJ,通过ZMMs途径形成I型交叉;也可以通过MUS81和FANCD2两种途径形成II型交叉,绝大多数通过SDSA途径形成非交叉。
1 起源与进化
基因家族起源于细胞生物祖先,由它分化为真细菌和古生菌与真核生物祖先两支(图2)[24]。在古生菌和真核生物分化之前由于基因复制产生了两个谱系——和,但是在真细菌中仍然是一个单拷贝基因(图2)。古生菌中有和两个基因拷贝,且古生菌比细菌与RAD51和DMC1的序列同源性更高[25]。在真核生物中,和基因都经历了多次复制事件,最终产生基因家族。在酵母中存在4个基因拷贝:、和;而在植物和动物中有7个拷贝,分别是、、、、、和。Lin等[24]将RAD51/RecA家族分为RADα、RADβ和RecA三个亚群,RADα亚群包括功能高度保守的初级重组酶RAD51和DMC1;RADβ亚群包括功能相对分化的RAD51B、RAD51C、RAD51D、XRCC2和XRCC3;而RecA亚群在真细菌和真核生物细胞器中发挥功能[24]。与RADα亚群相比,RADβ亚群基因快速进化,序列同源性较低,形成高度分化且呈现出丰富的基因多样性。RAD51、DMC1、RAD51C、XRCC3、RAD51B和RAD51D在进化树上非常接近,而XRCC2的进化距离很远,其表达谱也与其他基因不同,说明XRCC2可能与其他RAD51重组酶的分化较远[24]。
和在古生菌和真核生物分化之前由基因复制产生[26]。该基因复制事件使减数分裂同源重组得以实现,所以基因复制可能与减数分裂和有性生殖同时出现,或者允许了减数分裂和有性生殖的发生。不同生物减数分裂重组存在差异,这可能与这些生物体中的快速进化和的缺失有关[24]。研究表明,只有而没有的生物体可能基因组中存在,但在进化过程中逐渐丢失[27]。同时在基因缺失的生物中,RAD51的某些氨基酸被DMC1相对应的氨基酸所替代。
和在人体内处于严格的负选择状态,即突变的等位基因是有害的,在自然选择中处于劣势。而其他RAD51蛋白在人体内处于宽松的负选择状态,它们的表达谱与其所处环境的进化压力相关[16]。基因复制和垂直基因转移(vertical gene transfer)在驱动RAD51蛋白进化过程中发挥了重要作用。
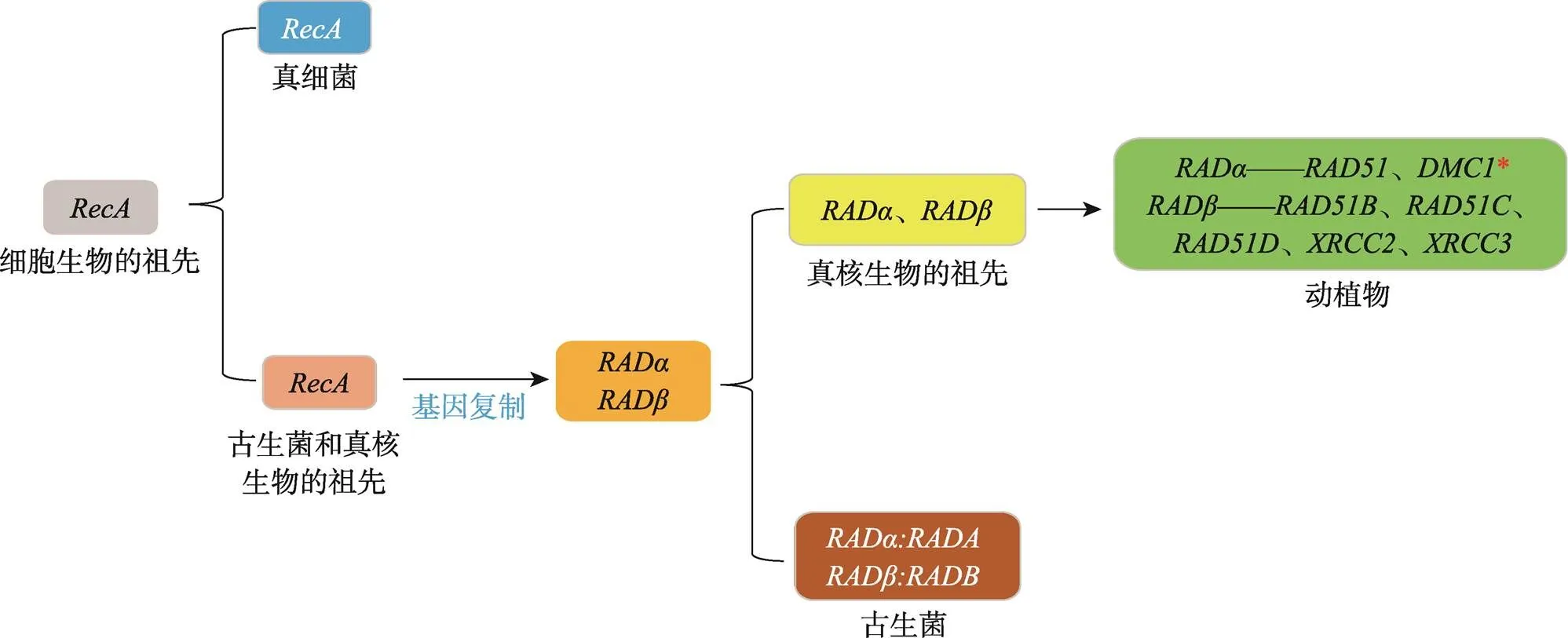
图2 RAD51/RecA家族进化模型
RAD51/RecA家族由细胞生物的祖先分化而来。古生菌和真核生物的祖先通过基因复制产生了两个谱系和。在真核生物中,和基因经历多次复制事件产生RAD51基因家族,在古生菌中它们仍然是单拷贝基因。大括号表示分化的方向,箭头表示基因复制事件。红色星号表示减数分裂特异基因。根据参考文献[24]修改绘制。
2 RAD51和DMC1序列和结构的保守与分化
2.1 序列上的保守性与差异性
RAD51和DMC1与其他水解ATP的蛋白相似,含有α/β ATP酶核心结构域,位于蛋白的N端,由Walker A (也称为NTP结合位点,XXXXGKT/S)和Walker B (也称为Mg2+螯合位点,R/K-XX-G-XX- LHHHD)组成(图3,A和B)。相比之下,C端氨基酸残基的保守性较差。研究表明,人RAD51 (HsRAD51) C端与HsRAD52的结合可以促进ssDNA与dsDNA的同源配对,且与HsRAD51或HsRAD52单独存在时相比,HsRAD51和HsRAD52同时存在时能够观察到更高的同源配对活性[28]。最近在裂殖酵母中发现,Rad51第205位谷氨酸(E205)、206位谷氨酸(E206)和209位天冬氨酸(D209)共同形成突出的酸性区域(protruding acidic patch, PAP)是促进与辅助因子相互作用的基本基序,因为将206位谷氨酸(E206)突变后,裂殖酵母Rad51与Rad55-Rad57不能相互作用[29]。PAP可能在酿酒酵母和人中发挥相似的作用,裂殖酵母第205位谷氨酸(E205)、206位谷氨酸(E206)和209位天冬氨酸(D209)对应于酿酒酵母Rad51的239位天冬氨酸(D239)、241位天冬氨酸(D241)和242位天冬氨酸(D242),及人RAD51的184位天冬氨酸(D184)和187位天冬氨酸(D187) (图3,B和D)。RAD51还含有一个“FXXA”基序(图3B),苯丙氨酸(F)和丙氨酸(A)在其中高度保守[30]。通过该基序可以与其他RAD51分子结合,同时再与DNA结合,形成更高阶的丝状结构。然而,在DMC1中没有发现在RAD51中类似的结构域。
在成核(nucleation)以及同源搜索和链交换的动态性质的相关研究中,取得了很大的进展,如最小成核大小(约6个重组酶)对稳定的核蛋白丝形成的重要性、碱基三联体(base triplet)在稳定的RecA/ Rad51/Dmc1-ssDNA结合模式中的重要性,以及链交换过程中8核苷酸大小的微同源性搜索等[31,32]。重组酶介导链入侵是以碱基三联体的步骤进行的,虽然RecA、RAD51和DMC1都能允许错配,但是只有DMC1能稳定异位双链DNA结合中单、双、三碱基位点,甚至稳定包含碱基内部三联体的错配[33]。RAD51和DMC1错配耐受性的差异主要是由Loop1和Loop2结构域决定的(图3C)。在里氏木霉() RAD51介导的同源性搜索过程中,Loop1和Loop2共同作用从而获得错配耐受性[34]。最新的研究也表明Loop1区域并不是独立工作的,Loop2通过与Loop1相互作用在链交换过程中行使校对检查点的功能[35]。对HsDMC1前联会(presynapsis)和后联会(postsynapsis)复合物的结构分析表明,Loop1中244位谷氨酰胺 Gln244 (在RAD51中为243位甲硫氨酸Met243)可能有助于稳定DNA主干,而Loop2 中274位脯氨酸Pro274和275位甘氨酸Gly275 (在RAD51中为273位缬氨酸Val273和274位天冬氨酸Asp274)可能提供了一个开放的错配耐受性“三联门”(图3C,图4)。一个紧密的门和一个松散的主干支持有助于RAD51的高保真度,而一个松散的门和一个紧密的主干支持有助于DMC1的错配耐受性[36]。将二者相对应的氨基酸位点突变发现含有DMC1 Loop1谱系特异性氨基酸(lineage-specific amino acids)的RAD51嵌合体能够稳定不匹配的碱基三联体,而含有RAD51 Loop1谱系特异性氨基酸的DMC1嵌合体则丧失了这种能力[37]。DMC1这种稳定不完全配对重组中间体的能力可能反映了两种真核生物重组酶的内在差异,暗示了DMC1介导的减数分裂重组能产生更多的遗传变异。此外,同源配对后,RAD51会迅速被一些蛋白质从重组位点上移除,而DMC1则不是。已知有几种蛋白质可以分解重组中间体,包括酿酒酵母解旋酶Srs2 (silver-russell syndrome 2)和Sgs1 (salivary gland secretion 1)。Srs2被认为是一种典型的反重组酶,它能拆除含有Rad51的重组中间体。Srs2通过分解Rad51-ssDNA和D-loop中间体发挥作用,从而引导重组中间体通过合成依赖链退火(SDSA)途径产生非交叉[37~41]。Dmc1则能抑制Srs2的ATP酶活性,这可能有助于促进减数分裂过程产生I型或II型交叉[42]。有假设认为含有碱基错配的中间体可能以某种方式改变核蛋白复合物的结构,从而使它们更容易受到解旋酶的破坏。这些酶或其他酶可能识别一些明显的错配依赖相关结构特征,使它们更容易作用于Rad51结合的中间体,而Dmc1可以保护不匹配的中间体免受这些酶的影响[37]。RAD51和DMC1对碱基错配容忍度的差异可能是由于在体细胞中RAD51介导的同源重组必须有一定的保真度,以免细胞基因突变对生物个体生存造成极大的损伤,而在减数分裂细胞中,DMC1这种对错配碱基的容忍度为生物体进化和变异提供物质基础,产生新的基因型以适应环境的变化。
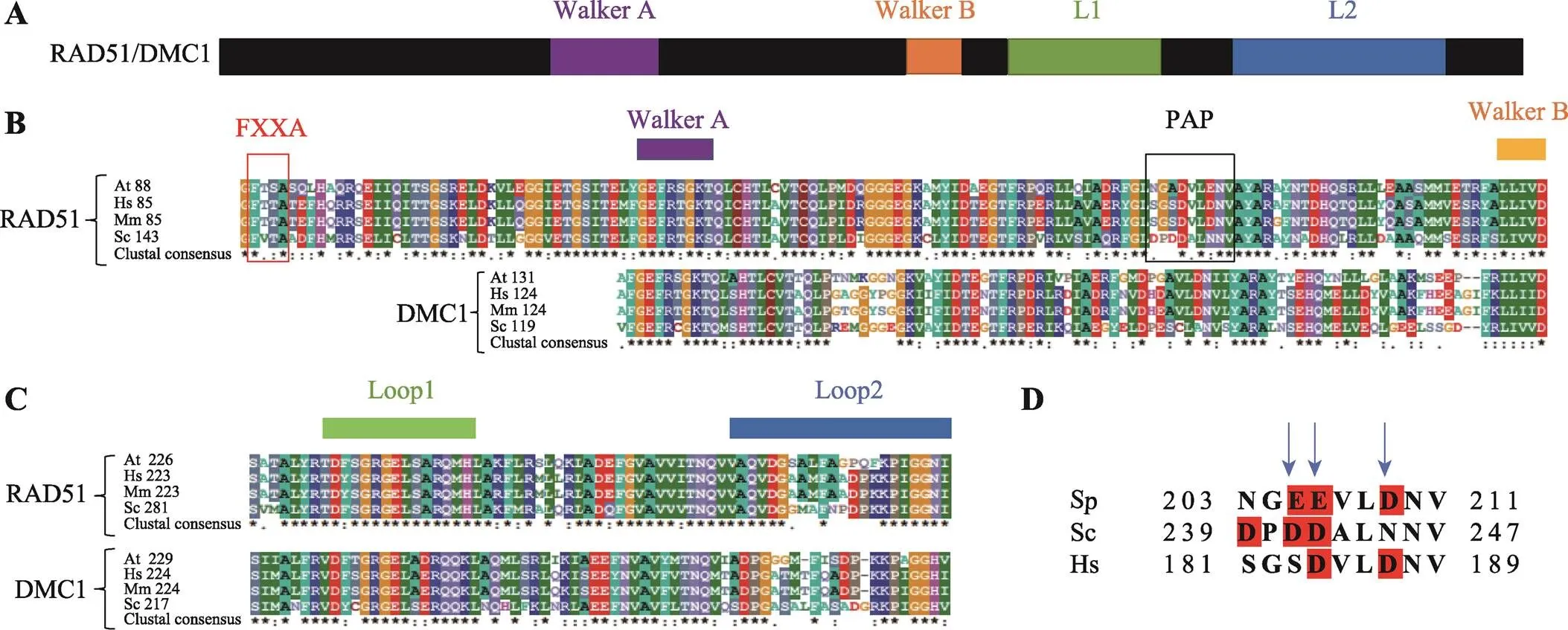
图3 代表物种RAD51和DMC1结构域的定位和序列
A:RAD51和DMC1蛋白结构域定位;B:代表物种RAD51的FXXA、Walker A、PAP和Walker B与DMC1的Walker A和Walker B同源序列对比;C:在代表物种中RAD51和DMC1的Loop1和Loop2同源序列对比;D:B图中PAP motif的放大图。At:拟南芥;Hs:人;Mm:小鼠;Sc:酿酒酵母;Sp:裂殖酵母。图中拟南芥对应的TAIR号为RAD51:AT5G20850.1,DMC1:AT3G22880.1。人、小鼠和酵母的蛋白序列在https://www.uniprot.org/网站下载,序列号分别为Hs RAD51 (Q06609)、Mm RAD51 (Q08297)、Sc RAD51 (P25454)、Hs DMC1 (Q14565)、Mm DMC1 (Q61880)和Sc DMC1 (P25453)。
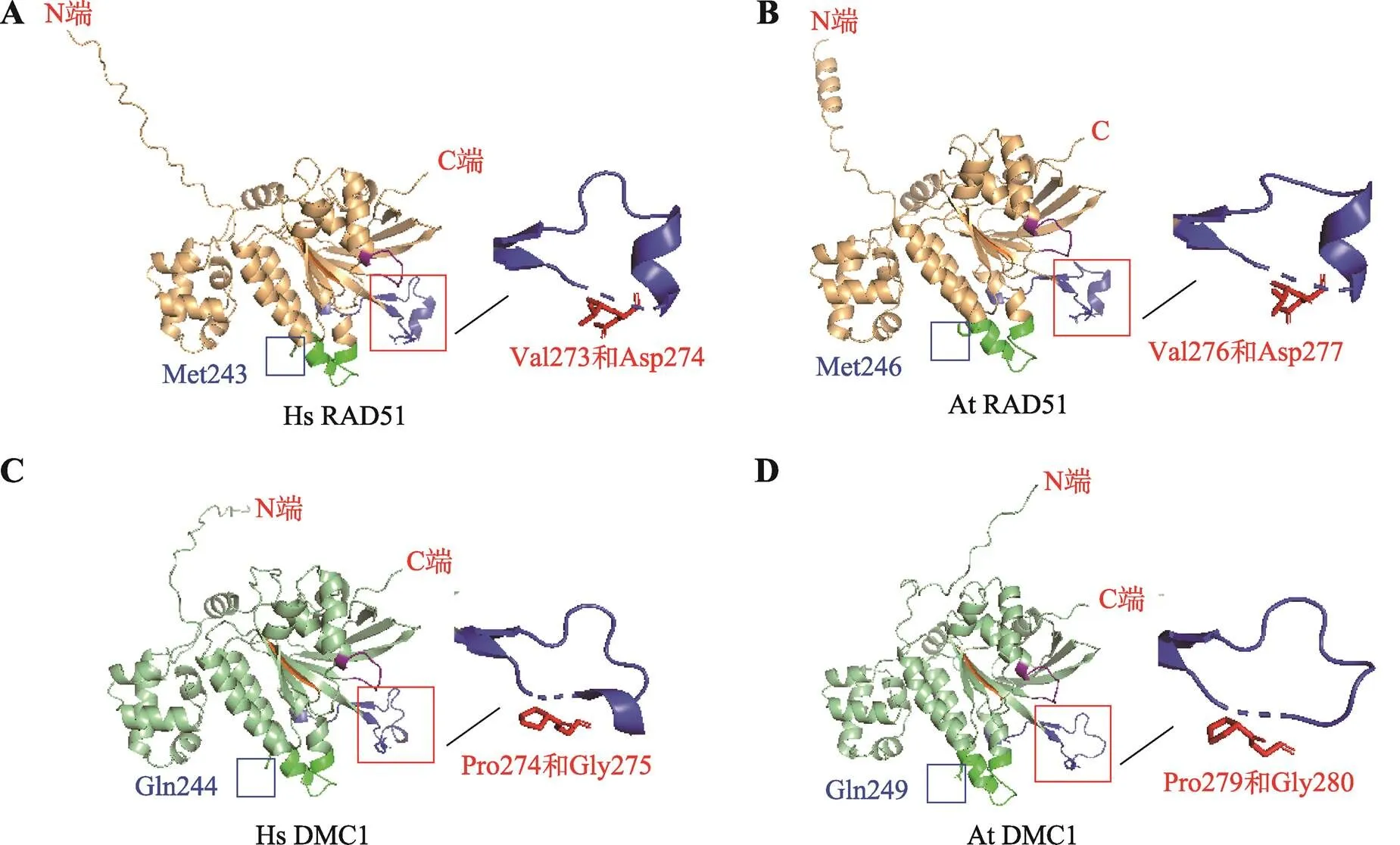
图4 人和拟南芥RAD51和DMC1晶体结构
A:人RAD51晶体结构;B:拟南芥RAD51晶体结构;C:人DMC1晶体结构;D:拟南芥DMC1晶体结构。图中紫色代表Walker A结构,橙色代表Walker B结构,绿色代表Loop1 (L1)结构,蓝色代表Loop2 (L2)结构。蓝色框代表Loop1中关键性谱系特异性氨基酸残基,红色框代表Loop2中关键性谱系特异性氨基酸残基。影响RAD51和DMC1对碱基错配耐受性的关键性残基分别用蓝色和红色标出。图中所用模型来自 AlphaFold 蛋白质结构数据库(Protein Structure Database) https://alphafold.ebi.ac.uk/,结构展示软件为PyMOL (version 2.5)。
总之,无论是RAD51和DMC1对碱基错配的容忍度,还是DMC1能抑制反重组酶的ATP酶活性免于重组中间体被分解的作用,都证明减数分裂特异性重组酶DMC1的存在是为了保证生物体通过减数分裂重组产生更丰富的多样性。
2.2 结构上的保守性和差异性
为了探究人和拟南芥RAD51及DMC1蛋白质结构的保守性与差异性,利用AlphaFold蛋白质结构数据库[43,44]获得它们的晶体结构(图4)。人与拟南芥中RAD51和DMC1的晶体结构非常相似,尤其是参与ATP结合和水解的Walker A和Walker B等保守结构域(图4,紫色和橙色显示)。此外,人和拟南芥RAD51的Loop2中缬氨酸(Val)和天冬氨酸(Asp)形成的构象十分相似(图4,放大显示)。虽然人和拟南芥DMC1 Loop2中脯氨酸(Pro)和甘氨酸(Gly)所形成的构象也十分相似,但是这种构象与RAD51明显不同。这种不同暗示着DMC1能允许新合成的DNA序列的碱基较为灵活,因而错配耐受性提高,保真度降低(图4,放大显示)。Xu等[35]最新研究为RAD51和DMC1的差异提供了新的解释,人RAD51的Loop2中273位缬氨酸(V273)和274位天冬氨酸(D274),对应于DMC1的274位脯氨酸(P274)和275位甘氨酸(G275),在DNA结合中起着至关重要的作用(图4)。此外,人RAD51的273位缬氨酸(V273)和238位亮氨酸(L238)可以形成一个V273:L238疏水/空间门(hydrophobic/steric gate),274位天冬氨酸(D274)和235位精氨酸(R235)可以形成一个D274:R235盐桥。它们共同作用可以“锁住”邻近的三碱基对,导致低容错性和高保真度;而在DMC1中,疏水/空间效应较小,没有盐桥,这可以“放松”对三碱基对的控制,使碱基对具有一定的构象灵活性,导致高容错性和低保真度[36]。
虽然RAD51和DMC1都是减数分裂重组所必需的,但是它们与DNA结合所形成的结构并不完全相同。在没有DNA的条件下,RAD51以由6个单体组成的环的形式存在,而DMC1形成八聚体环[45]。当DNA存在时,RAD51形成螺旋状细丝,DMC1也形成螺旋状细丝或堆叠的八聚体环[46]。酿酒酵母和裂殖酵母Dmc1蛋白也有与八聚体环相似的结构[31,47],这说明DMC1的环状结构从酵母到人类相对保守,但是RAD51却不以环的方式与DNA结合,只有螺旋状细丝的形式存在。
细菌RecA、酵母和人RAD51的结构通过x射线晶体学和冷冻电子显微镜进行了解析,这些结构表明,利用一个RAD51/RecA/DMC1和3个核苷酸的比例与ssDNA结合,ssDNA局部类似于B型DNA,因此可以与互补链进行碱基配对[48]。近年来对Rad51晶体结构的研究表明,Rad51-DNA相互作用调节了在核蛋白丝中组装的Rad51前聚合物的螺旋旋转和上升,这有助于解释Rad51突变影响核蛋白丝组装的机制[49]。酿酒酵母Dmc1 (ScDmc1)核蛋白丝在动力学上大大减少了成核步骤,比Rad51核蛋白丝更不稳定。与ScRad51相比,ScDmc1的成核速率较低是因为其与ssDNA亲和力较低。ScDmc1的成核主要在含有单链和双链DNA连接的DNA结构上发生,允许在5′到3′极性上延伸,而ScRad51的成核强烈依赖于ssDNA的长度。在哺乳动物中,RAD51和DMC1也保留了这种成核倾向。DMC1的成核可以通过结构组成(如DNA连接和蛋白质相互作用)和DNA极性促进[50]。另外,有研究发现RAD51-ssDNA复合物能有效地与核小体结合,但是核小体可能会阻碍RAD51-ssDNA复合物接近同源染色体,并且在没有核小体重塑因子RAD54的情况下无法克服这一阻碍[51]。相比之下,DMC1-ssDNA复合物与核小体的结合活性较低,表明DMC1-ssDNA复合物更易接近同源染色体,因而DMC1-ssDNA复合物优先靶向核小体缺失的区域,即减数分裂重组的热点区域[51]。这可能是减数分裂重组中主要由DMC1介导同源重组而不是RAD51介导的原因之一。
3 RAD51和DMC1功能的保守与分化
RAD51和DMC1广泛存在于绝大多数真核生物中,其功能有共性也有特性。RAD51主要介导姐妹染色单体为模板的DSB修复,DMC1则介导同源染色体为模板的DSB修复。有丝分裂DSB主要以姐妹染色单体为模板进行修复,而在减数分裂期间,姐妹和非姐妹染色单体都能够作为修复的模板[52]。这可能是RAD51同时参与有丝分裂和减数分裂而DMC1是减数分裂特异的原因之一。
如前所述,RAD51和DMC1都是在ssDNA上结合3个核苷酸,并将DNA扩展到大约1.5倍,以ATP依赖的方式形成一个右手螺旋丝[31,52,53]。同源重组的机制在细菌中的研究较为深入,包括核蛋白丝形成和链入侵两个关键步骤。为了确保其准确性并避免潜在的有害后果,这两个过程是可逆的并受到精确调控[54,55]。其中链交换反应以同源重组为中心,通过RecA核蛋白丝的组装、重排和拆卸催化DNA链的协调来进行。在连续进行链交换的同时,链入侵和蛋白质分离的速率相似,会导致一个恒定长度的DNA合成区域(80bp左右)沿着同源区域移动[56]。最新研究发现,RecA以降维的方式搜索找到同源DNA,因为这种搜索不是在三维立体空间,而发生在二维层面,这大大加快了搜索的速度。RecA- ssDNA丝状结构以降维的方式,将至少一个ssDNA片段定位在其同源染色体附近,这样同源dsDNA片段可以使用快速、短距离的搜索找到ssDNA片段。虽然是以降维的方式搜索同源DNA,但只要核蛋白丝长度随DNA数量的增加而变化,搜索时间就不受DNA数量增加的影响[57]。下面将从功能定位、生物学功能和分子机制3个方面介绍真核生物中RAD51和DMC1的功能的异同点。
3.1 RAD51和DMC1在减数分裂重组中的功能定位
RAD51和DMC1定位可以利用免疫荧光使用共聚焦显微镜评估二者位点在细胞核中的总体分布,也可以使用结构照明显微镜(structured illumination microscopy, SIM)和直接随机光学重建显微镜(direct stochastic optical reconstruction microscopy, dSTORM)相结合的方法,对减数分裂前期细胞核中RAD51和DMC1位点的纳米细节进行可视化。DMC1和RAD51在ssDNA丝上的定位已有报道。对酿酒酵母的研究表明,Rad51和Dmc1在切除的DSB末端存在并排定位[58]。酿酒酵母Dmc1和Rad51的体外实验表明,Dmc1和Rad51可以自发地在ssDNA上分离成离散的同源聚合物丝。RAD51和DMC1具有形成空间上不同的丝状体的能力,表明其他辅助因子可能不需要直接分离RAD51和DMC1丝状体[59]。在酿酒酵母突变体中,Dmc1核蛋白丝的形成发生改变,但在突变体中,Rad51定位正常。在拟南芥中,几乎没有发现RAD51和DMC1共同定位,RAD51的定位也不受RAD51B、XRCC2和DMC1的影响,但依赖于SWI1[60,61]。最近对小鼠()的研究表明,在同源搜索过程中DMC1在DNA断裂位点附近,而RAD51远离DNA断裂点,也就是说DMC1优先加载在ssDNA的剪切端,而RAD51优先加载在相对端,且ssDNA结合的DMC1数量多于RAD51[62]。这可能是因为在减数分裂重组过程中链交换是由DMC1介导而不是由RAD51介导,所以DMC1结合在剪切位点以便介导同源染色体DNA双螺旋链的搜索和交换。对小鼠的研究表明,与DMC1相比,RAD51蛋白位点会随着减数分裂前期的进展而变化,并且定位在更接近于介导同源染色体间物理连接(联会)的蛋白轴上[63]。
3.2 RAD51和DMC1在减数分裂重组中的生物学功能
在裂殖酵母中,Rad51的缺失强烈影响减数分裂重组,导致DSB无法修复和细胞周期阻滞。Dmc1的缺失会导致类似的表型,但能产生少量可育孢子,突变体中的缺陷可以通过Rad51的过表达实现部分恢复[52,64,65]。在拟南芥中,RAD51的缺失导致染色体配对和联会存在缺陷,并伴随大量的染色体碎裂[20,66],而在玉米()中RAD51位点缺失与配对缺陷相关[67]。这揭示了RAD51对减数分裂重组的多重影响。拟南芥突变体的同源染色体虽然也不能配对和联会,但是没有明显染色体碎片[68,69]。在单子叶植物大麦(L)突变体中,减数分裂过程中染色体异常,在后期和末期能够观察到染色体碎片和染色体桥[70]。与RAD51在植物中不影响营养生长而严重影响减数分裂进程截然不同的是[31],脊椎动物中突变体是致死性的。小鼠敲除突变体完全不育,出现同源染色体配对、联会和DSB修复缺陷[71,72]。同时突变两个重组酶会导致比任何一个单一的突变更为严重的表型[73,74],这说明二者功能存在部分冗余。
RAD51和DMC1在减数分裂过程中的必要性是不同的,比如催化活性较差的酵母突变体虽然缺乏催化链交换活性但对重组没有特别大的影响,在拟南芥中也有类似的结果[75]。表明在减数分裂重组中需要的是RAD51蛋白本身而不是其链交换活性,而失去链交换活性的突变体与缺失突变体具有相同的减数分裂前期阻滞[23,76]。这说明RAD51在有丝分裂和减数分裂过程中在主要功能上有差异,RAD51和DMC1同时存在时,DMC1在减数分裂重组中发挥链交换的功能。RAD51蛋白作为DMC1的辅助因子促进DMC1前联会核蛋白丝的组装,当DMC1缺失时,RAD51可以以姐妹单体为模板的DSB修复,最终产生非交叉[77]。
综上所述,RAD51和DMC1在功能上存在部分冗余和相对特异性。在减数分裂重组过程中,参与同源染色体间重组和链交换主要由DMC1介导并形成交叉,而RAD51辅助DMC1并介导姐妹染色单体间重组导致非交叉。
3.3 RAD51和DMC1在减数分裂重组中的分子机制
减数分裂重组通常倾向于使用同源染色体而不是姐妹染色单体,以便在第一次减数分裂中形成使染色体精确分离所必需的交叉,这种现象被称为“IH (inter homolog)偏爱”[78]。拟南芥ASY1 (meiotic asynaptic mutant 1)、ASY3 (meiotic asynaptic mutant 3)、HOP2 (homeodomain-only protein)和植物特异性蛋白SDS (SOLO DANCERS)促进DMC1介导的IH修复而不是RAD51介导IS (inter sister)修复[79~82],而ATM可能通过拟南芥IS重组促进RAD51介导的减数分裂DSB修复[83]。虽然有研究认为在DMC1介导同源染色体间重组的同时,也发生姐妹染色单体的重组[15],但是在此期间RAD51活性不高[84,85]。如果RAD51被过早激活也可以介导同源染色体间的重组,但是不如DMC1效率高[86,87]。这恰好解释了突变体中RAD51过表达可以部分回复异常,可能是由于RAD51被提前激活。
3.3.1 RAD51和DMC1可以与同一个辅助因子的不同区域相互作用
RAD51和DMC1都可以与BRCA2相互作用,促进同源交换和重组的稳定性。然而,人BRCA2的PhePP结构域只能特异结合DMC1,而BRC重复序列可以同时结合RAD51和DMC1[88]。在拟南芥中,BRCA2可以存在于一个三伴侣复合物中,与DSS1 (decreased sperm survival 1)和RAD51同时相互作用或与DSS1和DMC1同时相互作用[89]。RAD51和DMC1分别与BRCA2不同结构域相互作用,这可能与二者在同源重组中的功能特异性有关。此外,拟南芥FIGL1 (fidgetin-like protein 1)和FLIP (fidgetin- like 1 interacting protein)与BRCA2对RAD51/DMC1依赖的DNA动态交换具有拮抗作用,从而维持同源重组(homologous recombination, HR)的准确修复[90],且FIGL1比FLIP发挥更核心的作用[91](图5)。
3.3.2 RAD51和DMC1与不同的辅助因子相互作用
生化研究表明,酵母Dmc1主要与Rdh54/Tid互作,而Rad51主要与Rad54互作,与Dmc1的减数分裂特异性不同,Rdh54/Tid在体细胞中也有表达[92]。Rdh54/Tid1和Rad54共同存在与只有Rad54存在时相比,D-loop的形成和长度都受到抑制,可能是Rdh54/Tid1和Rad54在Rad51核蛋白丝中竞争潜在的结合位点。Rdh54/Tid1作为RAD54物理屏障,限制了D-loop的形成和长度[93]。遗传学研究表明,在拟南芥DMC1缺失的情况下,RAD54是RAD51修复减数分裂双链断裂所必需的[94]。人RAD54蛋白还可以将RAD51从dsDNA中分离出来,而不是ssDNA[95]。对真核生物减数分裂研究表明,在前联会复合物中加入Rad54和存在较少Rdh54的条件下,可以提高同源搜索率[96]。在酿酒酵母中,减数分裂特异蛋白Hed1与Rad51相互作用,可以阻断Rad54与Rad51的相互作用[97]。减数分裂特异蛋白Hed1对Rad51的限制作用会下调其活性,不利于以姐妹染色单体作为模板进行DNA合成,从而有利于Dmc1介导的同源染色体间重组发生[16,98]。在植物中还没有发现Hed1的同源物,但是可能存在相似的机制。ATR (serine/threonine kinase)在调控拟南芥DMC1丝形成方面非常重要,拟南芥双突变体允许DMC1组装和随后的联会、减数分裂中DSB的修复发生和交叉形成。

图5 重组酶RAD51和DMC1在植物中参与减数分裂重组的机制
蓝色和红色线条分别描绘了两条父母亲本双链DNA。重组酶DMC1和RAD51结合到ssDNA上由BRCA2和DSS1介导,RAD51C和XRCC3与RAD51结合可能改变了RAD51结构有助于RAD51的装载。SMC5/6复合物与RAD51和DMC1三者之间结合相互平衡,RAD51能抑制SMC5/6复合物与DMC1的结合,有助于DMC1进行同源重组。而FIGL1-FLIP与BRCA2拮抗作用,抑制RAD51和DMC1的装载。Hop2-Mnd1与RAD51和DMC1结合促进链交换。RAD54不仅参与了促进链交换同时也参与RAD51在dsDNA上的去除,但DMC1的降解机制还有待探索。
3.3.3 RAD51和DMC1与辅助因子相互作用所需要的条件不同
Hop2-Mnd1与RAD51和DMC1重组酶的相互作用是非常重要的,但Hop2-Mnd1与RAD51的结合依赖于ATP的催化,而Hop2-Mnd1与DMC1的结合不依赖于ATP的催化[99,100]。在机制上,裂殖酵母Swi5-Sfr1 (酿酒酵母Mei5-Sae3蛋白复合物)促进DMC1核蛋白丝的建立,参与成核或丝的延长[101,102],同时Mei5-Sae3和RAD51对DMC1丝形成的促进作用是相互独立的[103~105]。而Hop2-Mnd1定义了启动链交换的关键限速步骤。在执行这一功能之后,Swi5-Sfr1与Hop2-Mnd1随后促进了链交换[106]。Mnd1 (meiotic nuclear divisions 1)是DMC1的一种附属蛋白,在DMC1核蛋白丝形成后,DMC1的正常活性需要Mnd1,Mnd1也可能参与了DMC1的移除。例如,在没有Mnd1的情况下,DMC1核蛋白丝积累的时间更长[8]。同样,在拟南芥突变背景下,DMC1位点变多会导致不能完成DSB的修复[94]。
3.3.4 RAD51和DMC1与相同的辅助因子相互作用影响不同
在裂殖酵母中,Rad22 (酿酒酵母Rad52的同源物)激活Rad51 (也称为Rhp51),但抑制Dmc1[107]。最近的研究表明,拟南芥RAD51能通过抑制减数分裂过程中的SMC5/6复合物来促进DMC1在同源重组中的作用[108](图5)。
在具有DMC1的生物体中,染色体配对是通过重组依赖机制启动的[77,109],而不具有DMC1的生物体如秀丽隐杆线虫()在减数分裂过程中链交换功能由RAD51行使[110~112]。这说明基因可能不是有性生殖生物所必须的,但DMC1蛋白的功能在减数分裂中是必须的。然而,在RAD51和DMC1之间几乎没有其他差异,这可能有助于解释在有丝分裂和减数分裂中使用不同重组酶的可能起源或潜在的进化优势。
4 结语与展望
重组酶是DNA损伤修复和减数分裂重组所必需的酶,而RAD51与DMC1是真核生物中相对保守的重组酶,其功能的相似性和特异性在不同物种中均已报道。同时对RAD51和DMC1的研究也是DSB修复领域热点,比如RAD51可以作为癌症治疗的靶点,DMC1则成为生殖发育中配子形成和遗传多样性产生的关键点。二者对基因组完整性的维持都十分重要。
基因组完整性对于有丝分裂和减数分裂都至关重要。在有丝分裂中,基因突变与植物的发育和抗逆性,以及人类的癌症等密切相关。而有性生殖生物减数分裂过程中同源染色体的重组和分离对维持染色体数目恒定至关重要。在体细胞DSB修复过程中,SDSA和SSA是两个主要的 HR途径。减数分裂重组途径更加多样性,涉及到DSB产生后选择同源染色体或姐妹染色单体作为模板进行修复。尽管RAD51和DMC1重组酶具有共同祖先,在蛋白质序列和结构上都非常相似,但是在有丝分裂和减数分裂重组中的功能却大相径庭。其中RAD51保证严格的基因组稳定性,不允许碱基的错配;相比之下DMC1对碱基错配的包容性更高,大大提高了后代的遗传多样性,对物种的进化具有重要意义。
目前通过遗传学、生物化学、分子生物学、结构生物学等手段对RAD51和DMC1在同源重组中的功能和作用机制有了一定的认识,但对其调控机制和作用网络还不够清晰,例如RAD51和DMC1蛋白质的降解机制是什么?RAD51和DMC1蛋白质的修饰及与其功能之间的关系?RAD51和DMC1在细胞内的稳态平衡及与DNA结合能力?RAD51和DMC1及与其互作蛋白形成复合体的结构是什么?这些问题都有待深入研究。随着技术方法的发展,更多的新技术应用于生命科学研究中,例如原子力显微镜(atomic force microscopy, AFM)在研究DNA和蛋白质相互作用领域具有广泛的应用[113]。AFM可在空气和液体中对静态的DNA-蛋白复合体进行成像,也可在液体中实时观察DNA和蛋白质的反应过程,而且AFM还可获得DNA和蛋白分子间作用力的信息。AFM已揭示了许多基因调控的机制,也将在生命科学的研究中起到越来越重要的作用。除此之外,单细胞测序[114]、活细胞成像技术[115]及多组学技术的结合,未来能帮助人们更加深入的认识RAD51和DMC1功能的共性和特性。
[1] Osman K, Higgins JD, Sanchez-Moran E, Armstrong SJ, Franklin FC. Pathways to meiotic recombination in.2011, 190(3): 523– 544.
[2] Kuo P, Da Ines O, Lambing C. Rewiring meiosis for crop improvement.2021, 12: 708948.
[3] Taagen E, Bogdanove AJ, Sorrells ME. Counting on crossovers: controlled recombination for plant breeding.2020, 25(5): 455–465.
[4] Shim EY, Chung WH, Nicolette ML, Zhang Y, Davis M, Zhu Z, Paull TT, Ira G, Lee SE.Mre11/Rad50/Xrs2 and Ku proteins regulate association of Exo1 and Dna2 with DNA breaks.,2010, 29(19):3370–3380.
[5] Ghosal G, Muniyappa K. The characterization ofMre11/Rad50/Xrs2 complex reveals that Rad50 negatively regulates Mre11 endonucleolytic but not the exonucleolytic activity.2007, 372(4): 864–882.
[6] Waterworth WM, Altun C, Armstrong SJ, Roberts N, Dean PJ, Young K, Weil CF, Bray CM, West CE. NBS1 is involved in DNA repair and plays a synergistic role with ATM in mediating meiotic homologous recombination in plants.2007, 52(1): 41–52.
[7] Puizina J, Siroky J, Mokros P, Schweizer D, Riha K. Mre11 deficiency inis associated with chromosomal instability in somatic cells and Spo11- dependent genome fragmentation during meiosis., 2004, 16(8): 1968–1978.
[8] Wang YX, Copenhaver GP. Meiotic recombination: mixing it up in plants., 2018, 69: 577–609.
[9] Ma CJ, Gibb B, Kwon Y, Sung P, Greene EC. Protein dynamics of human RPA and RAD51 on ssDNA during assembly and disassembly of the RAD51 filament., 2017, 45(2): 749–761.
[10] Xia B, Sheng Q, Nakanishi K, Ohashi A, Wu JM, Christ N, Liu XG, Jasin M, Couch FJ, Livingston DM. Control of BRCA2 cellular and clinical functions by a nuclear partner, PALB2.2006, 22(6): 719–729.
[11] Vasianovich Y, Altmannova V, Kotenko O, Newton MD, Krejci L, Makovets S. Unloading of homologous recombination factors is required for restoring double- stranded DNA at damage repair loci., 2017, 36(2): 213–231.
[12] Lambing C, Franklin FCH, Wang CR. Understanding and manipulating meiotic recombination in plants., 2017, 173(3): 1530–1542.
[13] Li X, Zhang J, Huang JY, Xu J, Chen ZY, Copenhaver GP, Wang YX. Regulation of interference-sensitive crossover distribution ensures crossover assurance in.2021, 118(47): e2107543118.
[14] Cromie GA, Hyppa RW, Taylor AF, Zakharyevich K, Hunter N, Smith GR. Single holliday junctions are intermediates of meiotic recombination., 2006, 127(6): 1167–1178.
[15] Bonilla B, Hengel SR, Grundy MK, Bernstein KA. RAD51 gene family structure and function., 2020, 54: 25–46.
[16] Jiang S, Lin T, Xie QJ, Wang LJ. Network analysis of RAD51 proteins inand the evolutionary relationships with their archaeal homologs.2018, 9: 383.
[17] Xu Z, Zhang JX, Xu M, Ji W, Yu MM, Tao YJ, Gong ZY, Gu MH, Yu HX. Rice RAD51 paralogs play essential roles in somatic homologous recombination for DNA repair.2018, 95(2): 282–295.
[18] Su H, Cheng ZH, Huang JY, Lin J, Copenhaver GP, Ma H, Wang YX.RAD51, RAD51C and XRCC3 proteins form a complex and facilitate RAD51 localization on chromosomes for meiotic recombination., 2017, 13(5): e1006827
[19] Da Ines O, Degroote F, Amiard S, Goubely C, Gallego ME, White CI. Effects of XRCC2 and RAD51B mutations on somatic and meiotic recombination in., 2013, 74(6): 959–970.
[20] Da Ines O, Abe K, Goubely C, Gallego ME, White CI. Differing requirements for RAD51 and DMC1 in meiotic pairing of centromeres and chromosome arms in., 2012, 8(4): e1002636.
[21] Pradillo M, López E, Linacero R, Romero C, Cuñado N, Sánchez-Morán E, Santos JL. Together yes, but not coupled: new insights into the roles of RAD51 and DMC1 in plant meiotic recombination., 2012, 69(6): 921–933.
[22] Hong S, Sung YJ, Yu M, Lee M, Kleckner N, Kim KP. The logic and mechanism of homologous recombination partner choice., 2013, 51(4): 440–453.
[23] Da Ines O, Degroote F, Goubely C, Amiard S, Gallego ME, White CI. Meiotic recombination inis catalysed by DMC1, with RAD51 playing a supporting role., 2013, 9(9): e1003787.
[24] Lin ZG, Kong HZ, Nei M, Ma H. Origins and evolution of the recA/RAD51 gene family: evidence for ancient gene duplication and endosymbiotic gene transfer., 2006, 103(27): 10328–10333
[25] Prentiss M, Prévost C, Danilowicz C. Structure/function relationships in RecA protein-mediated homology recognition and strand exchange., 2015, 50(6): 453–476.
[26] Chintapalli SV, Bhardwaj G, Babu J, Hadjiyianni L, Hong YJ, Todd GK, Boosalis CA, Zhang ZH, Zhou XF, Ma H, Anishkin A, van Rossum DB, Patterson RL. Reevaluation of the evolutionary events within recA/ RAD51 phylogeny2013, 14: 240.
[27] Ramesh MA, Malik SB, Logsdon JM, Jr. A phylogenomic inventory of meiotic genes; evidence for sex inand an early eukaryotic origin of meiosis., 2005, 15(2): 185–191.
[28] Kurumizaka H, Aihara H, Kagawa W, Shibata T, Yokoyama S. Human Rad51 amino acid residues required for Rad52 binding.1999, 291(3): 537–548.
[29] Afshar N, Argunhan B, Palihati M, Taniguchi G, Tsubouchi H, Iwasaki H. A novel motif of Rad51 serves as an interaction hub for recombination auxiliary factors., 2021, 10: e64131.
[30] Scott DE, Marsh M, Blundell TL, Abell C, Hyvönen M. Structure-activity relationship of the peptide binding- motif mediating the BRCA2:RAD51 protein-protein interaction.2016, 590(8): 1094–1102.
[31] Lee JY, Terakawa T, Qi Z, Steinfeld JB, Redding S, Kwon Y, Gaines WA, Zhao WX, Sung P, Greene EC. DNA recombination. base triplet stepping by the Rad51/ RecA family of recombinases.015, 349(6251): 977–981.
[32] Qi Z, Redding S, Lee JY, Gibb B, Kwon Y, Niu H, Gaines WA, Sung P, Greene EC. DNA sequence alignment by microhomology sampling during homologous recombination., 2015, 160(5): 856–869.
[33] Lee JY, Steinfeld JB, Qi Z, Kwon Y, Sung P, Greene EC. Sequence imperfections and base triplet recognition by the Rad51/RecA family of recombinases., 2017, 292(26): 11125–11135.
[34] Li WC, Lee CY, Lan WH, Woo TT, Liu HC, Yeh HY, Chang HY, Chuang YC, Chen CY, Chuang CN, Chen CL, Hsueh YP, Li HW, Chi P, Wang TF.Rad51 tolerates mismatches in hybrid meiosis with diverse genome sequences., 2021, 118(8): e2007192118.
[35] Xu JF, Zhao LY, Peng SJ, Chu HY, Liang R, Tian M, Connell PP, Li GH, Chen CL, Wang HW. Mechanisms of distinctive mismatch tolerance between Rad51 and Dmc1 in homologous recombination., 2021, 49(22): 13135–13149.
[36] Luo SC, Yeh HY, Lan WH, Wu YM, Yang CH, Chang HY, Su GC, Lee CY, Wu WJ, Li HW, Ho MC, Chi P, Tsai MD. Identification of fidelity-governing factors in human recombinases DMC1 and RAD51 from cryo-EM structures., 2021, 12(1): 115.
[37] Steinfeld JB, Beláň O, Kwon Y, Terakawa T, Al-Zain A, Smith MJ, Crickard JB, Qi Z, Zhao WX, Rothstein R, Symington LS, Sung P, Boulton SJ, Greene EC. Defining the influence of Rad51 and Dmc1 lineage-specific amino acids on genetic recombination., 2019, 33(17–18): 1191–1207.
[38] Bugreev DV, Pezza RJ, Mazina OM, Voloshin ON, Camerini-Otero RD, Mazin AV. The resistance of DMC1 D-loops to dissociation may account for the DMC1 requirement in meiosis., 2011, 18(1): 56–60.
[39] Lorenz A. Modulation of meiotic homologous recombination by DNA helicases.2017, 34(5): 195–203.
[40] Branzei D, Szakal B. Building up and breaking down: mechanisms controlling recombination during replication., 2017, 52(4): 381–394.
[41] Spell RM, Jinks-Robertson S. Examination of the roles of Sgs1 and Srs2 helicases in the enforcement of recombination fidelity in., 2004, 168(4): 1855–1865.
[42] Crickard JB, Kaniecki K, Kwon Y, Sung P, Greene EC. Meiosis-specific recombinase Dmc1 is a potent inhibitor of the Srs2 antirecombinase.2018, 115(43): E10041–E10048.
[43] Varadi M, Anyango S, Deshpande M, Nair S, Natassia C, Yordanova G, Yuan D, Stroe O, Wood G, Laydon A, Žídek A, Green T, Tunyasuvunakool K, Petersen S, Jumper J, Clancy E, Green R, Vora A, Lutfi M, Figurnov M, Cowie A, Hobbs N, Kohli P, Kleywegt G, Birney E, Hassabis D, Velankar S. AlphaFold protein structure database: massively expanding the structural coverage of protein-sequence space with high-accuracy models., 2022, 50(D1): D439–D444.
[44] Jumper J, Evans R, Pritzel A, Green T, Figurnov M, Ronneberger O, Tunyasuvunakool K, Bates R, Žídek A, Potapenko A, Bridgland A, Meyer C, Kohl SAA, Ballard AJ, Cowie A, Romera-Paredes B, Nikolov S, Jain R, Adler J, Back T, Petersen S, Reiman D, Clancy E, Zielinski M, Steinegger M, Pacholska M, Berghammer T, Bodenstein S, Silver D, Vinyals O, Senior AW, Kavukcuoglu K, Kohli P, Hassabis D. Highly accurate protein structure prediction with AlphaFold., 2021, 596(7873): 583–589.
[45] Kinebuchi T, Kagawa W, Enomoto R, Tanaka K, Miyagawa K, Shibata T, Kurumizaka H, Yokoyama S. Structural basis for octameric ring formation and DNA interaction of the human homologous-pairing protein Dmc1., 2004, 14(3): 363–374.
[46] Shin DS, Pellegrini L, Daniels DS, Yelent B, Craig L, Bates D, Yu DS, Shivji MK, Hitomi C, Arvai AS, Volkmann N, Tsuruta H, Blundell TL, Venkitaraman AR, Tainer JA. Full-length archaeal Rad51 structure and mutants: mechanisms for RAD51 assembly and control by BRCA2., 2003, 22(17): 4566–4576.
[47] Sauvageau S, Stasiak AZ, Banville I, Ploquin M, Stasiak A, Masson JY. Fission yeast rad51 and dmc1, two efficient DNA recombinases forming helical nucleoprotein filaments.2005, 25(11): 4377–4387.
[48] Xu JF, Zhao LY, Xu YY, Zhao WX, Sung P, Wang HW. Cryo-EM structures of human RAD51 recombinase filaments during catalysis of DNA-strand exchange., 2017, 24(1): 40–46.
[49] Short JM, Liu Y, Chen SX, Soni N, Madhusudhan MS, Shivji MKK, Venkitaraman AR. High-resolution structure of the presynaptic RAD51 filament on single-stranded DNA by electron cryo-microscopy., 2016, 44(19): 9017–9030.
[50] Lan WH, Lin SY, Kao CY, Chang WH, Yeh HY, Chang HY, Chi P, Li HW. Rad51 facilitates filament assembly of meiosis-specific Dmc1 recombinase., 2020, 117(21): 11257–11264.
[51] Kobayashi W, Takaku M, Machida S, Tachiwana H, Maehara K, Ohkawa Y, Kurumizaka H. Chromatin architecture may dictate the target site for DMC1, but not for RAD51, during homologous pairing., 2016, 6: 24228.
[52] Hyppa RW, Smith GR. Crossover invariance determined by partner choice for meiotic DNA break repair., 2010, 142(2): 243–255.
[53] Bishop DK, Park D, Xu L, Kleckner N. DMC1: a meiosis-specific yeast homolog of E. coli recA required for recombination, synaptonemal complex formation, and cell cycle progression., 1992, 69(3): 439–456.
[54] Sehorn MG, Sigurdsson S, Bussen W, Unger VM, Sung P. Human meiotic recombinase Dmc1 promotes ATP-dependent homologous DNA strand exchange., 2004, 429(6990): 433–437.
[55] Heyer WD. Regulation of recombination and genomic maintenance., 2015, 7(8): a016501.
[56] van der Heijden T, Modesti M, Hage S, Kanaar R, Wyman C, Dekker C. Homologous recombination in real time: DNA strand exchange by RecA., 2008, 30(4): 530–538.
[57] Wiktor J, Gynnå AH, Leroy P, Larsson J, Coceano G, Testa I, Elf J. RecA finds homologous DNA by reduced dimensionality search., 2021, 597(7876): 426–429.
[58] Brown MS, Grubb J, Zhang AN, Rust MJ, Bishop DK. Small Rad51 and Dmc1 complexes often co-occupy both ends of a meiotic DNA double strand break, 2015, 11(12): e1005653.
[59] Crickard JB, Kaniecki K, Kwon Y, Sung P, Greene EC. Spontaneous self-segregation of Rad51 and Dmc1 DNA recombinases within mixed recombinase filaments., 2018, 293(11): 4191–4200.
[60] Kurzbauer MT, Uanschou C, Chen D, Schlögelhofer P. The recombinases DMC1 and RAD51 are functionally and spatially separated during meiosis in.2012, 24(5): 2058–2070.
[61] Mercier R, Armstrong SJ, Horlow C, Jackson NP, Makaroff CA, Vezon D, Pelletier G, Jones GH, Franklin FCH. The meiotic protein SWI1 is required for axial element formation and recombination initiation in.2003, 130(14): 3309–3318.
[62] Hinch AG, Becker PW, Li T, Moralli D, Zhang G, Bycroft C, Green C, Keeney S, Shi QH, Davies B, Donnelly P. The configuration of RPA, RAD51, and DMC1 binding in meiosis reveals the nature of critical recombination intermediates., 2020, 79(4): 689–701.e610.
[63] Slotman JA, Paul MW, Carofiglio F, de Gruiter HM, Vergroesen T, Koornneef L, van Cappellen WA, Houtsmuller AB, Baarends WM. Super-resolution imaging of RAD51 and DMC1 in DNA repair foci reveals dynamic distribution patterns in meiotic prophase., 2020, 16(6): e1008595.
[64] Rockmill B, Sym M, Scherthan H, Roeder GS. Roles for two RecA homologs in promoting meiotic chromosome synapsis.1995, 9(21): 2684–2695.
[65] Tsubouchi H, Roeder GS. The importance of genetic recombination for fidelity of chromosome pairing in meiosis., 2003, 5(6): 915–925.
[66] Li WX, Chen CB, Markmann-Mulisch U, Timofejeva L, Schmelzer E, Ma H, Reiss B. TheAtRAD51 gene is dispensable for vegetative development but required for meiosis., 2004, 101(29): 10596–10601.
[67] Franklin AE, Golubovskaya IN, Bass HW, Cande WZ. Improper chromosome synapsis is associated with elongated RAD51 structures in the maize desynaptic2 mutant.2003, 112(1): 17–25.
[68] Crismani W, Portemer V, Froger N, Chelysheva L, Horlow C, Vrielynck N, Mercier R. MCM8 is required for a pathway of meiotic double-strand break repair independent of DMC1 in., 2013, 9(1): e1003165.
[69] Couteau F, Belzile F, Horlow C, Grandjean O, Vezon D, Doutriaux MP. Random chromosome segregation without meiotic arrest in both male and female meiocytes of a dmc1 mutant of., 1999, 11(9): 1623–1634.
[70] Szurman-Zubrzycka M, Baran B, Stolarek-Januszkiewicz M, Kwaśniewska J, Szarejko I, Gruszka D. The dmc1 mutant allows an insight into the DNA double-strand break repair during meiosis in barley ()., 2019, 10: 761.
[71] Pittman DL, Cobb J, Schimenti KJ, Wilson LA, Cooper DM, Brignull E, Handel MA, Schimenti JC. Meiotic prophase arrest with failure of chromosome synapsis in mice deficient for Dmc1, a germline-specific RecA homolog.1998, 1(5): 697–705.
[72] Yoshida K, Kondoh G, Matsuda Y, Habu T, Nishimune Y, Morita T. The mouse RecA-like gene Dmc1 is required for homologous chromosome synapsis during meiosis., 1998, 1(5): 707–718.
[73] Bishop DK. RecA homologs Dmc1 and Rad51 interact to form multiple nuclear complexes prior to meiotic chromosome synapsis., 1994, 79(6): 1081–1092.
[74] Shinohara A, Gasior S, Ogawa T, Kleckner N, Bishop DK.recA homologues RAD51 and DMC1 have both distinct and overlapping roles in meiotic recombination., 1997, 2(10): 615–629.
[75] Singh G, Da Ines O, Gallego ME, White CI. Analysis of the impact of the absence of RAD51 strand exchange activity inmeiosis., 2017, 12(8): e0183006.
[76] Cloud V, Chan YL, Grubb J, Budke B, Bishop DK. Rad51 is an accessory factor for Dmc1-mediated joint molecule formation during meiosis.2012, 337(6099): 1222–1225.
[77] Brown MS, Bishop DK. DNA strand exchange and RecA homologs in meiosis., 2014, 7(1): a016659.
[78] Marsolier-Kergoat MC, Khan MM, Schott J, Zhu X, Llorente B. Mechanistic view and genetic control of DNA recombination during meiosis., 2018, 70(1): 9–20. e26.
[79] Azumi Y, Liu D, Zhao DZ, Li WX, Wang GF, Hu Y, Ma H. Homolog interaction during meiotic prophase I inrequires the SOLO DANCERS gene encoding a novel cyclin-like protein., 2002, 21(12): 3081–95.
[80] Sanchez-Moran E, Santos JL, Jones GH, Franklin FCH. ASY1 mediates AtDMC1-dependent interhomolog recombination during meiosis in., 2007, 21(17): 2220–2233.
[81] De Muyt A, Pereira L, Vezon D, Chelysheva L, Gendrot G, Chambon A, Lainé-Choinard S, Pelletier G, Mercier R, Nogué F, Grelon M. A high throughput genetic screen identifies new early meiotic recombination functions in., 2009, 5(9): e1000654.
[82] Ferdous M, Higgins JD, Osman K, Lambing C, Roitinger E, Mechtler K, Armstrong SJ, Perry R, Pradillo M, Cuñado N, Franklin FC. Inter-homolog crossing-over and synapsis inmeiosis are dependent on the chromosome axis protein AtASY3., 2012, 8(2): e1002507.
[83] Yao Y, Li XJ, Chen WL, Liu H, Mi LM, Ren D, Mo AW, Lu PL. ATM promotes RAD51-mediated meiotic DSB repair by inter-sister-chromatid recombination in., 2020, 11: 839.
[84] Argunhan B, Leung WK, Afshar N, Terentyev Y, Subramanian VV, Murayama Y, Hochwagen A, Iwasaki H, Tsubouchi T, Tsubouchi H. Fundamental cell cycle kinases collaborate to ensure timely destruction of the synaptonemal complex during meiosis., 2017, 36(17): 2488–2509.
[85] Prugar E, Burnett C, Chen XY, Hollingsworth NM. Coordination of double strand break repair and meiotic progression in yeast by a Mek1-Ndt80 negative feedback loop., 2017, 206(1): 497–512.
[86] Callender TL, Laureau R, Wan LH, Chen XY, Sandhu R, Laljee S, Zhou S, Suhandynata RT, Prugar E, Gaines WA, Kwon Y, Börner GV, Nicolas A, Neiman AM, Hollingsworth NM. Mek1 down regulates Rad51 activity during yeast meiosis by phosphorylation of Hed1., 2016, 12(8): e1006226.
[87] Lao JP, Cloud V, Huang CC, Grubb J, Thacker D, Lee CY, Dresser ME, Hunter N, Bishop DK. Meiotic crossover control by concerted action of Rad51-Dmc1 in homolog template bias and robust homeostatic regulation., 2013, 9(12): e1003978.
[88] Thorslund T, Esashi F, West SC. Interactions between human BRCA2 protein and the meiosis-specific recombinase DMC1., 2007, 26(12): 2915–2922.
[89] Dray E, Siaud N, Dubois E, Doutriaux MP. Interaction betweenBrca2 and its partners Rad51, Dmc1, and Dss1., 2006, 140(3): 1059– 1069.
[90] Kumar R, Duhamel M, Coutant E, Ben-Nahia E, Mercier R. Antagonism between BRCA2 and FIGL1 regulates homologous recombination., 2019, 47(10): 5170–5180.
[91] Fernandes JB, Duhamel M, Seguéla-Arnaud M, Froger N, Girard C, Choinard S, Solier V, De Winne N, De Jaeger G, Gevaert K, Andrey P, Grelon M, Guerois R, Kumar R, Mercier R. FIGL1 and its novel partner FLIP form a conserved complex that regulates homologous recombination., 2018, 14(4): e1007317.
[92] Nimonkar AV, Dombrowski CC, Siino JS, Stasiak AZ, Stasiak A, Kowalczykowski SC.Dmc1 and Rad51 proteins preferentially function with Tid1 and Rad54 proteins, respectively, to promote DNA strand invasion during genetic recombination., 2012, 287(34): 28727–28737.
[93] Shah SS, Hartono S, Piazza A, Som V, Wright W, Chédin F, Heyer WD. Rdh54/Tid1 inhibits Rad51- Rad54-mediated D-loop formation and limits D-loop length.2020, 9: e59112.
[94] Hernandez Sanchez-Rebato M, Bouatta AM, Gallego ME, White CI, Da Ines O. RAD54 is essential for RAD51-mediated repair of meiotic DSB in., 2021, 17(5): e1008919.
[95] Mason JM, Dusad K, Wright WD, Grubb J, Budke B, Heyer WD, Connell PP, Weichselbaum RR, Bishop DK. RAD54 family translocases counter genotoxic effects of RAD51 in human tumor cells., 2015, 43(6): 3180–3196.
[96] Renkawitz J, Lademann CA, Jentsch S. Mechanisms and principles of homology search during recombination., 2014, 15(6): 369–383.
[97] Busygina V, Sehorn MG, Shi IY, Tsubouchi H, Roeder GS, Sung P. Hed1 regulates Rad51-mediated recombination via a novel mechanism., 2008, 22(6): 786–795.
[98] Busygina V, Saro D, Williams G, Leung WK, Say AF, Sehorn MG, Sung P, Tsubouchi H. Novel attributes of Hed1 affect dynamics and activity of the Rad51 presynaptic filament during meiotic recombination.2012, 287(2): 1566–1575.
[99] Vignard J, Siwiec T, Chelysheva L, Vrielynck N, Gonord F, Armstrong SJ, Schlögelhofer P, Mercier R. The interplay of RecA-related proteins and the MND1-HOP2 complex during meiosis in., 2007, 3(10): 1894–1906.
[100] Zhao WX, Sung P. Significance of ligand interactions involving Hop2-Mnd1 and the RAD51 and DMC1 recombinases in homologous DNA repair and XX ovarian dysgenesis., 2015, 43(8): 4055–4066.
[101] Hayase A, Takagi M, Miyazaki T, Oshiumi H, Shinohara M, Shinohara A. A protein complex containing Mei5 and Sae3 promotes the assembly of the meiosis-specific RecA homolog Dmc1.2004, 119(7): 927–940.
[102] Tsubouchi H, Roeder GS. The budding yeast mei5 and sae3 proteins act together with dmc1 during meiotic recombination., 2004, 168(3): 1219–1230.
[103] Reitz D, Grubb J, Bishop DK. A mutant form of Dmc1 that bypasses the requirement for accessory protein Mei5-Sae3 reveals independent activities of Mei5-Sae3 and Rad51 in Dmc1 filament stability.2019, 15(12): e1008217.
[104] Ferrari SR, Grubb J, Bishop DK. The Mei5-Sae3 protein complex mediates Dmc1 activity in., 2009, 284(18): 11766–11770.
[105] Petukhova GV, Pezza RJ, Vanevski F, Ploquin M, Masson JY, Camerini-Otero RD. The Hop2 and Mnd1 proteins act in concert with Rad51 and Dmc1 in meiotic recombination.2005, 12(5): 449–453.
[106] Tsubouchi H, Argunhan B, Ito K, Takahashi M, Iwasaki H. Two auxiliary factors promote Dmc1-driven DNA strand exchange via stepwise mechanisms., 2020, 117(22): 12062–12070.
[107] Murayama Y, Kurokawa Y, Tsutsui Y, Iwasaki H. Dual regulation of Dmc1-driven DNA strand exchange by Swi5-Sfr1 activation and Rad22 inhibition., 2013, 27(21): 2299–2304.
[108] Chen HC, He CP, Wang CY, Wang XP, Ruan FY, Yan JJ, Yin P, Wang YX, Yan SP. RAD51 supports DMC1 by inhibiting the SMC5/6 complex during meiosis., 2021, 33(8): 2869–2882.
[109] Hunter N. Meiotic recombination: the essence of heredity., 2015, 7(12): a016618.
[110] Woglar A, Villeneuve AM. Dynamic architecture of DNA repair complexes and the synaptonemal complex at sites of meiotic recombination., 2018, 173(7): 1678–1691.e1616.
[111] Villeneuve AM, Hillers KJ. Whence meiosis?, 2001, 106(6): 647–650.
[112] Gerton JL, Hawley RS. Homologous chromosome interactions in meiosis: diversity amidst conservation., 2005, 6(6): 477–487.
[113] An YR, Manuguri SS, Malmström J. Atomic force microscopy of proteins.2020, 2073: 247–285.
[114] Peng YH, Qiao HY. The application of single-cell RNA sequencing in mammalian meiosis studies.2021, 9: 673642.
[115] Prusicki MA, Keizer EM, van Rosmalen RP, Komaki S, Seifert F, Müller K, Wijnker E, Fleck C, Schnittger A. Live cell imaging of meiosis in., 2019, 8: e42834.
Recent advances in functional conservation and divergence of recombinase RAD51 and DMC1
Yuxuan Guo1, Shunping Yan2, Yingxiang Wang1
Meiosis is a specialized cell division that occurs in reproductive cells during sexual reproduction. It contains once DNA replication following nucleus division twice, thus producing haploid gametes. Fusion of male and female gametes restores genome to the diploid level, which not only ensures the genome stability between generations during sexual reproduction, but also leads to genetic diversity among offspring. Meiosis homologous recombination (HR) is one of the crucial events during meiotic prophase I, and it not only ensures thesubsequently faithful segregation of homologous chromosomes (homologs), but also exchanges genetic information between homologs with greatly increasing the genetic diversity of progeny. RAD51 (RADiation sensitive 51)and DMC1 (disruption Meiotic cDNA 1)are essential recombinases for the HR process, and have certain commonalities and differences. In this review, we summarize and compare the conserved and differentiated features of RAD51 and DMC1 in terms of origin, evolution, structure, and function, we also provide an outlook on future research directions to further understand and study the molecular mechanisms in regulation of meiotic recombination.
meiosis; homologous recombination; RAD51; DMC1
2022-01-17;
2022-03-31;
2022-04-12
国家自然科学基金项目(编号:31925005)资助[Supported by the National Natural Science Foundation of China (No. 31925005)]
郭雨萱,在读博士研究生,专业方向:分子细胞生物学。E-mail: 21110700065@m.fudan.edu.cn
王应祥,博士,研究员,研究方向:植物减数分裂的分子机制,E-mail: yx_wang@fudan.edu.cn
10.16288/j.yczz.22-016
(责任编委: 史庆华)