杜仲LBD基因家族全基因组鉴定与进化表达分析
2022-05-24陈玉龙吴耀松任闪闪董诚明
刘 俊,李 龙,陈玉龙,吴耀松,刘 燕,任闪闪,董诚明
杜仲基因家族全基因组鉴定与进化表达分析
刘 俊1, 4, 5,李 龙3,陈玉龙1,吴耀松1,刘 燕1,任闪闪1,董诚明2
1.河南中医药大学 中医药科学院,河南省中医方证信号传导重点实验室,河南 郑州 450046 河南中医药大学 河南省道地药材生态种植工程技术研究中心,河南 郑州 450046 西北农林科技大学林学院,陕西 杨凌 712100 国际竹藤中心 国家林业和草原局竹藤科学与技术重点开放实验室,北京 100102 国际竹藤中心 安徽太平试验中心,安徽 黄山 245716
通过对杜仲基因家族进行全面鉴定,以期为基因功能深入研究提供依据。以杜仲基因组数据库为基础,利用生物信息学方法,对杜仲基因家族进行全面鉴定和表达模式分析。从杜仲基因组中共鉴定到19个,分为Class I与Class II 2大类,进一步划分为6个亚家族(Ia、Ib、Ic、Id与IIa、IIb)。理化性质分析显示:EuLBDs编码152~293个氨基酸,理论等电点分布于4.62~9.91,相对分子质量区域为17 420~31 820,均为亲水性蛋白,以α-螺旋和不规则卷曲为主,亚细胞定位于细胞核中。EuLBD基因家族含有类锌指、亮氨酸拉链和甘氨酸-丙氨酸-丝氨酸(GAS)保守结构域。表达模式分析显示,参与杜仲叶片发育,随着叶片发育,水平逐渐降低,基因表达不受叶片胶含量的影响。从杜仲基因组水平对基因家族进行了全面鉴定和生物信息学分析,为进一步研究杜仲基因功能奠定基础。
杜仲;LBD基因家族;系统进化;基因表达;
杜仲Oliver为杜仲科杜仲属落叶乔木,是中国特有的单科、单属、单种的名贵中药材,集“经济、生态、社会效益于一体[1],为我国二级保护植物。杜仲主要以树皮入药,具有抗疲劳、抗肿瘤、预防太空骨质疏松,还具有利胆止血、抗菌、利尿、免疫镇静等作用[2]。LBD蛋白(lateral organ boundaries domain/ASYMMETRIC LEAVES2- Like)是植物特有的一类转录因子[3-6],-末端含有由100个氨基酸残基3个基序(C-block、GAS-block和leu zipper coiled coil)组成的保守LOB结构域。C-block由4个半胱氨酸残基(CX2CX6CX3C)组成,主要参与DNA与顺式作用元件结合,GAS-block由49个氨基酸残基组成,以FX2VH序列开始,DP(V/I)序列结束[7],Leu zipper coiled coil包含1个“LX6LX3LX6C”序列的亮氨酸拉链结构,主要参与蛋白质二聚化过程[8]。基因家族分为Class I和Class II 2大类,研究报道显示,Class I类基因主要在侧生器官的原基边缘表达,调控根、叶、花和胚胎生长发育[9];Class II类基因主要参与植物花青素的合成以及氮代谢调控[10-17]。
自第一个基因从拟南芥中发现以后,其他物种LBD蛋白也相继被报道。研究显示,LBD转录因子参与植物侧生器官发育、激素调节、氮素代谢等多种生物学过程[5,18]。在拟南芥中,1(36)、4()和5(12)参与分生组织侧枝器官发育[8,19-20],29(16)、18(20)和16(18)主要在侧根中表达[21-22];18和33形成异源二聚体,激活下游基因转录[23],促使根部侧生分生组织细胞进入细胞周期,影响拟南芥侧根发育[24-25]。41在拟南芥叶片发育中发挥重要作用[20],2主要在幼嫩花器官近轴面特异表达[6,26],调控花器官发育[27]。基因特异性调控小麦组织发育,Class Ib亚家族主要参与盐胁迫响应[28]。丹参23异源转化拟南芥,导致转基因植株花青素和总酚酸含量降低,干扰植株叶中黄酮含量升高,根中黄酮含量降低;16导致转基因拟南芥花青素和总酚酸含量升高,由此说明,丹参基因参与花青素和总酚酸代谢调控[5,29]。
随着分子生物学的发展,基因已在多个物种中进行了研究(表1),然而杜仲基因家族还未报道。本研究以杜仲基因组数据[30]为基础,从基因组水平对基因家族进行全面鉴定、理化性质分析、进化关系、保守基序以及表达模式分析,以期为进一步探索杜仲基因功能奠定基础。
1 材料
杜仲材料种植于西北农林科技大学苗圃(陕西杨凌),经西北农林科技大学李龙博士鉴定为杜仲Oliver。取生长正常,长势一致的2年生“秦仲1号”“Qinzhong 1”杜仲幼苗的叶芽(茎尖)、生长叶(3 cm 长叶片)、幼叶(完全展开的新叶)、老叶(完全展开60 d叶片);采集高产胶杜仲品种“秦仲2号”“Qinzhong 2”和低产胶杜仲品种“小叶”“Xiaoye”成熟叶片,设置3个生物学重复,经液氮处理后冻存于−80 ℃ 冰箱,用于RNA提取。无菌水、乙醇、TRIzol、液氮、反转录试剂盒、qRT-PCR SYBR试剂盒等。

表1 不同物种LBD基因家族分布
2 方法
2.1 杜仲LBD基因家族鉴定及理化性质分析
从杜仲基因组数据库Genome Warehouse(https://bigd.big.ac.cn/gwh/Assembly/13/show)中下载LBD蛋白候选序列,利用NCBI在线分析软件[Conserved Domain Search Service(CD Search)]分析序列的结构域,保留含有完整LOB结构域的蛋白序列。通过在线软件ProtParam(http://web.expasy.org/protparam/)分析蛋白的理化性质,使用Plant-mPLoc(http://www.csbio.sjtu.edu.cn/bioinf/ plant-multi/)预测EuLBDs蛋白的亚细胞定位,利用在线工具ExPASY(https://www.expasy.org/tools)预测EuLBDs氨基酸数量、相对分子质量、理论等电点[45],通过Expasy(https://web.expasy.org/ protscale/)软件分析蛋白的亲疏水性,利用SOPMA(https://npsa-prabi.ibcp.fr/cgi-bin/npsa_automat.plpage= npsa_sopma.html)[46]和Phyre 2 (http://www.sbg.bio.ic.ac.uk/phyre2/html/page.cgi?id=index)[47]软件分别预测蛋白的二级结构和三级结构。
2.2 杜仲LBDs蛋白系统发育树构建
通过Clustal X1.83[48]软件对杜仲、水稻、拟南芥和毛果杨LBDs氨基酸序列进行多序列比对,利用MEGA 6.0的邻接法(neighbor-joining,NJ)构建系统发育树,重复次数设置为1000次,其他参数为默认值[49]。
2.3 保守基序及启动子分析
为了分析EuLBDs蛋白的保守基序,通过MEME(http://meme-suite.org/)在线软件对基因家族成员进行基序分析(参数为any number of Repetitions(anr),maximum number of Motifs=10,minimum width≥6,and maximum width≤50)[50]。为了解析启动子区域顺式作用元件,对基因ATG上游2000 bp的序列进行查找分离,利用Plant CARE(http://bioinformatics.psb.ugent.be/webtools/plantcare/htmL/)软件进行启动子分析[51]。
2.4 杜仲EuLBD基因家族表达模式分析
从NCBI的Short Read Arshive(SRA)数据库中下载杜仲叶片不同发育时期(叶芽、初生叶、幼叶、老叶)[52]及不同胶含量(低含量、高含量)[53]转录组数据,基因在不同组织中的相对表达丰度用FPKM值表示,对该数值取对数(Log2)进行统计分析,通过MeV4-9-0工具绘制基因表达图谱,表达量最高的用红色方框表示,表达量最低的用绿色方框表示。
2.5 杜仲LBDs蛋白互作网络预测
利用STRING软件(https://string-db.org/)上传EuLBDs蛋白序列,选择拟南芥数据库进行序列比对,根据已知拟南芥蛋白互作关系,通过Blast比对杜仲同源蛋白,通过Cytoscape 3.7.0软件进一步分析,对EuLBD基因家族蛋白互作信息进行评估和预测[54]。
3 结果与分析
3.1 杜仲LBD基因家族鉴定及理化性质分析
利用Genome Warehouse数据库,通过生物信息学方法,从杜仲基因组中鉴定到19个基因(表2),通过Pfam和NCBI的Conserved Domain Search在线分析软件对EuLBDs蛋白结构域进行检测。结果显示,19个EuLBDs蛋白均含有保守LOB特征结构域,分别命名为EuLBD1~EuLBD19。通过ExPASy工具,对EuLBD家族成员进行蛋白质理化性质分析,最长蛋白EuLBD12编码293个氨基酸,最短蛋白EuLBD3编码152个氨基酸,相对分子质量分布区域为17 420~31 820,等电点范围是4.62(EuLBD8)~9.91(EuLBD9)。亚细胞定位预测结果显示,EuLBDs均定位在细胞核中(表2)。
3.2 杜仲LBD转录因子亲疏水性分析
利用ProtScale在线软件分析杜仲LBDs转录因子的亲疏水性。结果如图1所示,19条EuLBDs蛋白均存在明确的亲疏水区域,亲水区域多于疏水区域,表明EuLBDs多肽链均为亲水性蛋白。EuLBD1的最低值为−2.133,在218位,最高值为2.011,位于第47氨基酸残基处;EuLBD2在145位处于最低值为−3.489,于88位氨基酸残基处达到最高值是1.922,其他蛋白的最低值、最高值及具体位置见图1。
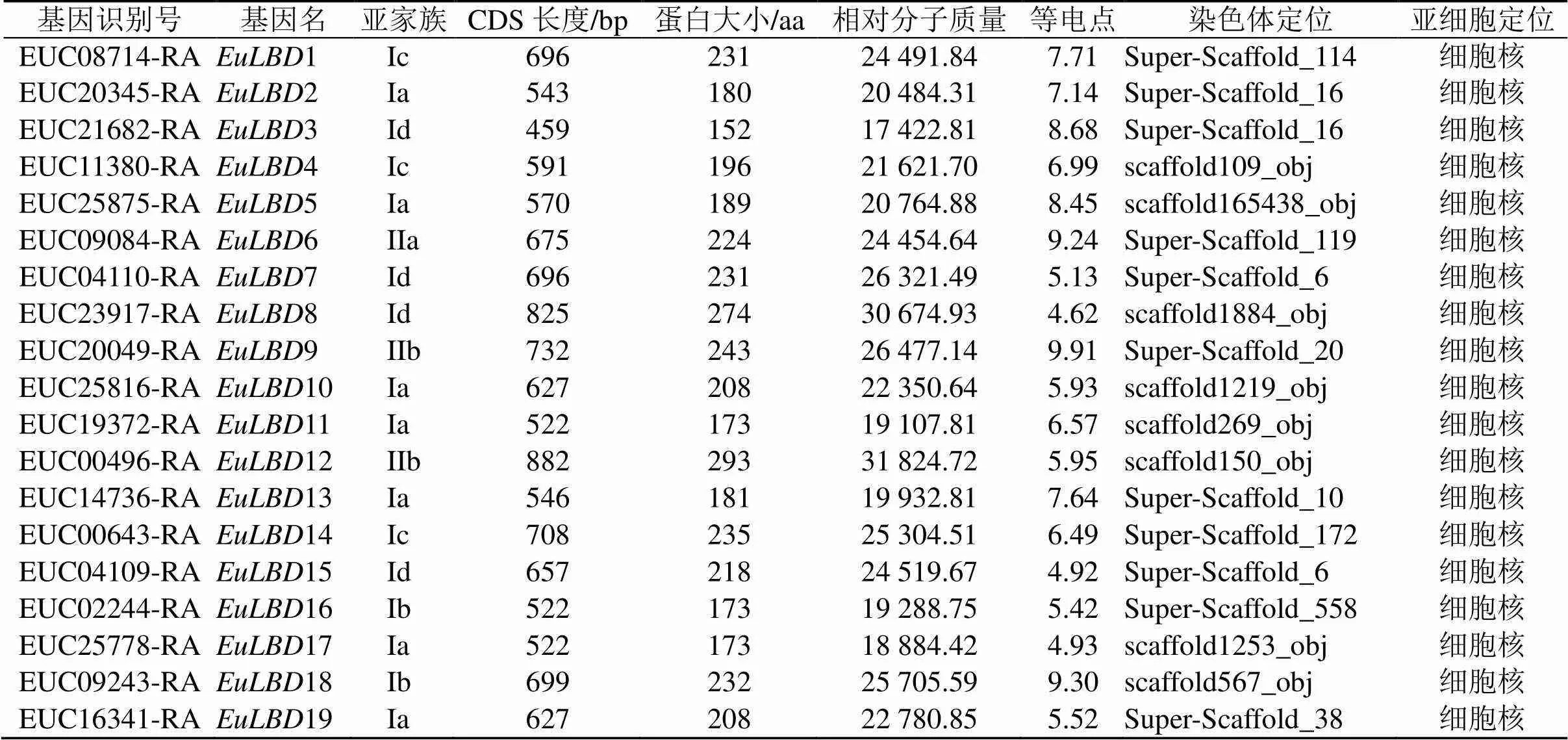
表2 杜仲LBD基因家族基本信息
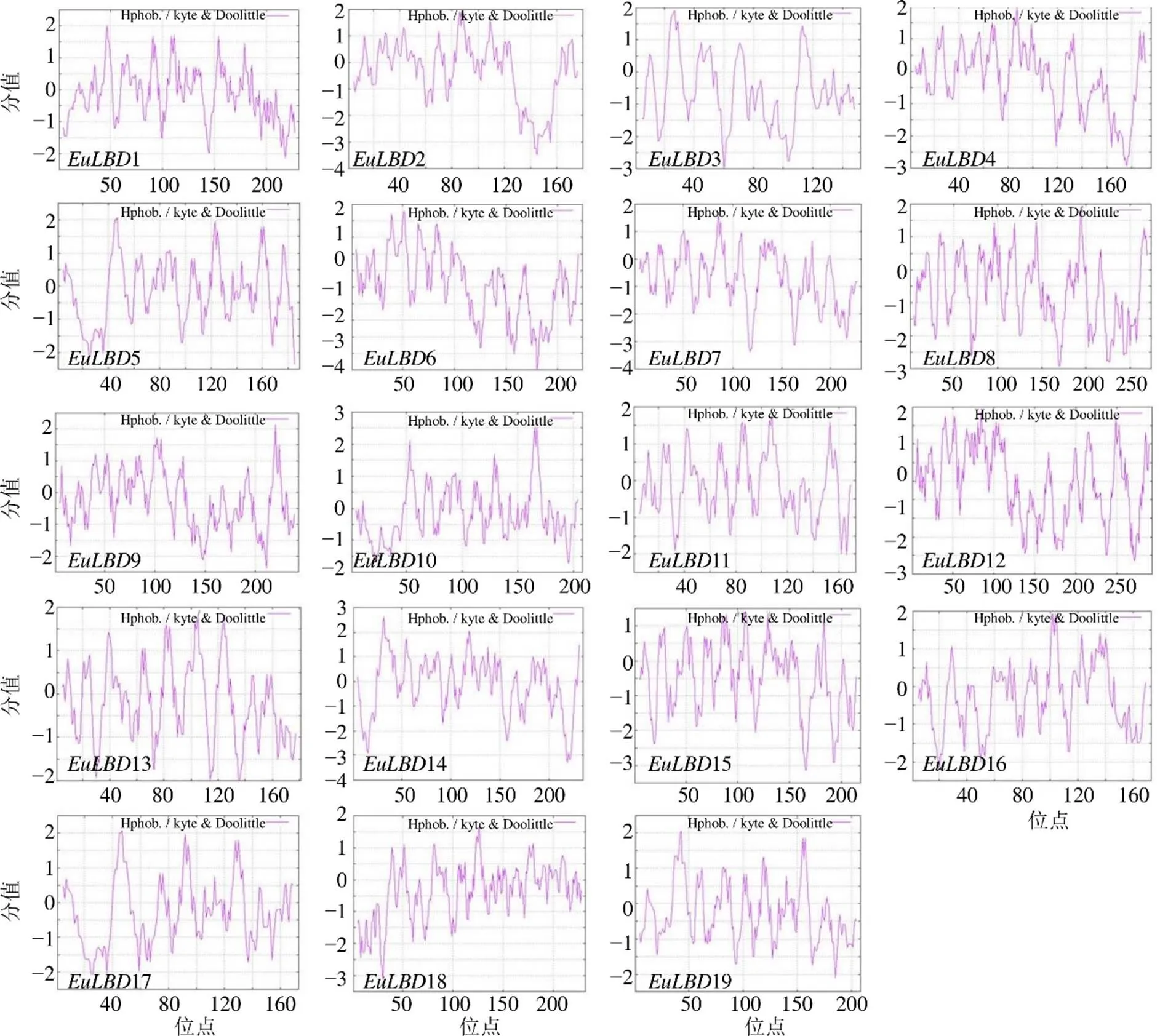
图1 杜仲LBDs转录因子亲疏水性预测
3.3 杜仲LBDs蛋白结构分析
EuLBDs蛋白二级结构分析显示,α-螺旋、无规则卷曲、延伸链和β-转角在杜仲LBD蛋白家族中均有分布。α-螺旋在DNA结合基序中发挥重要作用,无规则卷曲易受侧链相互作用,构成活性部位和功能部位。杜仲LBDs蛋白二级结构有2种类型,其中α螺旋>无规则卷曲>延伸链>β转角的数量最多,有11个基因,所占比例为57.89%;8个基因二级结构顺序为无规则卷曲>α螺旋>延伸链>β转角,比例是42.11%,具体情况见表3。
采用Phyre2在线软件进行杜仲LBDs蛋白高级结构同源建模预测,结果如图2所示,除EuLBD17三级结构出现特异性外,其余蛋白三级结构非常相似,均由α-螺旋、β-折叠和随机卷曲等组成,19个EuLBDs中随机卷曲所占比例最大,空间结构不同,决定EuLBDs蛋白功能的差异。
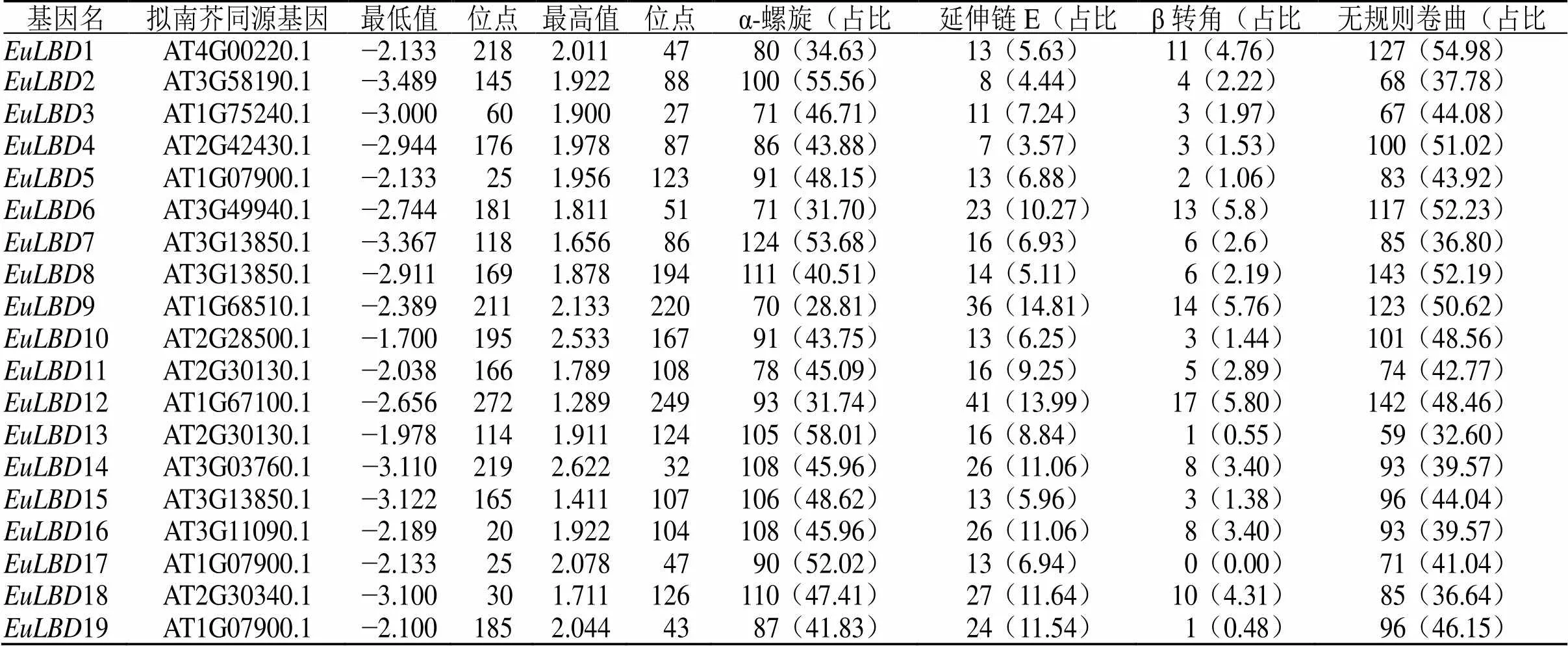
表3 杜仲LBD基因家族二级结构分析
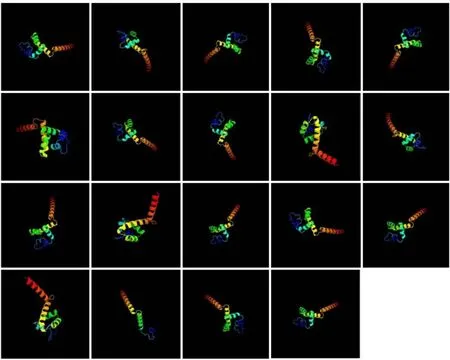
图2 杜仲LBDs蛋白三级结构预测
3.4 杜仲LBDs蛋白系统进化分析
利用MEGA 6.0分析软件,对19个杜仲EuLBDs,35个水稻OsLBDs[31],43个拟南芥AtLBDs[8]和57个毛果杨PtLBDs[55]蛋白进行1000次重复搜索,构建系统进化树。结果如图3所示,154个LBDs蛋白分为Class I和Class II 2个大亚家族,其中Class I亚家族进一步划分为:Ia、Ib、Ic、Id和Ie小亚家族;Class II亚家族划分为:IIa和IIb。154个LBDs蛋白共分为7个小亚家族(如图3所示),Class Ia亚家族所含LBDs成员数量最多,共有37个LBDs蛋白,分别包含3个EuLBDs,13个AtLBDs,8个OsLBDs和13个PtLBDs蛋白;其次是Class Id亚家族,由31个LBDs蛋白组成,Class Ib亚家族含有28个LBDs蛋白;29个LBDs蛋白属于Class Ic亚家族,Class Ie亚家族所含蛋白数量最少,仅有3个AtLBDs;Class IIa和Class IIb亚家族分别含有14、13个LBDs。EuLBDs在Class Ic亚家族中的分布最多,有7个蛋白,所占比例为36.8%,除Class Ic亚家族外,EuLBDs在7个亚家族中的蛋白数量最少,这可能与EuLBDs在4个物种中数量最少有关。
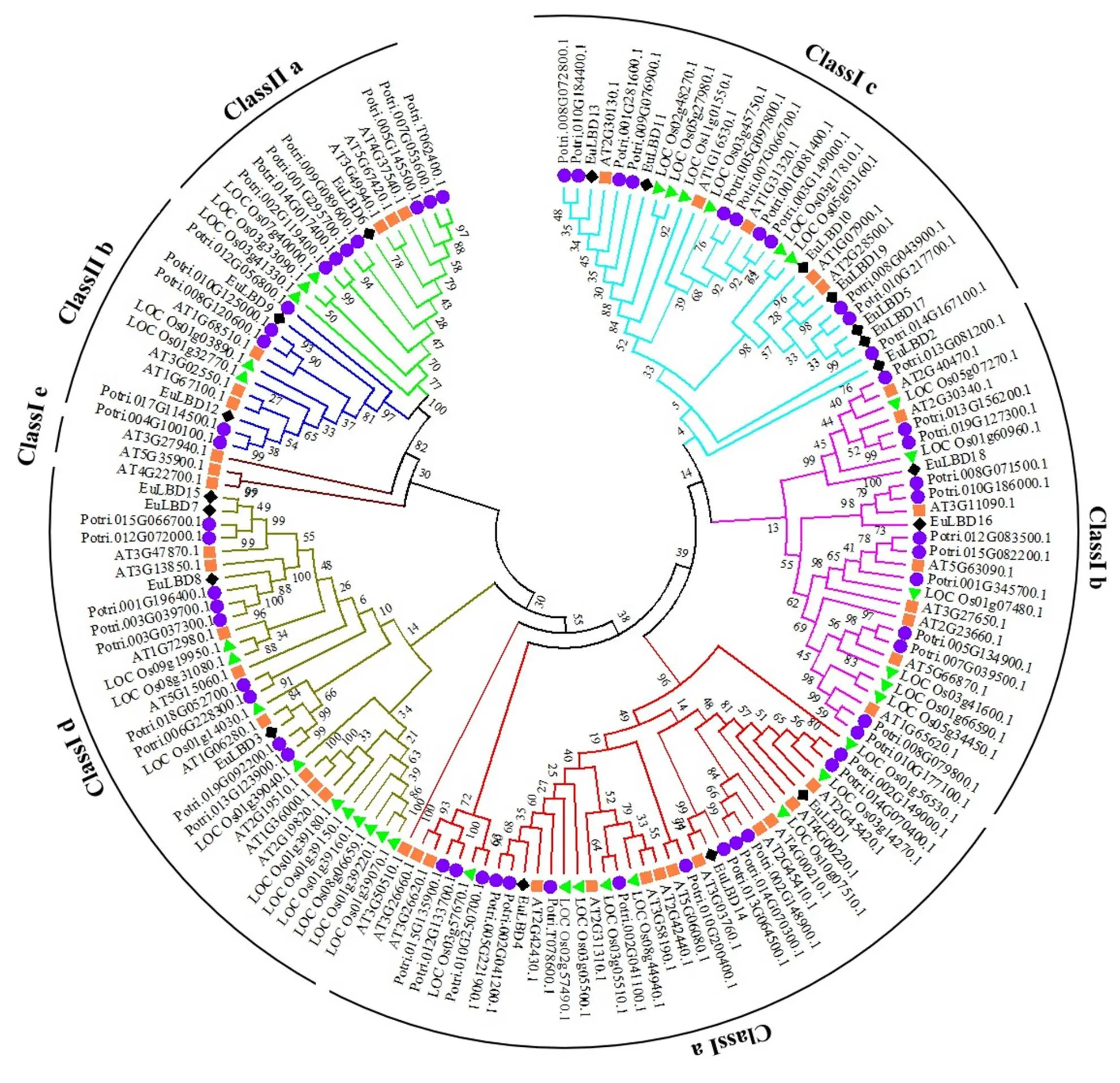
利用MEGA 6.0的邻接法构建系统进化树,不同物种的LBDs蛋白用不同颜色点标记,红色方形代表拟南芥,绿色三角形代表水稻,紫色圆形代表毛果杨,杜仲LBDs蛋白由黑色菱形标记
3.5 杜仲LBD转录因子基序分析
为了解析基因结构多样性和进化关系,利用MEME在线分析软件,对19条EuLBDs蛋白保守结构域进行分析。结果如图4所示,EuLBDs蛋白由1~10个Motifs组成(表4),均含有保守LOB结构域(Motif 1和Motif 2),表明19个EuLBDs均属于LBD家族成员。EuLBDs蛋白(除EuLBD5外)均含有Motif 3,有些Motifs只存在于特定亚家族,Motif 5仅在Class Ib亚家族中存在,Motif 4、Motif 7和Motif 10只存在于Class Ic亚家族,只有Class IIa亚家族含有Motif 9,这可能与不同亚家族功能不同相关。
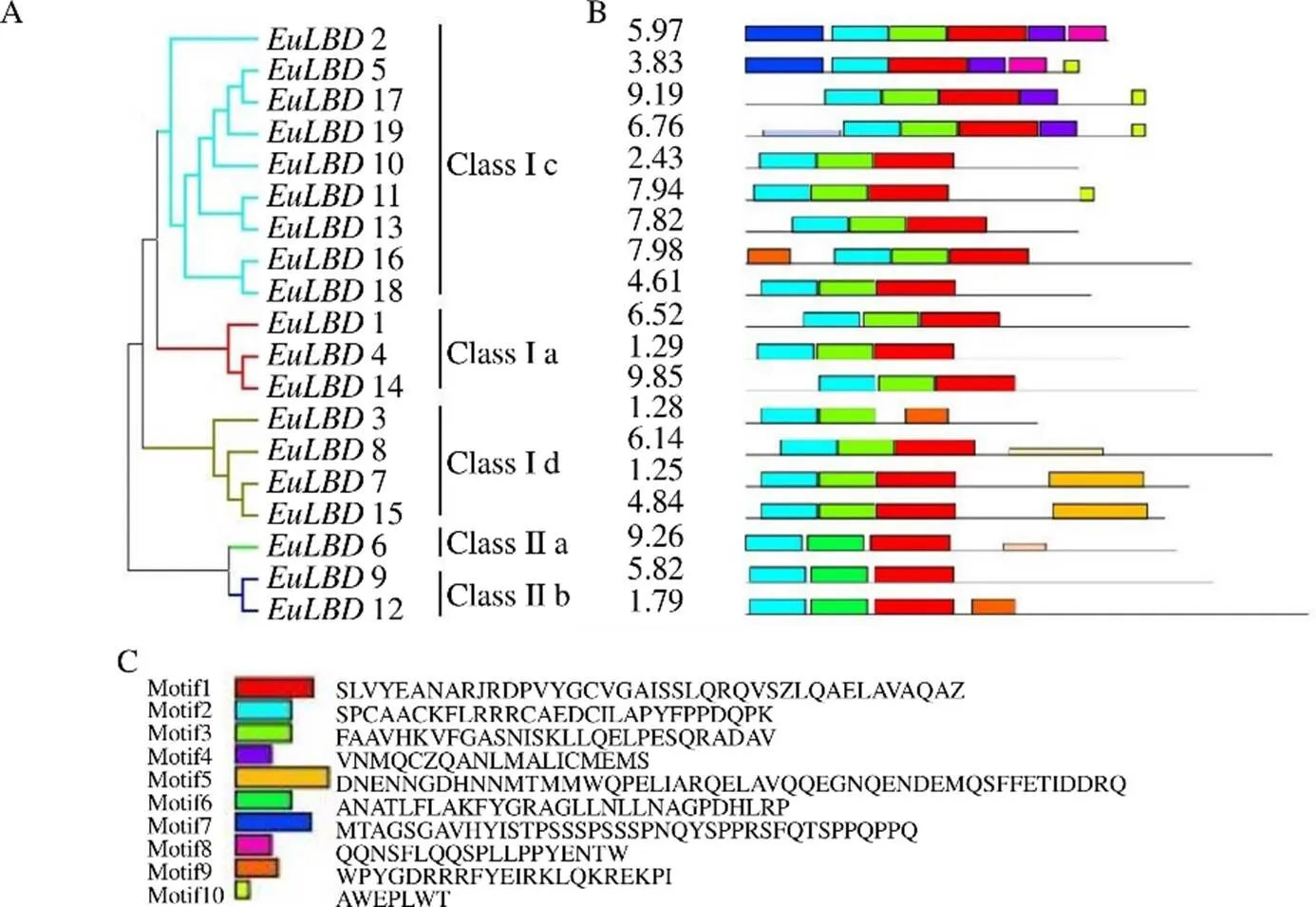
A-利用MEGA 6.0的邻接法构建EuLBD基因家族系统进化树B-EuLBDs基因结构分析 C-保守基序氨基酸分布

表4 杜仲LBDs蛋白保守基序分布
3.6 杜仲LBDs蛋白序列保守性分析
利用DNAMAN7.0工具对19个EuLBDs蛋白保守结构域进行比对,结果如图5所示,EuLBDs蛋白N端均含有1个由15个氨基酸残基组成的CX2CX6CX3C保守基序,暗示其可能与下游基因顺式元件的结合有关,Class II类蛋白C端含有赖氨酸组成的类似亮氨酸拉链(LX6LX3LX6L)二级结构(图5),参与转录因子二聚化。Class Ia、Ib和Ic亚家族含有1个GAS模块(GAS-block),该模块C端含有保守的脯氨酸(P)和甘氨酸(G),脯氨酸在LBD基因家族生物学功能中发挥重要作用[56]。

图5 杜仲LBDs蛋白保守结构域序列比对
3.7 杜仲LBDs启动子顺式作用元件分析
为了探索基因的功能和表达调控模式,利用PlantCARE在线软件对起始密码子(ATG)上游2000 bp进行顺式作用元件分析,结果如图6所示,启动子不仅含有基本顺式作用元件,还存在3种类型元件:(1)生长发育调控元件,如赤霉素响应元件ABRE;生长素响应元件AuxRR-core;水杨酸响应元件CGTCA-motif;(2)胁迫响应调控元件,如干旱胁迫响应元件MBS、低温响应元件LTR和厌氧胁迫相关元件ARE;(3)光响应元件,如GT1-motif、GATA-motif、Sp1、G-Box和ACE等,推测可能在杜仲生长发育、逆境胁迫以及光周期调控中发挥重要作用。基因中光响应元件数量最多,共有232个,包含55个Box 4,34个G-box元件,暗示s基因的转录可能受光周期调控。基因启动子区域含有48个ABRE元件,表明可能参与ABA调控。
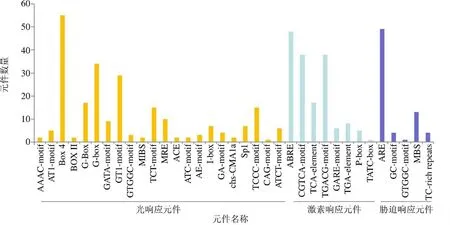
图6 杜仲LBDs启动子顺式作用元件分析
3.8 杜仲LBD基因家族表达模式分析
为了探索基因在杜仲叶片不同发育时期及杜仲胶形成中的功能,根据转录组数据,检测基因表达模式(图中显示基因编号与文中基因命名一致)。结果如图7和8所示,不同基因在杜仲叶片不同发育时期及杜仲胶含量中表达丰度存在显著差异,大部分基因在杜仲叶片发育中表达丰度较低,其中有4个基因(3、9、13和15)在杜仲叶片中不表达,表明3、9、13和15在杜仲叶片发育中不发挥作用。基因随着杜仲叶片发育,表达水平逐渐降低,12FPKM值在叶芽中是357.0,幼叶中为62.0,成熟叶中是2.2,老叶中仅为0.2,表明大部分基因参与杜仲叶片发育,特别在叶片发育的早期阶段。

T1-1~T1-3-叶芽 T2-1~T2-3-初生叶 T3-1~T3-3-幼叶 T4-1~T4-3-老叶,热图右侧为色标,绿色表示低表达丰度,红色表示高转录水平
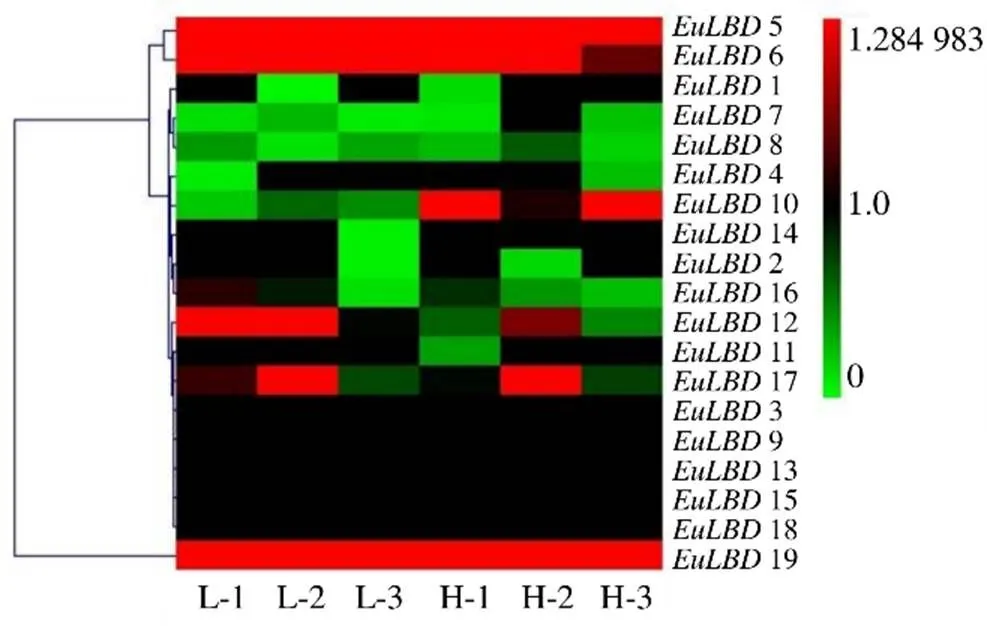
L-1~L-3:低胶含量叶片;H-1~H-3:高胶含量叶片。热图右侧为色标,绿色表示低表达丰度,红色表示高转录水平
为了检测在杜仲胶形成中发挥的作用,利用不同胶含量的转录组数据,检测的表达水平。结果如图8所示,基因在杜仲不同胶含量中的表达水平均较低,几乎检测不到,转录水平不随胶含量的高低而改变,由此说明在杜仲胶形成过程中发挥的作用较小,这可能与在杜仲成熟叶中表达量较低有关。
3.9 杜仲LBDs蛋白互作网络预测
表达模式分析显示,6和19在杜仲叶片不同发育阶段和杜仲胶形成中表达量较高,表明6和19在杜仲叶片发育和胶形成过程中发挥重要作用。为了检测EuLBD6和EuLBD19与其他蛋白的互作关系,利用STRING数据库,预测EuLBD6和EuLBD19互作蛋白。结果如图9-A所示,EuLBD6可以与10个蛋白发生相互作用,其中8个属于EuLBD家族,一个是MYB-like转录因子,另一个为NLP7蛋白;EuLBD19可与5个蛋白产生互作关系(图9-B),其中3个为EuLBD蛋白,一个Auxin-like转录因子,一个MYB-like蛋白,暗示EuLBD6和EuLBD19可能参与杜仲生长素信号调控。
为了探索基因家族潜在调控关系,构建了EuLBD家族共表达调控网络。结果如图10所示,调控网络中共有14个节点(代表EuLBDs蛋白)和19条边(代表蛋白质之间的相互作用),表明19个EuLBDs中有14个蛋白表现出共表达相关性,其中EuLBD12互作蛋白数量最多,可与8个EuLBDs蛋白发生互作关系,暗示EuLBD12可能是EuLBD基因家族的核心蛋白,EuLBD6、EuLBD4和EuLBD1,分别可以与4个EuLBDs蛋白互作,其余EuLBDs蛋白互作关系较少。
4 讨论
杜仲全基因组测序完成以后,已有多个基因家族进行了分析报道,例如[57]、[58]、[59]、[60]、[61]、[62]基因家族等。LBD转录因子是植物特有的一类转录因子,在植物侧生器官发育、激素响应、逆境胁迫、花青素和氮素代谢等途径中发挥了重要作用[63]。本研究以杜仲基因组数据为基础,通过全基因组查找和生物信息学分析,鉴定出19个含有完整LOB结构域的LBD转录因子,基因数量均少于二穗短柄草(28个)[34]、拟南芥(43个)[8]、水稻(35个)[31]、玉米(44个)[32]、杨树(57个)[6]、番茄(46个)[4]、辣椒(56个)[37]、烟草(45个)[40]、葡萄(98个)[42],杜仲基因组大小是1.18 Gb[64],是水稻(441 Mb)[65]的2倍多,为二穗短柄草(300 Mb)[66]的3倍多,是拟南芥(164 Mb)的7倍,为番茄(850 Mb)的1倍多,然而基因数量与基因组大小不成正比,这可能与杜仲和双子叶共同祖先发生分化后,进行全基因组复制(WGD)及基因丢失事件有关。
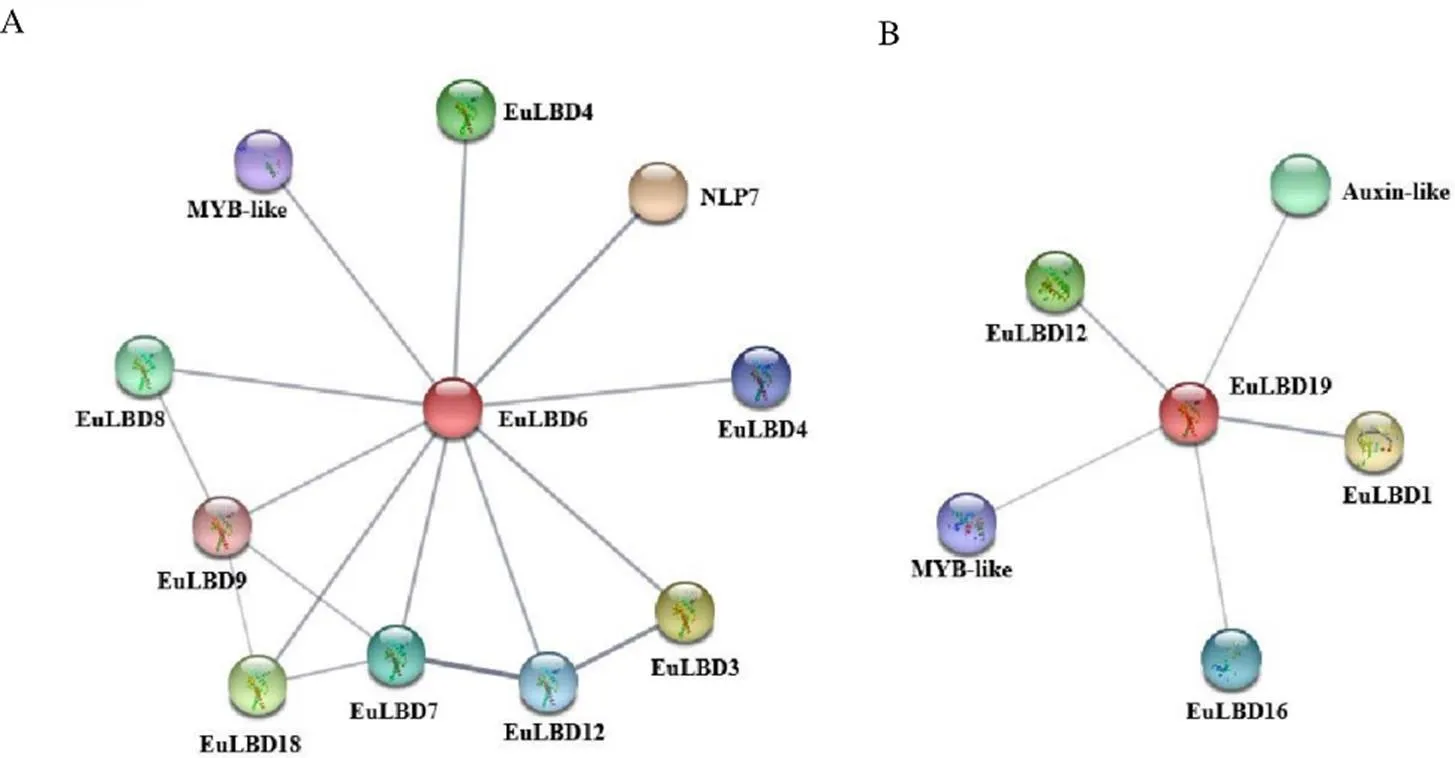
图9 EuLBD6 (A) 和EuLBD19 (B) 蛋白相互作用网络
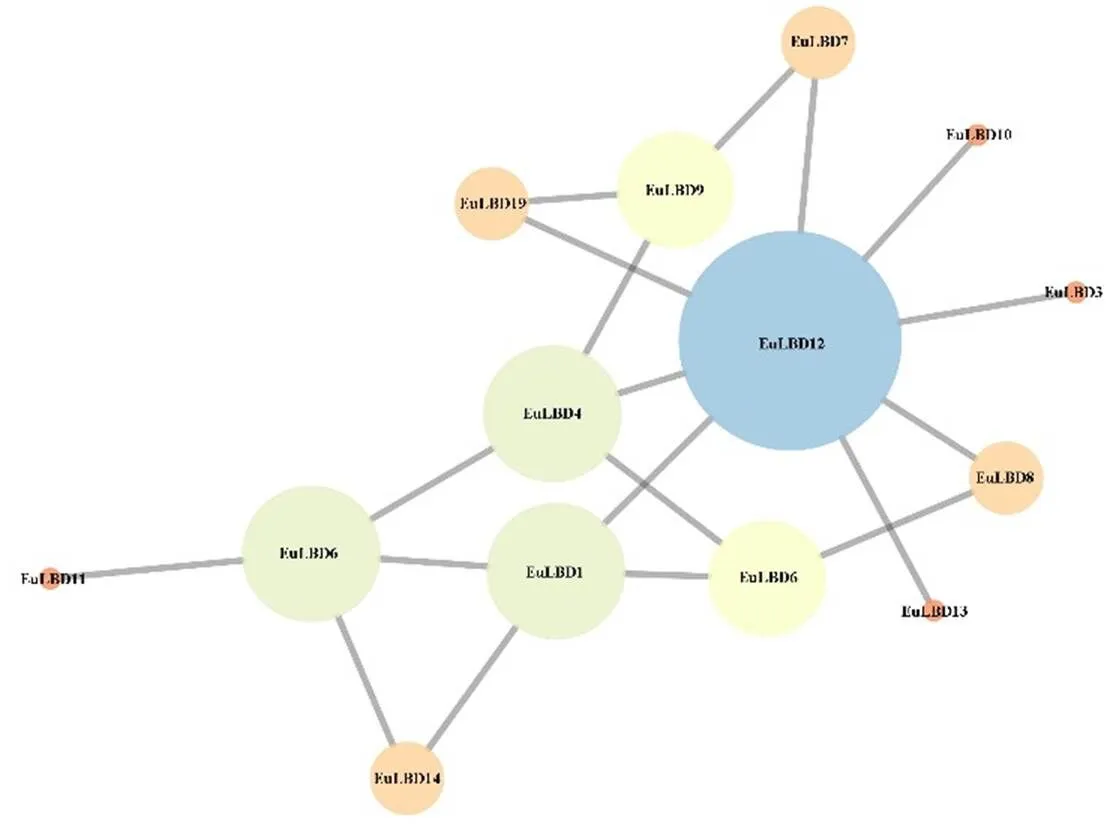
图10 EuLBD基因家族蛋白互作网络
拟南芥、水稻、杜仲和毛果杨LBD蛋白聚类分析结果显示,19个EuLBDs分为2个大亚家族,分别是Class I和Class II,进一步划分为7个小亚家族(Ia、Ib、Ic、Id、Ie、IIa和IIb)(图3)。Class I大亚家族含有16个EuLBDs蛋白,Ia亚家族中所含EuLBDs蛋白数量最多,有7个EuLBDs,Ie亚家族不含EuLBDs。Class II大亚家族含有3个EuLBDs(分别是EuLBD6、EuLBD9、EuLBD12)。系统进化结果显示,杜仲EuLBD5、EuLBD6、EuLBD11和EuLBD15与拟南芥AtLBD37(AtASL39)、AtLBD38(AtASL40)和AtLBD39(AtASL41)聚为一支,属于Class II大亚家族。37、38和39参与拟南芥花青素合成和氮代谢[5,13,23],推测5、6、11和15可能具有功能相似性。14是6的同源基因,6(2)参与拟南芥花序发育调控[67],暗示14在杜仲花序发育中可能发挥重要作用。16(18)、17(15)和9(16)与杜仲1和13互为同源基因,16(18)、17(15)和9(16)诱导愈伤组织形成,参与植物的再生过程[68-69],表明1和13在杜仲愈伤组织形成中可能具有相似的功能。
基因结构决定基因功能,EuLBDs蛋白结构分析发现,同一亚家族含有相似的结构域,LX6LX3LX6L结构域(Motif 1)存在于所有EuLBDs转录因子中,表明EuLBDs转录因子结构具有保守性。不同EuLBDs转录因子结构存在差异,除EuLBD4和EuLBD11不含CX2CX6CX3C结构域(Motif 2)外,其他EuLBDs蛋白均含有Motif 2,Class I大亚家族基因结构较为复杂,不仅含有保守的LOB结构域,还含有特异结构域,这可能与基因功能的多样性有关。
研究报道,水稻、小麦、拟南芥、毛果杨、番茄、辣椒、巨桉等物种LBD蛋白参与植物叶片发育、侧生根形成、次生木质部和韧皮部发育、细胞分裂素、生长素以及非生物胁迫等生物学过程[15, 24, 43, 70-71]。基因表达与其功能特征密切相关,利用转录组数据,分析基因在杜仲叶片不同发育时期(图7)以及杜仲胶含量中的表达模式(图8)。结果显示,基因在杜仲叶芽中转录水平最高,随着叶片发育,表达水平逐渐减低,推测参与杜仲叶片发育,特别是叶芽形成期。6、12和16在叶片4个发育阶段表达量均较高,表明6、12和16在杜仲叶片生长发育中发挥重要作用,这为进一步探索基因在杜仲叶片发育中的功能提供了重要的候选基因。4、9、13、15和18转录水平较低,在各发育阶段表达丰度几乎为0,暗示4、9、13、15和18在杜仲叶片中发挥的作用较小。6参与拟南芥旗叶形成,超表达6导致转基因植株叶片细小、卷曲[12]。在水稻中,降低37和38表达水平,影响水稻抽穗时间关键基因1的表达,导致水稻延迟抽穗[72]。在巨桉中,37在韧皮部高量表达[6],超表达37导致转基因植株次生木质部增厚,韧皮纤维增多[73],表明LBD基因家族在植物叶片发育、花序形成、次生发育等过程发挥重要作用。
进化分析显示,6是拟南芥38的同源基因,在N/NO3缺失条件下,超表达38强烈抑制花青素合成关键调控因子PAP1和PAP2,以及类黄酮合成过程中花青素特异基因的表达;相反,38突变体在N/NO3充足条件下积累花青素,组成性表达花青素生物合成基因,38抑制多种氮应答基因表达,表明38是花青素生物合成和氮有效性利用的新抑制因子[74]。蛋白互作网络结果显示,EuLBD6不仅可以与LBD蛋白发生相互作用,还可与MYBL2蛋白互作,在拟南芥中mir858a通过翻译抑制花青素生物合成关键负调控因子MYBL2的表达,是拟南芥幼苗花青素生物合成的正调控因子[75],推测6在杜仲花青素合成中可能也发挥重要作用。19与1互为同源基因,1在韧皮部和形成层中表达量最高,显性抑制因子PtaLBD1通过与抑制因子SRDX区域融合,减少杨树直径增长,抑制韧皮部发育,并且高度不规则,表明1在杨树次生木质部生长过程中具有广泛的调控作用[76],暗示19在杜仲次生生长过程中可能也发挥重要作用。
综上所述,本研究以杜仲全基因组数据为背景,对杜仲LBD转录因子家族进行了全面的生物信息学分析,共鉴定出19个基因,分为Class I与Class II 2大类,划分为6个亚家族(Ia、Ib、Ic、Id与IIa、IIb)。理化性质分析显示:EuLBDs编码152~293个氨基酸,理论等电点分布于4.62~9.91,相对分子质量区域为17 420~31 820,均为亲水性蛋白,以α-螺旋和无规则卷曲为主,亚细胞定位于细胞核中。EuLBDs蛋白含有类锌指、亮氨酸拉链和甘氨酸-丙氨酸-丝氨酸(GAS)保守结构域。表达模式分析显示,参与杜仲叶片发育,随着叶片发育,转录水平逐渐降低,基因表达不受叶片胶含量的影响,本研究为深入研究杜仲基因功能奠定了理论基础。
利益冲突 所有作者均声明不存在利益冲突
[1] 程娜.剥皮对杜仲生长期内生理特性及次生代谢物含量的影响 [D].杨凌: 西北农林科技大学, 2009.
[2] Li Z Y, Gu J, Yan J,.Hypertensive cardiac remodeling effects of lignan extracts fromOliv.bark: A famous traditional Chinese medicine [J]., 2013, 41(4): 801-815.
[3] Husbands A, Bell E M, Shuai B,.LATERAL ORGAN BOUNDARIES defines a new family of DNA-binding transcription factors and can interact with specific bHLH proteins [J]., 2007, 35(19): 6663-6671.
[4] 王小非, 刘鑫, 苏玲, 等.番茄LBD基因家族的全基因组序列鉴定及其进化和表达分析 [J].中国农业科学, 2013, 46(12): 2501-2513.
[5] 王美艳.两种LBD转录因子及其DNA结合结构域的表达与纯化 [D].杨凌: 西北农林科技大学, 2013.
[6] 芦强.巨桉、毛果杨、雷蒙德氏棉LBD基因家族的研究 [D].北京: 中国林业科学研究院, 2017.
[7] Lin W C, Shuai B, Springer P S.Thelateral organ boundaries-domain gene asymmetric leaves2 functions in the repression ofgene expression and in adaxial-abaxial patterning [J]., 2003, 15(10): 2241-2252.
[8] Shuai B, Reynaga-Peña C G, Springer P S.The lateral organ boundaries gene defines a novel, plant-specific gene family [J]., 2002, 129(2): 747-761.
[9] 任杨柳.玉米侧生器官分化基因转化拟南芥的功能分析 [D].郑州: 河南农业大学, 2017.
[10] Semiarti E, Ueno Y, Tsukaya H,.Thegene ofregulates formation of a symmetric, establishment of venation and repression of meristem-related homeobox genes in leaves [J]., 2001, 128(10): 1771-1783.
[11] Taramino G, Sauer M, Stauffer J L Jr,.The maize (L.)gene encodes a LOB domain protein that is a key regulator of embryonic seminal and post-embryonic shoot-borne root initiation [J]., 2007, 50(4): 649-659.
[12] Matsumura Y, Iwakawa H, Machida Y,.Characterization of genes in the asymmetric leaves2/lateral organ boundaries (As2/lob) family in, and functional and molecular comparisons between As2 and other family members [J]., 2009, 58(3): 525-537.
[13] Rubin G, Tohge T, Matsuda F,.Members of the LBD family of transcription factors repress anthocyanin synthesis and affect additional nitrogen responses in[J]., 2009, 21(11): 3567-3584.
[14] Albinsky D, Kusano M, Higuchi M,.Metabolomic screening applied to rice FOXlines leads to the identification of a gene-changing nitrogen metabolism [J]., 2010, 3(1): 125-142.
[15] Bell E M, Lin W C, Husbands A Y,.lateral organ boundaries negatively regulates brassinosteroid accumulation to limit growth in organ boundaries [J]., 2012, 109(51): 21146-21151.
[16] Thatcher L F, Powell J J, Aitken E A B,.The lateral organ boundaries domain transcription factor LBD20 functions inwilt susceptibility and jasmonate signaling in[J]., 2012, 160(1): 407-418.
[17] Kim M J, Kim M, Lee M R,.Lateral organ boundaries domain (lbd)10interacts with sidecar pollen/lbd27 to control pollen development in[J]., 2015, 81(5): 794-809.
[18] 汤玮婧.LBD转录因子家族的起源与进化 [D].杨凌: 西北农林科技大学, 2013.
[19] Nakazawa M, Ichikawa T, Ishikawa A,.Activation tagging, a novel tool to dissect the functions of a gene family [J]., 2003, 34(5): 741-750.
[20] Chalfun-Junior A, Franken J, Mes J J,.ASYMMETRIC LEAVES2-LIKE1 gene, a member of the/family, controls proximal-distal patterning inpetals [J]., 2005, 57(4): 559-575.
[21] 张娇娇, 江力, 杨杰, 等.拟南芥抗旱突变体的筛选和鉴定 [J].合肥工业大学学报: 自然科学版, 2012, 35(4): 531-535.
[22] Berckmans B, Vassileva V, Schmid S P C,.Auxin-dependent cell cycle reactivation through transcriptional regulation ofE2Fa by lateral organ boundary proteins [J]., 2011, 23(10): 3671-3683.
[23] 李艳艳.红豆杉韧皮部分化、发育关键基因的克隆及功能分析 [D].北京: 中国林业科学研究院, 2015.
[24] Okushima Y, Overvoorde P J, ARIMA K,.Functional genomic analysis of the auxin response factor gene family members in: Unique and overlapping functions of arf7 and arf19 [J]., 2005, 17(2): 444-463.
[25] Laplaze L, Parizot B, Baker A,.GAL4-GFP enhancer trap lines for genetic manipulation of lateral root development in[J]., 2005, 56(419): 2433-2442.
[26] Lee H W, Kim N Y, Lee D J,.LBD18/ASL20 regulates lateral root formation in combination with LBD16/ASL18 downstream of ARF7 and ARF19 in[J]., 2009, 151(3): 1377-1389.
[27] Lee H W, Kim N Y, Lee D J,.LBD18/ASL20 regulates lateral root formation in combination with LBD16/ASL18 downstream of ARF7 and ARF19 in[J]., 2009, 151(3): 1377-1389.
[28] 孙亭亭.烟草分枝异常类型突变体基因鉴定及相关家族分析[D].北京: 中国农业科学院, 2015.
[29] Iwakawa H, Ueno Y, Semiarti E,.The ASYMMETRIC LEAVES2 gene of, required for formation of a symmetric flat leaf, encodes a member of a novel family of proteins characterized by cysteine repeats and a leucine zipper [J]., 2002, 43(5): 467-478.
[30] 邢光伟.小麦LBD转录因子家族的鉴定、系统进化及表达分析 [D].杨凌: 西北农林科技大学, 2018.
[31] 冯书超.丹参转录因子SmLBD23和SmLBD16的克隆及功能研究 [D].西安: 陕西师范大学, 2018.
[32] Wuyun T N, Wang L, Liu H M,.The hardy rubber tree genome provides insights into the evolution of polyisoprene biosynthesis [J]., 2018, 11(3): 429-442.
[33] Yang Y, Yu X B, Wu P.Comparison and evolution analysis of two rice sub species lateral organ boundaries domain gene family and their evolutionary characterization from[J]., 2006, 39(1): 248-262.
[34] Zhang Y M, Zhang S Z, Zheng C C.Genomewide analysis of lateral organ boundaries domain gene family in[J]., 2014, 93(1): 79-91.
[35] Guo B J, Wang J, Lin S,.A genome-wide analysis of the asymmetric leaves2/lateral organ boundaries (As2/lob) gene family in barley (L.) [J]., 2016, 17(10): 763-774.
[36] Gombos M, Zombori Z, Szécsényi M,.Characterization of the LBD gene family in: A phylogenetic and transcriptional study [J]., 2017, 36(1): 61-79.
[37] Wu J, Liu S Y, Guan X Y,.Genome-wide identification and transcriptional profiling analysis of auxin response-related gene families in cucumber [J]., 2014, 7: 218.
[38] Liu H Z, Cao M X, Chen X L,.Genome-wide analysis of the lateral organ boundaries domain (LBD) gene family in[J]., 2019, 20(21): 5360.
[39] 郑忠凡, 张亚利, 胡灿, 等.辣椒全基因组中LBD转录因子的鉴定与表达分析 [J].园艺学报, 2016, 43(4): 683-694.
[40] Yang T Q, Fang G Y, He H,.Genome-wide identification, evolutionary analysis and expression profiles of lateral organ boundaries domain gene family inand[J]., 2016, 11(8): e0161901.
[41] 刘同金, 张晓雪, 张晓辉, 等.萝卜全基因组中LBD基因家族成员的鉴定与分析 [J].植物遗传资源学报, 2019, 20(1): 168-178.
[42] 孙亭亭, 龚达平, 张磊, 等.普通烟草LBD基因家族的全基因组序列鉴定与表达分析 [J].植物遗传资源学报, 2016, 17(2): 316-325.
[43] Luo Y W, Ma B, Zeng Q W,.Identification and characterization of lateral organ boundaries domain genes in mulberry,[J]., 2016, 8: 44-50.
[44] Grimplet J, Pimentel D, Agudelo-Romero P,.The lateral organ boundaries domain gene family in grapevine: Genome-wide characterization and expression analyses during developmental processes and stress responses [J]., 2017, 7(1): 15968.
[45] Wang X F, Zhang S Z, Su L,.A genome-wide analysis of the lbd (lateral organ boundaries domain) gene family inwith a functional characterization of mdlbd11 [J]., 2013, 8(2): e57044.
[46] Yu Q, Hu S M, Du J C,.Genome-wide identification and characterization of the lateral organ boundaries domain gene family invar.[J]., 2020, 42(1): 52-60.
[47] Gasteiger E, Gattiker A, Hoogland C,.ExPASy: The proteomics server for in-depth protein knowledge and analysis [J]., 2003, 31(13): 3784-3788.
[48] Geourjon C, Deléage G.SOPMA: Significant improvements in protein secondary structure prediction by consensus prediction from multiple alignments [J]., 1995, 11(6): 681-684.
[49] Kelley L A, Sternberg M J E.Protein structure prediction on the Web: A case study using the Phyre server [J]., 2009, 4(3): 363-371.
[50] Larkin M A, Blackshields G, Brown N P,.Clustal W and clustal X version 2.0 [J]., 2007, 23(21): 2947-2948.
[51] Tamura K, Stecher G, Peterson D,.MEGA6: Molecular evolutionary genetics analysis version 6.0 [J]., 2013, 30(12): 2725-2729.
[52] Bailey T L, Williams N, Misleh C,.MEME: Discovering and analyzing DNA and protein sequence motifs [J]., 2006, 34: W369-W373.
[53] Higo K, Ugawa Y, Iwamoto M,.Plant-acting regulatory DNA elements (PLACE) database: 1999 [J]., 1999, 27(1): 297-300.
[54] Li L, Liu M H, Shi K,.Dynamic changes in metabolite accumulation and the transcriptome during leaf growth and development in[J]., 2019, 20(16): 4030.
[55] Liu J, Cheng Z C, Li X Y,.Expression analysis and regulation network identification of the-like gene family in moso bamboo () under photoperiod treatments [J]., 2019, 38(7): 607-626.
[56] Lee H W, Kim M J, Park M Y,.The conserved proline residue in the LOB domain of LBD18 is critical for DNA-binding and biological function [J]., 2013, 6(5): 1722-1725.
[57] Jing T, Wang L, Liu H M,.Genome-wide identification of mitogen-activated protein kinase cascade genes and transcriptional profiling analysis during organ development in[J]., 2017, 7(1): 17732.
[58] 朱利利, 庆军, 杜庆鑫, 等.杜仲脂氧合酶基因家族全基因组鉴定及其表达特性研究 [J].植物研究, 2019, 39(6): 927-934.
[59] 王淋, 乌云塔娜, 叶生晶.杜仲EuHMGRS基因家族的鉴定及生物信息学分析 [J].经济林研究, 2013, 31(4): 16-24.
[60] 乌云塔娜, 王淋, 叶生晶.杜仲甲羟戊酸激酶(EuMK)基因鉴定及生物信息学分析 [J].经济林研究, 2014, 32(1): 6-12.
[61] 乌云塔娜, 王淋, 叶生晶.杜仲EuACOTS基因家族的鉴定及生物信息学分析 [J].经济林研究, 2013, 31(4): 1-8.
[62] 王淋, 乌云塔娜, 叶生晶.杜仲EuHMGS基因鉴定及生物信息学分析 [J].经济林研究, 2014, 32(1): 13-20.
[63] 贾喜涛, 刘文献, 谢文刚, 等.蒺藜苜蓿LBD转录因子基因家族全基因组分析 [J].西北植物学报, 2014, 34(11): 2176-2187.
[64] 王淋.杜仲基因组分析及适应性和橡胶生物合成的分子基础研究 [D].北京: 中国林业科学研究院, 2017.
[65] Burr B.Mapping and sequencing the rice genome [J]., 2002, 14(3): 521-523.
[66] International B I.Genome sequence analysis of the model grass: insights into grass genome evolution [J]., 2009, 463(7282): 763-768.
[67] Xu B, Li Z Y, Zhu Y,.genes AS1, AS2, and JAG negatively regulate boundary-specifying genes to promote sepal and petal development [J]., 2008, 146(2): 566-575.
[68] Fan M Z, Xu C Y, Xu K,.Lateral organ boundaries domain transcription factors direct callus formation inregeneration [J]., 2012, 22(7): 1169-1180.
[69] Liu J C, Sheng L H, Xu Y Q,.WOX11 and 12 are involved in the first-step cell fate transition during de novo root organogenesis in[J]., 2014, 26(3): 1081-1093.
[70] Naito T, Yamashino T, Kiba T,.A link between cytokinin and asl9 (asymmetric leaves 2 like 9) that belongs to the As2/lob (lateral organ boundaries) family genes in[J]., 2007, 71(5): 1269-1278.
[71] Zentella R, Zhang Z L, Park M,.Global analysis of della direct targets in early gibberellin signaling in[J]., 2007, 19(10): 3037-3057.
[72] Li C N, Zhu S S, Zhang H,.OsLBD37 and OsLBD38, two class II type LBD proteins, are involved in the regulation of heading date by controlling the expression of Ehd1 in rice [J]., 2017, 486(3): 720-725.
[73] Lu Q, Shao F J, MacMillan C,.Genomewide analysis of the lateral organ boundaries domain gene family inreveals members that differentially impact secondary growth [J]., 2018, 16(1): 124-136.
[74] Rubin G, Tohge T, Matsuda F,.Members of the LBD family of transcription factors repress anthocyanin synthesis and affect additional nitrogen responses in[J]., 2009, 21(11): 3567-3584.
[75] Wang Y L, Wang Y Q, Song Z Q,.Repression of MYBL2 by both microRNA858a and HY5 leads to the activation of anthocyanin biosynthetic pathway in[J]., 2016, 9(10): 1395-1405.
[76] Yordan S, Yordanov, Sharon R,.Members of the LATERAL ORGAN BOUNDARIES DOMAIN transcription factor family are involved in the regulation of secondary growth in populus [J]., 2010, 22: 3662-3677.
Identification, evolution and expression analysis ofgene family in
LIU Jun1, 4, 5, LI Long3, CHEN Yu-long1, WU Yao-song1, LIU Yan1, REN Shan-shan1, DONG Cheng-ming2
1.Henan Key Laboratory of TCM Prescription and Syndrome Signaling, Academy of Chinese Medicine, Henan University of Chinese Medicine, Zhengzhou 450046, China Henan Province Genuine Medicinal Materials Ecological Planting Engineering Technology Research Center, Henan Universityof Chinese Medicine, Zhengzhou 450046, China College of Forestry, Northwest Agriculture and Forestry University, Yangling 712100, China Key Open Laboratory of Bamboo and Rattan Science and Technology, International Center for Bamboo and Rattan, China National Forestry and Grassland Administration, Beijing 100102, China Anhui Taiping ExperimentalStation of International Center for Bamboo and Rattan, Huangshan 245716, China
In order to provide a basis for further research on the function ofgene, thegene family ofwas comprehensively identified in this study.Based ongenome database, this study used bioinformatics methods to identify and analyzegene family.A total of 19were identified fromgenome, which were divided into Class I and Class II groups and further divided into six subfamilies (Ia, Ib, Ic, Id and IIa, IIb).EuLBDs encodes 152—293 amino acids, and the theoretical isoelectric points distribution were 4.62—9.91, molecular weight between 17 420—31 820 by physicochemical analysis, all of EuLBD proteins were hydrophilic proteins.α-Helix and irregular curl were the main structure of the secondary structure in thegene family which was mainly expressed in nucleus.gene family had sinusoidal structure, the leucine zipper and the glycine-alanine-serine (GAS) structure domain.Expression pattern analysis showed thatinvolved inleaves development, and the transcription level ofgradually decreased with leaf development, which were not affected by the leaf gum content.In this study, the members of the LBD family were comprehensively identified and bioinformatics analysis from the genomic level of,which laid a foundation for further functional research ofgenes.
Oliver; LBD gene family; phylogeny analysis; gene expression;
R286.12
A
0253 - 2670(2022)10 - 3142 - 14
10.7501/j.issn.0253-2670.2022.10.025
2021-10-03
河南省高等学校重点科研项目(22A360005);国家林业和草原局/北京市共建竹藤科学与技术重点实验室开放基金(ICBR-2020-05);国际竹藤中心安徽太平试验中心开放课题基金(1632021006-4);河南中医药大学博士科研基金资助项目(RSBSJJ2019-04)
刘 俊(1990—),女,助理研究员,博士,主要从事药用植物分子生物学研究。E-mail: liujun_0325@163.com
[责任编辑 时圣明]
