面向信息SNP选择的聚类算法∗
2021-11-08周从华张付全蒋跃明
邢 斌 周从华 张付全 张 婷 蒋跃明
(1.江苏大学计算机科学与通信工程学院 镇江 212013)(2.无锡市精神卫生中心 无锡 214151)(3.无锡市妇幼保健院 无锡 214002)(4.无锡市第五人民医院 无锡 214073)
1 引言
遗传病是由致病基因所控制的疾病,这种由于遗传物质发生改变引起的疾病病种多,目前已发现的遗传病超过3000种,而且具有发病率高、先天性、终生性、家族性等特点,对人类健康产生巨大影响。近年来随着世界范围内人类基因组研究(Ge⁃nome-Wide Association Study,GWAS)开展实施,基因测序技术依靠对遗传特征的有效挖掘,在疾病诊断、预测和治疗等方面发挥着更加重要作用[2],而GWAS是在单核苷酸多态(Single Nucleotide Poly⁃morphism,SNP)性的基础上展开研究的,单核苷酸多态性主要是指基因组水平上的单核苷酸变异导致脱氧核糖核酸(DNA)序列多态性的现象,是人类最常见的一种遗传变异,研究表明SNP对人类疾病有直接或间接的联系,并且SNP具有稳定性好、频率高、采集容易等优点,因此对SNP数据进行研究有重要意义。在此背景下,许多机器学习算法在SNP数据中得到了广泛的应用。但是由于SNP数量过大,维数过高,数据中存在冗余和噪声,研究中必须要考虑“维数灾难”问题。因此,SNP数据分析的初始阶段一般是选择SNP中信息量最大的子集即信息SNP子集,以提高算法的性能,降低时间要求。
信息SNP子集的选择本质上是特征选择,特征选择(Feature Selection,FS)是在保持原始数据准确表示的同时,显著降低特征空间的维数的过程。然而由于SNP数据之间的非相互独立的特点,现有的特征选择方法难以挖掘SNP位点之间的相关性,从而漏掉重要的遗传信息,最终降低算法的分类效果。鉴于上述问题,本文采用基于信息论的新的相似度度量方法,并将其应用到K-means中,把SNP依据一定的关系划分为若干簇,然后使用粒子群算法从每个簇中选出一个或多个能够代表整个簇的信息SNP,构造出最终的信息SNP子集。
2 相关工作
2.1 SNP选择研究现状
目前SNP选择主要有两种方法,一种是基于统计检验的关联研究,分两个步骤,第一步,通过生物实验技术在全基因组上扫描筛选有效位点,第二步查验SNP位点基因分型,通过关联分析识别信息SNP;另一种是基于机器学习的SNP选择,这种SNP选择本质上是特征选择问题,可分为过滤式和封装式两种。过滤式设置评价指标给每个SNP打分,优点是计算量小,但忽略了SNP之间的内在联系,不容易得到最优的特征子集;而包裹式方法虽然计算量大,却可以把学习器和评价指标结合起来,最终得到最优解。近年来,许多人尝试对这两种方法进行改进,Raid Alzubi等[1]将过滤式和包裹式方法结合在一起提出一种混合特征选择方法来检测信息SNP并选择最优SNP子集,取得了不错的效果。为解决SNP数据高维问题,Cong等[2]提出了一种基于主成分分析的基因数据降维算法,用于低维空间SNP位点的聚类。上述两篇文献虽然在结果上较前人有一定的进步,但是仍忽略了SNP与SNP子集之间的相关性问题,Liao[3]将信息论引入到SNP选择中,用于衡量SNP子集之间的相关性,该方法显著提高了标签SNP选择的效率和预测精度,但该方法没有将SNP之间的相似度和聚类有效结合在一起。
2.2 K-means聚类算法
K-means算法是一种迭代求解的无监督聚类算法,通过选取K个对象作为初始聚类中心计算每个样本点与聚类中心的距离即相似度,将每个样本点分配到最近的聚类中心,最终使得相同簇内的样本点相似度尽可能高,不同簇间的相似度尽可能低。
2.2.1 欧氏距离
欧氏距离是衡量两个同一样本集样本对象差异性,距离越大样本差异度越大。欧氏距离公式如下:
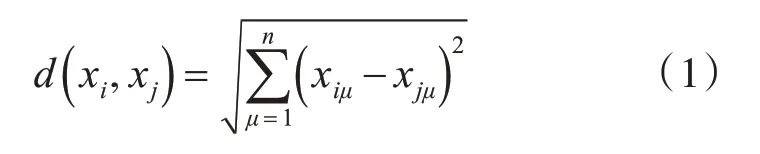
式中,xiμ为样本对象xi的第μ个属性。
2.2.2 簇内误差平方和
在欧氏距离基础上,进行变形,将其中一个样本对象换为簇中心得到簇内误差平方和,簇内误差平方和用来度量聚类效果的好坏。
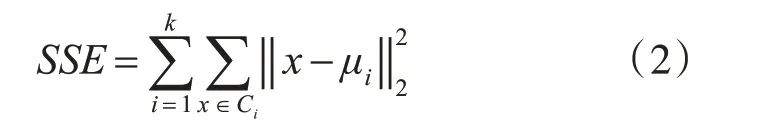
3 基于改进的K-means算法的SNP选择方法
3.1 传统K-means在SNP选择上的缺陷
传统的K-means聚类使用SNP之间的欧氏距离来度量SNP之间的相似度,这并不能挖掘出SNP位点之间生物学上的联系性,鉴于上述问题[1]将互信息引入到聚类中,用作SNP相似度度量,虽然起到很好的效果,但仍没解决传统K-means算法中另一缺陷,只考虑两个SNP位点之间的相似度,然而在实际SNP数据集中,单个SNP往往和某个SNP子集有着强关联性,鉴于上述两个问题,提出一种新的基于信息论的SNP选择算法K-MIGS。
3.2 基于信息论的相似度度量
3.2.1 信息熵与互信息
信息熵用来衡量信息的不确定性,单个SNP位点的信息熵可表示为
假设样本S中某一SNPX有T个可能取值,其中p(xi)表示位点X中第i类样本出现的频率。Tx表示位点X所有属性的个数。联合信息熵:
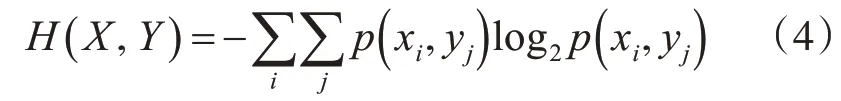
则SNP位点X和Y的互信息可表示为
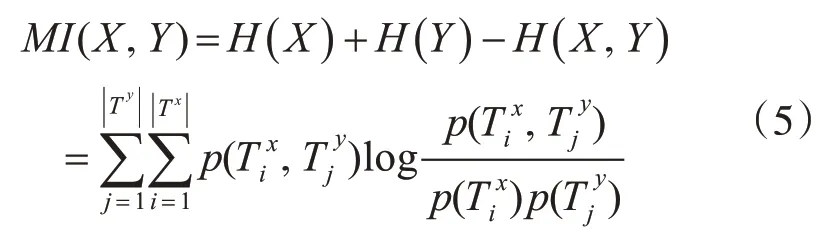
我们使用两个位点的互信息来衡量它们之间的相似度,互信息越大,表示相似度越大,每个特征与初始簇中心的距离度量公式表示如下:
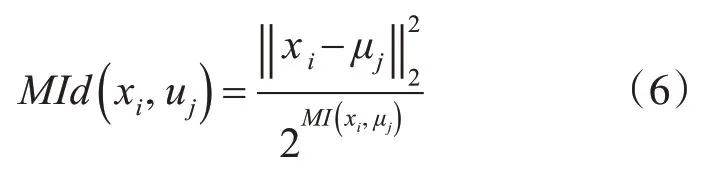
接下来我们试图利用MI(S1;SNPX)和MI(S2;SNPX)来构建SNPx与SNP子集之间的相似度,有下式:
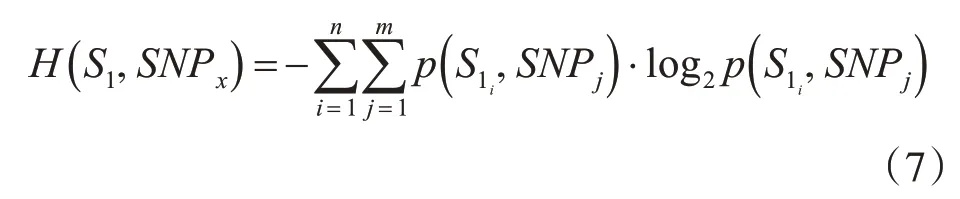
假设n个SNP位点上存在m种,那么SNP子集内联合熵可表示为
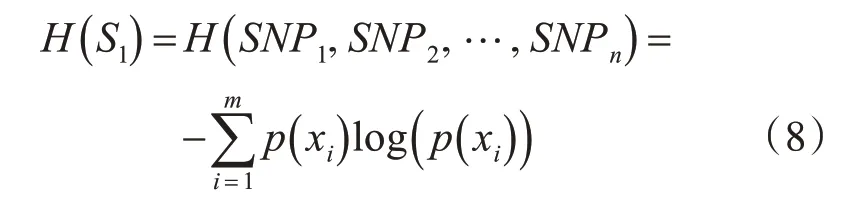
但是如果S1、S2两个子集SNP个数是不相等的,那么I(S1;SNPX)和I(S2;SNPX)将没有可比性,因为单个SNP和大的SNP子集会得到大的互信息值,为了解决这个问题,我们结合了互信息和信息熵提出了一种单个SNP和SNP子集的相似度度量方式MIGS,公式如下:
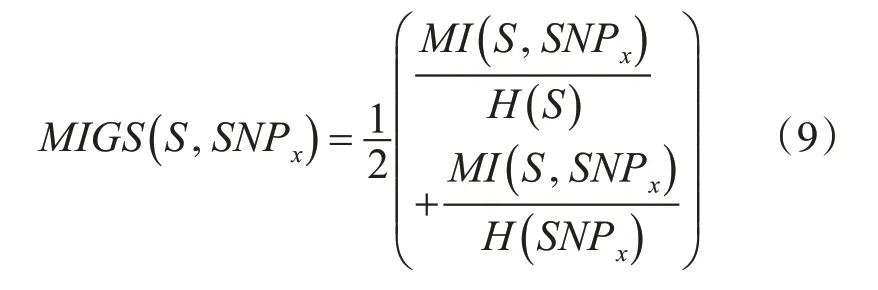
其中,H(S)、H(SNPX)分别表示SNP子集S和单个SNP的信息熵,MI(S;SNPX)表示它们之间的互信息,考虑到SNP位点之间存在强相关性的特性,我们在原K-means聚类的距离度量欧氏距离中引入互信息的概念,则第一轮迭代计算中,每个特征与初始簇中心的距离度量公式表示如下:
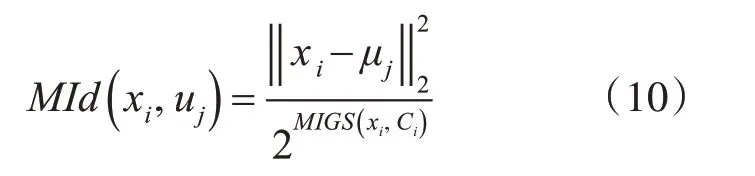
其中MId(xi,uj)表示特征xi与初始簇中心uj的相似度,表示特征与初始簇中心的欧氏距离,MIGS(xi,Ci)表示特征x i与初始簇i之间的相似度。
3.2.2 簇中心的更新
在K-MIGS中,初始的簇中心的选择与传统K-means一致,不同的是在簇中心更新时,传统K-means采用均值向量的方式更新簇中心,这并不适合用在K-MIGS算法中,因此我们采用新的簇更新方式,选取与均值向量最近的一个SNP作为簇中心。
3.3 算法K-MIGS步骤
结合章节3.1和3.2,则算法K-MIGS的整体步骤如算法1所示。
算法1:K-MIGS算法
输入:数据集D={x1,x2,…,xm}聚类簇数kMAX_N
1)从数据集D={x1,x2,…,xm}中随机选择k个样本作为初始均值向量(μ1,μ2,…,μk)
2)for j=1 to k do
3) forj=1 j=1 to mdo
4)若簇CiSNP数为1,根据式(6)、(7)计算与μj的相似度距离,否则根据式(10)、(11)计算SNP子集S与SNPi的相似度距离MId(S1,SNPi)
5) end for
6)end for
7)for i=1 to m do
8)将xi划入与其相似度距离最小的簇
9)end for
10)repeat
11)for i=1 to k do
12)计算新的均值向量,并通过式(1)计算x( )x∈Ci与μ'i的距离
13)选择最小的d(x,μ'i)作为簇Ci新的簇中心
14)end for
15)until迭代次数达到阈值或簇中心不再更新
其中,2~6行根据式(6)、(9)、(10)分两种情况计算SNP之间的相似度距离,分为当前簇SNP数为一和SNP数不为一;7~9行将所选SNP划分到相似度距离最小的簇,11~13行根据式(1)将簇中心更新为与均值向量欧氏距离最小的SNP。
3.4 K-MIGS算法在SNP选择中的应用
本节将结合K-MIGS算法和粒子群算法对每个簇中的SNP进行选择。
粒 子 群 算 法(Particle Swarm Optimization,PSO)是一种群体智能算法,该算法模仿鸟群群体合作来最快获得食物的基本原理。在粒子群算法中,每一个候选解都可以看作是“鸟群中的个体”,即一个粒子。在粒子运动过程中每个粒子根据自身经验和相邻粒子经验,通过对速度进行改变来调整自己的位置。粒子通过最佳粒子流在问题空间内运动,迭代一定次数或者达到给定的最小误差得到最优解。
将粒子群算法应用到SNP选择中包括两个主要部分,种群初始化和粒子更新。
我们为SNP选择问题设计了种群:
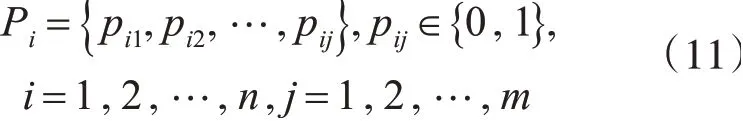
其中n代表种群P的大小,m代表SNP的数量,pij=1表示选择了第i个粒子的第j个SNP。
通常,在种群的初始化过程中每个SNP的选择为信息SNP的概率由确定,其中k代表选择的SNP的数量,m代表SNP的总数。在每次迭代中依据pbest和gbest的参数对粒子进行更新,每个粒子根据以下方程式更新:
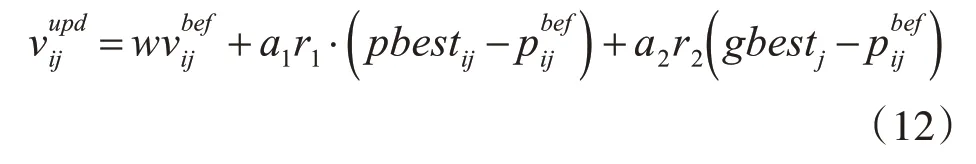
其中w是惯性权重,a1和a2是加速学习因子,r1和r2为0~1的随机数,和为更新前和更新后的粒子i速度矢量的第j维分量,为更新前粒子i位置矢量的第j维分量,pbestij=(pi1,pi2,…,pij)表示粒子i个体经历过的最好位置,gbestj=(g1,g2,…,gj)表示种群所经历过的最好位置。
将实验所需原始数据进行预处理,并使用K-MIGS算法进行聚类,最后使用粒子群算法从每个簇中选择得到最终的信息SNP。
4 实验
4.1 实验环境和数据
实验环境:编译工具Python 3.7.3,操作系统Windows10 64位,处 理 器Intel(R)Core(TM)i7-6700HQ,CPU@2.60GHz 2.59GHz,GPU Nvidia GeForce GTX 1060 3G,运行内存16G,硬盘1.25T。
实验数据集:实验数据来自于无锡精神卫生中心,包括两种SNP数据集EN1000(9445SNPS)、E144(2514825SNPS),另外还包含样本的基因型,患病与否的标记,具体描述如表1所示。

表1 数据集描述
4.2 实验评价指标
1)SNP选择评价指标
本文使用SNP选择常用评价指标,信息SNP子集对非信息SNP子集重构准确度来评价SNP选择效果的好坏。
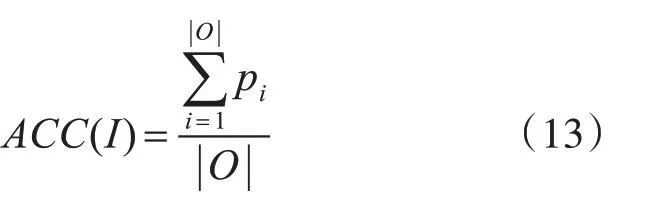
其中pi为信息SNP子集对非信息SNP位点i的重构准确度,||O是非信息SNP的数量。重构准确度越高,SNP选择效果越好。
2)分类预测评价指标
本文使用分类实验常用的评价指标预测的准确率(Accuracy)及F-Measure对分类结果进行评价,分类结果的“混淆矩阵”如表2所示。

表2 预测类别与实际类别的“混淆矩阵”
根据上表分类结果评价指标可由式(14~17)计算:

4.3 数据预处理
SNP数据预处理分为两个子阶段,数据编码和数据更新,原始SNP具有三种基因型,纯合子基因型(AA),杂合子基因型(Aa)以及纯合变异基因型(aa),这种基因型数据不利于后续聚类操作,需要对数据进行编码,分别将AA、Aa、aa编码为0、1、2;数据编码后对数据进行更新,更新又包括缺失数据填充及不符合标准数据的删除。依据上述原则处理后,数据集G1000和E144分别剩余9298和214935条有效的SNP。
4.4 实验结果与分析
4.4.1 聚类实验及分析
此部分实验包括四种算法比较实验,分别为K-means、特征加权K-means、模糊聚类算法FCM和K-MIGS,主要评价指标为最后得到不同簇下信息SNP子集的重构准确度。在数据集G1000和E144上,分别用四种算法进行聚类实验,使用粒子群算法对每个簇进行信息SNPs提取,最后计算信息SNP子集对非信息SNP子集的重构度。实验结果如图1和图2所示。
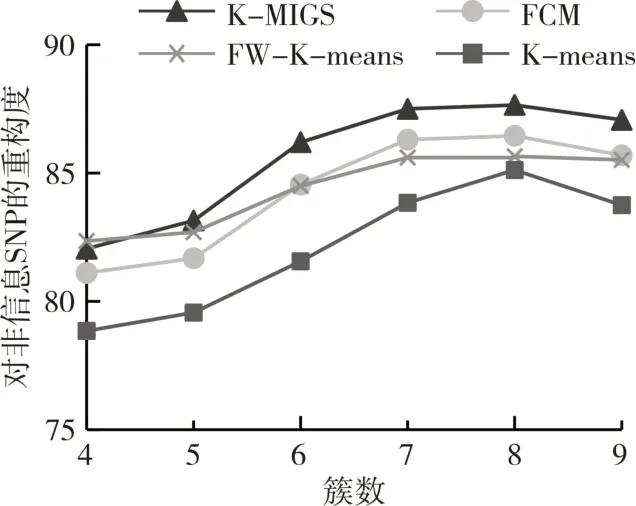
图1 G1000上算法选出的信息SNP对非信息SNP的重构度图

图2 E144上算法选出的信息SNP对非信息SNP的重构度图
由图1、2可看出,使用K-MIGS/粒子群算法最终提取的信息SNP在两个数据集上对非信息SNP具有更好的重构度,并且在聚类簇数为8时重构度达到最大值,因此后续分类实验采用簇数为8。
4.4.2 分类实验及分析
分类实验的目的是更进一步检测K-MIGS/粒子群算法所选择的信息SNP子集包含信息的重要程度,此实验中,采用K-means/粒子群算法、K-MIGS/粒子群算法、特征加权K-means/粒子群算法(FW-K-means/粒子群)、ReliefF和MCMR算法进行信息SNP子集的筛选,使用SVM、DT和神经网络(Neural Networks,NN)为分类器,主要评价指标为分类准确率Acc和F1-measure。实验结果如表3所示。
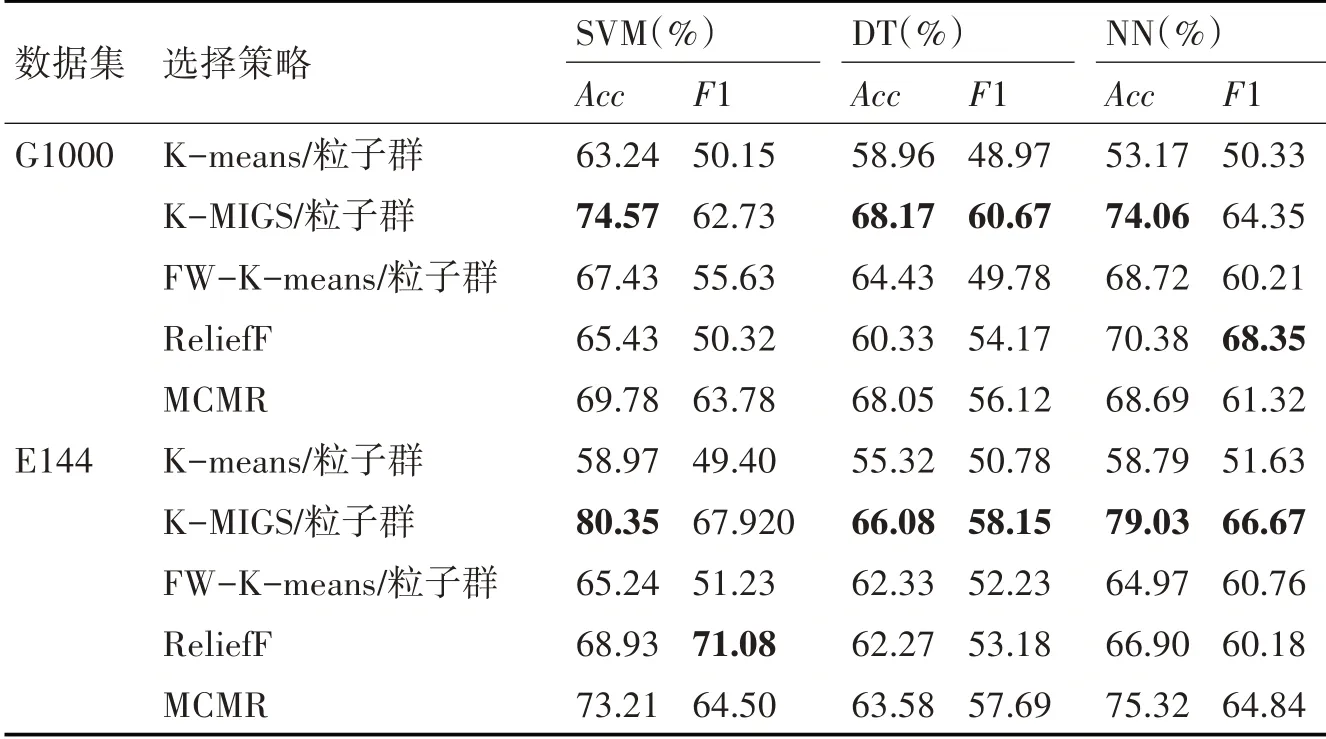
表3 不同的分类器进行SNP子集评价的结果
5 结语
针对SNP本身具有的高维少样本特性以及自身存在的遗传规律,本文提出了一种基于K-means的算法K-MIGS并将其应用到SNP选择中。对数据进行预处理后,使用提出的方法对SNP数据进行聚类,最后使用粒子群算法构造最终的SNP子集。在聚类效果评估和分类实验中均表明,该方法很大地提升了SNP选择的有效性。本文提出的方法相比其他SNP选择方法优势在于同时考虑了单个SNP与SNP子集的相似度对聚类结果的影响。本文的后续工作主要有两点:一是继续优化算法以减少单个SNP与SNP子集的相互关联时引入的额外时间复杂度;二是继续优化粒子群算法,使其在每个簇中选择出信息量更大的SNP。
