中国东北和华北地区紫花苜蓿根瘤菌遗传多样性研究
2021-09-02李昕芫娄金秀刘清源胡健张英俊
李昕芫,娄金秀,刘清源,胡健,张英俊
中国东北和华北地区紫花苜蓿根瘤菌遗传多样性研究
李昕芫,娄金秀,刘清源,胡健,张英俊
南京农业大学草业学院,南京 210095
【】紫花苜蓿(L.)被誉为牧草之王,近年来,中国东北和华北地区种植面积不断扩大,但紫花苜蓿质量与产量仍不足以满足中国畜牧业发展的需求。本研究旨在分析中国东北和华北地区紫花苜蓿根瘤菌遗传多样性,为紫花苜蓿高效固氮根瘤菌的筛选与应用提供参考。采用表面消毒和平板划线法从根瘤中分离纯化根瘤菌单菌落;使用BOX-PCR方法对供试菌株进行基因型划分;选取代表菌株进行持家基因(、和)和共生基因(和)的系统发育分析。从中国东北和华北19个采样地共分离纯化了499株根瘤菌,BOX-PCR可将供试菌株分为37种BOX型,BOX型存在显著的地理分布现象,同时寄主品种对根瘤菌基因型具有一定的选择作用。97.60%(487/499)的根瘤菌为。其余12株分别为、、、、、sp.和sp.,这12株根瘤菌仅在中国东北地区发现,华北地区根瘤菌均为。3个持家基因在紫花苜蓿根瘤菌种间系统发育分析结果一致,但其揭示优势种的种内多样性存在差异。共生基因系统发育结果显示,在根瘤菌属间和属内发生了共生基因的水平转移现象。在种内显示出比更丰富多样性。中国东北和华北地区紫花苜蓿根瘤菌具有较丰富的遗传多样性,且根瘤菌存在显著的地理分布特征和寄主选择现象。
紫花苜蓿;根瘤菌;遗传多样性;系统发育;水平转移
0 引言
【研究意义】紫花苜蓿(L.)产量高,适口性好,常作刈割草地,为家畜提供青贮和干草资源,也可与禾本科草混播作放牧利用,被称为牧草之王[1],近年来,中国紫花苜蓿种植面积不断扩大,但仍不足以满足中国畜牧业发展的需求,如何提高紫花苜蓿产量与质量,是当前亟待解决的问题[2]。根瘤菌剂的应用被作为提高豆科植物产量及质量的有效手段[3-4],据统计,全世界有70多个国家在种植豆科植物时使用根瘤菌剂,尤其在美国、巴西等西方国家,根瘤菌接种率更是高达70%左右[5]。而中国由于根瘤菌研究起步较晚,菌剂质量、应用范围及应用效果等问题未能很好地解决。中国豆科作物的根瘤菌接种面积尚不足其播种面积的3%,豆科牧草接种面积更少[5]。紫花苜蓿根瘤菌遗传多样性的研究对根瘤菌剂的筛选和应用具有重要的参考意义[6-7]。【前人研究进展】目前,苜蓿根瘤菌多样性的研究主要集中在中国西部地区[6-11],张荣娟[6]对中国西部苜蓿根瘤菌进行调查研究,发现中国西部根瘤菌主要有和,其中是中国西部苜蓿根瘤菌的主要种群,另外少数则出现在西南部偏酸性的土壤中;KANG等[7]通过对甘肃紫花苜蓿根瘤菌多样性分析,发现主要有、sp.、和;同样,张小甫[8]研究发现甘肃地区紫花苜蓿上还有另外一些根瘤菌,如和sp.。【本研究切入点】近年来,随着紫花苜蓿种植范围的扩大,中国东北和华北地区也成为了紫花苜蓿种植的重要地区,而关于这些地区紫花苜蓿根瘤菌多样性的研究则鲜有开展。不同地区紫花苜蓿品种及其种植的环境不尽相同,其根瘤菌遗传多样性可能存在差别[12-13]。【拟解决的关键问题】本研究于2015—2017年重点采集了中国东北及华北19个地区紫花苜蓿根瘤菌499株,采用BOX-PCR指纹图谱对紫花苜蓿根瘤菌多样性进行分析,进一步选用3个持家基因(、和)和2个共生基因(和)对各BOX型代表菌株进行系统发育分析,以期了解中国东北和华北地区紫花苜蓿根瘤菌的遗传多样性,为紫花苜蓿根瘤菌优质菌种资源的筛选及应用提供科学参考。
1 材料与方法
1.1 样品的采集
试验选择种植成片的紫花苜蓿地进行样品采集,东北地区种植的紫花苜蓿品种主要有公农5号、东苜1号、公农1号、龙牧803、龙牧806、WL319和敖汉等,华北地区种植的主要有三得利、M343、M354、劲能5010、中苜1号、中苜3号、甘农3号和WL363等(表1)。部分采样地在相邻地块种植了不同的紫花苜蓿品种,如新乡、沧州、胶州、衡水和辽阳等,本文分别进行了样品的采集(表1)。采样地紫花苜蓿种植年限普遍在2—5年。采样地除五河外,均未有根瘤菌接种的情况。样品采集选择在5—6月进行,一般在紫花苜蓿初花期左右时间,此时根瘤菌活性一般较好,易获得促生效果好的菌种。
使用铁锹将健康的紫花苜蓿植株和根一起挖出,轻抖去根系上的土壤,将根瘤连根一起剪下,置于含有蓝色变色硅胶粒及滤纸片的50 mL离心管中。每个采样点随机选择5—10株长势相似的紫花苜蓿进行取样,每个植株至少保持>10 m的距离,随机从每个植株根部不同部位采集10—15个根瘤,以满足采集样品的代表性。当大部分蓝色硅胶粒变色时,需要及时更换,以保持离心管处于干燥状态。
1.2 根瘤菌的分离纯化、保存
取出干燥的根瘤样品,用灭菌蒸馏水冲洗其表面后,使用无菌生理盐水,4℃浸泡1—2 h,至根瘤吸水恢复原状,用于根瘤菌的分离,每个根瘤最多分离保存1株根瘤菌。使用Wang等[13]方法进行根瘤的表面消毒和破碎、根瘤菌的分离和纯化,并稍作改动。使用酵母甘露醇琼脂培养基(yeast mannitol agar,YMA)平板进行根瘤菌的分离和纯化[8]。将具有根瘤菌形态特征的细菌一份短期保存于YMA斜面培养基上[8],置于4℃备用,另一份使用2 mL冻存管,装有0.8 mL胰蛋白酵母(tryptone yeast,TY)液体培养基[9],将菌液摇培至生长对数期,加入等体积的50%甘油,置于-80℃长期保存。
1.3 根瘤菌DNA的提取
将1 mL灭菌TY液体培养基加入2 mL灭菌离心管中,使用接种环挑取YMA平板上的根瘤菌单菌落接入TY培养基,将离心管置于28℃恒温摇床上以180 r/min的转速摇培2—4 d。待培养至对数生长期时,使用离心机以13 500 r/min的转速离心5 min,收集菌体。收集的菌体按照天根细菌基因组DNA提取试剂盒(DP302)说明书进行根瘤菌基因组DNA的提取,-20℃保存备用。
1.4 BOX-PCR指纹图谱分析
以根瘤菌基因组DNA为模板,使用BOX-PCR引物(BOXAIR:5′-CTACGGCAAGGCGACGCTGA CG-3′)进行扩增[14]。25 μL BOX-PCR反应体系为12.5 μL Master Mix(TaKaRa)、1 μL BOX A1R(10 μmol·L-1)、0.2 μL BSA(10 mg·ml-1)、2.5 μL DMSO、1 μL DNA和7.8 μL ddH2O。BOX-PCR反应程序为95℃ 7 min;94℃ 1 min,52℃ 1 min,65℃ 8 min,30个循环;65℃ 22 min。用1.8%的琼脂糖凝胶检测,60 V电压电泳3 h,对BOX-PCR产物进行分离。使用凝胶成像仪将电泳结果进行拍照保存,分析BOX-PCR指纹图谱并对根瘤菌进行BOX型划分。
1.5 持家基因和共生基因PCR扩增
选取每种BOX型的代表菌株,用于持家基因(、和)及共生基因(和)的PCR扩增。扩增引物信息和PCR反应程序参考已有文献报道[7, 15-18]。所有基因PCR反应体系均为30 μL:15 μL Master Mix(TaKaRa)、正向和反向引物(10 μmol·L-1)各0.5 μL、1 μL模板DNA和13 μL ddH2O。对PCR产物进行100 V凝胶电泳20 min,成功扩增出目的片段且条带清晰的送至上海生工有限公司测序。
1.6 系统发育分析
将代表菌株各基因测序结果提交至GenBank数据库中,其中基因序列号为MZ165036—MZ165071,基因序列号为MZ165072—MZ165105,基因序列号为MZ165106—MZ165138,基因序列号为MZ165139—MZ165170,基因序列号为MZ165171—MZ165202。同时在NCBI网站进行序列同源性比对(BLAST search),下载数据库中相似性高的基因序列及信息作为参照,使用MEGA7.0软件的Clustal W进行序列比对[6],并采用Kimura 2- parameter模型,bootstrap值为1 000,构建Neighbour- Joining系统发育树[7]。
2 结果
2.1 BOX-PCR指纹图谱
2015—2017年采集了中国19个地区紫花苜蓿根瘤样品(表1),共分离出499株根瘤菌,按区域可将所有采样地区划分为两大区(东北和华北)(表1),对所有菌株进行BOX-PCR扩增并对指纹图谱进行分析,可将供试的499株根瘤菌分为37种BOX型(表2),中国东北和华北地区共有的BOX型15个,东北地区特有的BOX型16个,其中,除BOX13菌株数为15个外,其余东北地区特有的BOX型菌株数量均不超过3个。华北地区特有的BOX型6个,其中BOX24和BOX33的菌株数为8和19个,其余华北地区特有的BOX型菌株数均不超过2个。在15个共有的BOX型中,菌株数占比在7%以上的有BOX1(7.62%)、BOX7(18.64%)、BOX19(7.20%)、BOX22(11.62%)和BOX23(28.26%)。这5个BOX型中,BOX1(36/38)和BOX22(53/58)主要分布在东北地区,BOX7(72/93)、BOX19(31/35)和BOX23(132/141)主要分布在华北地区。所有菌株BOX型分布信息如表2所示。
2.2 紫花苜蓿根瘤菌鉴定
选取37种BOX型的代表菌株,进行持家基因的扩增、测序和比对分析(BOX3使用进行序列比对),以鉴定各BOX型的根瘤菌种类,结果显示,大部分BOX型(28种)菌株为,有9种BOX型为sp.(相似度91.34%,BOX3)、(100%,BOX6和BOX11)、(100%,BOX16)、(99.67%,BOX21和BOX31)、(98.01%,BOX30)、(99.33%,BOX36)、(92.72%,BOX32)(表2)。所有这9种BOX型均来自中国的东北:白城(4种BOX型)、通榆(1种BOX型)和沈阳(4种BOX型),BOX3和BOX32可能为尚未明确分类地位的新种(表2)。
2.3 根瘤菌系统发育分析
持家基因、和的系统发育结果具有类似的特征,3个持家基因揭示根瘤菌种间多样性的结果一致,但揭示种内多样性的结果存在差异(图1)。持家基因均可将BOX16与聚在一起(100%相似度);BOX21和BOX31与聚在一起,但和聚类显示其与GenBank数据库中的形成明显的分支;BOX30与聚为一类,但与GenBank数据库中的参考序列形成明显的分支(EU120727、DQ310809和KF206971),BOX6和BOX11可与聚在一起,但将它们聚为2个分支;BOX36与聚在一起,但和将二者聚为2个分支。3个持家基因可将所有为的BOX型与显著的分开(图1)。在揭示种内多样性方面,3个持家基因均可将分为4个分支(图1),但分支内BOX型存在差别,仅BOX24在3个持家基因中均为单独分支。可将BOX18、BOX26和BOX27聚在一起,和则只能将BOX18和BOX27聚在一起。将其余代表菌株分类2个分支,但和则无法有效将其分开(图1)。
共生基因和在BOX3、BOX21、BOX32和BOX36中不能有效的扩增出来,对其他BOX型代表菌株的和进行系统发育分析,结果显示,二者揭示相同的特征。BOX16的和型与属聚在一起,其余BOX型代表菌株的和均与的聚在一起(图2)。BOX6(,100%)、BOX30(,98.01%)和BOX31(,99.67%)与分属不同的属和种,但和都聚在一起。在种内显示出比丰富的多样性(图2)。

表1 本文供试的紫花苜蓿根瘤菌菌株信息

表2 紫花苜蓿根瘤菌BOX型种类及其分布

续表2 Continued table 2
a:BOX3选取进行序列比对,其余BOX型选取进行序列比对;b:采样地后面括号里的数字代表菌株数,采样地后面无数字则代表只有1株菌株。NE:东北;N:华北
a:BOX3 was aligned withgene, the rest BOX types were aligned withgene;b: The number in the bracket after sampling site represented the number of strains, sampling site with no number represented only one strain in this sampling site. NE: Northeast; N: North
2.4 紫花苜蓿品种对根瘤菌基因型的影响
对部分采样地相邻地块不同紫花苜蓿品种共生的根瘤菌基因型进行分析,结果显示,新乡地区,品种M343的根瘤菌中,BOX23占据优势(占比62.12%,23/37),而品种M354中,BOX7则占优势(90%,19/21);衡水地区,品种中苜1号的根瘤菌中,BOX7占优势(90.32%,28/31),品种WL363中,BOX23占优势(64.71%,11/17);胶州地区,与品种三得利共生结瘤的优势根瘤菌基因型为BOX19(66.67%,8/12),与中苜3号结瘤的基因型主要为BOX23(66.67%,8/12);沧州和辽阳地区,不同品种的根瘤菌占优势的基因型则相同,分别为BOX23和BOX1(表3)。
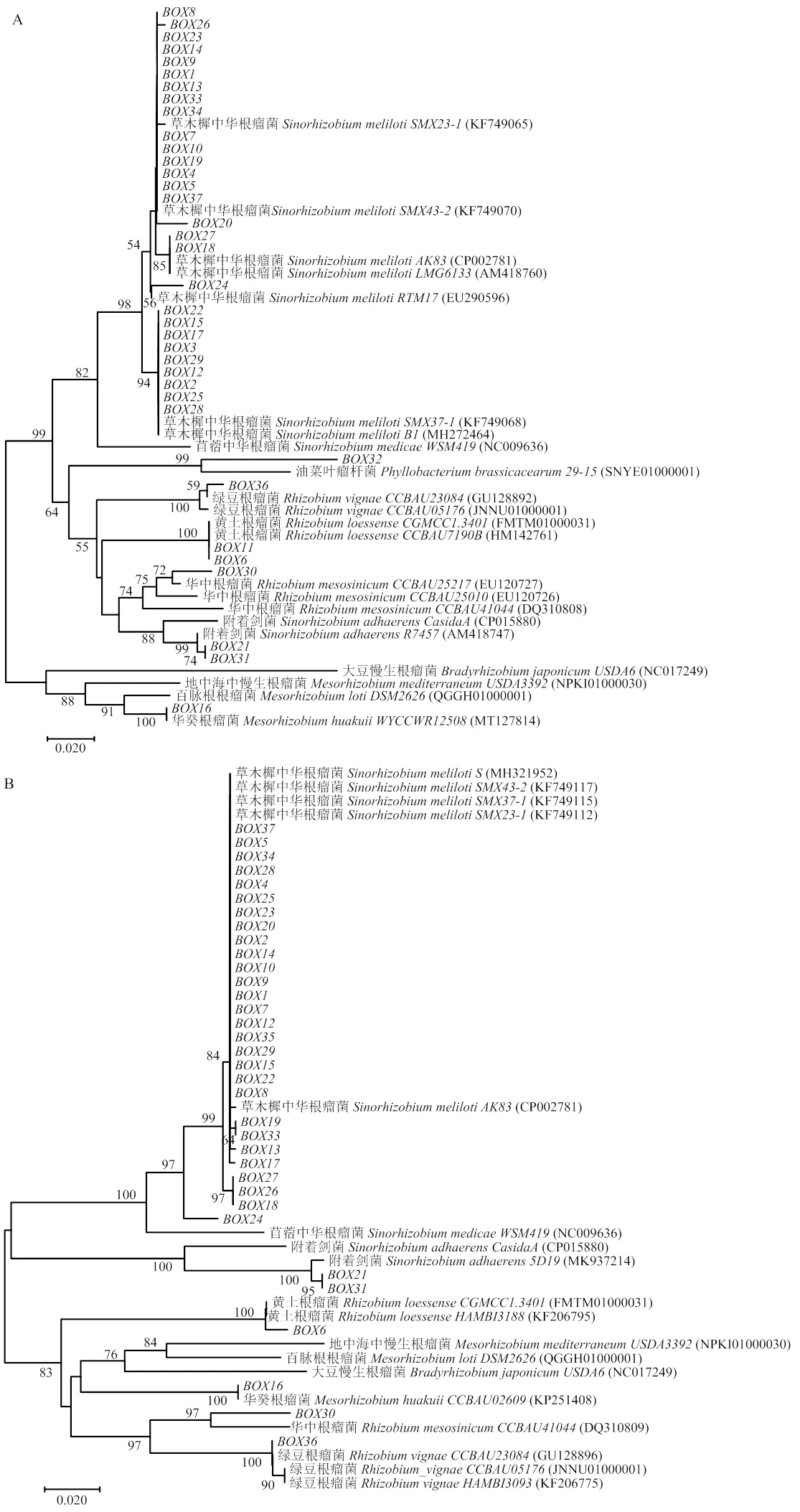

表3 栽培品种对紫花苜蓿根瘤菌基因型的影响
a:同一采样地的紫花苜蓿不同品种种植在相邻的地块,其生境相似;b:BOX型后面括号里的数字代表菌株数,BOX型后面无数字则代表只有1株菌株
a: Cultivars from the same sampling site were planted adjacently, they had similar habitat type;b: The number in the bracket after BOX type represented the number of strains, BOX type with no number represented only one strain in this BOX type

图2 共生基因nodC(A)和nifH(B)的系统发育分析
3 讨论
BOX-PCR与16sRNA-RFLP、IGS-RFLP等方法常用作大样本量根瘤菌的基因型初步鉴定,有研究显示BOX-PCR方法在揭示根瘤菌基因型多样性方面具有更好的效果[19]。Wang等[13]使用BOX-PCR可将青藏高原地区581株紫花苜蓿根瘤菌分为124种BOX型。本研究采用BOX-PCR方法将中国东北及华北地区499株紫花苜蓿根瘤菌分为37种BOX型。持家基因、和的系统发育分析也进一步验证了BOX-PCR在揭示根瘤菌基因型多样性方面的能力(图1)。
紫花苜蓿根瘤菌BOX型存在一定的地理分布现象,在所有37个BOX型中,为东北和华北特有的分别为14和6个。特有的BOX型占比一般都较少,仅华北特有的BOX33占比超过3.8%,表明特有BOX型虽然能在局部区域定植,但其在环境适应方面存在一定的劣势(表2)。BOX型占比前5的在中国东北和华北均广泛分布,但优势的BOX型存在一定的地理偏好性,BOX23(占比28.26%)和BOX7(占比18.64%)主要分布在中国的华北地区,而BOX22(占比11.62%)则主要分布在东北地区(表2)。研究表明中国东北和华北地区的紫花苜蓿根瘤菌存在地理分化现象,这与张荣娟[6]对中国西部苜蓿根瘤菌的研究结果相似,且已有很多研究表明苜蓿根瘤菌存在着显著的地理学分布[20-21]。本文也发现,紫花苜蓿不同品种对根瘤菌基因型选择存在一定的寄主偏好性,在新乡和衡水地区,这种现象尤为明显(表3)。这与很多相关文献报道植物品种对根际微生物具有选择性相一致[22-23],紫花苜蓿不同品种在种植的过程中,可能选择了特定基因型的根瘤菌与其发生共生关系。因此,本研究结果表明,在筛选根瘤菌菌剂时,应当充分考虑地理和寄主品种的因素,选择在特定区域与特定品种高效结瘤共生的优势基因型的根瘤菌,以提高其在田间竞争结瘤的能力。接下来可通过盆栽试验,进一步探究寄主和环境对根瘤菌基因型选择的机制,以及优势BOX型的竞争能力是否是其广泛分布的重要原因,同时可通过比较基因组分析并揭示根瘤菌优势基因型广适应性的分子机制[24]。
本研究对紫花苜蓿根瘤菌进行了鉴定,发现中国东北和华北地区优势根瘤菌种仍然是,这与其他研究报道类似。张荣娟[6]对中国西部苜蓿根瘤菌多样性分析显示其主要也为,少数是,主要来自于西南亚热带气候区呈酸性的土壤中,表明与苜蓿共生的根瘤菌中为优势菌种;在国外,Talebi等[25]对伊朗地区982紫花苜蓿根瘤菌进行分析,发现仅有7株为,其余均为;Silva等[26]发现墨西哥15个采样点分离的176株苜蓿根瘤菌中仅有6株为。本研究的499株根瘤菌中未发现有,可能与采样地的土壤理化特性有关。本文的采样点基本上都属于中性或偏碱的环境(未发表数据),而已有研究显示倾向于偏酸的环境[6, 25-26]。研究也发现中国东北地区根瘤菌存在丰富的种间多样性,所有非优势种的根瘤菌均来自于东北地区,而华北地区根瘤菌则均为。目前与紫花苜蓿共生的根瘤菌种间多样性的报道较少。张小甫[8]对从甘肃地区筛选的 31 株苜蓿根瘤菌进行分析,发现根瘤菌存在多个种(、、和sp.),但这些种主要为内生菌。冯春生[9]对西北地区天蓝苜蓿根瘤菌分析,发现供试菌株少数可归属于根瘤菌属()、土壤杆菌属()、中华根瘤菌属(),大部分菌株为。Wang等[13]发现581株分离至西藏紫花苜蓿根瘤的根瘤菌仅1株为未确定的种sp.。本文在中国东北地区发现紫花苜蓿根瘤菌较丰富的种间多样性可能与共生基因在逆境胁迫下发生基因的水平转移有关。结瘤和固氮基因可在根瘤菌种间进行水平转移,是根瘤菌进化的重要机制[27]。基因的水平转移使一些腐生的根瘤菌获得与豆科植物共生结瘤以及固氮的能力,并形成与寄主的协同进化。而在逆境胁迫下,根瘤菌受到的选择压力可能使得基因水平转移现象更为频繁[28-29]。东北地区的根瘤菌大多采自盐碱地,这为共生基因在根瘤菌间的水平转移创造了逆境条件。通过与紫花苜蓿共生的各根瘤菌基因组的比较分析将有助于揭示中国东北地区紫花苜蓿根瘤菌种间多样性的分子机制;同时加大逆境胁迫环境条件下苜蓿根瘤菌的采样和分析有助于更清楚地了解环境胁迫在根瘤菌的基因水平转移中所发挥的作用[30]。
本文采用了3个持家基因(、和)和2个共生基因(和)对供试紫花苜蓿根瘤菌多样性进行了系统的分析。目前,可用于根瘤菌多样性分析的持家基因很多,但有很多研究报道不同持家基因揭示多样性的能力存在差别[5]。本研究结果显示,3种持家基因在进行紫花苜蓿种间多样性的系统发育分析时具有一致的效果,但在种内多样性的分析时则存在部分不一致的结果(图1)。当多基因联合分析时,种内多样性呈现比较杂乱的状态,因此,本研究没有将3种持家基因进行多基因联合分析。研究表明是中国东北和华北地区优势根瘤菌,未来需要进一步评估各种分子标记在根瘤菌种内多样性研究中的作用。随着测序技术的进步和测序费用的下降,根瘤菌染色体基因组范围内的SNPs分子标记的开发和利用也将有助于紫花苜蓿根瘤菌种内多样性的分析及根瘤菌种内分化机制的揭示[31-32]。根瘤菌种内多样性的分析对筛选高效结瘤、固氮和促生的根瘤菌应用于紫花苜蓿的生产具有重要的意义。本研究的共生基因系统发育结果表明,共生基因在一些根瘤菌中发生了属内和属间的水平转移现象,和的与的具有一样的系统发育分支(图2)。根瘤菌属内共生基因水平转移常见报道,但属间则报道较少[27]。此外,本研究还发现保留了其和,但仍可与紫花苜蓿进行结瘤共生。根瘤菌早期被认为是与紫云英(与紫花苜蓿结瘤发生共生的原因[34]。
本研究还发现了2种与紫花苜蓿结瘤共生的潜在新种(sp.和sp.),需要通过进一步的分析以明确其分类地位[35]。本研究明确了中国东北和华北部分地区紫花苜蓿根瘤菌的遗传信息,获得了丰富的种间和种内多样性的根瘤菌菌株,可为高效根瘤菌剂的筛选和应用提供有价值的菌种资源和参考信息。此外,在逆境胁迫条件下(如盐碱),紫花苜蓿根瘤菌种间和种内的共生效率及其相互之间的竞争或合作关系,仍不清楚。厘清根瘤菌种间及种内互作机制,是根瘤菌高效应用的重要环节,未来可以充分利用本研究获得的盐碱地中与紫花苜蓿结瘤共生的多个根瘤菌种以及优势种丰富的种内多样性,开展相关的研究,为盐碱地高效根瘤菌剂的筛选和利用提供参考。
4 结论
中国东北和华北地区紫花苜蓿根瘤菌具有较丰富的遗传多样性,且存在显著的地理分布特征及寄主品种选择现象。为优势根瘤菌种,占总数的97.60%。其余12株分别为、、、和,以及2个潜在的新种(sp.和sp.)均分布于东北地区。根瘤菌属间和属内发生了共生基因的水平转移现象。
[1] 徐丽君, 徐大伟, 逄焕成, 辛晓平, 金东艳, 唐雪娟, 郭明英. 中国苜蓿属植物适宜性区划. 草业科学, 2017, 11(11): 2347-2358.
Xu L J, Xu D W, Pang H C, Xin X P, Jin D Y, Tang X J, Guo M Y. Chinese alfalfa habitat suitability regionalization. Pratacultural Science, 2017, 11(11): 2347-2358. (in Chinese)
[2] 郭婷, 薛彪, 白娟, 孙启忠. 刍议中国牧草产业发展现状: 以苜蓿、燕麦为例. 草业科学, 2019, 36(5): 1466-1474.
GUO T, XUE B, BAI J, SUN Q Z. Discussion of the present situation of China’s forage grass industry development: An example using alfalfa and oats. Pratacultural Science, 2019, 36(5): 1466-1474. (in Chinese)
[3] 刘鹏, 田颖哲, 钟永嘉, 廖红. 酸性土壤上花生高效根瘤菌的分离及应用. 中国农业科学, 2019, 52(19): 3393-3403.
LIU P, TIAN Y Z, ZHONG Y J, LIAO H. Isolation and application of effective rhizobium strains in peanut on acidic soils. Scientia Agricultura Sinica, 2019, 52(19): 3393-3403. (in Chinese)
[4] 苗阳阳, 师尚礼, 康文娟. 赤霉素对根瘤菌运移、定殖及苜蓿幼苗生长的影响. 中国农业科学, 2017, 50(23): 4545-4557.
MIAO Y Y, SHI S L, KANG W J. Effects of gibberellin on migration and colonization of rhizobia and seedling growth of alfalfa. Scientia Agricultura Sinica, 2017, 50(23): 4545-4557. (in Chinese)
[5] 管凤贞, 邱宏端, 陈济琛, 林新坚. 根瘤菌菌剂的研究与开发现状. 生态学杂志, 2012, 31(3): 755-759.
GUAN F Z, QIU H D, CHEN J C, LIN X J. Rhizobium inoculants: Research progress and development status. Chinese Journal of Ecology, 2012, 31(3): 755-759. (in Chinese)
[6] 张荣娟. 中国西部苜蓿根瘤菌的生物地理分布及其抗逆机理研究[D]. 北京: 中国农业大学, 2011.
ZHANG R J. Biogeography and stress adaptation mechanisms ofassociated within Western China [D]. Beijing: China Agricultural University, 2011. (in Chinese)
[7] KANG W J, SHI, S L, XU L. Diversity and symbiotic divergence of endophytic and non-endophytic rhizobia of. Annals of Microbiology, 2018, 68: 247-260.
[8] 张小甫. 甘肃不同生态区域苜蓿根瘤菌筛选菌株常规鉴定与分子鉴定[D]. 兰州: 甘肃农业大学, 2008.
ZHANG X F. Identification of alfalfa rhizobium strains and analysis of genetic diversity in different ecological regions in Gansu [D]. Lanzhou: Gansu Agricultural University, 2008. (in Chinese)
[9] 冯春生. 西北地区天蓝苜蓿根瘤菌系统发育研究[D]. 杨凌: 西北农林科技大学, 2008.
FENG C S. Phylogeny of rhizobia isolated fromin Northwest of China [D]. Yangling: Northwest Agricultural and Forestry University, 2008. (in Chinese)
[10] 位秀丽, 付芸芸, 韦革宏. 西北部分重金属矿区天蓝苜蓿根瘤菌生理生化特性及16S rDNA PCR-RFLP分析. 干旱地区农业研究, 2009, 27(2): 223-226.
WEI X L, FU Y Y, WEI G H. Physiological and biochemical characters and 16S rDNA PCR-RFLP of rhizobia isolated fromin mining regions of Northwest China. Agricultural Research in the Arid Areas, 2009, 27(2): 223-226. (in Chinese)
[11] 葛勇. 川西高寒地区苜蓿和无翅山黧豆根瘤菌遗传多样性及系统发育地位研究[D]. 成都: 四川农业大学, 2013.
GE Y. Genetic diversity and phylogeny of rhizobia isolated fromandin the west plateau of Sichuan, China [D]. Chengdu: Sichuan Agricultural University, 2013. (in Chinese)
[12] STEFAN A, CAUWENBERGHE J V, ROSU C M, STEDEL C. LABROU N, FLEMETAKIS E, EFROSE R C. Genetic diversity and structure ofpopulations associated with clover plants are influenced by local environmental variables. Systematic and Applied Microbiology, 2018, 41(3): 251-259.
[13] WANG X L, CUI W J. FENG X Y, ZHONG Z M, LI Y, CHEN W X, CHEN W F, SHAO X M, TIAN C F. Rhizobia inhabiting nodules and rhizosphere soils of alfalfa: A strong selection of facultative microsymbionts. Soil Biology and Biochemistry, 2018, 116: 340-350.
[14] VERSALOVIC J, SCHNEIDER M, DE BRUIJN F J, LUPSKI J R. Genomic fingerprinting of bacteria using repetitive sequence-based polymerase chain reaction. Methods in Molecular and Cellular Biology, 1994, 5: 25-40.
[15] VINUESA P, SILVA C, WERNER D, MARTINEZ-ROMERO E. Population genetics and phylogenetic inference in bacterial molecular systematics: the roles of migration and recombination inspecies cohesion and delineation. MolecularPhylogenetics and Evolution, 2005, 34(1): 29-54.
[16] VINUESA P, ROJAS-JIMENEZ K, CONTRERAS-MOREIRA B, MAHNA S K, PRASAD B N, MOE H, SELVARAJU S B, THIERFELDER H, WERNER D. Multilocus sequence analysis for assessment of the biogeography and evolutionary genetics of fourspecies that nodulate soybeans on the Asiatic Continent. Applied and Environmental Microbiology, 2008, 74(22): 6987-6996.
[17] SARITA S, SHARMA P K, PRIEFER, U B, PRELL J. Direct amplification of rhizobialsequences from soil total DNA and comparison todiversity of root nodule isolates. FEMS Microbiology Ecology, 2005, 54(1): 1-11.
[18] YAO Y, WANG R, LU J K, SUI X H, WANG E T, CHEN W X. Genetic diversity and evolution ofpopulations nodulating, an evergreen tree indigenous to the southern subtropical region of China. Applied Environmental Microbiology, 2014, 80(19): 6184-6194.
[19] LIU J, WANG E T, CHEN W X. Diverse rhizobia associated with woody legumes,andgrown in the temperate zone of China. Systematic and Applied Microbiology, 2005, 28(5): 465-477.
[20] ZHANG X X, GUO H J, JIAO J, ZHANG P, XIONG H Y, CHEN W X, TIAN C F. Pyrosequencing ofuncovers a significant biogeographical pattern of rhizobial species in soybean rhizosphere. Journal of Biogeography, 2017, 44(7): 1491-1499.
[21] XIONG H Y, ZHANG X X, GUO H J, JI Y Y, LI Y, WANG X L, ZHAO W, MO F Y, CHEN J C, YANG T, ZONG X X, CHEN W X, TIAN C F. The epidemicity of facultative microsymbionts in faba bean rhizosphere soils. Soil Biology and Biochemistry, 2017, 115: 243-252.
[22] BROWN S P, GRILLO M A, PODOWSKI J C, HEATH K D. Soil origin and plant genotype structure distinct microbiome compartments in the model legume. Microbiome, 2020, 8: 139.
[23] WAGNER M R, LUNDBERG D S, DEL RIO T G, TRINGE S G, DANGL J, MITCHELL-OLDS T. Host genotype and age shape the leaf and root microbiomes of a wild perennial plant. Nature communications, 2016, 7: 12151.
[24] PORTER S S, CHANG P L, CONOW C A, DUNHAM J P, FRIESEN M L. Association mapping reveals novel serpentine adaptation gene clusters in a population of symbiotic. The ISME Journal, 2017, 11(1): 248-262.
[25] TALEBI M B, BAHAR M, SAEIDI G, MENGONI A, BAZZICALUPO M. Diversity ofstrains nodulatingfrom different Iranian regions. FEMS Microbiology Letters, 2008, 288(1): 40-46.
[26] SILVA C, KAN F L, MARTÃNEZ-ROMERO E. Population genetic structure ofandisolated from nodules ofspp. in Mexico. FEMS Microbiology Ecology, 2007, 60(3): 477-489.
[27] LING J, WANG H, WU P, LI T, TANG Y, NASEER N, ZHENG H M, MASSON-BOIVIN C, ZHONG Z T, ZHU J. Plant nodulation inducers enhance horizontal gene transfer ofsymbiosis island. Proceedings of the National Academy of Sciences of the United States of America, 2016, 113(48): 13875-13880.
[28] XIE J B, DU Z L, BAI L Q, TIAN C F, ZHANG J Z, XIE J Y, WANG T S, LIU X M, CHEN X, CHENG Q, CHEN S F, LI J L. Comparative genomic analysis of N2-fixing and non-N2-fixingspp.: organization, evolution and expression of the nitrogen fixation genes. PLoS Genetics, 2014, 10(3): e1004231.
[29] TIAN C F, ZHOU Y J, ZHANG Y M, LI Q Q, ZHANG Y Z, LI D F, WANG S, WANG J, GILBERT L B, LI R Y, CHEN W X. Comparative genomics of rhizobia nodulating soybean suggests extensive recruitment of lineage-specific genes in adaptations. Proceedings of the National Academy of Sciences of the United States of America, 2012, 109(22): 8629-8634.
[30] TORO N, MARTINEZ-ABARCA F, MOLINA-SANCHEZ M D, GARCIA-RODRIGUEZ F M, NISA-MARTINEZ RAFAEL. Contribution of mobile group II introns togenome evolution. Frontier in Microbiology, 2018, 9: 627.
[31] ACOSTA J L, EGUIARTE L E, SANTAMARIA R I, BUSTOS P, VINUESA P, MARTINEZ-ROMERO E, DAVILA G, GONZALEZ V. Genomic lineages ofrevealed by the extent of nucleotide polymorphisms and low recombination. BMC Evolutionary Biology, 2011, 11: 1-13
[32] EPSTEIN B, ABOU-SHANAB R A I, TAYLOR M R, GUHLIN J, BURGHARDT L T, NELSON M, SADOWSKY M J, TIFFIN P. Genome-wide association analyses in the model rhizobium. mSphere, 2018, 3(5): e00386- e00418.
[33] WANG S W, HAO B H, LI J R, GU H L, PENG J L, XIE F L, ZHAO X Y, FRECH C, CHEN N S, MA B G, LI Y G. Whole-genome sequencing of7653R provides molecular insights into host specificity and symbiosis island dynamics. BMC Genomics, 2014, 15(1): 440.
[34] REMIGI P, ZHU J, YOUNG J P W, MASSON-BOIVIN C. Symbiosis within symbiosis: evolving nitrogen-fixing legume symbionts. Trends in Microbiology, 2016, 24(1): 63-75.
[35] REJILI M, OFF K, BRACHMANN A, MARIN M.sp. nov., isolated fromgrowing in the northern region of Tunisia. International Journal of Systematic and Evolutionary Microbiology, 2020, 70(10): 5539-5550.
Genetic Diversity Analysis of Rhizobia Associated withCultivated in Northeast and North China
LI Xinyuan, LOU JinXiu, LIU QingYuan, HU Jian, ZHANG YingJun
College of Agro-grassland Science, Nanjing Agricultural University, Nanjing 210095
【】is reputed to be the “Queen of Forages”, its cultivated area has increased in Northeast and North China in recent years. However, the quality and yield ofstill do not meet the needs of livestock husbandry development in China. The objective of this study was to analyze the genetic diversity of rhizobia associated withfrom Northeast and North China, which will provide valuable reference for screening and applying high efficient nitrogen fixing rhizobia from【】Surface sterilization and plate streaking method were used to recover and purify the rhizobia fromroot nodules; BOX-PCR was applied to identify the genotypes of the tested rhizobia; Three housekeeping (,and) and two symbiotic genes (and) were selected for phylogenetic analysis of the representative genotypes. 【】A total of 499 rhizobia strains were obtained from 19 sampling regions located in Northeast and North China. All strains were classified into 37 BOX types. The BOX types showed a strong geographic distribution, and host cultivars imposed a certain selection pressure to the genotypes of rhizobia. 97.60% (487/499) of the strains was identified as, while the rest 12 strains were identified as,,,,,sp. andsp.. All the 12 strains were collected from Northeast China, while all strains from North China were. The housekeeping genes revealed similar phylogenetic trends when they were used for inter-species analysis, but revealed differently for intra-species analysis. Phylogeny of symbiotic genes revealed that horizontal gene transfer happened between genera and species within genus.showed high genetic diversity thaninstrains.【】 Rhizobia associated withfrom Northeast and North China showed high genetic diversity, moreover, they had a strong characteristics of geographic distribution and host selection.
;; genetic diversity; phylogeny; horizontal transfer

10.3864/j.issn.0578-1752.2021.16.003
2021-02-03;
2021-05-02
中央高校基本科研业务费(KJQN201836)
李昕芫,E-mail:nalxy0806@163.com。通信作者胡健,E-mail:jaffyhu@njau.edu.cn
(责任编辑 李莉)
