Microbulbifer sp. A4B-17菌株的磷酸烯醇式丙酮酸羧激酶与烯醇化酶研究
2021-08-02刘曹彤陆依琳徐慧彭学
刘曹彤 陆依琳 徐慧 彭学
摘要:对羟基苯甲酸(4-Hydroxybenzoate,4HBA)应用广泛,在工业领域上被当作前体合成各种芳香族化合物,其中包括液晶材料、农药等;在食品和化妆品等领域上被当作防腐剂。微泡菌属(Microbulbifer sp.) A4B-17是一种能将葡萄糖合成4HBA的海洋细菌,为了提高4HBA的合成效率对合成莽草酸途径前体的2个关键酶:GM004356编码的磷酸烯醇式丙酮酸羧激酶(Phosphoenolpyruvate carboxykinase,PEPCK)和GM0031333编码的烯醇化酶(Enolase)进行研究。通过构建蛋白系统进化树发现,PEPCK与Microbulbifer sp. GL-2的PEPCK相似度为95.15%,Enolase与Microbulbifer variabilis的Enolase相似度为98.13%。利用低温表达载体在大肠杆菌中对2个酶进行高效表达,提取和纯化后,采用BCA法蛋白定量计算后得到纯化后的PEPCK和Enolase分别占各自粗酶液的1.304%和1.123%。酶活采用PK/LDH(丙酮酸激酶/乳酸脱氢酶)偶联检测法检测NADPH的减少量。根据米氏常数双倒数法求得PEPCK和Enolase的Km值分别为0.041 mmol/L和0.056 mmol/L,PEPCK反应的最适温度为30 ℃,最适pH值为7,Enolase的最适温度为 30 ℃,最适pH值为7。以上结果为高效生产4HBA提供了理论依据。
关键词:对羟基苯甲酸;磷酸烯醇式丙酮酸;烯醇化酶;磷酸烯醇式丙酮酸羧激酶
中图分类号:S188+.3 文献标志码: A
文章编号:1002-1302(2021)12-0045-06
收稿日期:2020-11-04
基金项目:江苏师范大学科研创新计划校立项目(编号:2020XKT485)。
作者简介:刘曹彤(1996—),女,江苏淮安人,硕士研究生,主要从事环境微生物学研究。E-mail:3220859786@qq.com。
通信作者:彭学,博士,教授,主要从事环境微生物学研究。E-mail:pengxue@jsnu.edu.cn。
对羟基苯甲酸(4-Hydroxybenzoate,4HBA)及其酯类物质,不仅能抑制真菌和细菌的生长,还能调节一些酶的代谢,应用非常广泛[1],覆蓋了衣食住行各个方面,比如用于食品与化妆品的防腐剂、农业中的杀菌剂与化肥、手机电脑等的液晶制品等方面[2-6]。4HBA的毒性较低,抑菌效果也好,所以在2002年,对羟基苯甲酸酯类物质已经被我国批准作为食品防腐剂添加使用[7]。
目前大部分4HBA通过石油分馏[8]得到,也有报道利用植物[9]或者重组大肠杆菌[10]合成4HBA,但是这些方法存在很多的缺陷,植物合成得到的4HBA提纯难度大[11]。微泡菌属(Microbulbifer sp.) A4B-17菌株是能将葡萄糖合成4HBA及其酯类的海洋细菌,笔者所在课题组已经完成了A4B-17菌株的基因组测序和注释[12]。合成4HBA的途径有2条:葡萄糖降解途径和莽草酸途径,在后者途径中磷酸烯醇式丙酮酸(PEP)和4-磷酸赤藓糖缩合反应生成3-脱氧-D-阿拉伯庚酮糖酸-7-磷酸合成酶(DAHP),PEP的合成量较低成为莽草酸途径的限速步骤。A4B-17菌株合成PEP的途径有2条:2-酮-3-脱氧-6-磷酸葡糖酸途径(ED途径)和糖酵解途径(EMP途径)。在ED途径中,磷酸烯醇式丙酮酸羧激酶(PEPCK)将草酰乙酸(OAA)脱羧生成PEP;在EMP途径中烯醇化酶(Enolase)将2-磷酸甘油酸(OAA)脱水生成PEP。EMP途径和ED途径[13]的共同点是将合成PEP作为通路的最后一步反应。同时在之后的合成途径中,PEP作为起始物质,4HBA及其酯类合成多少完全取决于它的积累量[14]。可以说要想大量合成4HBA及其酯类,就需要大量合成PEP。A4B-17菌株的PEPCK和Enolase这2个酶分别由编号为GM004356和GM001333的基因编码,而且在该细菌体内,有且只有这2种酶能够合成PEP,因此对这2个基因编码的蛋白酶的研究是非常有必要的。对这2个酶的研究不仅能了解它们的理化性质,还能够为提高PEP的产量提供理论基础,从而为提高4HBA的产量打下基础[15]。
1 材料与方法
1.1 试验材料
Microbulbifer sp. A4B-17菌株(由笔者所在课题组从海水中分离得到,并保存于江苏师范大学生命科学学院);大肠杆菌DH5α(笔者所在实验室长期保存于 -80 ℃ 超低温冰箱中);pET-21a(+)载体(购买自武汉淼灵生物科技有限公司)。
1.2 试验试剂
Q5 High-Fidelity DNA Polymerase,5×Q5 High GC Enhancer(optional),5×Q5 Reaction Buffer,dNTP Mix,dd Water,LB肉汤,100 mg/mL Ampicillin(Amp)母液,50×TAE Buffer(pH值8.5),1%琼脂糖凝胶,DNA marker,DNA loading buffer,GoldView核酸染料,质粒提取相关试剂[16],磷酸缓冲液PBS母液(0.2 mol/L),0.1 mol/L异丙基-β-D-硫代半乳糖苷(IPTG),30% (质量浓度)丙烯酰胺溶液(Acr-Bis),10%(质量浓度)过硫酸铵,5×Tris-Glycine Buffer(电泳缓冲液),1 mol/L Tris-HCl(pH值为6.8),1.5 mol/L Tris-HCl(pH值为8.8),10% SDS考马斯亮蓝R-250染色液,考马斯亮蓝脱色液,5×SDS-PAGE loading Buffer,β-巯基乙醇(β-ME),蛋白纯化试剂盒等。
1.3 GM004356和GM001333编码基因的克隆与鉴定
1.3.1 目的片段的扩增
首先分别设计GM004356和GM001333这2个目的基因的引物,引物设计见表1,引物序列交由上海捷瑞生物工程有限公司合成。设计PCR反应体系,加入PCR管中,混匀,放入PCR仪,设置好反应条件,运行程序。35次循环。
1.3.2 载体酶切
选择pET-21a(+)载体,大小为5 443 bp,酶切位点为BamHⅠ和XhoⅠ,采用双酶切,载体酶切后,需要将目的基因与载体进行连接,形成重组质粒。选择的酶连试剂盒为Ready-to-Use Seamless Cloning Kit。总反应体系为 50 μL,混合均匀后于37 ℃水浴5 min。
1.3.3 阳性转化子质粒提取
将重组质粒转入大肠杆菌,培养结束后,挑取部分单菌落进行液体培养,然后利用十二烷基硫酸钠(SDS)碱裂法提取质粒。
1.3.4 阳性转化子酶切鉴定
将酶切体系加到PCR管中,混合均匀,放入37 ℃水浴锅中孵育5~10 min,然后琼脂糖凝胶电泳验证结果。
1.4 PEPCK和Enolase的表达与酶活探究
1.4.1 粗酶提取
将活化好的菌落接入含氨苄青霉素(Amp)的LB三角瓶中,37 ℃,180 r/min,培养至吸光度为0.4~0.6,再加入异丙基-β-D-硫代半乳糖苷(IPTG)低温低速诱导培养12~24 h,使目的蛋白充分表達。将培养好的菌液5 000 r/min,低温离心10 min,弃上清,用pH值为7.4的PBS缓冲液(磷酸缓冲盐溶液)重悬细胞,再离心,再重悬,用超声波破碎仪破碎细胞,4 ℃ 离心,上清即为粗酶混合液。
1.4.2 蛋白纯化
提取出的粗酶中,绝大部分是不需要的杂蛋白,想要研究目的蛋白的性质,需要将目的蛋白提取出来,选择蛋白纯化试剂盒进行纯化。为了给后续试验提供一个定量结果,需要计算出粗酶中PEPCK和Enolase的含量。采用BCA(聚氰基丙烯酸正丁酯)蛋白定量法,根据吸光度与蛋白浓度成正比,绘制蛋白浓度标准曲线,从而能够计算出目的蛋白占粗酶的百分比。
1.4.3 PEPCK和Enolase酶活检测方法
PEPCK和Enolase酶活检测方法采用PK(丙酮酸激酶)/LDH(乳酸脱氢酶)偶联检测法,即将不方便检测的磷酸烯醇式丙酮酸,经PK和LDH催化后,生成丙酮酸,然后与还原型辅酶Ⅱ(NADPH)反应生成NADP+,而NADPH容易被检测,NADPH浓度的减少量与PEPCK和Enolase的活性成正相关,通过测定在340 nm处NADPH吸光度的减少,来反映酶的活性。
根据PK/LDH偶联检测法探究2个酶的最适温度和最适pH值。研究PEPCK的最适温度,要控制pH值、反应时间、调控剂不变。设置不同反应温度,固定pH值不变;研究PEPCK最适pH值时,要控制温度、反应时间、调控剂不变,各反应成分与研究最适温度时一致,只是改变缓冲液的pH值不同。用同样的方法来探究Enolase的最适温度和最适pH值。
2 结果与分析
2.1 系统进化树分析
在Microbulbifer sp. A4B-17菌株中找到编号为GM004356和GM001333的全部碱基序列,经NCBI数据库比对分析,确定这2个基因分别编码PEPCK和Enolase。GM004356编码的PEPCK与Microbulbifer sp. GL-2、Microbulbifer sp. THAF38和Microbulbifer variabilis编码的PEPCK相似度较高,相似度都为95.15%,系统进化树见图1。GL-2菌株分离自硬骨鱼肠道中[17],是一个新的降解纤维素的菌株。Microbulbifer variabilis分离自太平洋海藻,能够产生抗癌吩嗪抗生素[18]。关于THAF38菌株还没有相关报道。GM001333编码的Enolase与Microbulbifer variabilis和Microbulbifer sp. THAF38的Enolase相似度较高,相似度都为98.13%,系统进化树见图2。
2.2 高效表达质粒的构建
2个目的基因GM004356和GM001333分别进行PCR,琼脂糖凝胶成像分析(图3)。选用pET-21a(+)载体,采用双酶切,与目的基因相连,构成重组质粒,导入大肠杆菌体内。通过验证,获得阳性转化子,并测序,与原始目的基因比对,发现重组质粒中的目的基因未发生突变或缺失,与原始基因完全一致,可以利用该菌落进行后续的蛋白研究。
2.3 蛋白质的提取和纯化
低温低速诱导目的蛋白表达,提取粗酶,电泳初步判断转入大肠杆菌的目的基因成功表达,接着对粗酶进行纯化,电泳验证纯化结果,发现目的蛋白PEPCK和Enolase均能被纯化,且效果明显,可以进行后续的定性分析,具体结果见图4和图5。
将粗酶液稀释10倍后用BCA法测得样品吸光度为2.526 4,通过标准曲线, 计算出粗酶液中蛋白总浓度为3.298 mg/mL。纯化之后目的蛋白PEPCK吸光度为0.337,根据标准曲线,计算出纯化后的PEPCK浓度为0.043 mg/mL,占粗酶液的1.304%。同样的方法,得到Enolase占粗酶液的1.123%。
2.4 PEPCK和Enolase米式常数的研究
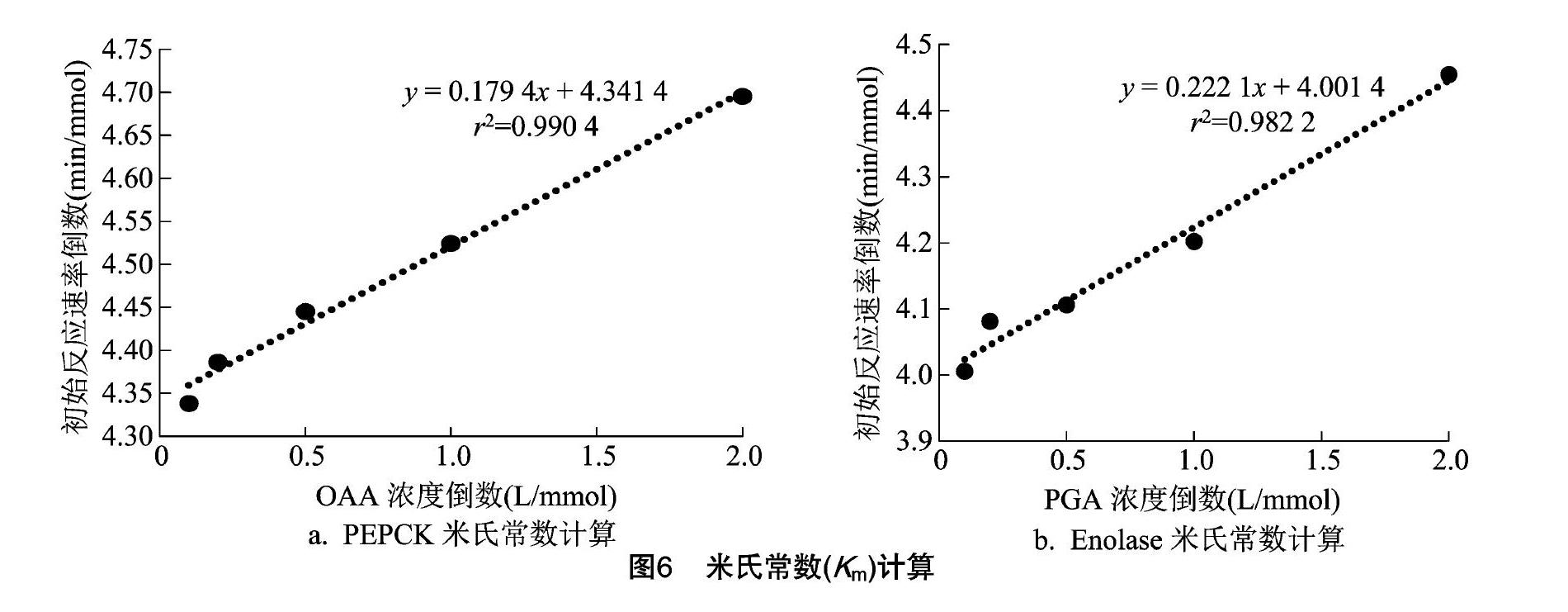
米氏常数(Km)定义为当酶促反应达最大速度(vm)一半时底物(S)的浓度。对Km的研究,能揭示其在参加反应时的重要性质,为该酶应用到工业生产中提供重要的理论依据。根据米氏常数双倒数求法的计算公式:1/v=Km/(vmax[S])+1/vmax,可以看成y=ax+b的形式,需要做出初始反应速率的倒数,以及反应底物浓度的倒数,设定反应时间为 2 min,底物浓度足够大。因为当y=0时,横截距即为-1/Km,得到PEPCK的Km值为0.041 mmol/L,如图6-A所示。Enolase的Km值为0.056 mmol/L,如图6-B所示。2个Km值都较低,说明PEPCK与OAA,Enolase与底物2-磷酸甘油(PGA)的酶促反应很容易进行,从而转化生产磷酸烯醇式丙酮酸(PEP),因此更有利于重要中间产物PEP在菌体中的积累。
2.5 最适温度和最适pH值
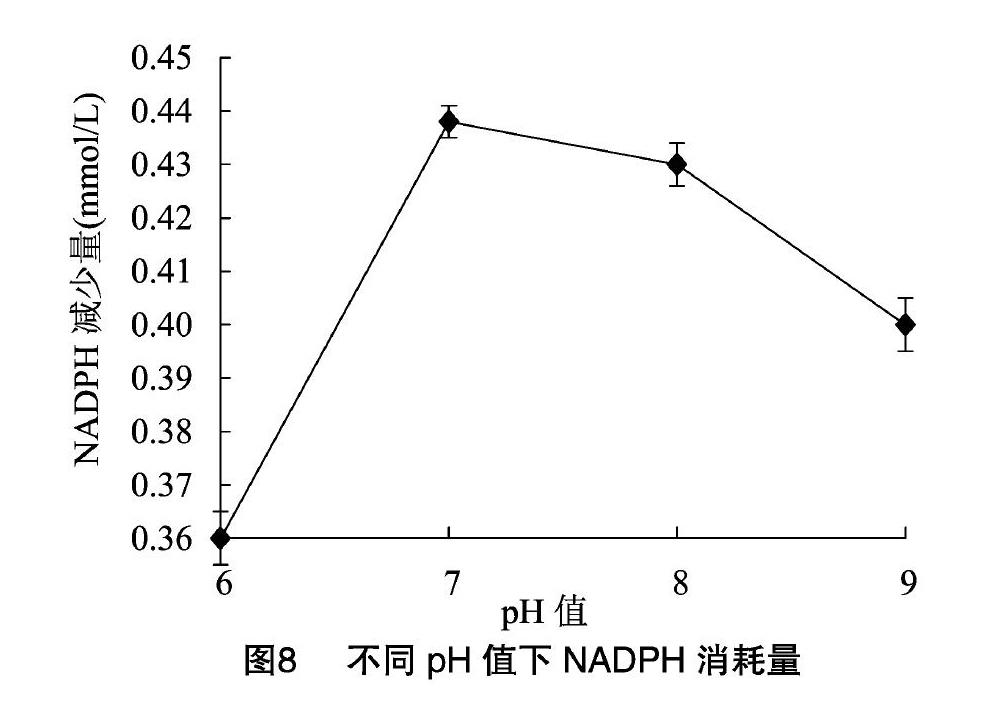
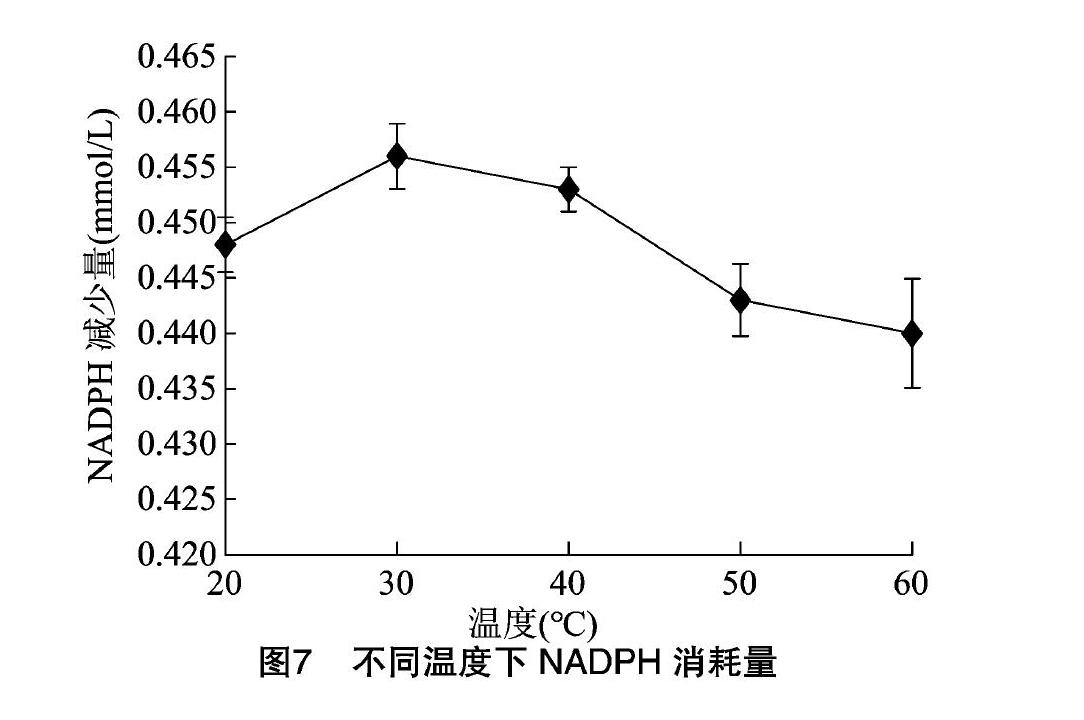
探究PEPCK的最适温度,设置不同反应温度,固定pH值保持不变,反应结束后测定NADPH的吸光度,结果见图7,发现在30 ℃左右时,PEPCK活性最高。探究最适pH值时,设置不同pH值,保持温度为30 ℃,发现pH值为7时酶活最高(图8)。
同样的方法,分别设置Enolase的反应温度和缓冲液pH值,发现Enolase的最适反应温度为30 ℃,最适pH值为7,结果如图9和图10所示。
3 讨论与结论
Microbulbifer sp. A4B-17菌株的特点是本身就可以合成对羟基苯甲酸及其酯类,而且产量较高,不会产生对人体有毒有害的物质。本研究对细菌细胞内编号为GM004356和GM001333的2个目的基因进行转化克隆,高效表达PEPCK和Enolase,通过提取纯化计算出纯化之后目的蛋白PEPCK占粗酶液的1.304%,纯化后的Enolase占粗酶液的1.123%。PEPCK酶反应的最适温度为30 ℃。最适pH值为7;Enolase的最适温度为30 ℃,最适pH值为7。PEPCK和Enolase的米氏常数分别是0.041、0.056 mmol/L,较低的Km值更有利于酶促反应的快速进行。通过蛋白系统进化树分析,PEPCK与Microbulbifer sp. GL-2的PEPCK相似度为95.15%;Enolase与Microbulbifer variabilis的Enolase相似度为98.13%。本研究对PEPCK和Enolase 2种酶进行深入研究,为将来2个酶的优化研究建立标准。笔者所在课题组期望通过对该菌株的深入研究,改变传统的生产方式,利用可再生资源合成4HBA等工业原料。
本试验所采用的酶活性研究方法均为PK/LDH酶偶联法,该方法虽操作简单,思路清晰,但是在偶联法中,PK以及LDH的活性会对整个试验结果产生较大影响,而且最终得到的米氏常数其实是目的蛋白酶与PK、LDH共同作用的结果。目前对PEP的检测暂无有效可行的方法,开辟一种新的检测方法还需要大量工作,后续可以在本研究的基础上进行拓展研究。
参考文献:
[1]郭 阳,臧 埔,郜 玉,等. 化妆品防腐剂的使用现状及进展[J]. 中南药学,2018,16(9):1258-1263.
[2]石金娥,刘静秋,尚淑霞,等. 对羟基苯甲酸酯类防腐剂在酱油、食醋中应用状况分析[J]. 中国调味品,2011,36(7):11-12.
[3]李欣航,张金龙,毕永贤,等. 无尼泊金酯类化妆品防腐体系的应用研究[J]. 日用化学品科学,2018,41(8):14-21.
[4]陈建文,厉华明,周荣荣. 食品中对羟基苯甲酸酯类的应用现状与检测方法[J]. 中国酿造,2008(8):4-5.
[5]胡 梅. 化妆品防腐及防腐体系的构建[C]//第九届中国化妆品学术研讨会论文集. 北京:品观网,2012:105-107.
[6]朱 莉,许超艳,李晶晶,等. 生物合成对羟基苯甲酸的研究进展[J]. 生物工程学报,2015,31(3):328-337.
[7]林忠洋,马万里,齐 迹,等. 对羟基苯甲酸酯类防腐剂的人体暴露[J]. 化学进展,2015,27(5):614-622.
[8]Okai N,Miyoshi T,Takeshima Y,et al. Production of protocatechuic acid by Corynebacterium glutamicum expressing chorismate-pyruvate lyase from Escherichia coli[J]. Applied Microbiology and Biotechnology,2016,100(1):135-145.
[9]董秀梅,晁 青,王柏臣. 植物磷酸烯醇式丙酮酸羧激酶(PEPCK)研究进展[J]. 植物学报,2013,48(3):320-328.
[10]Zhang H R,Pereira B,Li Z J,et al. Engineering Escherichia coli coculture systems for the production of biochemical products[J]. Proceedings of the National Academy of Sciences of the United States of America,2015,112(27):8266-8271.
[11]Sommer S,Heide L. Expression of bacterial chorismate pyruvate-lyase in tobacco:evidence for the presence of chorismate in the plant cytosol[J]. Plant & Cell Physiology,1998,39(11):1240-1244.
[12]Tian J,Zhu L,Wang W J,et al. Genomic analysis of Microbulbifer sp. strain a4B-17 and the characterization of its metabolic pathways for 4-hydroxybenzoic acid synthesis[J]. Frontiers in Microbiology,2018,9:3115.
[13]張丽平,汪文君,李 智,等. 对羟基苯甲酸甲酯降解菌的初步研究[J]. 江苏农业科学,2018,46(20):337-340.
[14]郭 晶,高菊芳,唐振华. 羧酸酯酶及其在含酯类化合物代谢中的作用[J]. 农药,2007,46(6):365-368.
[15]Fiske M J,Whitaker R J,Jensen R A. Hidden overflow pathway to L-phenylalanine in Pseudomonas aeruginosa[J]. Journal of Bacteriology,1983,154(2):623-631.
[16]李载平. 分子克隆实验指南[J]. 科学通报,2002,47(24):1888.
[17]Sugimoto Y,Ohnishi K I,Suzuki S. Complete genome sequence of cellulase-producing Microbulbifer sp. strain GL-2,isolated from marine fish intestine[J]. Microbiology Resource Announcements,2020,9(32):720-746.
[18]Nishijima M,Takadera T,Imamura N,et al. Microbulbifer variabilis sp. nov. and Microbulbifer epialgicus sp. nov.,isolated from Pacific Marine algae,possess a rod-coccus cell cycle in association with the growth phase[J]. International Journal of Systematic and Evolutionary Microbiology,2009,59(7):1696-1707.
