编码酿酒酵母丙酮酸脱羧酶(Pdc6)基因克隆及其生物信息学分析
2021-04-27王长丽叶广彬葛菁萍马毓坚宾晓芸
王长丽,廖 巍,叶广彬,葛菁萍,刘 磊,马毓坚,黄 霞,宾晓芸
(1右江民族医学院,广西百色533000;2黑龙江大学生命科学学院微生物省高校重点实验室,哈尔滨150080;3农业微生物技术教育部工程研究中心,哈尔滨150500)
0 引言
近年来,通过基因工程和代谢工程的手段改造野生型酿酒酵母菌株积累丙酮酸和生产高级醇被广泛关注,酿酒酵母(Saccharomyces cerevisiae)具有较高潜在应用价值,它不仅是GRAS模式生物,且自身具有工业化生产丙酮酸和高级醇的优势。临床研究证实[1],丙酮酸在治疗和预防因碳水化合物和能量代谢紊乱引起的糖尿病、心脑血管疾病等疾病上效果显著。但野生型酵母积累丙酮酸较少、代谢高级醇能力不强,故基因工程策略成为关注热点[2-3]:一方面增加缬氨酸生物合成的支链高级醇的产量,过表达由丙酮酸合成高级醇代谢途径的关键酶基因;另一方面,抑制或消除乙醇的通量,积累丙酮酸。无论哪种途径,丙酮酸脱羧酶(pyruvate decarboxylase,Pdc)活性在整个代谢路径中,都起着决定性作用。众所周知,有3个编码Pdc的结构基因(pdc1、pdc5、pdc6),前2个基因在基因工程改造过程中被广泛关注,而pdc6基因自身表达较弱,往往被研究者所忽略[4]。因此,克隆酵母细胞中pdc6基因并对该基因进行生物信息学分析,可以从基因序列和Pdc6蛋白结构预测上阐明其在影响Pdc酶活上的理论依据。
李亿等[5]发现,XY-6(△pdc6)菌株与原始菌株相比,Pdc酶活力为对照组的92%,乙醇合成不受影响、丙酮酸积累少。刘磊等[6]发现,△pdc5菌株与对照组相比,乙醇产量下降了17.54%。吴满珍等[3]发现,△pdc1△pdc1菌株与原始菌株相比,丙酮酸积累达2.0g/L,乙醇产量降低了63.8%。Lian等[7]构建的△pdc6△pdc5△pdc1菌株,无乙醇合成,高级醇积累显著增高。故可以证明酿酒酵母细胞中,单个结构基因的缺失,确实在不同程度上影响Pdc酶活力[8],但当单基因缺失菌株因生存环境改变时,三者又是如何调控Pdc的酶活,其翻译的Pdc1、Pdc5、Pdc6蛋白结构具有的功能又发生怎样变化,这也需要我们明确阐述,进而对基因工程改造更具理论支撑。
因此,本研究以此为切入点,通过PCR克隆pdc6基因序列,利用软件对其进行分析,进一步明确pdc6与pdc1和pdc5结构基因在编码蛋白Pdc上的差异和作用机理,为今后课题组构建酿酒酵母△pdc菌株奠定理论基础,能够从分子水平上证明编码Pdc的3种结构基因敲除的先后顺序,从理论代谢流向上最大程度积累丙酮酸或提高高级醇产量提供依据。
1 材料与方法
1.1 试验材料
酿酒酵母(S.cerevisiae)H5、大肠杆菌(Escherichia coli)DH5α均由黑龙江大学微生物实验室提供;限制性内切酶SacI和BamH I,pMD18-T购自北京天玺瑞至有限公司;TaqDNA聚合酶、pfuDNA聚合酶购自TaKaRa 公 司 ;TIANamp Yeast DNA Kit、BioSpin Plasmid DNA Extraction Kit购自北京天根和杭州博日科技术有限公司。
1.2 培养基
LB Medium:Tryptone 10 g/L,Yeast Extract 5 g/L,NaCl 10 g/L,pH 7.0,121℃、15 min湿热灭菌后,作为E.coliDH5α菌株的培养备用。
YPD Medium:Peptone 2 g/L,Yeast Extract 1 g/L,Glucose 2 g/L,pH自然,108℃、20 min湿热灭菌后,作为S.cerevisiaeH5菌株的培养备用。
1.3 引物设计
根据NCBI中序列号为NC_001139.9的S.cerevisiaeS288C Chromosome gene VII的pdc6序列,用Primer 5.0和Oligo6.0软件设计引物对pdc6-Forward和pdc6-Reverse。
pdc6-Forward:5’-GGAGAGCTCATGTCTGAAA TTAC-3’;
pdc6-Reverse:5’-CCGGGATCCTTATTGTTTGG C-3’。
其中,引物5端和3端分别引入SacI和BamH I酶切位点(下划线),用于克隆S.cerevisiaeH5 genomic DNA中目的核苷酸序列。
1.4 目的基因克隆与鉴定
利用酵母DNA提取试剂盒提取S.cerevisiaeH5 genomic DNA,以其为模板克隆获得pdc6序列。PCR反应程序:95℃预变性5 min;95℃变性30 s;58℃退火30 s;72℃延伸2 min;72℃终延伸10 min。PCR反应体系:S.cerevisiaeH5 DNA(25 ng/μm)10 μL、dNTP混合液(2.5 mmol/L)4 μL、ddH2O 20 μL、含Mg2+的pfuDNA Polymerase Buffer 5 μL、pdc6-up(1 pmol/μL)5 μL、pdc6-down(1 pmol/μL)5 μL、pfuDNA Polymerase(2.5 U/μL)1 μL。
PCR结束后,50μLPCR产物加0.25μLTaq(5U/mL)DNA聚合酶,72℃反应10 min加A尾,便于后续“TA”连接,随之取 45 μL 反应产物,加 5 μL 10× loading buffer,1.0%琼脂糖凝胶电泳检测目的基因条带。
将PCR产物利用试剂盒纯化后与pMD18-T载体(温度16℃)连接,转化至E.coliDH5α Competent Cells中,蓝白筛选法挑取白色菌落的阳性克隆子,利用菌液PCR和酶切验证(SacI/BamH I)验证后送交Sangon测序并命名为pTCL-pdc6,测序后登录NCBI,运用BLAST数据库进行序列比较分析。
1.5 生物信息学分析
利用BLAST对所测序后的pdc6序列进行ORF Finder和同源性分析;利用软件翻译获得该蛋白质的氨基酸序列,分析其相对分子质量(MW)、理论等电点(pI)、氨基酸(aa)组成、原子组成、消光系数、半衰期、稳定性指数、脂肪族氨基酸指数、亲(疏)水性的平均值及磷酸化位点(http://www.cbs.dtu.dk/services/NetPhos/;https://web.expasy.org/protparam/);对该基因序列进行RPS-Blast分析(http://www.ncbi.nlm.nih.gov/Structure/cdd/wrpsb.cgi),Pdc6蛋白跨膜区及重复序列分析(http://www.cbs.dtu.dk/services/TMHMM-2.0/;http://www.ebi.ac.uk/Radar/),预测蛋白质二、三级结构模型(http://npsa-pbil.ibcp.fr/cgibin/npsa_automat.pl?page=/NPSA/npsa_hnn.html)(http://swissmodel.expasy.org/workspace/index.php?func=modelling_simple1),进行亚细胞定位分析(http://www.psort.org/psortb/index.html)。
2 结果与分析
2.1 S.cerevisiae H5 genomic DNA提取及pdc6基因的克隆与鉴定
电泳结果显示15000 bp处有清晰条带,说明H5 genomic DNA提取成功(图1 A);PCR扩增获得的核苷酸序列大小与预期片段相符,约为1692 bp(图1 B);菌液PCR电泳验证和SacI/BamH I酶切电泳验证(图1 C和D),结果片段与预期均相符,说明pdc6克隆成功。
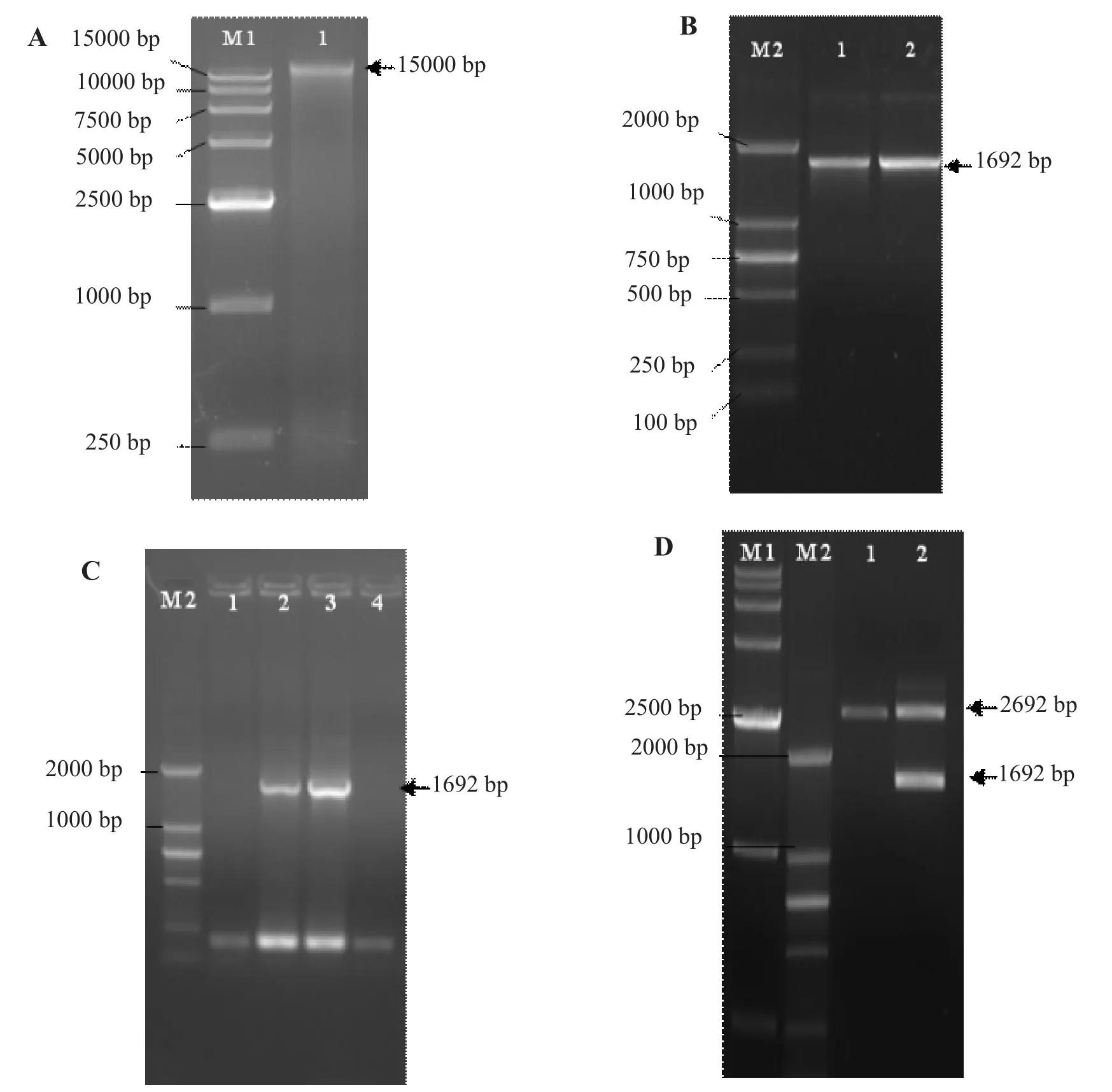
图1 S.cerevisiae H5 genomic DNA(A);pdc6的PCR扩增产物(B);菌液PCR产物(C);质粒pTCL-pdc6双酶切产物(D)
2.2 pdc6的生物信息学分析
经Sangon测序后的pdc6核苷酸序列长度约为1692 bp,利用BLAST分析该核酸序列和氨基酸序列。结果发现,所测序列与S.cerevisiaeySR 128染色体VII基因(NCBI:CP036485.1)序列比较有100%序列一致性,与 SY14 染色体 I(NCBI:CP029160.1)基因序列比较有100%序列一致性,与BY4742染色体VII(NCBI:CP026294.1)基因序列比较有100%序列一致性,与DBVPG6765染色体VII(CP020163.1)比较有99.94%的序列一致性。因此,确定该核苷酸序列为酿酒酵母的pdc6序列(图2)。

图2 Pdc6的生物信息学分析结果
经ORF Finder分析发现,该pdc6基因序列拥有完整开放阅读框(Open Reading Frame,ORF),起始密码子ATG,终止密码子TAA,且编码563个氨基酸。对其编码蛋白分子量进行预测约为62 kDa,理论等电点pI=5.8。由此可知该序列编码的蛋白Pdc6为酸性蛋白质。且该氨基酸序列中Leu含量相对较高(11.4%),整个序列中强酸(60)、强碱性(51)氨基酸残基数量相差不大,预测Pdc6氨基酸化学分子式:C2796H4406N720O830S6,原子总数为8758个。预测其在OD280水溶液中的消光系数为6.383×104;半衰期预估在20 h以上;预测其脂肪族氨基酸指数为100.28;亲水性分析(图2A)发现,Pdc6蛋白疏水性最大值为2.32,亲水性平均值(-0.049)。蛋白质稳定性分析表明,Pdc6不稳定指数(30.51)较高,是一种不稳定蛋白质;在PROSITE数据库中检索到Pdc6的功能性基序和位点在427~446之间,为硫胺焦磷酸酶。磷酸化位点分析(图2B)预测可知,该Pdc6序列中有25个Ser、23个Thr、8个Tyr。蛋白重复序列分析(图2E),结果表明在Pdc6中可能存在3个重复序列,但这些氨基酸序列存在差异性。
亚细胞定位预测该Pdc6可能存在于细胞外基质(9.13%),极低的可能位于细胞质膜(0.05%),0.82%的可能位于细胞壁,因此,预测Pdc6蛋白定位于细胞外基质。跨膜结构域(图2C)分析显示,该Pdc6不含跨膜区。rps-blast分析(图2D)显示该片段属于Pdc1超家族,具有指导碳水化合物和辅酶转运和代谢的功能。
利用Hopfield神经网络(HNN)预测Pdc6蛋白结构[9],发现其二级结构主要由无规则卷曲、α-螺旋及延伸链构成(图2F)。其中,无规则卷曲、α-螺旋、延伸链分别占40.14%、27.71%、8.70%(蓝色:α-螺旋、红色:延伸链、紫色:无规则卷曲),在得到Pdc6二级结构信息基础上,预测构建该蛋白质的三级结构(图2G)。
3 讨论与结论
酿酒酵母作为真核模式生物,近年来,各种组学的理论研究随着全基因组测序的完成已经日渐透彻[10],其应用领域逐渐呈多元化趋势,不单单在食品生产中[11-12]被广泛应用,在学科研究[13-14]、疾病模式探索[15-17]、能源探索与环境保护[18-19]等领域也备受关注,其在遗传调控方面的研究也越来越清晰,但是仅针对Pdc的研究,更多是停留在pdc1/pdc5基因缺陷菌株的构建和发酵工业化生产高级醇产量的层面上[20],忽视pdc6分子机制调控蛋白表达上作用,对pdc6单基因缺失菌株表型上的探究较少。针对此问题,预测pdc6和Pdc6蛋白结构则是探究其分子水平基因表达调控机制和表型上变化的出发点。因此,笔者利用PCR技术克隆pdc6基因序列,并通过信息学软件对其序列进行分析,为后续试验探究基因表达调控机制夯实基础[21]。
核苷酸序列结果阐明,该基因全长为1692 bp,这与Hohmann等[22]等报道结果一致。拥有完整ORF,编码563个氨基酸,且核苷酸比对结果同源性达99%~100%。同时,rps-blast分析显示该基因序列属于Pdc1超家族,具有指导碳水化合物、辅酶转运和代谢的功能,但pdc6能够产生的分子生物学功能需后续试验进行验证。
氨基酸序列分析结果可知,Pdc6的MW≈62 kDa,等电点pI=5.8,属酸性蛋白质,该Pdc6的氨基酸序列,以疏水性氨基酸为主;蛋白质稳定分析表明,Pdc6不稳定指数较高,且预测蛋白质结构中存在蛋白激酶位点;亚细胞定位预测,该蛋白质Pdc6存在于细胞外基质中可能较大,属于单一亚基、不稳定酸性蛋白,一般作为单体与其他蛋白一起发挥其功能。Iding等[23]研究表明,酵母Pdc分子总量约250 kDa,是由同源四聚体、辅酶因子构成的、性质稳定的全酶,单一亚基性质不稳定、含有563个氨基酸残基、包含3个结构紧凑的域,这与本研究Pdc6预测蛋白分子量和三级预测结果相符。有研究阐明[24],酵母Pdc是由pdc1、pdc5、pdc6,3个结构基因共同编码的蛋白,且编码过程受调节因子pdc2调控,研究提到pdc6是一种在野生型酿酒酵母细胞中,表达较弱的核苷酸序列。pdc6基因表达过程中分子调控机理较为有趣[25]:野生型酿酒酵母中Pdc的活性主要取决于Pdc1,Pdc5和Pdc6对于全酶影响不大。而在△pdc1缺失型菌株中,由于pdc1基因序列被置换或缺失,为了维持细胞正常代谢,其核苷酸序列上启动子将会调控pdc6第二次复制、进而表达Pdc1-Pdc6融合蛋白,它的功能几乎取代pdc1正常编码下的功能,但同时自发融合蛋白又受到pdc5表达调控影响。可见当细胞在外界环境发生变化时,Pdc6的作用机理也发生着变化。因此,对pdc6的探索不仅有利于酵母丙酮酸脱羧酶的研究,同时对于结构基因pdc1相关融合蛋白Pdc1-Pdc6更深层次的研究也十分关键。关于编码Pdc的结构基因pdc1和pdc5同源基因的pdc6的研究并不多,但在探索工程菌株高级醇或丙酮酸积累方面,单基因pdc1或pdc5缺失后,Pdc酶活力并未像预期一样在缺失型菌株中完全被抑制,故笔者从对单基因缺失菌株的遗传表达方面[26]上,进行pdc6和Pdc6预测结构分析其相关调控系统[27],期望揭示出Pdc6的潜在功能,也使得酵母Pdc的研究更加完善。
综上,本研究成功克隆得到全长为1692 bp的pdc6基因片段,没有发生碱基缺失与突变,该基因序列可以编码563个氨基酸,且与Genebank数据库中S.cerevisiae的pdc6基因序的同源性接近100%,对pdc6生物信息学分析预测Pdc6蛋白质属于酸性蛋白,定位于细胞外基质,不含有跨膜区;二级结构预测表明Pdc6含有40.14%的无规则卷曲、27.71%的α-螺旋、8.70%的延伸链;三级结构预测表明该蛋白模型符合一般的丙酮酸脱羧酶结构特性。预测pdc6片段属于Pdc1超家族,具有指导碳水化合物转运和代谢、辅酶转运和代谢的功能,也从侧面证实与实际在基因工程改造中,代谢流向高级醇产量上理论相符。结果表明,加大对该基因的研究可以使研究者更好地利用这一基因,进一步开展对于丙酮酸脱羧酶的研究。
