2017-2019年四川地区猪A群轮状病毒的分子流行病学调查
2021-03-26周群陈小飞阚蕊慈李玉曹慧彭艳伶张斌
周群,陈小飞,阚蕊慈,李玉,曹慧,彭艳伶,张斌,4
2017-2019年四川地区猪A群轮状病毒的分子流行病学调查
1西南民族大学生命科学与技术学院,成都 610041;2华南农业大学兽医学院,广州 510642;3西昌市畜牧局,四川西昌 615000;4青藏高原动物遗传资源保护与利用教育部/四川省重点实验室,成都 610041
【】通过对四川地区规模化猪场A群轮状病毒(RVA)的分子检测,从而了解RVA流行情况及分子特征,为猪RVA疫苗研制提供理论基础。2017—2019年从四川省14个地区40个猪场采集303份仔猪腹泻样本,用荧光定量RT-PCR方法调查RVA的分子流行率,用RT-PCR方法对RVA阳性样本进行分型和VP4和VP7全基因组的扩增,用RotaC2.0软件确定相应毒株的基因型,用MegAlign软件进行同源性分析,通过MEGA 7.0软件用邻近法构建系统进化树,用SimPlot和RDP4软件进行重组分析。303份仔猪腹泻样本检出RVA阳性样本98份,阳性率为32.34%(98/303,95%CI=27.1%—37.9%)。从98份RVA阳性样本中扩增出39个VP7片段,G9为优势基因型(41%),G4、G5、G26和G3各占23%、28.2%、5.1%和2.7%。扩增出的59个P型中以P[13]型为主,占40.7%,其次为P[6]、P[23]和P[1]型,分别占30.5%、23.7%和5.1%。30株毒株成功鉴定出G/P基因型,G9P[23](23.3%)为优势组合基因型。其他基因型为G4P[6](16.7%)、G9P[13](13.3%)、G5P[23](10%)、G5P[13](10%)、G9P[6](6.7%)、G26P[13](6.7%)、G4P[13](6.7%)、G4P[23](3.3%)和G3P[13](3.3%)。基因型G5P[23]、G4P[13]、G9P[6]、G26P[13]和G4P[23]为国内首次鉴定。此外,重组分析表明4株毒株在VP7或VP4基因上存在重组现象。四川地区腹泻仔猪粪便中RVA的流行率高且基因型复杂,优势组合基因型为G9P[23]。该结果丰富了四川地区RVA的流行病学资料,对四川地区猪RVA的防控提供了重要参考。
猪A群轮状病毒;分子流行病学;基因组研究;重组
0 引言
【研究意义】A群轮状病毒(rotavirus A, RVA)为呼肠孤病毒科轮状病毒属成员,是引起婴幼儿及其他多种幼龄动物病毒性腹泻的主要病原体之一[1]。RVA基因组分为11个基因节段,分别编码6种结构蛋白(VP1—VP4,VP6和VP7)和5或6种非结构蛋白(NSP1—NSP5/6),RVA的11个基因之间均易发生重配事件[2]。根据VP6蛋白的抗原性可将RV分类为10个血清群(A—J)[3-4],A—C、E和H群RV已经被证实可感染猪,其中感染人类和动物最常见的为A群轮状病毒[5-6]。RVA感染仔猪呈世界流行,各国猪场中RVA的阳性率为3.3%—67.3%,对世界养猪业产生了严重的经济损失[7]。研究表明猪RVA是人源RVA感染的潜在来源[8-11],因此了解猪RVA的流行情况和分子特征对RVA的防控以及防止猪RVA传播给人类至关重要。【前人研究进展】VP4和VP7蛋白共同组成RVA的外衣壳,二者都是重要的中和抗原,可以诱导机体产生中和抗体[12]。结构蛋白VP4和VP7分别决定RVA的P型和G型,且G型和P型之间会产生多种组合,不同组合的血清型之间交叉保护性低[2]。在猪RVA中,已经鉴定出了12个G型(G1—G6,G8—G12和G26)和16个P型(P[1]—P[8],P[13],P[19],P[23],P[26],P[27],P[28],P[32],P[34]),其中G3、G4、G5、G9和G11被认为是最常见的G型,通常与P[5]、P[6]、P[7]、P[13]和P[28]组合在一起[13-15]。国内的病原学检测结果显示RVA在我国规模化猪场中普遍存在,仔猪腹泻粪便中RVA的阳性率为7.69%—28.76%[16-19]。近年来中国大陆陆续报道了G11P[13]、G9P[23]和G9P[7]等组合基因型的RVA[20-22],然而四川地区RVA的流行情况及遗传多样性资料相对匮乏。【本研究切入点】2017—2019年分别从四川省14个地区40个猪场采集腹泻样本303份,对其进行RVA检测及基因型的鉴定,以期了解四川地区猪群中RVA的感染情况和流行趋势。【拟解决的关键问题】为研究四川地区猪RVA的遗传进化特征提供基础研究资料,预测四川地区猪RVA的流行趋势,从而为猪RVA疫苗的研制提供理论基础。
1 材料与方法
试验于2018年3月至2019年12月在西南民族大学生命科学与技术学院动物医学实验室进行。
1.1 材料
1.1.1 病料采集 对四川省不同地区、不同时间和不同猪群的腹泻样本进行主动采集,2017—2019年采集了四川省绵阳、泸州、眉山和西昌等14个地区40个猪场共303份仔猪腹泻样本,发病猪均表现为产房哺乳仔猪腹泻、消瘦,腹泻病死率为35%—85%。样本置于-80℃冰箱保存备用。
1.1.2 主要试剂与仪器 Trizol试剂(RNAiso Plus)、PrimeScriptTMRT试剂盒、pMD19-T Vector、MarkerⅡ和TB Green Premix ExTaqⅡ购自宝生物工程(大连)有限公司;Quick Taq HS DyeMix购自东洋纺(上海)生物科技有限公司;PCR产物纯化试剂盒和胶回收试剂盒购自OMEGA公司;DH5α购自北京天根生化科技有限公司;LB培养基购自青岛海博试剂有限公司;高速离心机5804购自Eppendor公司;普通PCR仪、核酸蛋白电泳仪、凝胶成像系统VersaDoc2000均购自Bio-Rad公司。
1.1.3 引物 用西南民族大学生命科学与技术学院动物医学实验室基于RVA VP6基因建立的荧光定量PCR方法检测303份腹泻样本中的RVA;RVA分型引物信息见文献[23—24];用于RVA阳性样本G型和P型全基因扩增的引物信息见文献[23—25]。以上所有引物均由上海生工生物工程技术服务有限公司合成。
1.2 方法
1.2.1 病毒核酸的提取 将适量的粪便样本加入装有400 μL生理盐水的1.5 mL EP管中,根据生产商说明,使用RNAios Plus从400 μL粪便混悬液中提取病毒RNA。反转录按照PrimeScriptTMRT试剂盒说明书进行,反转录产物于-20℃保存。
1.2.2 病料样本的检测分型及序列测定 以303份样本的cDNA为模板,使用TB Green Premix ExTaqⅡ和Quick Taq HS DyeMix进行RVA的检测和分型,具体方法参照说明书。将PCR扩增产物通过琼脂凝胶核酸电泳及凝胶成像仪进行检测与分析。PCR产物纯化后与pMD19-T载体连接,转化到DH5α感受态细胞中,筛选阳性重组质粒由上海生工生物有限公司双向测序。对测序结果进行序列拼接,并在NCBI中对拼接的基因组序列进行比对。
1.2.3 RVA VP7和VP4基因型鉴定及序列分析 将测序获得的RVA VP7和VP4基因的核苷酸序列使用RotaC2.0自动基因分析软件进行序列分析以确定相应毒株的基因型;应用Lasergene中MegAlign软件进行同源性分析;并通过MEGA 7.0软件用邻近法构建系统进化树,Bootstrap值选择1 000重复;应用SimPlot和RDP4软件对全基因组序列进行重组分析。
2 结果
2.1 腹泻样本RVA的检测和G/P型分型结果
303份仔猪腹泻的样本RVA检测结果显示:共检出RVA阳性样本98份(32.34%,95% confidence interval(CI)=27.1%—37.9%),40个猪场中有25个猪场检出RVA阳性,检出率为62.5%(95% CI=45.8%—77.3%)。对98份RVA阳性样本分别测定P基因型和G基因型,共得到47株RVA VP4部分基因、30株RVA VP7部分基因、12株RVA VP4全基因组和9株RVA VP7全基因组,GenBank登录号为MT198708—MT198805,将测序获得的RVA VP7和VP4基因的核苷酸序列使用RotaC2.0自动基因分析软件进行序列分析以确定相应毒株的基因型。结果显示:G型存在G9(16/39)、G5(11/39)、G4(9/39)、G3(1/39)和G26(2/39)五种型;P型存在P[13](24/59)、P[23](14/59)、P[6](18/59)和P[1](3/59)4种型。有30份样本成功测定出G/P组合基因型,共包括10种组合,分别为:G9P[23](7/30);G4P[6](5/30);G26P[13](2/30);G9P[13](4/30);G5P[23] (3/30);G4P[23](1/30);G3P[13](1/30);G5P[13](3/30);G4P[13](2/30);G9P[6](2/30),RVA的检测结果及详细分布见表1。
2.2 RVA VP7 基因部分序列遗传进化分析
将本研究中获得的30株RVA VP7部分基因和9株全基因组序列与其他已知G基因型的代表毒株通过MEGA 7.0软件进行遗传进化分析,结果显示(图1):所获得的G3型毒株与四川猪源毒株SCQL-5-3(GenBank No. MG066590)亲缘关系最近;四川地区G4型RVA分为两个不同的分支,来自同一地区4个毒株和RVA/Pig/CHN/SCMS-69与日本猪源毒株GUB71(GenBank No. AB573647)聚为一个分支,另外4株G4型RVA毒株分别来自两个地区,在分支的上端与中国猪源和人源毒株聚为一支;在G5型RVA分支中,来自同一地区的7株毒株聚为一个分支,来自同一地区的4株G5型RVA分别与中国台湾猪源毒株和河北人源RVA毒株聚为一支;在G9型RVA分支中,RVA/Pig/CHN/SCSN-96和RVA/Pig/CHN/SCRS-142虽来自不同地区,但都与中国猪源的毒株HBHP(GenBank No. KX831114)同源性最高为98.3%,并与另外2个地方的5株毒株聚为一支,剩余来自5个地区的7株毒株与湖北猪源毒株(GenBank No. KX831100)同源性最高为95.9%—97.1%,来自内江的2株毒株单独聚为一支;本研究获得的两株来自同一地区的G26型RVA毒株与尼泊尔人源毒株亲缘关系最近。RVA VP7基因同源性分析见表2。
2.3 RVA VP4 基因部分序列遗传进化分析
将本研究中获得的47株RVA VP4部分基因和12株全基因组序列与其他已知P基因型的代表毒株通过MEGA 7.0软件中邻近法构建系统进化树。结果显示(图2):3株P[1]型RVA毒株与人源、牛源和羊源的毒株聚为一个大支;15株来自同一猪场的P[6]型RVA毒株在进化树上端单独聚为一支,其余3株均与斯里兰卡人源毒株R1207(GenBank No. LC389888)的同源性最高为95.7%—98.6%;本研究中的P[13]型毒株RVA/Pig/CHN/SCYB/Y1单独分为一支,其余23株分散在4个亚支中,分支Ⅰ由来自8个地区的11株毒株与四川猪源毒株(GenBank No. MK410284)和人源毒株(GenBank No. MK597985)组成;分支Ⅱ进一步分为2个亚支,分别由RVA/Pig/CHN/SCLZ/FB与日本猪源毒株FGP36(GenBank No. AB573878)和本研究中其余2株毒株与中国猪源毒株LNCY(GenBank No. MF462324)组成;分支Ⅲ由本研究中分别来自3个地区的4株毒株与之前获得的1株四川地区的毒株和1株日本猪源毒株(GenBank No. AB573650)组成;亚支Ⅳ中5株毒株与泰国猪源毒株CMP178(GenBank No. DQ536362)亲缘关系最近。在P[23]型RVA毒株进化树中来自同一个猪场的7个毒株聚为一个小支,位于进化树的最上端,来自2个地区的3株毒株聚为一个小支。RVA/Pig/CHN/SCSN-96和来自同一个地区的2株毒株在进化树的最下端分别聚为一个小支。RVA VP4基因同源性分析见表3。
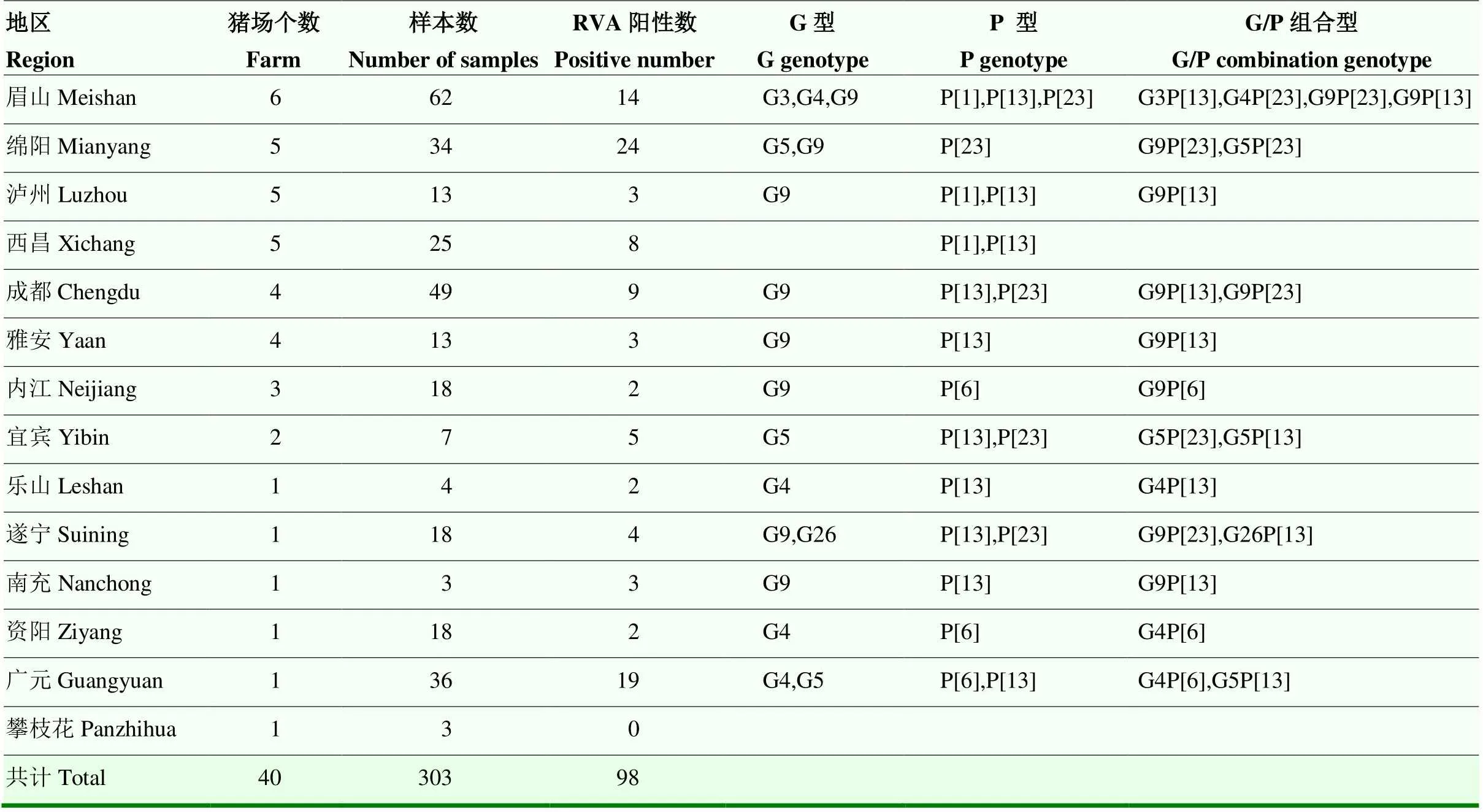
表1 四川地区猪场RVA感染率及基因型的分布

表2 RVA VP7 基因同源性分析
表中毒株名均为删除毒株病毒名、宿主、地区、年份和基因型之后的简称,括号里为GenBank号。下同
The strain names in the table are short names after deleting the strain virus name, host, region, year and genotype, the GenBank number is in parentheses. The same as below
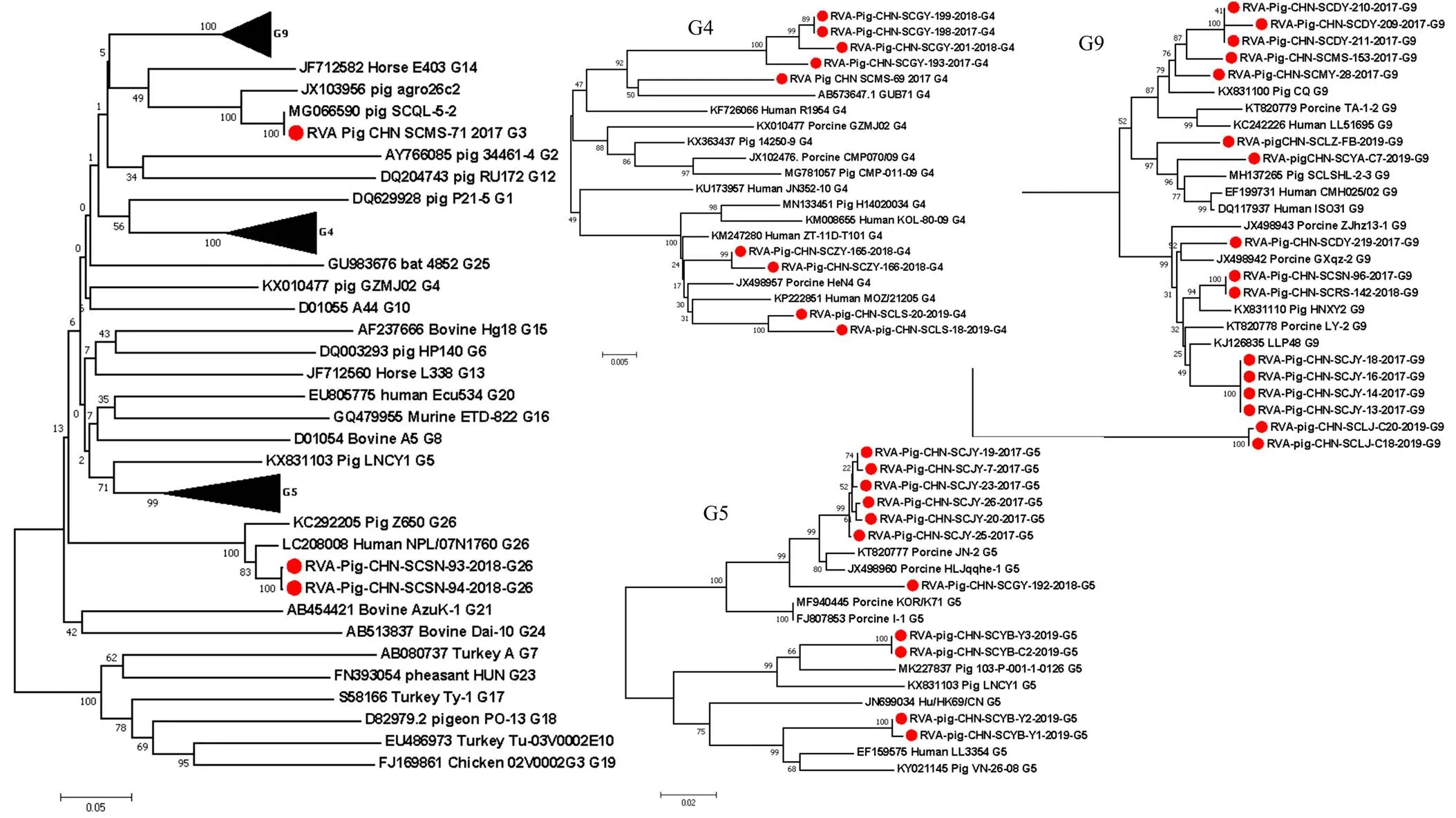
:本研究中的毒株 Strains in this study。下同 The same as below
2.4 RVA VP7基因和VP4基因重组分析
应用SimPlot和RDP4软件对12株RVA VP4全基因组和9株RVA VP7全基因组序列进行重组分析。结果显示:RVA/Pig/CHN/SCLJ/C18和RVA/Pig/CHN/ SCLJ/C20的VP7基因,RVA/Pig/CHN/SCYB/Y1和RVA/Pig/CHN/SCYB/Y2的VP4基因均发生了重组。RVA/Pig/CHN/SCLJ/C20和RVA/Pig/CHN/SCLJ/C18(图略)均为猪源G9型YN(GenBank No. KJ466985)与人源G4型KOL-80-09(GenBank No. KM008655)的重组毒株,预测重组值为0.692和0.703,预测重组断点位于253和707 bp处(图3);RVA/Pig/CHN/ SCYB/Y1为猪源P[23]型14150-53(GenBank No. KX363351)与鼠源P[13]型SCLS-M(GenBank No. MK606442)的重组毒株,预测重组值为0.692,预测重组断点位于860 bp(图3);RVA/Pig/CHN/SCYB/Y2为猪源P[23]型14175-24(GenBank No. KX363359)与猪源P[13]型SWU-1C(GenBank No. MK410284)的重组毒株,预测重组值为0.634,预测重组断点位于1 604 bp(图略)。本研究分别对每个毒株重组断点前后区域构建了进化树,结果显示毒株重组断点前后均表现出不一致的遗传进化关系,进一步证实了本研究中毒株的重组事件。

表3 RVA VP4 基因同源性分析
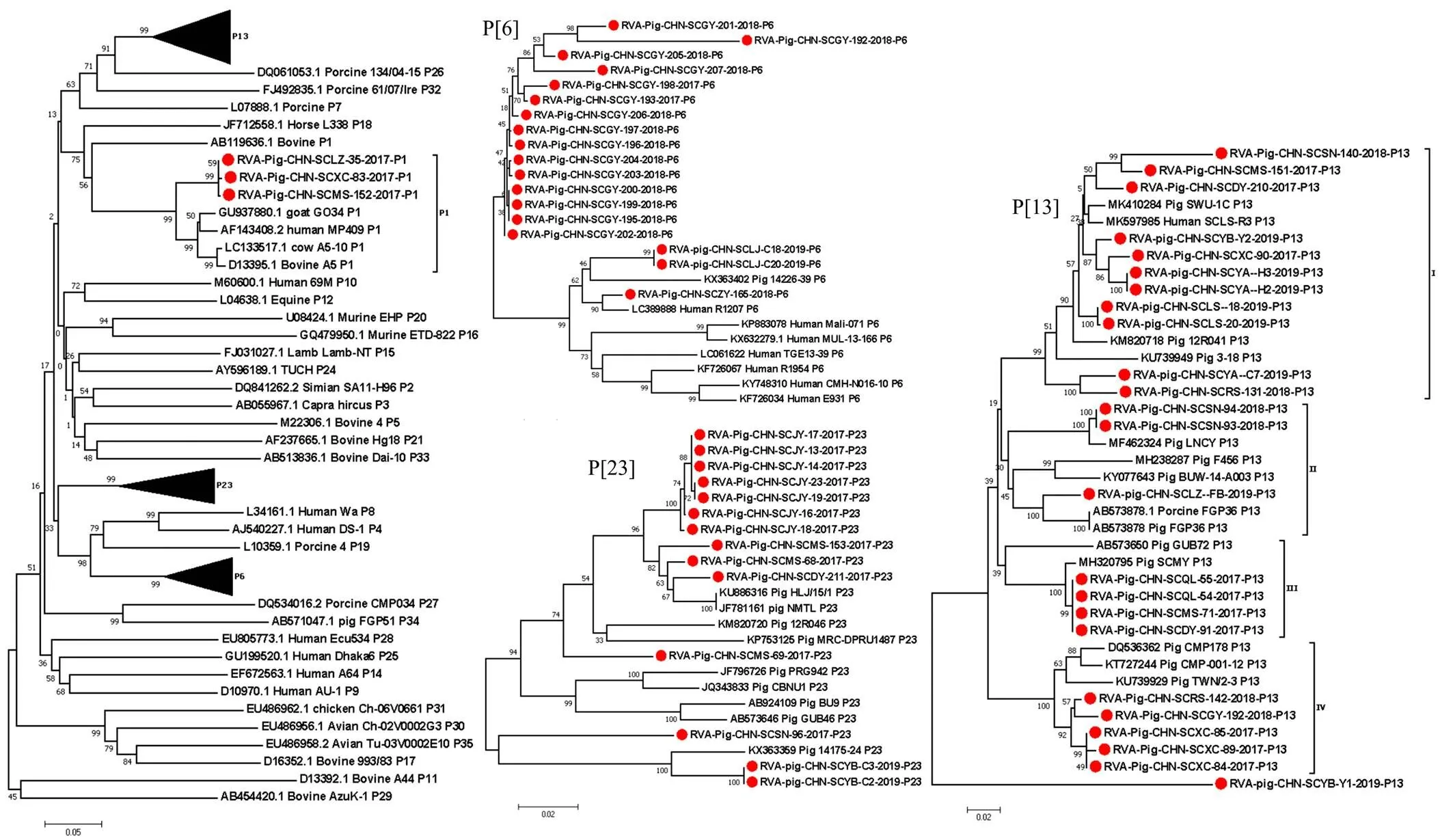
:右侧P[6],P[13]和 P[23]进化树的缩略图 Thumbnails of the right P[6], P[13] and P[23] phylogenetic trees
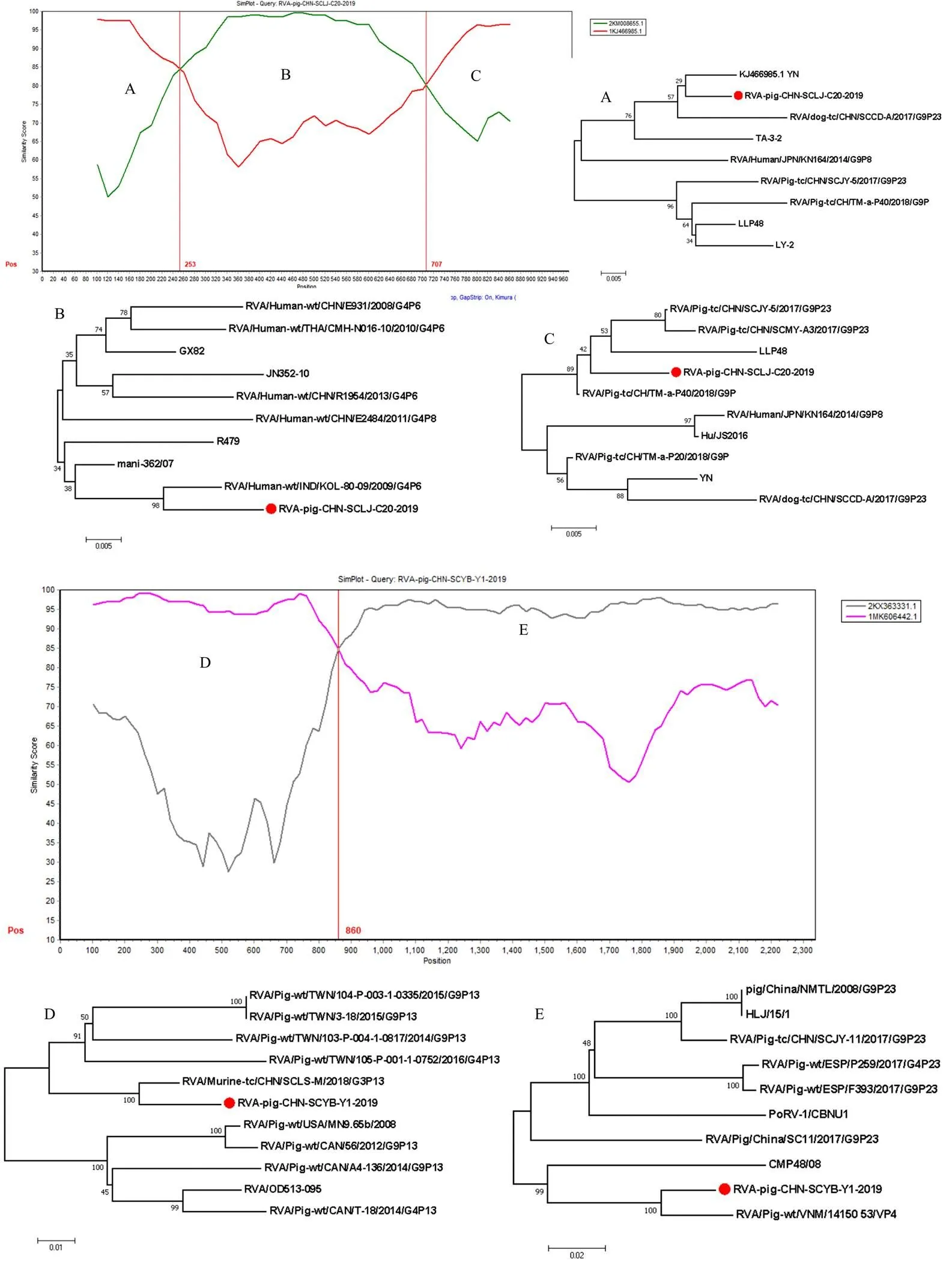
纵轴表示查询毒株与其他参考毒株核苷酸序列的相似性(%),水平轴表示核苷酸位置。A、B、C为Simplot软件分析毒株RVA/Pig/CHN/ SCLJ/C20/2019 VP7基因重组区域及基于A、B、C重组区域构建的进化树;D、E为Simplot软件分析毒株RVA/Pig/CHN/SCYB/Y1/2019 VP4基因重组区域及基于D、E重组区域构建的进化树
3 讨论
RVA是引起婴幼儿以及多种幼龄动物急性肠胃炎的重要病原[26-28],对养殖业造成重大的经济损失。国内病原学检测结果显示RVA在我国规模化猪场中普遍存在,仔猪腹泻粪便中RVA的阳性率为7.69%—28.76%[16-19]。本研究采用荧光定量RT-PCR的方法对2017—2019年四川遂宁、宜宾、资阳、西昌和眉山等14个地区共40个规模化猪场的303份仔猪腹泻粪便样本进行了RVA的检测,结果显示猪场RVA阳性率为62.5%(25/40),RVA个体阳性率为32.34%(98/303),较2014—2016年四川地区RVA的阳性率(7.69%—26.7%)明显增高[16-17]。由于不同猪场的卫生条件和地理位置不同,不同猪场中RVA阳性率区别较大,在四川不同地区的猪场中RVA个体阳性率也有区别,绵阳、宜宾和广元地区的RVA阳性率较高,均超过了50%,而内江和资阳等地的RVA阳性率相对较低,为10%左右。本研究表明RVA在四川地区猪场中感染严重且分布广泛。因此,有必要加强对四川地区RVA的监测,本研究结果对四川地区RVA的防控提供了重要的科学依据。
VP7和VP4分别决定RVA的G型和P型,且G型与P型之间会产生不同的组合,不同组合型的RVA毒株诱导的交叉保护性低[2,12]。因此,加强对RVA不同G/P型的监测和研究对了解RVA的遗传进化和疫苗研究非常重要。在本研究中,四川地区RVA中G9型、G5型和G4型为最为流行的G型,分别占分型毒株的41%、28.2%和23%;P[13]型、P[6]型和P[23]型为最为流行的P型,分别占分型毒株的40.7%、30.5%和23.7%,而其中P[13]型RVA最具多样性和复杂性;优势G/P基因型为G9P[23],占分型样本的23.3%,其中眉山地区的组合基因型最为复杂,存在3种不同的基因组合型(G3P[13]、G4P[23]和G9P[23])(表1)。本研究还在四川地区仔猪中检测出了G4P[23]、G4P[13]、G9P[6]、G26P[13]和G5P[23] 5种首次在国内出现的组合基因型。此外,对39株RVA VP7基因片段和59株RVA VP4基因片段同源性分析表明(表2、表3),本研究所获毒株与东南亚地区的猪源和人源毒株具有高度同源性。在30株成功鉴定出G/P组合基因型的毒株中,有13株毒株存在猪RVA与人RVA基因重配现象,推测在我国和东南亚地区猪RVA与人RVA存在交叉传播,且存在大量的基因重配毒株。本研究结果对四川地区猪RVA的防控提供了重要参考,其中首次报道的在国内新出现的RVA组合基因型丰富了国内RVA的资料,有利于对国内RVA的监测。对了解四川地区猪RVA的遗传多样性提供了科学依据,丰富了四川地区RVA的流行病学资料。
研究表明猪RVA与人RVA具有高度的相似性,且常发生重组和重配事件[1,29-30]。近年来,四川地区RVA重组事件陆续被报道[21,31],本研究中获得的12株RVA VP4全基因组和9株RVA VP7全基因组中分别存在2株RVA VP4和2株RVA VP7重组毒株。其中G型重组毒株为G4型和G9型重组,P型重组毒株为P[13]型与P[23]型重组,表明在四川地区RVA感染率升高的同时,RVA流行基因型之间重组频率也正在逐渐增高,提示应加强对四川地区RVA分子流行病学的监测和对猪RVA的综合防控。两株P型重组毒株来自同一猪场,均为猪源毒株与猪源毒株发生重组,但两株毒株预测的重组断点和亲本株存在差异。两株G型重组毒株来自内江某猪场,为人源毒株与猪源毒株重组,但两株毒株预测的重组断点相近,并且亲本株一致。推测两株P型重组毒株所在场内猪群存在多种猪RVA毒株混合感染,而两株G型重组毒株所在场内猪群可能存在跨种间传播,但该场内具体的传播途径还需要根据具体情况进行进一步调查研究。
4 结论
通过对2017—2019年间在四川省不同地区采集的猪腹泻样本进行猪RVA的检测和分型鉴定,揭示了近年来四川省猪A群轮状病毒的主要流行毒株,G9型和P[13]型为分别为优势G基因型和P基因型,G9P[23]为优势组合基因型。总体看来,A群轮状病毒在四川地区猪群中的分布广泛且基因型多样,与人源毒株关系密切,并且个别毒株存在基因重组现象。
[1] Theingi W M, Hlaing M T, Ye M K, Khin M A, Mo M W, Htin L, Thin T S, Win M, Khin K O, Kyaw Z T. Sentinel surveillance for rotavirus in children <5 years of age admitted for Diarrheal illness to Yangon Children's Hospital, Myanmar, 2009-2014. Vaccine, 2018, 36(51): 7832-7835. doi:10.1016/j.vaccine.2017.11.002.
[2] Hoshino Y, Sereno M M, Midthun K, Flores J, Kapikian A Z, Chanock R M. Independent segregation of two antigenic specificities (VP3 and VP7) involved in neutralization of rotavirus infectivity. Proceedings of the National Academy of Sciences of the United States of America, 1985, 82(24): 8701-8704. doi:10.1073/pnas.82.24.8701.
[3] Mihalov K E, Gellért Á, Marton S, Farkas S L, Fehér E, Oldal M, Jakab F, Martella V, Bányai K. Candidate new rotavirus species in sheltered dogs, Hungary. Emerging Infectious Diseases, 2015, 21(4): 660-663. doi:10.3201/eid2104.141370.
[4] Matthijnssens J, Otto P H, Ciarlet M, Desselberger U, Van R M, Johne R. VP6-sequence-based cutoff values as a criterion for rotavirus species demarcation. Archives of Virology, 2012, 157(6): 1177-1182. doi:10.1007/s00705-012-1273-3.
[5] Tonietti P O, Hora A S, Silva F D F, Ruiz V L A, Gregori F. Phylogenetic analyses of the VP4 and VP7 genes of porcine group A rotaviruses in Sao Paulo State, Brazil: first identification of G5P[23] in piglets. Journal of Clinical Microbiology, 2013, 51(8): 2750-2753. doi:10.1128/JCM.01175-13.
[6] Vlasova A N, Amimo J O, Saif L J. Porcine Rotaviruses: Epidemiology, immune responses and control strategies.Viruses, 2017, 9(3): 48. doi:10.3390/v9030048.
[7] Kozyra I, Kozyra J, Dors A, Rzeżutka A. Molecular characterisation of porcine group A rotaviruses: Studies on the age-related occurrence and spatial distribution of circulating virus genotypes in Poland. Veterinary Microbiology, 2019, 232: 105-113. doi:10.1016/j.vetmic.2019.03.026.
[8] Wakuda M, Ide T, Sasaki J, Komoto S, Ishii J, Sanekata T, Taniguchi K. Porcine rotavirus closely related to novel group of human rotaviruses. Emerging Infectious Diseases, 2011, 17(8): 1491-1493. doi:10.3201/eid1708.101466.
[9] Malasao R, Khamrin P, Kumthip K, Ushijima H, Maneekarn N. Complete genome sequence analysis of rare G4P[6] rotavirus strains from human and pig reveals the evidence for interspecies transmission. Infection, Genetics and Evolution, 2018, 65: 357-368. doi:10.1016/j.meegid.2018.08.019.
[10] Ghosh S, Varghese V, Samajdar S, Bhattacharya S K, Kobayashi N, Naik T N. Evidence for independent segregation of the VP6- and NSP4- encoding genes in porcine group A rotavirus G6P[13] strains. Archives of Virology, 2007, 152(2): 423-429. doi:10.1007/s00705-006-0848-2.
[11] Agbemabiese C A, Nakagomi T, Gauchan P, Sherchand J B, Pandey B D, Cunliffe N A, Nakagomi O. Whole genome characterisation of a porcine-like human reassortant G26P[19] Rotavirus A strain detected in a child hospitalised for diarrhoea in Nepal, 2007. Infection, Genetics and Evolution, 2017, 54: 164-169. doi:10.1016/j.meegid.2017.06.026.
[12] Greenberg H, McAuliffe V, Valdesuso J, Wyatt R, Flores J, Kalica A, Hoshino Y, Singh N. Serological analysis of the subgroup protein of rotavirus, using monoclonal antibodies. Infection and Immunity, 1983, 39(1): 91-99. doi: 10.1128/ IAI.39.1.91-99.
[13] Papp H, László B, Jakab F, Ganesh B, De G S, Matthijnssens J, Ciarlet M, Martella V, Bányai K. Review of group A rotavirus strains reported in swine and cattle. Veterinary Microbiology, 2013, 165: 190-199. doi:10.1016/j.vetmic. 2013.03.020.
[14] MATTHIJNSSENS J, CIARLET M, MCDONALD S M, ATTOUI H, BÁNYAI K, BRISTER J R, BUESA J, ESONA M D, ESTES M K, GENTSCH J R, et al. Uniformity of rotavirus strain nomenclature proposed by the Rotavirus Classification Working Group (RCWG). Archives of Virology, 2011,156(8): 1397-1413.doi:10.1007/s00705- 011-1006-z.
[15] Jing Z, Zhang X, Shi H, Chen J, Shi D, Dong H, Feng L. A G3P[13] porcine group A rotavirus emerging in China is a reassortant and a natural recombinant in the VP4 gene. Transboundary and Emerging Diseases, 2018, 65(2): e317-e328. doi:10.1111/tbed.12756.
[16] 曹恭貌, 张斌, 岳华, 费磊, 任玉鹏. 四川部分猪场PEDV、TGEV、GARV和PKV感染状况调查. 动物医学进展, 2016, 37(1): 118-122.
CAO G M, ZHANG B, YUE H, FEI L, REN Y P. Investigation of PEDV, TGEV, GARV and PKV infection in some pig farms in Sichuan Province. Progress in Veterinary Medicine, 2016, 37(1): 118-122. (in Chinese)
[17] 杨文宇, 周远成, 韩燕, 陈蕾, 廖春燕, 付梦瑾, 季洪伟, 蔡雨涵, 朱玲, 徐志文, 郭万柱.猪A群轮状病毒和C群轮状病毒以及猪星状病毒多重RT-PCR同时检测方法的建立及应用. 中国兽医科学, 2014, 44(4): 394-400.
YANG W Y, ZHOU Y C, HAN Y, CHEN L, LIAO C Y, FU M J, JI H W, CAI Y H, ZHU L, XU Z W, GUO W Z. Establishment and clinical application of a multiplex reverse transcription-PCR for simultaneous detection of porcine group A rotrvirus, porcine group C rotavirus and porcine astrovirus. Chinese Veterinary Science, 2014, 44(4): 394-400. (in Chinese)
[18] Xue R, Tian Y, Zhang Y, Zhang M, Li Z, Chen S, Liu Q. Diversity of group A rotavirus of porcine rotavirus in Shandong province China. Acta Virologica, 2018, 62(3): 229-234. doi:10.4149/ av_2018_216.
[19] 周玲, 陈桂华, 伍子娴, 麦凯杰, 李迪, 王胜阳, 马静云, 张祥斌. 广东地区2016-2017年规模化猪场腹泻病原调查分析. 中国兽医杂志, 2017, 53(10): 3-5+9+49.
ZHOU L, CHEN G H, WU Z X, MAI K J, LI D, WANG S Y, MA J Y, ZHANG X B. Investigation on porcine diarrhea pathogens in Guangdong province form 2016 to 2017. Chinese Journal of Veterinary Medicine, 2017,53(10):3-5+9+49. (in Chinese)
[20] 库旭钢, 张坤, 刘羽茜, 何启盖. 猪A群轮状病毒的分离与鉴定. 畜牧兽医学报, 2012, 43(2):275-281.
KU X G, ZHANG K, LIU X X, HE Q G. Isolation and identification of group A porcine rotavirus. Acta Veterinaria et Zootechnica Sinica, 2012, 43(2): 275-281. (in Chinese)
[21] 李玉, 穷达, 张敏, 阚蕊慈, 汤承, 岳华, 张斌. 猪A群轮状病毒RVA/Pig-tc/CHN/SWU-1C/2018/G9P[13]株的分离与鉴定. 中国预防兽医学报, 2019, 41(11): 1170-1173.
LI Y, QIONG D, ZHANG M, KAN R C, TANG C, YUE H, ZHANG B. Isolation and identification of the porcine group A rotavirus strain of RVA/Pig-tc/CHN/SWU-1C/2018/G9P[13]. Chinese Journal of Preventive Veterinary Medicine, 2019, 41(11): 1170-1173. (in Chinese)
[22] Zhang H, Zhang Z, Wang Y F, Wang X, Xia M Q, Wu H. Isolation, molecular characterization and evaluation of the pathogenicity of a porcine rotavirus isolated from Jiangsu Province, China. Archives of Virology, 2015, 160(5): 1333-1338. doi:10.1007/s00705-015- 2347-9.
[23] Amimo J O, Vlasova A N, Saif L J. Detection and genetic diversity of porcine group A rotaviruses in historic (2004) and recent (2011 and 2012) swine fecal samples in Ohio: Predominance of the G9P[13] genotype in nursing piglets. Journal of Clinical Microbiology, 2013, 51(4), 1142-1151. doi:10.1128/JCM.03193-12.
[24] DiStefano D J, Kraiouchkine N, Mallette L, Maliga M, Kulnis G, Keller P M, Clark H F, Shaw A R. Novel rotavirus VP7 typing assay using a one-step reverse transcriptase PCR protocol and product sequencing and utility of the assay for epidemiological studies and strain characterization, including serotype subgroup analysis. Journal of Clinical Microbiology, 2005, 43(12): 5876-5880. doi:10.1128/JCM.43.12.5876-5880.
[25] Yan N, Tang C, Kan R C, Feng F, Yue H. Genome analysis of a G9P[23] group A rotavirus isolated from a dog with diarrhea in China. Infection, Genetics and Evolution, 2019, 70: 67-71. doi:10.1016/j. meegid.2019.02.020.
[26] Wang Y H, Pang B B, Zhou X, Ghosh S, Tang W F, Peng J S, Hu Q, Zhou D J, Kobayashi N. Complex evolutionary patterns of two rare human G3P[9] rotavirus strains possessing a feline/canine-like H6 genotype on an AU-1-like genotype constellation. Infection, Genetics and Evolution2013, 1: 103-112. doi:10.1016/j. meegid.2013.01.016.
[27] Timurkan M Ö, Alkan F. Identification of rotavirus A strains in small ruminants: first detection of G8P[1] genotypes in sheep in Turkey.Archives of Virology2020, 165(2): 425-431. doi:10.1007/ s00705-019-04476-7.
[28] Mishra N, Reslan L, El-Husseini M, Raoof H, Finianos M, Guo C, Thakkar R, Inati A, Dbaibo G, Lipkin W I, Zaraket H. Full genome characterization of human G3P[6] and G3P[9] rotavirus strains in Lebanon. Infection, Genetics and Evolution2020, 78: 104-133. doi:10.1016/j.meegid.2019.104133.
[29] Li D D, Duan Z J, Zhang Q, Liu N, Xie Z P, Jiang B M, Steele D, Jiang X, Wang Z S, Fang Z Y. Molecular characterization of unusual human G5P[6] rotaviruses identified in China. Journal of Clinical Virology, 2008, 42(2): 141-148. doi:10. 1016/j.jcv.2007.12.013.
[30] Ramani S, Iturriza G M, Jana A K, Kuruvilla K A, Gray J J, Brown D W, Kang G. Whole genome characterization of reassortant G10P[11] strain (N155) from a neonate with symptomatic rotavirus infection: Identification of genes of human and animal rotavirus origin. Journal of Clinical Virology, 2009, 45(3): 237-244. doi:10.1016/j.jcv.2009.05.003.
[31] Chen D Y, Zhou L, Tian Y M, Wu X, Feng L, Zhang X P, Liu Z H, Pang S R, Kang R M, Yu J F, Ye Y G, Wang H N, Yang X. Genetic characterization of a novel G9P[23] rotavirus A strain identified in southwestern China with evidence of a reassortment event between human and porcine strains. Archives of Virology, 2019, 164(4): 1229-1232. doi:10.1007/s00705-019-04188-y.
Molecular Epidemiological Investigation of Porcine Group A Rotavirus in Sichuan from 2017 to 2019

1College of Life Science and Technology, Southwest University for Nationalities, Chengdu 610041;2College of Veterinary Medicine, South China Agricultural University, Guangzhou 510642;3Animal Husbandry Bureau of Xichang City, Xichang 615000, Sichuan;4Key Laboratory of Qinghai-Tibetan Plateau Animal Genetic Resource Reservation and Utilization of Ministry of Education/Sichuan Province, Chengdu 610041
【】The aim of this study was to investigate the prevalence and molecular characterization of porcine group A rotavirus (RVA) in large-scale pig farms in Sichuan, to provid a theoretical basis for the development for porcine RVA vaccine. 【】From 2017 to 2019, the 303 samples were collected from 40 pig farms in 14 regions of Sichuan province. Prevalence of RVA was detected by real-time RT-PCR method, and RVA positive samples were typed by RT-PCR. At the same time, the whole genomes of RVA VP4 and VP7 gene were amplified from some positive samples. The genotypes of the corresponding strains were determined by RotaC2.0classification tool. Sequence homology was analyzed by MegAlign. Phylogenetic tree was constructed by Neighbor-Joining method through MEGA 7.0. SimPlot and RDP4 softwares were applied for recombination analysis. 【】Of the 303 samples examined, 32.34% (98/303, 95%CI=27.1%-37.9%) were positive for RVA. Among the 39 G types, G9 (41%) was the dominant genotype, while G4, G5, G26, and G3 were detected in 23%, 28.2%, 5.1% and 2.7%, respectively. The P[13] genotype (40.7%) was dominant among the 59 P types, followed by P[6], P[23] and P[1] in 30.5%, 23.7% and 5.1%, respectively. Furthermore, the 30 strains successfully identified the G/P combination genotype, and the dominant combination genotype was G9P[23] (23.3%), the other combinations were G4P[6] (16.7%), G9P[13] (13.3%), G5P[23] (10%), G5P[13] (10%), G9P[6] (6.7%), G26P[13] (6.7%), G4P[13] (6.7%), G4P[23] (3.3%) and G3P[13] (3.3%). Notably, the G5P[23], G4P[13], G9P[6], G26P[13] and G4P[23] were first identified in China. In addition, the recombination analysis showed that four strains had recombination on VP7 or VP4 genes. 【】The results demonstrated that the prevalence of RVA in diarrhea piglet feces was high and the genotypes were complex in Sichuan. The dominant genotype of RVAs was G9P[23]. The results of this study enriched the epidemiological data of RVA and provided an important reference for the prevention and control of porcine RVA in Sichuan province.
porcine group A rotavirus; molecular epidemiology; genomic research; recombination

10.3864/j.issn.0578-1752.2021.05.017
2020-04-13;
2020-07-29
国家自然科学基金(31772766)、四川应用基础研究计划(2020YJ0247)、西南民族大学中央高校基本科研业务费专项资金(2020NYB21)
周群,E-mail:2297248747@qq.com。陈小飞,E-mail:chenxf-1@163.com。周群与陈小飞为同等贡献作者。通信作者张斌,E-mail:binovy@sina.com
(责任编辑 林鉴非)
