针对非洲猪瘟病毒MGF360-13L基因的TaqMan荧光定量PCR的建立
2021-03-26王涛韩玉潘力王冰孙茂文王翌罗玉子仇华吉孙元
王涛,韩玉,潘力,王冰,孙茂文,王翌,罗玉子,仇华吉,孙元
针对非洲猪瘟病毒MGF360-13L基因的TaqMan荧光定量PCR的建立
王涛,韩玉,潘力,王冰,孙茂文,王翌,罗玉子,仇华吉,孙元
中国农业科学院哈尔滨兽医研究所/兽医生物技术国家重点实验室,哈尔滨 150069
【】建立一种以MGF360-13L基因为靶标的实时荧光定量PCR检测非洲猪瘟病毒(African swine fever virus, ASFV)的方法,为非洲猪瘟(African swine fever, ASF)的诊断、基因缺失毒株鉴别、病毒分离鉴定、基因功能研究提供技术支持。首先,以ASFV中国流行毒株(GenBank: MK333180.1)的基因序列为靶标设计并筛选了1对特异性引物和探针,建立其荧光定量PCR检测方法。设计引物13L-F/13L-R,扩增,并将其克隆至pOK12载体,挑取阳性克隆并测序验证,构建重组质粒标准品。将重组质粒标准品连续10倍梯度稀释,以稀释后的各梯度质粒标准品为模板,配制反应体系和设置反应条件后进行荧光定量PCR检测,建立标准曲线,并对其敏感性和重复性进行评价;其次,以连续10倍梯度稀释的重组质粒标准品为模板,利用引物13L-F/13L-R进行常规PCR检测,以比较荧光定量PCR的敏感性。最后,运用本检测方法和本研究组已建立的针对ASFV的荧光定量PCR检测方法同时对黑龙江某猪场发生ASF时采集的30份临床样品进行检测,以比较两种检测方法的符合率。另外,应用该方法对感染原代巨噬细胞的ASFV野毒株和MGF基因缺失毒株进行鉴别检测。利用引物13L-F/13L-R可扩增出一条800 bp左右的特异性目的条带,而阴性对照无条带,成功构建出标准品。本研究建立的实时荧光定量PCR检测方法在质粒标准品为1.56×101-1.56×108拷贝/μL时,呈现出良好的线性关系,线性回归方程为:=-3.295 lg() + 45.995,线性相关系数2为0.997,对ASFV核酸最低检测限为15.6拷贝/μL。在特异性检验中,除ASFV核酸外,猪瘟病毒、伪狂犬病病毒、猪繁殖与呼吸综合征病毒、猪传染性胃肠炎病毒、猪圆环病毒1型、猪圆环病毒2型等病毒核酸均未出现扩增曲线,表明该方法的特异性良好。与最低检测限为1.56×104拷贝/μL病毒核酸的常规PCR检测方法相比,本研究建立的TaqMan荧光定量PCR检测方法高出其约3个数量级,具有较高的敏感性。在临床样品检测中,两种检测方法经过McNemar检验,= 0.5>0.05,表明两种检测方法的结果无统计学差异;经过Kappa检验,Kappa = 0.867>0.75,<0.001,提示两种检测方法符合率较好,能够有效区分ASFV野毒株和基因缺失毒株。建立的TaqMan荧光定量PCR特异、敏感、重复性好,不仅丰富了ASFV现有检测靶标,也为后续功能、缺失毒株的鉴定及相应基因缺失疫苗株的鉴别诊断提供技术支持。
非洲猪瘟病毒;;荧光定量PCR;鉴别诊断
0 引言
【研究意义】非洲猪瘟(African swine fever, ASF)是由非洲猪瘟病毒(African swine fever virus, ASFV)感染猪引起的一种接触传染性、广泛出血性、烈性传染病,最急性和急性型感染死亡率可高达100%,是养猪业的头号杀手[1-3]。该病已呈现出跳跃式传播,自2007年再次传入欧洲后,迅速扩散至俄罗斯、乌克兰、拉脱维亚、波兰等十多个东欧国家[4]。2018年8月传入中国后[5-6],不到一年时间扩散至蒙古、朝鲜、以及越南、柬埔寨、老挝、缅甸等多个东南亚国家。已初步呈现出全球流行趋势,对全球养猪业的健康发展造成了重大威胁[7-8]。截至目前,我国已累计暴发ASF疫情176起,扑杀猪只超过数百万头,保守估计造成直接经济损失数百亿元。由于无商品化疫苗可用,加之ASFV污染面积较广,我国ASF的防控和根除形势异常严峻[9-12]。因此,建立可鉴别不同ASFV毒株的荧光定量PCR方法对于该病的早期检测,以及深入研究ASFV致病机制和将来疫苗株与野毒株感染的鉴别检测都至关重要。【前人研究进展】ASFV是目前所知的唯一虫媒DNA病毒[2],也是非洲猪瘟相关病毒科的唯一成员,成熟的病毒粒子结构复杂,主要感染猪单核巨噬细胞[1-3]。ASFV基因组为170—194 kb的双链线性DNA,可编码近200个蛋白,末端含有5个多基因家族,其中和对ASFV的复制、免疫逃逸及致病性有重要作用。有研究表明,和可增强ASFV在软蜱细胞内的复制,也可抑制Ⅰ型干扰素的产生[13]。ASFV Malawi Lil-20/1株缺失和可降低病毒毒力[14-15],缺失ASFV Georgia 2007株和()的6个基因(和)可使病毒显著致弱,并可对亲本强毒株的致死性攻击提供完全保护[13]。我国分离的ASFV SY18株,缺失可以显著降低病毒毒力,与国外报道一致;在此基础上进一步缺失后表现出了较好的安全性和保护效果[16]。中国农业科学院哈尔滨兽医研究所构建的与多基因缺失病毒,能对中国流行的ASFV强毒株攻击提供100%的免疫保护,可作为安全、有效的ASF候选疫苗株[17]。由此可见,缺失ASFV毒株具有较好的研发和应用前景[13, 16-17]。现阶段,ASF的防控只能依靠加强生物安全措施、早期检测和严格的扑杀。针对ASF的检测技术主要有间接免疫荧光试验、红细胞吸附试验、病毒分离、聚合酶链式反应(PCR)和实时荧光定量PCR等[18-19]。【本研究切入点】目前,ASF的PCR及荧光定量PCR检测主要是针对ASFV B646L(p72)基因,诊断靶标相对单一[20]。因此,本研究以ASFV中国流行毒株为对象,选择多基因家族中相对保守又具研究和应用前景的MGF360-13L基因为靶标,建立ASFVTaqMan实时荧光定量PCR检测方法。【拟解决的关键问题】为后续MGF360-13L基因功能研究、缺失病毒的鉴定及相关基因缺失疫苗株的鉴别提供技术支持。
1 材料与方法
1.1 病毒与细胞
ASFV分离株ASFV HLJ/18株(GenBank登录号:MK333180.1)由中国农业科学院哈尔滨兽医研究所猪烈性传染病创新团队(本研究组)保存,ASFV MGF基因缺失毒株(ASFV-DMGF)由本研究组构建及鉴定(暂未发表数据);无特定病原体(SPF)猪肺泡巨噬细胞(PAM)由本研究组分离并保存。
特异性和敏感性质控样本的建立:猪瘟病毒(CSFV)石门株(GenBank登录号:AF092448.2)和伪狂犬病病毒(PRV)TJ株(GenBank登录号:KJ789182.1)均由本研究组增殖与保存;猪繁殖与呼吸综合征病毒(PRRSV)、猪传染性胃肠炎病毒(TGEV)、猪圆环病毒1型(PCV1)和猪圆环病毒2型(PCV2)阳性抗凝血各一份由本研究组鉴定与保存。取感染CSFV、PRV、ASFV的细胞培养物或者PRRSV、TGEV、PCV1、PCV2感染猪抗凝血各200 μL,提取核酸并对RNA病毒核酸进行反转录,于-20℃冻存,用于本研究的特异性和稳定性评价。本研究涉及的ASFV活病毒操作及相关阳性样品的处理均是在中国农业科学院哈尔滨兽医研究所生物安全3级实验室进行,符合ASF相关生物安全的要求。
1.2 主要仪器与试剂
DNA凝胶回收试剂盒、AxyPrepTMBody Fluid viral DNA/RNA提取试剂盒均购自康宁生命科学(吴江)有限公司;Premix PrimeSTAR HS、Premix Ex Taq™荧光定量试剂盒均购自宝生物工程(大连)有限公司;DH5α感受态、pOK12载体、质粒小提试剂盒均购自天根生化科技(北京)有限公司。荧光定量PCR仪(QuantStudio3&5)购自美国Thermo公司;紫外凝胶成像仪(AlphaImager HP)购自美国ProteinSimple公司;超微量分光光度计(NanoPhotmeter®)购自德国Implen公司;ClonExpress II One Step Cloning Kit一步同源重组试剂盒购自Vazyme公司。Antibiotic- Antimycotic和RPMI 1640含L-谷氨酰胺培养基购自赛默飞世尔科技(中国)有限公司。
1.3 引物和探针
根据GenBank中收录的ASFV全基因组(登录号:MK333180.1),选择序列设计扩增引物,13L-F:5′-ggtacccgggagctcgaattcCCTCTTCAAAGGCA TTACCA-3′;13L-R:5′-gtctgcagaagcttcgaattcCATTTT GAAATATTGTGGCCTA-3′(小写字母为左右同源臂序列);扩增片段大小800 bp。以序列为靶标,根据荧光定量PCR引物设计原则,利用Oligo7根据保守片段设计引物和探针并进行结构分析,经验证筛选出扩增曲线良好,电泳结果显示目的条带清晰的引物,序列为:F:5′-TCCTCTAGAGTTT CTTTGG-3′,R:5′-TCGTGTGGCATTAATAATAG-3′,扩增片段为159 bp;双标记探针信息如下:5′-(FAM)CAACTCTCGGATGTGCTTCGTATTG(TAMRA)-3′;以上引物和探针均由宝生物工程(大连)有限公司合成与验证。
1.4 阳性质粒标准品的构建
以本研究组保存的ASFV核酸为模板,利用引物对13L-F/13L-R扩增,反应体系为50 μL:Premix PrimeSTARHS 25 μL、ddH2O 21 μL、上、下游引物各1 μL、模板2 μL;反应条件为:预变性95℃ 5 min,变性95℃ 30 s,退火55℃ 30 s,延伸72℃ 1 min,扩增30个循环;将PCR扩增产物进行1%琼脂糖凝胶电泳;回收目的条带,利用限制性核酸内切酶RI切割pOK12载体,利用一步同源重组试剂盒将目的基因连接至pOK12载体上,并转化至DH5α感受态中,挑取阳性克隆提取质粒并进行测序验证。
1.5 荧光定量PCR标准曲线的建立及其特异性和敏感性
采用超微量分光光度计测定标准品浓度,根据公式:(6.02×1023拷贝/mol)×DNA量(g)/[DNA长度(碱基数)×660 g/(mol·碱基)] = 拷贝数,计算标准品拷贝数。利用ddH2O将标准品连续10倍梯度稀释至10-10,将其作为模板按照已优化的条件进行荧光定量PCR检测。反应体系为25 μL:Premix Ex Taq(Probe qPCR)12.5 μL、ddH2O 8.25 μL、上、下游引物各0.5 μL、探针1.0 μL、模板2 μL、ROX 0.25 μL;反应条件为:预变性95℃ 30 s,变性95℃ 5 s,退火55℃ 10 s,延伸72℃ 20 s,40个循环,其他条件为仪器默认设置。
另取CSFV、PRV、PRRSV、TGEV、PCV1、PCV2和ASFV的核酸按已优化的方法同时进行TaqMan荧光定量PCR检测,以评价其特异性。此外,将构建的质粒标准品梯度稀释后,利用本方法以及引物对13L-F/13L-R进行常规PCR进行检测,然后用1%琼脂糖凝胶电泳检测,以比较所建立的ASFVTaqMan荧光定量PCR检测方法的灵敏度。
1.6 临床样品检测
对发生ASF猪场采集的30份抗凝血灭活后每份取200 μL按照AxyPrepTMBody Fluid Viral DNA/RNA提取试剂盒说明书操作提取DNA。利用本研究组已建立的方法[20]及本方法同时对样品进行检测,利用SPSS 13.0软件进行一致性和配对卡方检验,比较两种检测方法结果之间的符合率。另对这30份样品进行了3次重复检测,对检测结果进行统计学分析,以评价该检测方法的稳定性与重复性。
1.7 ASFV MGF基因缺失毒株与野毒株感染PAM细胞检测鉴定
复苏的PAM细胞使用含L-谷氨酰胺、2%双抗和10%胎牛血清的RPMI 1640培养基培养于6孔板中,以3 MOI的ASFV HLJ/18与ASFV-∆MGF分别进行感染并做未接毒对照,48 h后收取病毒并保存。按照1.6方法提取DNA,每个样品进行3次重复,利用本方法进行检测。
2 结果
2.1 标准品的制备
利用引物13L-F/13L-R以ASFV基因组为模板进行PCR扩增,可以得到一条大小为800 bp的目的条带(图1)。利用一步克隆试剂盒将目的片段克隆至pOK12载体上,构建标准品,经测序验证后,提取标准品质粒并测得浓度为504 ng·µL-1,经计算每微升拷贝数为1.56×1011。
2.2 ASFV MGF360-13L TaqMan荧光定量PCR检测方法的建立
2.2.1 标准曲线的建立 对1.56×101—1.56×108拷贝/μL质粒标准品进行荧光定量PCR检测,以Ct值为纵坐标,标准品拷贝数的对数为横坐标,利用QuantStudioTMDesign & Analysis Software v1.4.3分析软件绘制其标准曲线(图2)。建立的ASFV荧光定量PCR线性回归方程为:= -3.295lg() + 45.995,2为0.997,扩增效率为98.12%,标准曲线为系统自动生成。
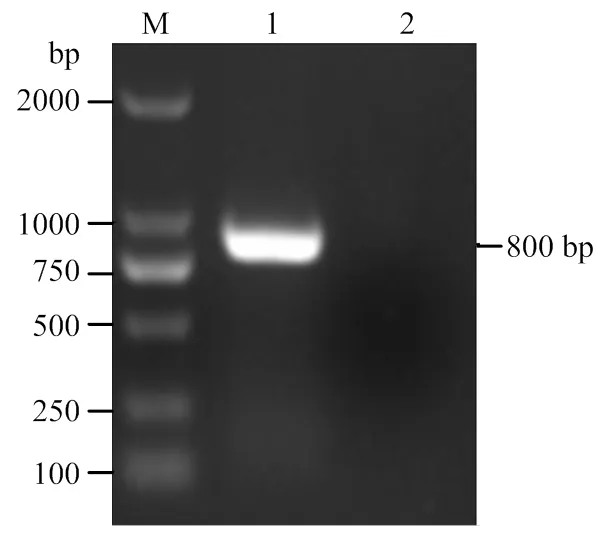
M: 2000 bp 分子量标准;1:MGF360-13L基因;2:阴性对照
2.2.2 ASFV荧光定量PCR的特异性和敏感性 以本研究建立的TaqMan荧光定量PCR对CSFV、PRV、PRRSV、TGEV、PCV1、PCV2和ASFV的核酸(cDNA/DNA)进行检测,除ASFV的DNA外,其余病毒核酸及阴性对照孔均未出现扩增曲线,未检测到Ct值(图3)。结果表明,本研究建立的ASFVTaqMan荧光定量PCR检测方法扩增效果良好,特异性强。
2.2.3 ASFV荧光定量PCR的重复性 选择10倍梯度稀释的质粒标准品的4个稀释度(1.56×109—1.56×106拷贝/μL)为模板,按照相同的条件进行3次组内和组间重复试验。结果显示(表1),组内变异系数(CV)均小于1%,组间变异系数(CV)均小于2%,表明本检测方法具有良好的重复性和稳定性。
2.2.4 ASFV荧光定量PCR的灵敏度 将10倍梯度稀释的标准品,进行常规PCR扩增,并将PCR产物进行1%琼脂糖凝胶电泳,反应条件和体系参照1.4。结果表明,使用Premix PrimeSTAR HS进行的常规PCR最低能够检测到约1.56×104拷贝/μL的病毒核酸(图4)。因此,本研究建立的TaqMan荧光定量PCR检测方法相比常规PCR检测方法在敏感性上高出约2—3个数量级。结果在1.56×1010拷贝/μL至1.56×101拷贝/μL各浓度可检出Ct值,而更低拷贝数时未检出Ct值。表明本方法最低可检测到15.6拷贝/μL的病毒核酸。
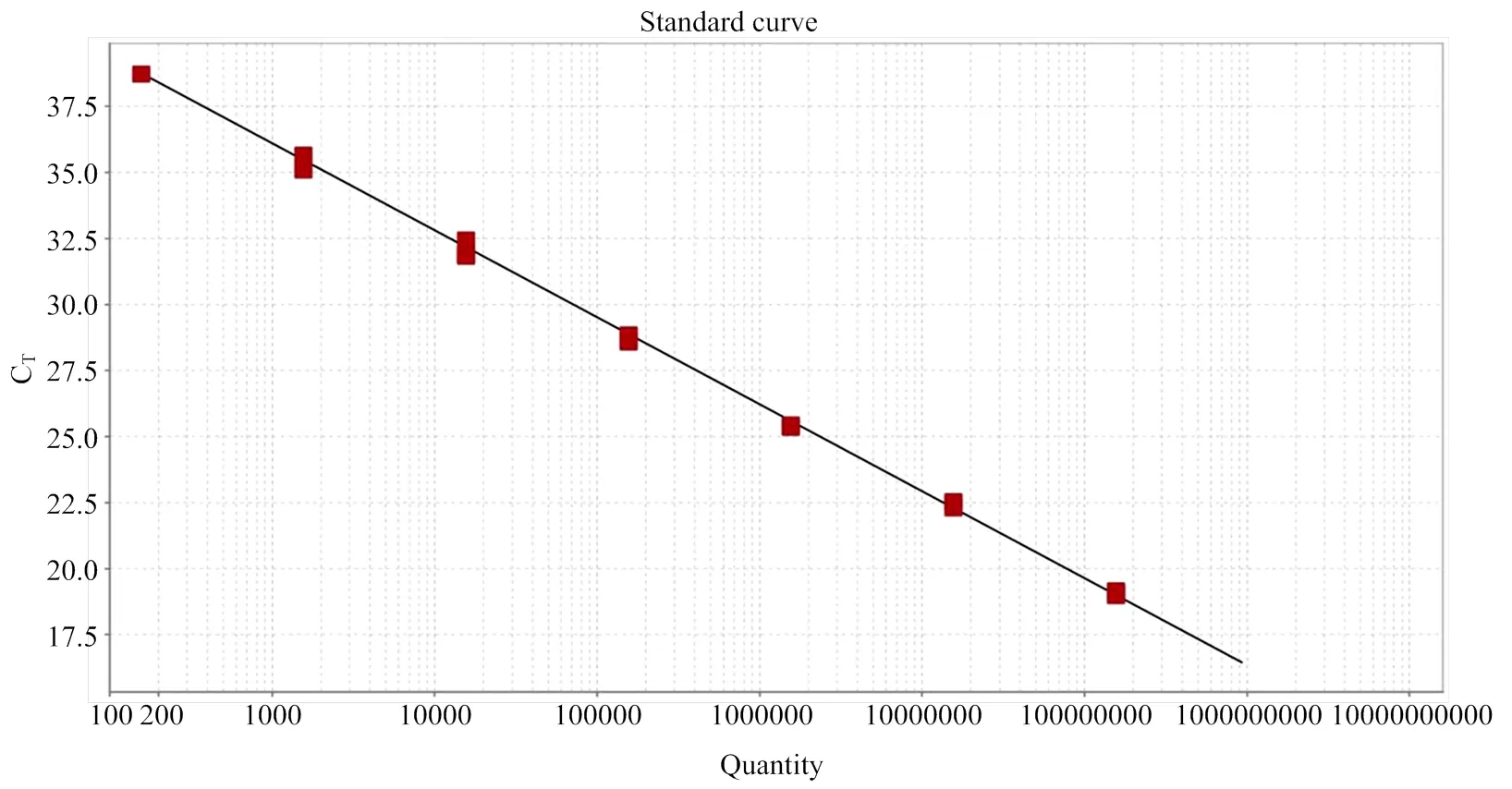
图2 ASFV MGF360-13L荧光定量PCR标准曲线

图3 ASFV MGF360-13L荧光定量PCR特异性检测
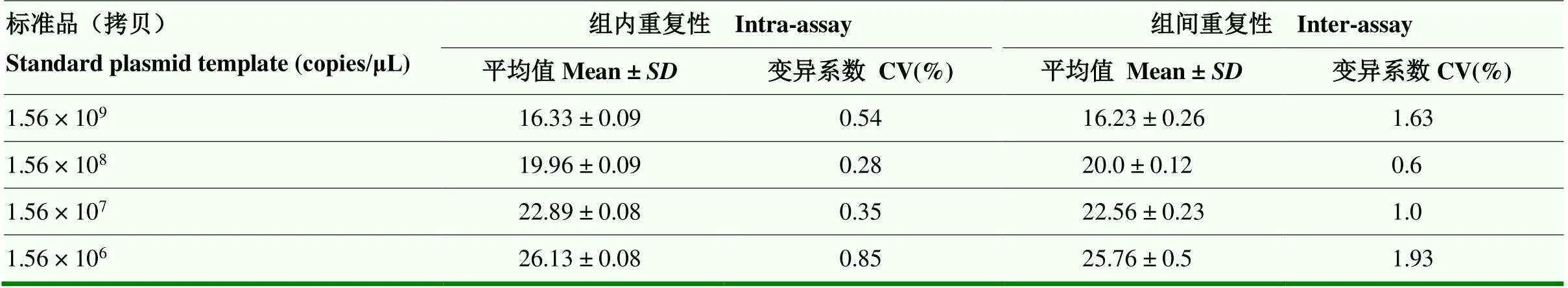
表1 MGF360-13L标准品TaqMan荧光定量PCR的批间、批内重复性
2.3 临床样品检测及符合率比较
用本研究建立的方法从30份临床样品中检出阳性样品14份,检出率为46.7%,未出现假阳性。McNemar检验的结果,=0.5>0.05,表明两种方法的检测结果无统计学差异;Kappa检验结果显示,Kappa=0.867>0.75,<0.001,提示两种检测方法符合率较好。利用本方法进行的3次重复试验,阳性样品检出均为14个,无假阳性;组内和组间变异系数均小于2.5%,表明本检测方法具有良好的稳定性和重复性。
2.4 ASFV疫苗毒与野毒感染检测
利用本方法检测感染ASFV野毒的PAM细胞,平均Ct值为23.5,对照组和感染ASFV-DMGF的PAM细胞无扩增曲线和Ct值(图5);表明该方法可以有效地区分ASFV野毒与缺失病毒感染。
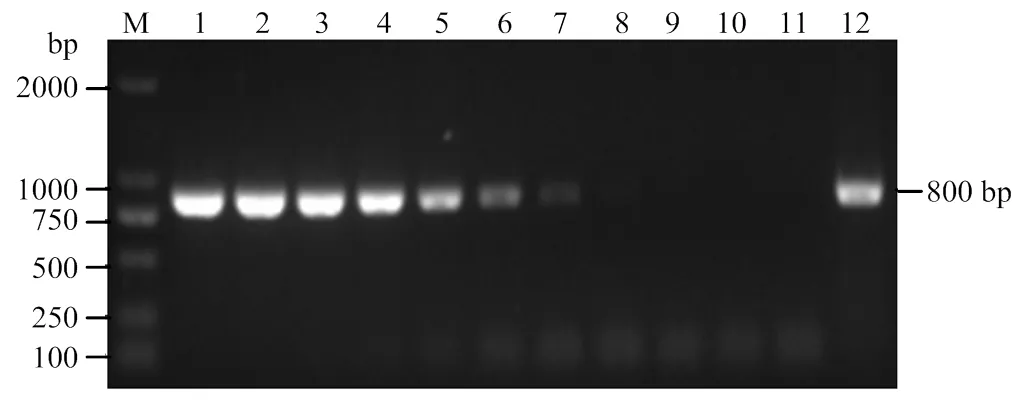
M: 2000 bp 分子量标准M:DL2000 DNA Marker;1-10:质粒标准品1.56×1010-1.56×101拷贝/μL 1.56×1010-1.56×101 copies/μL standard plasmid;11:阴性对照 Negative control;12:阳性对照 Positive control
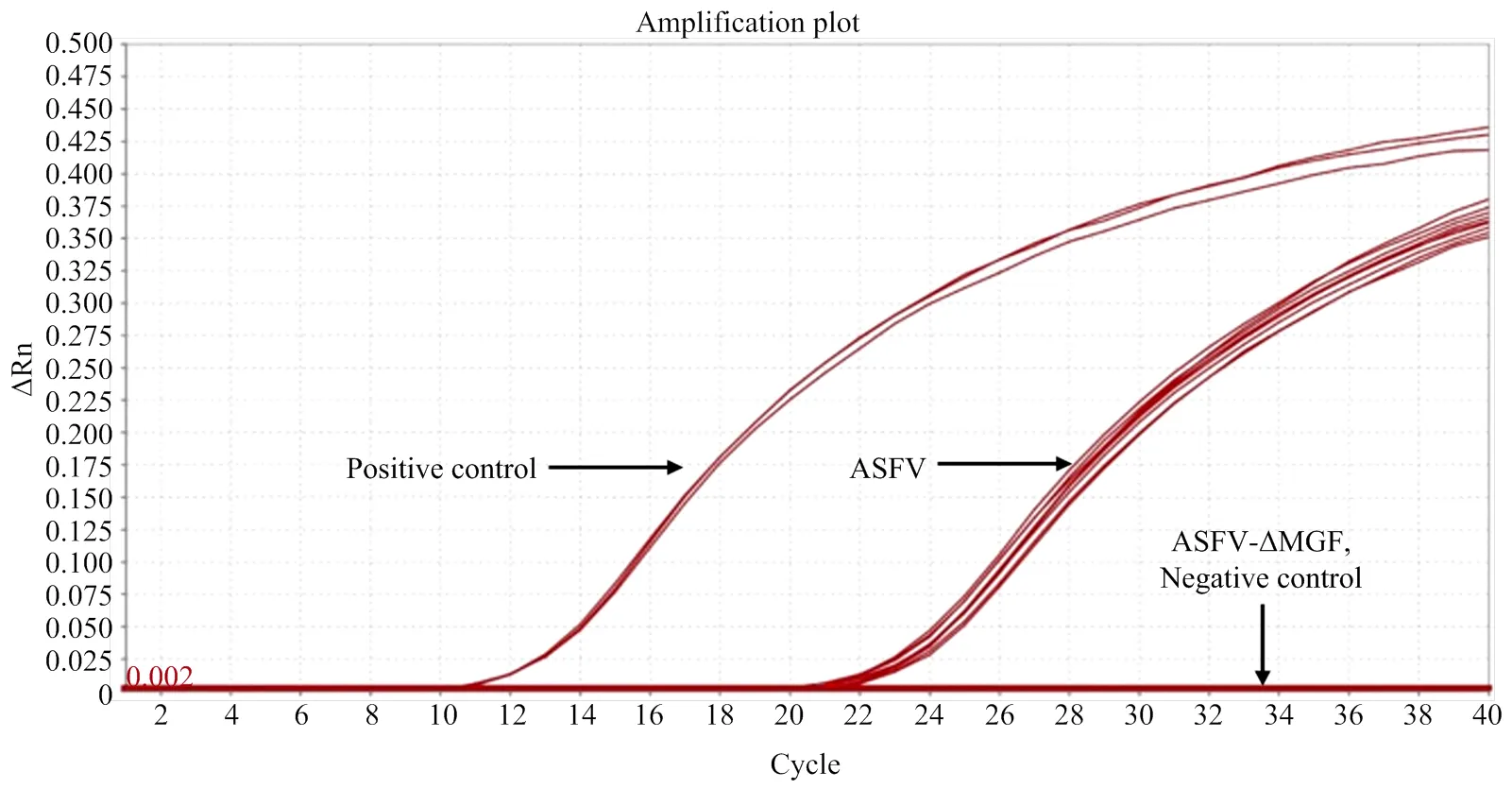
图 5 ASFV MGF基因缺失毒株与野毒株感染PAM细胞检测扩增曲线
3 讨论
ASFV结构复杂、基因组庞大,绝大部分基因功能未知严重制约了ASF疫苗和相关研究的开展[21]。已有研究表明,MGF家族的6个基因(和)为ASFV的重要毒力因子。国内相关研究也已证实,该部分基因也是我国ASFV流行毒株的毒力基因,该部分基因缺失的毒株将有较大的研究和应用前景[16-17]。有研究报道,将ASFV和替换为增强绿色荧光蛋白(Enhanced green fluorescent protein,EGFP)构建的荧光报告病毒不影响病毒在细胞中的复制,同时具备和亲本毒株相似的致病性,可应用于研究ASFV的致病机制及其与宿主之间的相互作用[22]。也有研究表明,MGF360-13L可抑制cGAS-STING信号通路[23],但具体机制仍不清楚。序列比对结果显示,在中国及东欧多个国家流行的11个毒株中同源性高达100%。基于以上分析,本研究选择为靶基因,建立其TaqMan实时荧光定量PCR检测方法。
ASF对养猪业危害严重且无安全有效疫苗及治疗药物可用,因此亟需开展综合防控技术及疫苗研发[24]。由于ASF的临床症状与多种病毒及细菌性疾病感染极为相似。因此,其确诊必须借助实验室检测[25]。实时荧光定量PCR具有极高的灵敏性和特异性,广泛应用于包括ASFV在内的多种病原检测[26]。经实验条件的优化,本研究建立的TaqMan荧光定量PCR检测方法可实现对最低15.6拷贝/μL ASFV核酸的检测,具有较高的灵敏性;同时,与CSFV、PRV、PRRSV、TGEV、PCV1和PCV2等多种病原不存在交叉反应,具有潜在应用价值。本研究建立的TaqMan荧光定量PCR检测方法相比使用高保真酶Premix PrimeSTAR HS扩增800 bp目的片段的常规PCR在敏感性上高出约2—3个数量级,与广泛应用的针对ASFV的荧光定量检测方法相比,具有较高的符合率,因此也可应用该方法对可疑样品进行二次确认。ASFV作为一个结构复杂、基因组庞大、危害严重的DNA病毒,除p72基因外,目前已建立的ASF荧光定量检测靶标主要有早期基因[27]、[28]和[29]等,本研究不仅丰富了ASFV现有检测靶标,也能够为区分ASFV MGF基因缺失毒株与野毒提供技术支持。ASF传入我国的2年多时间内已经造成了巨大经济损失,对养猪业及相关行业产生了巨大冲击[2,30]。由于ASFV在我国的污染面积大,加之养猪业生物安全水平较低,因此ASF的防控和根除将是一场持久战。十分有必要对ASFV展开系统全面的基础和应用研究,为我国防控和根除该病提供理论和技术支持[31],本研究也将为后续功能探索以及缺失病毒纯度鉴定等提供技术支持。
4 结论
本研究选择目前我国流行的非洲猪瘟病毒多基因家族中相对保守、功能重要又兼具研究前景的MGF360- 13L基因为靶标,建立其TaqMan荧光定量PCR检测方法。该方法特异性强、灵敏度高、重复性好,与已有的经典检测方法符合率高,不仅丰富了ASFV现有检测靶标,也能够鉴别ASFV MGF基因缺失毒株与野毒株感染。
[1] DIXON L K, CHAPMAN D A, NETHERTON C L, UPTON C. African swine fever virus replication and genomics. Virus Research, 2013, 173(1): 3-14.
[2] GALINDO I, ALONSO C. African swine fever virus: a review. Viruses, 2017, 9(5): 103.
[3] PENRITH M L, VOSLOO W. Review of African swine fever: transmission, spread and control.Journal of the South African Veterinary Association, 2009, 80(2): 58-62.
[4] SÁNCHEZ-VIZCAÍNO J M, MUR L, MARTÍNEZ-LÓPEZ B. African swine fever (ASF): five years around Europe. Veterinary Microbiology, 2013, 165(1/2): 45-50.
[5] ZHOU X T, LI N, LUO Y Z, MIAO F M, CHEN T, ZHANG S F, CAO P, LIN X D, TIAN K G, QIU H J, HU R L. Emergence of African swine fever in China, 2018. Transboundary and Emerging Diseases, 2018, 65(6): 1482-1484.
[6] GE S Q, LI J M, FAN X X, LIU F X, LI L, WANG Q H, REN W J, BAO J Y, LIU C J, WANG H, LIU Y T, ZHANG Y Q, XU T G, WU X D, WANG Z L. Molecular characterization of African swine fever virus, China, 2018. Emerging Infectious Diseases, 2018, 24(11): 2131-2133.
[7] WANG T, SUN Y, QIU H J. African swine fever: an unprecedented disaster and challenge to China. Infectious Diseases of Poverty, 2018, 7(1): 1-5.
[8] PIKALO J, ZANI L, HÜHR J, BEER M, BLOME S. Pathogenesis of African swine fever in domestic pigs and European wild boar - Lessons learned from recent animal trials. Virus Research, 2019, 271: 197614.
[9] MAZUR-PANASIUK N, WOŹNIAKOWSKI G, NIEMCZUK K. The first complete genomic sequences of African swine fever virus isolated in Poland. Scientific Reports, 2019, 9(1): 4556.
[10] BAO J, WANG Q, LIN P, LIU C, LI L, WU X, CHI T, XU T, GE S, LIU Y, LI J, WANG S, QU H, JIN T, WANG Z. Genome comparison of African swine fever virus China/2018/Anhui XCGQ strain and related European p72 genotype II strains. Transboundary and Emerging Diseases, 2019, 66(3): 1167-1176.
[11] 王涛, 孙元, 罗玉子, 仇华吉. 非洲猪瘟防控及疫苗研发: 挑战与对策. 生物工程学报, 2018, 34(12): 1931-1942.
WANG T, SUN Y, LUO Y Z, QIU H J. Prevention, control and vaccine development of African swine fever: Challenges and countermeasures. Chinese Journal of Biotechnology, 2018, 34(12): 1931-1942. (in Chinese)
[12] DIXON L K, ISLAM M, NASH R, REIS A L. African swine fever virus evasion of host defences. Virus Research, 2019, 266: 25-33.
[13] O'DONNELL V, HOLINKA L G, GLADUE D P, SANFORD B, KRUG P W, LU X, ARZT J, REESE B, CARRILLO C, RISATTI G R, BORCA M V. African swine fever virus Georgia isolate harboring deletions of MGF360 and MGF505 genes is attenuated in swine and confers protection against challenge with virulent parental virus. Journal of Virology, 2015, 89(11): 6048-6056.
[14] NEILAN J G, ZSAK L, LU Z, KUTISH G F, AFONSO C L, ROCK D L. Novel swine virulence determinant in the left variable region of the African swine fever virus genome. Journal of Virology, 2002, 76(7): 3095-3104.
[15] AFONSO C L, ZSAK L, CARRILLO C, BORCA M V, ROCK D L. African swine fever virus NL gene is not required for virus virulence. Journal of General Virology, 1998, 79(10): 2543-2547.
[16] 张艳艳, 周鑫韬, 齐宇, 缪发明, 王立冬, 米立娟, 杨金梅, 张静远, 张守峰, 杨金金, 王述超, 蒋依倩, 陈腾, 扈荣良. 非洲猪瘟病毒基因缺失疫苗构建和免疫保护特性研究, 中国兽医学报, 2019, 39(8): 1421-1427.
ZHANG Y Y, ZHOU X T, QI Y, MU F M, WANG L D, MI L J, YANG J M, ZHANG J Y, ZHANG S F, YANG J J, WANG S C, JIANG Y Q, CHEN T, HU R L. Construction and immune protective characterization of gene deleted African swine fever virus vaccine candidates. Chinese Journal of Veterinary Science, 2019, 39(8): 1421-1427. (in Chinese)
[17] 步志高, 陈伟业, 赵东明, 何希君, 刘任强, 柳金雄. 基因缺失的减毒非洲猪瘟病毒及其作为疫苗的应用: CN110093324A[P]. 2019-04-26.
BU Z G, CHEN W Y, ZHAO D M, HE X J, LIU R Q, LIU J X. Attenuated African swine fever virus with gene deletion and its application as vaccine: CN110093324A[P]. 2019-04-26. (in Chinese)
[18] ARIAS M, DE LA TORRE A, DIXON L, GALLARDO C, JORI F, LADDOMADA A, MARTINS C, PARKHOUSE R M, REVILLA Y, RODRIGUEZ F A J, SANCHEZ-VIZCAINO. Approaches and perspectives for development of African swine fever virus vaccines. Vaccines(Basel), 2017, 5(4): 35.
[19] OURA C A, EDWARDS L, BATTEN C A. Virological diagnosis of African swine fever comparative study of available tests. Virus Research, 2013, 173(1): 150-158.
[20] LUO Y, ATIM S A, SHAO L, AYEBAZIBWE C, SUN Y, LIU Y, JI S, MENG X Y, LI S, LI Y, MASEMBE C, STÅHL K, WIDÉN F, LIU L, QIU H J. Development of an updated PCR assay for detection of African swine fever virus. Archives of Virology, 2017, 162(1): 191-199.
[21] SÁNCHEZ EG, PÉREZ-NÚÑEZ D, REVILLA Y. Development of vaccines against African swine fever virus. Virus Research, 2019, 265: 150-155.
[22] BORCA M V, O'DONNELL V, HOLINKA L G, SANFORD B, AZZINARO P A, RISATTI G R, GLADUE D P. Development of a fluorescent ASFV strain that retains the ability to cause disease in swine. Scientific Reports, 2017, 7: 46747.
[23] 王西西. 非洲猪瘟病毒蛋白对cGAS-STING信号通路抑制作用研究[D]. 北京: 中国农业科学院, 2019.
WANG X X. Inhibition mechanisms of African swine fever virus protein on cGas-STING-mediated signaling pathway [D]. Beijing: Chinese Academy of Agricultural Sciences, 2019. (in Chinese)
[24] 仇华吉. 非洲猪瘟对我国养猪业的影响与防控建议. 中国兽药杂志, 2018, 52(11): 1-4.
QIU H J. Impacts of African swine fever on the Chinese pig industry and suggestions for the prevention and control of the disease. Chinese Journal of Veterinary Drug, 2018, 52(11): 1-4. (in Chinese)
[25] 张丽, 罗玉子, 王涛, 孙元, 仇华吉. 非洲猪瘟诊断技术发展现状与需求分析. 中国农业科技导报, 2019, 21(9): 1-11.
ZHANG L, LUO Y Z, WANG T, SUN Y, QIU H J. Current progress and demand analysis of technologies for African swine fever. Journal of Agricultural Science and Technology, 2019, 21(9): 1-11. (in Chinese)
[26] 姜睿姣, 张鹏飞, 朱光恒, 罗梓丹, 王印, 杨泽晓, 姚学萍, 罗燕. 非洲猪瘟检测技术进展. 病毒学报, 2019,35(3):523-532.
JIANG R J, ZHANG P F, ZHU G H, LUO Z D, WANG Y, YANG Z X, YAO X P, LUO Y. Diagnostic assays for African swine fever. Chinese Journal of Virology, 2019, 35(3): 523-532. (in Chinese)
[27] 崔贝贝, 李霆, 仇松寅, 梅琳, 韩雪清, 吴绍强, 林祥梅. 非洲猪瘟病毒E184L基因实时荧光定量PCR检测方法的建立. 动物医学进展, 2019, 41: 1-7.
CUI B B, LI T, QIU S Y, MEI L, HAN X Q, WU S Q, LIN X M. Establishment of a real-time PCR for detection of African swine virusgene. Progress In Veterinary Medicine, 2019, 41: 1-7. (in Chinese)
[28] 郭少平, 刘建, 吴绍强. 非洲猪瘟病毒实时荧光定量PCR检测技术的研究与评价. 中国畜牧兽医, 2010, 37(4): 76-80.
GUO S P, LIU J, WU S Q. Research and assessment of real-time quantitative PCR assay for the detection of African swine fever virus. Chinese Animal Husbandry and Veterinary Medicine, 2010, 37(4): 76-80. (in Chinese)
[29] 王建华, 董志珍, 赵丹, 王玉玲, 肖妍, 张俊哲, 陈小金, 王乃福, 陈本龙, 赵祥平. 一种基于CP530R序列非洲猪瘟病毒TaqMan- MGB探针实时荧光PCR检测方法的建立. 黑龙江畜牧兽医, 2016(3): 22-26.
WANG J H, DONG Z Z, ZHAO D, WANG Y L, XIAO Y, ZHANG J Z, CHEN X J, WANG N F, CHEN B L, ZHAO X P. Establishment of a TaqMan-MGB probe real-time fluorescence PCR method for detection of African swine fever virus based ongene sequences. Heilongjiang Animal Science and Veterinary Medicine, 2016(3): 22-26. (in Chinese)
[30] 赵启祖, 王琴. 非洲猪瘟紧急预防控制技术需求. 中国兽药杂志, 2018, 52(10): 1-4.
ZHAO Q Z, WANG Q. Technical requirements in African swine fever emergency prevention and control. Chinese Journal of Veterinary Drug, 2018, 52(10): 1-4. (in Chinese)
[31] 聂赟彬, 乔娟. 非洲猪瘟发生对我国生猪产业发展的影响. 中国农业科技导报, 2019, 21(1): 11-17.
NIE Y B, QIAO J. Impact of African swine fever on the development of pig industry in China.Journal of Agricultural Science and Technology2019, 21(1): 11-17. (in Chinese)
Development of a TaqMan Real-Time PCR Targeting the MGF360-13L Gene of African Swine Fever Virus
WANG Tao, HAN Yu, PAN Li, WANG Bing, SUN MaoWen, WANG Yi, LUO YuZi, QIU HuaJi, SUN Yuan
Harbin Veterinary Research Institute, Chinese Academy of Agricultural Sciences/Key Laboratory of Veterinary Biotechnology, Harbin 150069
【】The objective of this study is to develop a TaqMan real-time PCR for detection of African swine fever virus (ASFV)gene, providing technical support for diagnosis, virus purification, differential diagnosis ofdeletion ASFV strains, and gene function research of ASFV.【】In this study, the TaqMan real-time PCR primers and probes were designed based on the ASFV(GenBank: MK333180.1), and the TaqMan real-time quantitative PCR assay of ASFV was established. A pair of specific primers, 13L-F/13L-R was designed to construct ASFV standard plasmid. The standard curve was generated for quantitative analysis using the ten-fold dilution of the ASFV standard plasmid, and the sensitivity and repeatability of the system were also evaluated. The ten-fold dilution of the ASFV standard was also detected by PCR to determine the sensitivity of the method. Thirty clinical samples collected from an outbreak ASF pig farm in Heilongjiang province were simultaneously tested by this TaqMan real-time PCR, and another one previously established in our laboratory to compare the coincidence rate of these two detection methods. Differential diagnosis of parent ASFV andgene deletion strain after infection with PAM cell【】A specific band of about 800 bp was amplified using primer 13L-F/13L-R, and no band was found in the negative control, and the standard plasmid was successfully constructed. The standard curve had a good linear relationship between real-time PCR cycle threshold (Ct) and template copies. The linear regression equation was:= -3.295 lg() + 45.995 with correlation coefficient of 0.997, and the minimum detection limit was 15.6 copies/μl of ASFV nucleic acid. There was no cross-reaction with classical swine fever virus, pseudorabies virus, porcine reproductive and respiratory syndrome virus, porcine transmissible gastroenteritis virus, porcine circovirus type 1, and porcine circovirus type 2. In the clinical sample test, the results of the two methods in McNemar test,= 0.5>0.05, indicating that there was no statistical difference between the two methods and in Kappa test, Kappa = 0.867>0.75,<0.001, suggesting it was a good agreement and could effectively identify parent ASFV orgene deletion ASFV infection. 【】The TaqMan real-time PCR for detection of ASFVestablished in this study had highly specific, sensitive, reproducible, and high coincidence rate. It not only enriched the detection method of ASFV, but also provided technical support for researching the gene function of, identification of thegene-deleted ASFV, and differential diagnosis of related gene deletion vaccine strains.
African swine fever virus;; TaqMan real-time PCR; detection

10.3864/j.issn.0578-1752.2021.05.018
2019-12-31;
2021-01-11
国家重点研发计划项目(2017YFD0502300)
王涛,E-mail:wangtaocaas@163.com。通信作者仇华吉,Tel:0451-51051708;E-mail:qiuhuaji@caas.cn。通信作者孙元,Tel:0451-51051709;E-mail:sunyuan@caas.cn
(责任编辑 林鉴非)
