3种木材DNA提取方法比较研究
2021-03-03余敏魏宇创张浩刘盛全
余敏 魏宇创 张浩 刘盛全


摘 要:以微凹黄檀木材标本为研究对象,分别采用BATB法、PTB法和SDS法对微凹黄檀不同部位木材进行DNA提取试验,并扩增DNA条形码序列ITS2、trnH-psbA和matK,分析不同DNA提取方法对微凹黄檀木材DNA提取和DNA条形码序列扩增的影响。结果表明:采用BATB法从微凹黄檀木材心、边材组织中提取的DNA浓度以及DNA条形码扩增效率均要高于其他2种方法,说明BATB法更适合从长期存储木材中提取DNA。
关键词:微凹黄檀;DNA提取;DNA条形码;木材识别
中图分类号 S781.1 文献标识码 A文章编号 1007-7731(2021)03-0006-04
Comparison of Three Methods of DNA Extraction from Wood
YU Min et al.
(School of Forestry & Landscape Architecture, Anhui Agricultural University, Hefei 230036,China; Key Lab of State Forest and Grassland Administration on “Wood Quality Improvement & High Efficient”, Hefei 230036, China)
Abstract:The xylarium wood specimens of Dalbergia retusa were selected from the Wood Collection. Three methods, i.e., BTAB, PTB and SDS were employed to extract DNA in sapwood and heartwood for further PCR amplification of three potential DNA barcode sequences (ITS2, trnH-psbA, and matK). The effects of different methods on DNA extraction and PCR for target sequences were analyzed. The results showed that the concentration of DNA and efficacy of PCR using BTAB method was higher than that of PTB and SDS methods from sapwood and heartwood. The BTAB method is more suitable for extracting DNA from long-stored wood.
Key words:Dalbergia retusa Hemsl.; DNA extraction; DNA barcodes; Wood identification
相較于新鲜、幼嫩且具有分生能力的植物组织而言,从木材组织中提取和扩增DNA要困难得多,尤其是木材经过干燥和长期存储,进一步影响了DNA提取和扩增成功率[1]。随着DNA条形码技术在木材识别研究上的深入,研究人员发现从木材组织中提取足够质量和数量的DNA,是顺利应用DNA条形码技术识别和鉴定木材的基础和保证[2]。虽然已有研究报道了采用常规DNA提取方法提取木材组织DNA,但提取结果常常不能满足后续试验的需要[3]。因此,在传统的DNA提取方法基础上进一步优化木材DNA提取步骤,提高木材DNA提取效率和有效性,是长期存储木材DNA提取亟待解决的问题。为此,笔者以标本馆馆藏微凹黄檀木材标本为研究对象,分别采用BATB法、PTB法和SDS法对微凹黄檀木材进行DNA提取试验,并扩增DNA条形码序列ITS2、trnH-psbA和matK,分析不同DNA提取方法对微凹黄檀木材DNA提取和DNA条形码序列扩增的影响,探索适合长期存储木材基因组DNA提取的方法,以期为进一步开展长期存储木材DNA条形码识别研究奠定基础。
1 材料与方法
1.1 试验材料 微凹黄檀木材标本来自安徽农业大学木材标本馆。由于木材组织疏松多孔,运输和储存过程中易被其他外源植物组织污染,因此在进行木材DNA提取试验操作之前,需用无菌刀片去除木材表面部分,并将试样切成小片放置在全自动冷冻研磨仪(Retsch CryoMill)中研磨成木粉,避免木材组织在研磨过程中因温度过高造成DNA二次降解。
1.2 试验方法
1.2.1 木材DNA提取 分别采用BATB法、SDS法和PTB法对微凹黄檀标本心、边材进行提取。BATB法:向500mg木粉中加入4.5mL BoTab buffer、225μL DTT(1mol/L)、90μL蛋白酶K(20mg/mL),涡旋混匀;55℃水浴5h,期间颠倒混匀;加入4.5mL氯仿∶异戊醇(24∶1),涡旋混匀;用旋转混匀仪室温下混匀10min;12000rpm,4℃离心15min;用5mL枪头小心吸取上层水相到新50mL离心管中,加入等体积氯仿∶异戊醇(24∶1),上下颠倒混匀;12000r,4℃离心5min;移取上层水相,加入2.5倍体积的醋酸钠(3mol/mL,pH 5.2);加入与上层水相等体积的预冷异丙醇和糖原,混合均匀;-20℃低温放置2h;12000r,4℃离心15min,丢弃上清;加入5mL 70%无水乙醇洗涤白色沉淀物,轻弹离心管底数次。12000r,4℃离心15min;小心去除上清液,不要使白色沉淀流失。加入200μL无菌水,轻离心后置于4℃低温冰箱溶解1h,放入-20℃保存备用。SDS法[4]和PTB法[5]参照已发表文献方法进行DNA提取操作。
1.2.2 基因组DNA的纯化 前期研究表明[6],木材DNA提取原液中常含有未能彻底去除的色素等杂质,这些物质不仅干扰核酸的紫外吸收值,并且对后续PCR扩增反应具有强烈的抑制作用。因此,需要对提取后的DNA原液进行纯化处理,以满足下一步PCR扩增的需要。木材DNA纯化采用PCR扩增模板纯化试剂盒(High Pure PCR Template Preparation Kit,Roche)进行操作。
1.2.3 DNA纯度和浓度检测 用DeNovix DS-11FX核酸定量仪检测纯化后样品的A260和A280紫外吸收值。
1.2.4 DNA条形码片段的PCR扩增 为分析不同DNA提取方法对PCR扩增反应的影响,分别以3种方法提取的微凹黄檀木材不同部位纯化后的基因组DNA为模板,扩增3条DNA条形码候选片段:核基因组内转录间隔片段ITS2、叶绿体基因间隔区域trnH-psbA以及叶绿体基因片段matK。3条DNA条形码序列扩增引物信息和反应体系详见表1。
2 结果与分析
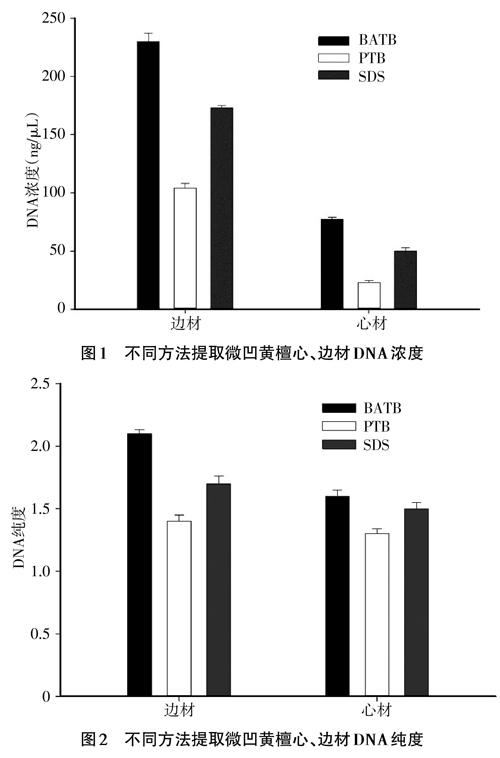
2.1 不同DNA提取方法提取微凹黄檀木材DNA的浓度和纯度 分别采用3种DNA提取方法提取微凹黄檀心、边材DNA。提取后的木材DNA原液经纯化后检测样品在260nm和280nm下的紫外吸收值,计算A260/A280的比值,测算纯化后的基因组DNA浓度。由图1可知,BATB法从心、边材组织中提取的DNA浓度均高于其他2种方法,PTB法提取的木材DNA浓度最低;3种DNA提取方法提取的边材DNA浓度均高于心材部分。由图2可知,BATB法提取的微凹黄檀心、边材DNA的A260/A280值均接近2.0,纯化后纯度较好;PTB法提取的微凹黄檀心、边材DNA的A260/A280值均小于1.5,纯化后的DNA仍存在蛋白质污染。
BATB法在经典的CTAB法基础上增加了糖原作为共沉剂。糖原作为惰性载体,离心时可使核酸分子聚于其上达到共沉淀的效果。试验结果表明,糖原能够有效地帮助降解木材的DNA小片段富集沉淀,提高DNA提取浓度,并进一步增加核酸沉淀效率。后续的纯化操作采用试剂盒进行操作,不仅减少了氯仿等有机试剂的使用,而且纯化小柱能够有效减少和避免DNA纯化过程中的污染。
2.2 不同DNA提取方法对微凹黄檀DNA条形码序列扩增的影响 以纯化后的微凹黄檀心、边材DNA为模板,分别扩增核基因组内转录间隔片段ITS2、叶绿体基因间隔区域trnH-psbA以及叶绿体基因片段matK,分析不同DNA提取方法对微凹黄檀木材DNA条形码序列扩增的影响。由图3可知,采用BATB法和SDS法提取的DNA模板均可以扩增出微凹黄檀ITS2片段,而以PTB法提取的DNA为模板扩增ITS2序列失败。ITS2片段在细胞核基因组中具有丰富的拷贝数量,能够为已降解的DNA模板提供足够数量的扩增模板。本研究中采用PTB法提取的微凹黄檀心、边材DNA的A260/A280值均小于1.5,表明其存在蛋白质等污染,而这些物质会干扰PCR扩增反应的顺利进行。
由图4可知,采用BATB法和SDS法提取的微凹黄檀心、边材DNA为模板可以成功扩增出trnH-psbA片段;而采用PTB法提取的微凹黄檀边材DNA为模板可以扩增出trnH-psbA片段,从心材中提取的DNA为模板无法扩增成功,较低的浓度无法满足扩增trnH-psbA序列的要求,从而导致扩增失败。
由图5可知,3种DNA提取方法提取微凹黄檀心、边材DNA扩增matK序列,仅有BATB法提取的邊材和心材部分可以成功扩增出matk片段。
3 结论与讨论
试验结果表明:BATB法从微凹黄檀心、边材组织中提取的DNA浓度和纯度均高于其他2种方法,PTB法提取的木材DNA浓度最低;以BATB法提取的微凹黄檀心、边材DNA为模板,扩增效果要优于其他2种方法。通过比较3种DNA提取方法对木材标本心、边材DNA的提取效果和扩增效率可知,BATB法和SDS法均要优于PTB法,且BATB法更适合长期存储的木材DNA提取。
相较于新鲜、幼嫩的植物叶片而言,从木材组织获取DNA并扩增出相应的基因片段存在着诸多不利因素,主要表现在:(1)木质部细胞编程性死亡过程中,DNA失去原生质体的保护,多降解成小片段且数量和质量均发生变化[7]。(2)木材中含有大量的厚壁管状分子,在提取木材组织中的DNA时需要处理纤维、导管类的坚硬组织,这会导致产生高温热量,而这些热量将导致不可逆的DNA分子降解[8]。(3)木材组织中的次生代谢物质和多酚类化合物易于氧化,氧化的多酚极易结合核酸分子导致DNA进一步降解。此外,木材中存在的蛋白质、酚类、多糖、单宁和色素等物质会影响DNA聚合酶的活性,干扰引物与模板的结合,从而导致PCR扩增的失败[9]。(4)木材存储环境和时间的影响,如潮湿环境下极易造成木材腐朽和真菌污染,导致DNA的进一步降解和污染。木材组织中的DNA也会随着储存时间的延长逐渐发生降解[10]。(5)木材加工过程中通常会有紫外线照射、化学处理和热处理等,这些处理将导致木材组织中残存的DNA进一步降解[11]。
参考文献
[1]Xiaoshu Tang, Guangjie Zhao, Liyan Ping. Wood identification with PCR targeting noncoding chloroplast DNA [J]. Plant Molecular Biology, 2011, 77(6):609-617.
[2]Lee Hong Tnah, Soon Leong Lee, Kevin Kit Siong Ng, et al. DNA extraction from dry wood of Neobalanocarpus heimii (Dipterocarpaceae) for forensic DNA profiling and timber tracking [J]. Wood Science and Technology, 2012, 46(5):813-825.
[3]Lichao Jiao, Yafang Yin, Fuming Xiao, et al. Comparative analysis of two DNA extraction protocols from fresh and dried wood of Cunninghamia lanceolata (Taxodiaceae) [J]. IAWA Journal, 2012, 33(4):441-456.
[4]王娟.不同温度干燥处理下的降香黄檀木材DNA提取方法的研究[D].合肥:安徽农业大学,2016.
[5]余敏,张浩,刘盛全,等.降香黄檀木材DNA提取及rDNA-ITS序列条形码分子鉴定[J].林产化学与工业, 2014, 34(5):103-108.
[6]Min Yu, Kai Liu, Liang Zhou, et al.Testing three proposed DNA barcodes for the wood identification of Dalbergia odorifera T. Chen and Dalbergia tonkinensis Prain[J]. Holzforschung, 2016,70(2):127-136.
[7]M.F. Deguilloux, M.H. Pemonge, R. Petit.Novel perspectives in wood certification and forensics:dry wood as a source of DNA [J]. Proceedings of the Royal Society of London. Series B:Biological Sciences, 2002, 269(1495):1039-1046.
[8]K. Weggler, M. Dobbertin, E. Juengling, et al.Dead wood volume to dead wood carbon:the issue of conversion factors [J]. European Journal of Forest Research, 2012, 131(5):1423-1438.
[9]Lichao Jiao, Tuo He, Eleanor E. Dormontt, et al. Applicability of chloroplast DNA barcodes for wood identification between Santalum album and its adulterants[J].Holzforschung, 2019, 73(2):209-218.
[10]Yanti Rachmayanti, Ludger Leinemann, Oliver Gailing, et al.DNA from processed and unprocessed wood:factors influencing the isolation success[J].Forensic Science International:Genetics, 2009, 3(3):185-192.
[11]Mervyn Shepherd, Michael Cross, Rhonda L Stokoe, et al. High-throughput DNA extraction from forest trees[J]. Plant Molecular Biology Reporter, 2002, 20(4):425.
(責编:徐世红)
基金项目:安徽省国际科技合作计划项目(1704e1002226);安徽省自然科学基金项目(1908085QC111)。
作者简介:余敏(1985—),男,河南信阳人,讲师,研究方向:木材DNA识别。 *通讯作者 收稿日期:2021-01-08
