克氏原螯虾PcCyc基因克隆及其对光周期的表达响应
2021-01-28蒋琦辰周开亚
谢 伟,蒋琦辰,孙 宾,杨 颖,李 鹏,严 洁,周开亚
(1.南京师范大学 生命科学学院,江苏 南京 210023; 2.江苏省淡水水产研究所,江苏 南京 210017 )
周期循环蛋白基因(Cyc基因)是生物钟基因的核心基因之一。生物钟是机体内部周期性接近24 h自我维持的时钟。这个时钟由分子振荡器组成,分子振荡器由转录激活因子和时钟本身及其他基因的抑制剂组成,分别形成正负循环[1]。时钟控制的生理和行为过程中受环境影响(如光照周期或食物摄入)[2]。Cyc基因是生物钟环路中的一个重要成员,通常其产物蛋白BMAL1蛋白与CLOCK蛋白形成异源二聚体,结合到period(Per)基因的 E-box盒上激活Per基因转录,在时钟基因调控网络中起到正调节的作用[3-5]。Cyc基因在脊椎动物中的同源基因被称为Bmal1基因,Bmal1基因于1997年被克隆获得,是调节昼夜节律钟的核心转录因子之一。PAS(PER-ARNT-SIM)结构域存在于多种蛋白质中,这些蛋白质在发育和适应环境中起作用[6]。PAS结构域也常见于具有碱性螺旋—环—螺旋(bHLH)结构域的蛋白质中,并且它们成对地充当异二聚体转录因子。Cyc基因包含bHLH/PAS转录因子[7-8]。bHLH/PAS作为转录因子,在中央神经系统的发育中起到至关重要的作用[9-10]。Cyc基因作为重要的生物钟转录基因,有许多重要的生理功能。已有研究发现,Bmal1基因的缺失会导致小鼠完全丧失昼夜节律,并完全破坏体内葡萄糖和甘油三酯水平的周期节律。另外特异性敲除小鼠肝胰腺Bmal1基因,会导致葡萄糖转运蛋白2的表达丧失昼夜节律性[11],并且小鼠在禁食期间存在低血糖风险[12]。
克氏原螯虾(Procambarusclarkii),属节肢动物门、甲壳纲、软甲亚纲、十足目、爬行亚目、螯虾科、原螯虾属,俗称小龙虾,在欧美部分地区也被称为红沼泽螯虾。近些年在中国随着克氏原螯虾需求量的增加,克氏原螯虾的人工繁殖培育不断发展,养殖面积逐年扩大,克氏原螯虾已成为中国水产品市场重要的淡水经济物种[13]。随着生物技术的发展,时钟基因的研究已证实,生物体内相关节律钟基因的紊乱可能导致代谢疾病、癌症、甚至死亡[14-16]。同时也发现生物的生理行为、新陈代谢、营养获取等都存在着明显的节律。认清节律,遵循节律的生活方式可以减少患病的风险[17-18]。在养殖过程中,明确经济物种的行为、生理、摄食和繁殖等的规律及其分子调控机理可以为健康养殖模式探索提供理论依据。虽然时钟基因在模式动物中的研究较多,但在水生甲壳动物中的调节机制尚不明确,自甲壳动物发现时钟基因以来,对其研究多集中于如何保持节律振荡及对生命活动节律的影响方面[19-21]。笔者克隆克氏原螯虾Cyc基因并进行生物信息学分析,探究不同光照模式下其mRNA组织差异表达情况,探究Cyc基因的节律振荡表达模式,以期了解克氏原螯虾生物钟基因对其生长发育及代谢的调控作用。
1 材料与方法
1.1 试验材料与处理
试验用克氏原螯虾样品于徐州(云龙区周边水域)野外捕捉采集,为除去性别的干扰,在进行试验时全部选取体长9~11 cm的雄性个体。活体样本采集完毕后,需要进行光照驯化。在实验室循环水箱中饲养15 d,温度恒定25 ℃,24 h充氧,箱底铺满沙石,并放置尼龙管和石块,模拟克氏原螯虾自然生境躲避模式。用全光谱日光灯模拟室外光照,光暗比12L∶12D(12LD)。驯化15 d之后将样品分成3组:第1组是驯化15 d之后的样品直接用于试验;第2组从中选取健康的雄性个体54只转入24 h持续黑暗中0L∶24D(24DD);第3组选取健康的雄性个体54尾转入72 h的持续黑暗中0L∶72D(72DD)。不同光照条件下的克氏原螯虾均在同一水箱中培养。在不同的光照模式下分别培养结束后,于时间点7:00、11:00、15:00、19:00、23:00、翌日03:00、7:00进行取样。每个时间点每组取2尾,共取3个平行对照组。使用的试验操作程序经南京师范大学生物医学研究伦理委员会[SOXR(江苏)2015-028]批准。
试验所用引物名称及序列见表1,引物合成及测序均由生工生物工程(上海)股份有限公司完成。
1.2 方法
1.2.1 RNA的提取及 cDNA的合成
将克氏原螯虾的肝胰腺、脑、眼柄组织按照Trizol试剂说明书提取总RNA,用Thermo NanoDrop 2000/2000C(Thermo Fisher Scientific,USA)分光光度计和1.0%的琼脂糖凝胶电泳检测RNA的质量和纯度,使用反转录试剂盒PrimeScriptTMRT Master Mix(TaKaRa,大连)对克氏原螯虾的RNA进行反转录,得到cDNA第一链。

表1 试验所用的引物序列
1.2.2 PcCyc基因表达序列标签序列的克隆和测序
根据从克氏原螯虾转录组中(Accession code: SRP044128)[22]获得的PcCyc基因部分序列设计相关引物,引物见表1。将逆转录合成的cDNA稀释10倍作为模板扩增EST序列。反应体系包括:Taq Master Mix 10 μL,ddH2O 7 μL,Cycle-ESF 1 μL,Cycle-ESR 1 μL,cDNA 1 μL。扩增条件为:95 ℃预变性5 min;95 ℃ 30 s,62 ℃ 30 s,72 ℃ 1 min,共29个循环;最后72 ℃ 10 min。PCR产物经1.0%琼脂糖凝胶电泳检测,回收后与pMD20-T Vector载体连接,连接产物转化DH-5α感受态细胞,通过蓝白斑筛选挑取阳性克隆菌落,经菌落PCR检测后于生工生物工程(上海)股份有限公司测序。将测序所得序列在美国国立生物技术信息中心上采用Blast工具进行比对、验证。
1.2.3 PcCyc基因全长cDNA的克隆
选取已验证的EST序列,用Primer Premier 5.0软件设计3′RACE和5′RACE的特异性引物和巢式PCR反应的特异性引物。先对5′RACE PCR进行前处理再进行反转录。对提取的总RNA进行CIAP、TAP处理,与5′RACE Adaptor连接后反转录合成cDNA。之后进行巢式PCR反应。同样对3′RACE PCR进行前处理,合成cDNA时要设立M-MLV(-)对照。反应体系:Total RNA(1 μg/μL) 1 μL,3′RACE Adaptor(5 μmol/L) 1 μL,dNTP Mixture(10 mmol/L each) 1 μL,RNase Free deionized H2O 4.5 μL。反应条件:70 ℃,10 min。上述反应结束后立即置于冰上2 min,然后加入下列组分。反应体系:5×M-MLV Buffer 2 μL,RNase Inhibitor (40 U/μL) 0.25 μL,Reverse Transcriptase M-MLV (200 U/μL) 0.25 μL。反应条件:42 ℃ 60 min,70 ℃ 15 min。之后进行巢式PCR反应。将3′RACE和5′RACE的扩增产物经琼脂糖凝胶电泳,切胶纯化并且连接到PMD18-T载体,转化大肠杆菌(Escherichiacoli)DH-5α感受态细胞,筛选阳性克隆并送生工生物工程(上海)股份有限公司测序。
1.2.4 RACE PCR序列验证
为验证RACE PCR扩增的序列是否正确,以EST序列验证时的cDNA为模板,使用高保真酶,Cycle-QCF/Cycle-QCR 为引物,进行PCR 扩增。反应体系及条件同 EST 序列验证。反应结束后,取PCR产物于1%的琼脂糖凝胶上电泳检测。根据检测结果确定使用 PCR 产物直接纯化还是凝胶回收[使用MiniBEST Agarose Gel DNA Extraction Kit Ver.3.0 (TaKaRa,大连)切胶回收 PCR 产物],用 Cycle-QCF/Cycle-QCR 对 PCR 产物进行测序。
1.2.5 生物信息学分析
利用美国国立生物技术信息中心(http:∥www.ncbi.nlm.nih.gov/BLAST) Blast验证PcCyc基因的cDNA全长。用ORF Finder查找PcCyc基因的开放阅读框。在线服务器ExPASy的ProtParam工具分析PcCYC蛋白相关的理化性质。在线软件Motif Scan可以用来搜索PcCYC蛋白的motif。分别用在线服务器ExPASy的ProtScale工具和在线软件SignalP分析PcCYC蛋白的疏水性和预测分析信号肽。TMHMM Server v.2.0预测分析PcCYC蛋白跨膜区。在线软件PSORT Ⅱ Prediction预测分析PcCYC蛋白结构域。PcCYC蛋白二级结构预测使用PSIPRED。三级结构预测分析使用在线服务器Zhang Lab。
1.2.6 不同昼夜节律表达模式分析
克氏原螯虾不同光照的样本用Trizol法进行总RNA提取;并各取500 ng用反转录试剂盒PrimeScriptTMRT Master Mix(TaKaRa,大连)进行逆转录合成cDNA。同时选定克氏原螯虾β-actin作为内参基因检测cDNA模板并设计荧光引物,所有的引物见表1。用SYBR®Premix Ex TaqTMⅡ(TaKaRa,大连)试剂在荧光定量仪ABI StepOnePlusTM(Applied Biosystems)上对不同时期的各个组织的基因表达情况进行测定。反应体系:20 μL,包括10 μL 的SYBR®Premix Ex-TaqTMⅡ、0.8 μL 的正反向引物、6.4 μL的PCR反应水、2 μL的cDNA模板和0.4 μL ROX。反应程序:95 ℃ 10 min;95 ℃ 10 s, 60 ℃ 30 s,40 个循环; 95 ℃ 15 s,60 ℃ 1 min, 95 ℃ 15 s。样本和内参均设置3个重复,反应结束后分析相关数据,采用比较Ct值的方法(基因相对表达量=2-△△Ct)来分析PcCyc基因的相对表达水平。PcCyc基因表达数据的统计分析使用STATISTIC 10.0进行单因子方差分析和Duncan′s多重比较分析,显著性水平以α=0.05表示。
2 结 果
2.1 cDNA序列及生物信息学分析
5′RACE PCR和3′RACE PCR产物经测序分别得到312 bp的5′端序列,1185 bp和1129 bp的3′端序列。经测序验证结果表明获得的RACE PCR产物序列是根据已知EST序列扩增所得(图1)。PcCyc基因开放阅读框序列扩增验证见图2。

图1 PcCyc基因RACE PCR序列验证Fig.1 RACE PCR sequence verification of the PcCyc gene M.DL5000 DNA marker; 1、2.PcCyc 基因. M.DL5000 DNA marker; 1,2.cDNA full-length amplified PCR products.
PcCyc基因的cDNA部分序列分析结果显示,它具有一个143 bp的5′端非翻译区(UTR),本试验中并未获得3′端非翻译区,其开放阅读框包含2010个碱基(GenBank登录号MN908586),编码669个氨基酸。PcCyc基因的开放阅读框中还包含一个典型的bHLH/PAS转录因子结构。翻译过程中起始密码子为ATG(位于144~146位),终止密码子为TGA,在本试验中获得的序列A+T含量为56.5%,G+C含量为43.5%。

图2 克氏原螯虾PcCyc基因cDNA序列及氨基酸序列分析Fig.2 cDNA sequence and amino acid sequence analysis of PcCyc gene in red swamp crayfish P. clarkii N表示糖基化位点; *表示终止密码子; □表示 CK2; ﹏表示 PKC; 蓝色标注表示扩增过程中所用到的引物序列. N denotes Glycosylation site; * denotes termination codon; □ denotes CK2; ﹏ denotes PKC and the primer sequence used in the amplification process is shown in blue.
自美国国立生物技术信息中心下载Cyc基因产物蛋白的同源序列,使用在线软件Portal CIPES基于最大似然法构建系统发生树,发现PcCyc基因与甲壳类的Cyc基因聚为一支,且与十足目通讯螯虾(Pacifastacusleniusculus)的Cyc基因极为相似,氨基酸相似度为93%(图3),表明本试验中扩增获得的PcCyc序列属于Cyc基因家族。
2.2 PcCYC蛋白性质分析
2.2.1 蛋白质理化特性
PcCYC蛋白的理化性质分析使用在线ProtParam工具进行,对克氏原螯虾PcCYC蛋白质的理化参数分析显示,PcCYC蛋白的分子量约为73.963 ku,理论等电点为6.56,原子总数为10 222个,化学分子式为C3192H5035N929O1036S30;在组成PcCYC蛋白的20多种氨基酸中,丝氨酸所占的比例最高,达到13.5%,色氨酸所占的比例最低,为1.2%;脂肪指数为69.07;不稳定指数达到50.54。
2.2.2 PcCYC蛋白功能结构域分析与Motif搜索
PcCYC蛋白结构域的预测分析采用SMART在线软件进行,分别给出了PcCYC蛋白结构功能域及其相关信息(图4)。预测结果显示,PcCYC蛋白含有BHLH/PAS结构域,包括1个HLH结构域和2个PAS结构域。这些结构域可以相互作用形成异二聚体,并结合下游基因的E-Box区域,行使其生物学功能。PcCYC蛋白的Motif搜索利用在线软件Myhits Motif Scan进行。结果显示,PcCYC蛋白可能包含19个酪蛋白激酶Ⅱ(CK2)磷酸化位点,14个蛋白激酶C(PKC)磷酸化位点,12个豆蔻酸化位点等(图2)。
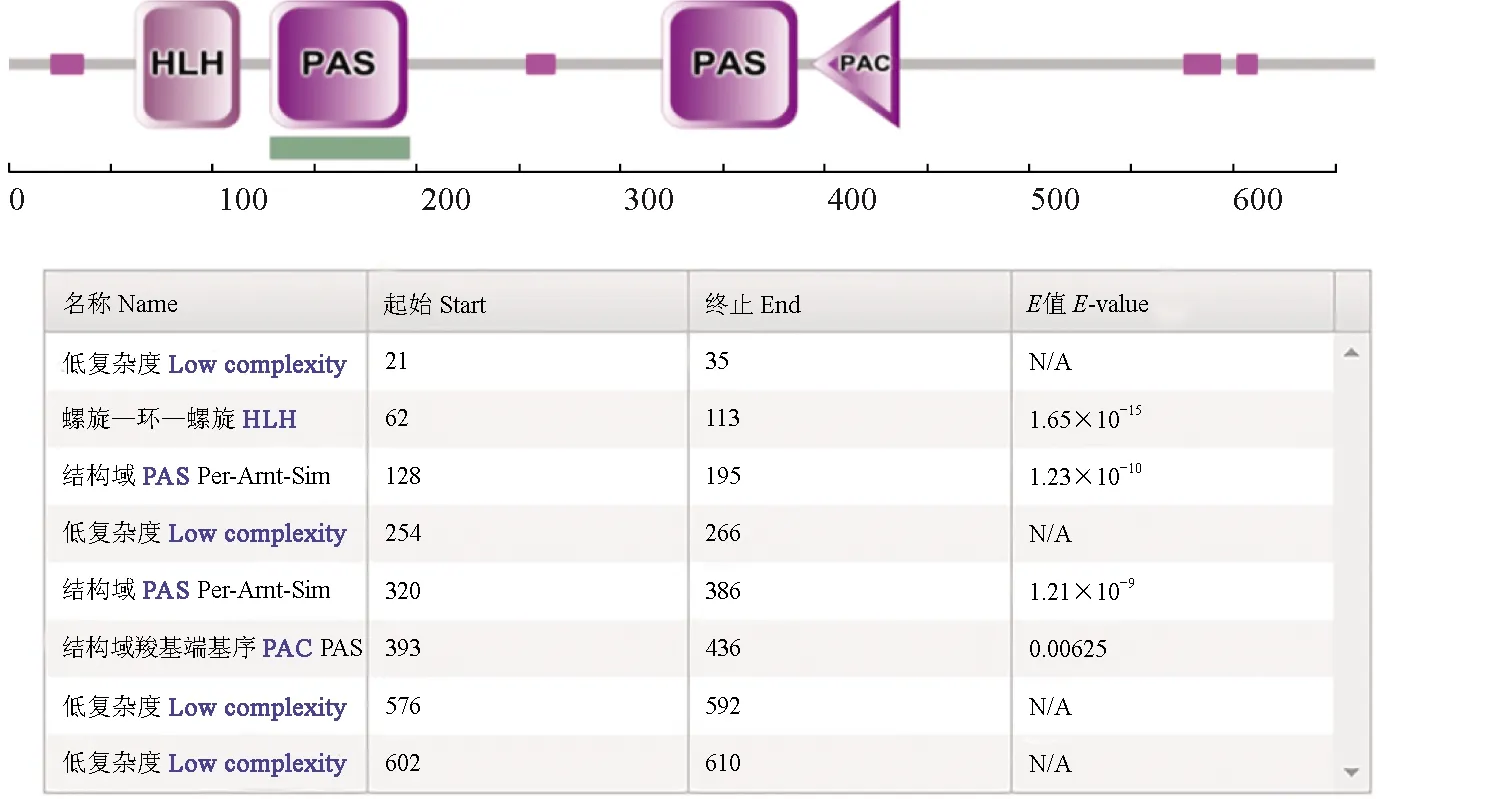
图4 PcCYC蛋白结构功能域分析预测Fig.4 Analysis and prediction of structure and functional domain of PcCYC protein
2.2.3 PcCYC信号肽预测
SignalP 4.1 Server工具分析PcCYC蛋白并没有发现信号肽,因此该蛋白为非分泌蛋白,会直接进入细胞质,易于富集,在细胞核中将与PcCLK蛋白形成异二聚体行使其生物学功能。
2.2.4 PcCYC蛋白亚细胞定位分析
采用PSORT Ⅱ Prediction在线服务器对PcCYC蛋白进行亚细胞定位预测。结果显示,PcCYC蛋白位于细胞核的可能性为95.7%,定位于细胞质的可能性为4.3%,因此PcCYC蛋白为核蛋白,主要存在细胞核中。
2.2.5 PcCYC蛋白空间结构预测
采用PSIPRED在线服务器预测PcCYC蛋白的二级结构,结果显示,PcCYC蛋白含有11个α-螺旋、12个β-折叠、24个无规则卷曲。采用在线服务器Zhang Lab对PcCYC蛋白进行三维结构预测。图5中黄色代表β-折叠,蓝色代表α-螺旋,红色代表无规则卷曲;同时PcCYC蛋白含有2个PAS结构域、一个BHLH结构域被分别标出,它们是PcCYC蛋白与其他蛋白互作行使功能的结构域。

图5 PcCYC蛋白三级结构预测Fig.5 The predicted tertiary structure of PcCYC protein
2.2.6 蛋白质疏水性
采用在线服务器ExPASy的ProtScale工具对PcCYC蛋白的疏水性进行预测,结果显示,PcCYC蛋白的最高峰值为2.554,最低峰值为-3.578,表明PcCYC蛋白为疏水性蛋白。
2.3 不同光照下PcCyc基因在不同组织中mRNA的表达分析
以β-actin基因作为内参,使用实时荧光定量PCR(qPCR)技术检测了不同光照条件下PcCyc基因在脑、肝胰腺和眼眪3种组织中的mRNA表达水平,结果表明,3种光照条件下,PcCyc基因在眼柄和肝胰腺中都基本遵循着24 h的节律振荡,而在大脑中的节律振荡随光照周期改变被打破(图6)。12LD的光照条件下,大脑与眼柄中PcCyc基因基本上有相似的表达规律,但在24DD的光照条件下,相似的规律被打乱了。在24DD光照条件下克氏原螯虾眼柄中PcCyc基因表达高峰比12LD条件下的提前了4 h,在72DD条件下,节律振荡恢复,但是与12LD的节律规律略有区别。
3 讨 论
生物钟一直是科学领域研究的热点问题,它是生命对地球光照以及温度等环境因子周期变化长期适应而演化的内在自主计时机制[23]。本试验主要采用RACE PCR技术首次获得了克氏原螯虾生物钟核心基因PcCyc的部分cDNA序列,包括完整的开放阅读框,共2010个碱基,可编码669个氨基酸(即PcCYC蛋白)。截至目前,甲壳类Cyc基因,仅有斑点海虱、通讯螯虾、沙滩跳钩虾(Talitrussaltator)和南极磷虾(Euphausiasuperba)4个物种被报道[8,24-25]。
3.1 克氏原螯虾PcCyc基因的生物信息学分析
本试验中使用PcCyc基因翻译的氨基酸序列对PcCYC蛋白进行相关的理化性质、功能结构域分析。其中功能结构域分析PcCYC蛋白含有典型的bHLH/PAS转录因子结构域,在CYC蛋白中该结构域的主要功能是与CLK蛋白结合成异二聚体在时钟调控网中行使功能[26-27]。对斑点海虱Cyc基因的研究也显示,其产物蛋白同样包含BHLH/PAS结构域[8]。这个结构域在果蝇、小鼠、人类等的CYC蛋白中普遍存在[26-28],是CYC蛋白公认的结构域之一,也是CYC蛋白的主要功能结构域。表明了CYC蛋白结构在进化过程中的保守性,也预示了其功能的保守性。已有研究显示,甲壳动物bHLH-PAS家族的成员通过幼年激素信号传导调控生长和繁殖[29],通过昼夜节律计时和对氧张力变化的响应来调节运动[30]。这对于养虾业来说非常重要,水产养殖业经常面临可能会影响养殖产量的环境条件波动的压力。

图6 克氏原螯虾PcCyc基因mRNA表达水平分析Fig.6 Analysis of mRNA expression levels of PcCyc gene in red swamp crayfish P. clarkii
3.2 克氏原螯虾PcCyc基因在不同光周期下的组织表达模式分析
Escamilla-Chimal等[31]利用抗体检测了克氏原螯虾CLK蛋白在不同光照下不同组织中的表达情况,结果表明,CLK 蛋白在12LD 的光照条件下,眼柄组织中于12:00出现蛋白表达高峰,在24DD的光照条件下,脑组织中于16:00出现蛋白表达高峰,在调控网络中 CYC 蛋白与CLK 蛋白形成异二聚体共同行使功能,因此推测这两种蛋白基因可能具有相似的mRNA表达水平。在时钟基因调控网络中,时钟基因蛋白的积累会比mRNA的生成延迟6~8 h[32-34]。本试验通过实时荧光定量PCR技术检测PcCyc基因在不同光照下(12LD、24DD和72DD)克氏原螯虾不同组织(脑、眼柄和肝胰腺)中的mRNA表达水平(图6)。结果显示,克氏原螯虾PcCyc基因在眼柄组织中的mRNA表达水平与之符合,但是脑组织的表达有4 h的出入,原因有待进一步研究。在24DD与72DD条件下,脑组织中该基因的mRNA的表达与 Escamilla-Chimal等[31]的结果相比基本上符合蛋白积累延迟8 h的规律。在克氏原螯虾肝胰腺中的PcCyc基因,由于肝胰腺属于外周振荡器,不同于眼柄中靠近视网膜上的光感受器,肝胰腺中生物钟基因的节律振荡主要调节生物的激素水平[35],参与调控机体的生理生化反应振荡。虽然最终要恢复起初的振荡水平,但由于生理生化的调节很复杂,可能需要更长的时间去恢复原有的节律振荡,以使生物适应外界环境的改变。结果分析显示,在改变光照的条件下,PcCyc基因慢慢地会趋于遵循24 h的昼夜周期振荡节律,并且在脑组织中有较高的起伏变化,以此证明时钟基因PcCyc有显著的内源性自由运转节律,并受脑组织支配。在斑点海虱脑组织中Cyc基因的mRNA表达虽受到潮汐节律的影响,但还是遵循着近日24 h的节律振荡,且节律振荡独立由脑控制,外部环境条件只会影响节律,而不会控制节律[8]。
4 结 论
在本试验中,采用RACE PCR技术克隆了克氏原螯虾的核心生物钟基因PcCyc的部分cDNA序列(2153个碱基),包括2010个碱基的开放阅读框(编码669个氨基酸);同时利用实时荧光定量PCR技术检测PcCyc基因在不同光照模式下(12LD、24DD和72DD)克氏原螯虾不同组织(脑、眼柄、肝胰脏)中的差异表达。在改变光照的条件下,PcCyc基因慢慢地会趋于遵循24 h的昼夜周期振荡节律,在大脑中的节律振荡随光照周期改变被打破。试验结果表明,克氏原螯虾的时钟基因PcCyc有显著的内源性自由运转节律,并受脑组织支配。本试验结果为研究克氏原螯虾生物钟基因对克氏原螯虾生长发育及代谢的调控作用奠定了一定的基础。
