乳酶生生产用菌种的全基因组序列分析研究
2020-12-17王珊珊石继春杜宗利石刚龙新星徐颖华叶强
王珊珊,石继春,杜宗利,石刚,龙新星,徐颖华,叶强
·论著·
乳酶生生产用菌种的全基因组序列分析研究
王珊珊*,石继春*,杜宗利,石刚,龙新星,徐颖华,叶强
102629 北京,中国食品药品检定研究院卫生部生物技术产品检定方法及其标准化重点实验室
了解我国乳酶生的生产用菌种全基因组分子特征。
将乳酶生的生产用菌种屎肠球菌 CMCC32901 培养后,依次进行形态观察、生化鉴定与 16S rRNA基因序列测定,并应用高通量测序技术,对生产用菌种 CMCC32901 进行全基因组序列测定,分析基因组基本特征、基因功能、同源性及致病性。
分析结果显示,发现 CMCC32901 菌株与 NCBI 数据库中收录的不同屎肠球菌菌株的 16S rRNA基因序列相似度大于 99.5%,与其他肠球菌菌株之间的 16S rRNA 基因序列相似度均大于 92.0%。屎肠球菌 CMCC32901 染色体全基因组序列全长为 2 394 726 bp,平均 GC 含量为 39.1%,共含有 2718 个基因,其中具有 COG 功能 2059 个,参与 KEGG 代谢通路 1402 个。同源性分析显示其基因组中编码 2589 个屎肠球菌特异性蛋白。细菌的致病性预测分析发现在 CMCC32901 菌株中发现 5 种对人潜在致病性相关蛋白。
首次获得了我国乳酶生生产用菌种——屎肠球菌 CMCC32901 全基因组的基本特征,为后续乳酶生的生产工艺优化和生产用菌种的质量控制奠定了基础。
乳酶生; 屎肠球菌; 生产用菌种; 全基因组序列; 质量控制
乳酶生是一种含有屎肠球菌的传统活菌制剂,主要利用其所含的活菌可在宿主肠道内分解糖类生产乳酸,使肠道内酸度增高,从而抑制肠道内其他一些菌群的生长繁殖。在临床上乳酶生作为助消化药,广泛用于肠内异常发酵引起的肠胀气,消化不良引起的腹泻等症状[1]。在国家药监局数据库查询发现,当前在全国范围内有近 20 家生产企业获得批准文号 36个,生产不同剂型的乳酶生。
生产用菌种是药品研究、生产和检定的物质基础,其质量直接与药物终产品安全和有效性密切相关[2]。目前在 2020 版《中国药典》二部“乳酶生”专论和三部“微生态活菌制品总论”中对乳酶生的生产用菌种质量控制主要包括形态观察、培养特性、产酸活性和安全性等比较传统的方法,缺乏菌株水平的分子特征方面的评价[3-4]。此外,我国乳酶生的生产用菌种 CMCC32901(原编号:140623)自 20 世纪 50 年代用于生产以来,随着细菌鉴定水平提高以及命名规范化,从最初菌种名称——乳酸肠球菌,历经多次命名的变化,确定为屎肠球菌[5]。因此,基于药品生产用菌种的菌株溯源、制剂工艺优化以及检定等方面考虑,进一步加强药品生产用菌种的质量控制,了解该生产用菌种菌株的分子特征和遗传背景就显得尤为重要。本研究首次应用高通量测序技术,对乳酶生的生产用菌种 CMCC32901 进行全基因组序列测定,分析其基因组基本特征、基因功能、同源性及致病性,为后续的药物质量控制研究奠定基础。
1 材料与方法
1.1 材料
1.1.1 菌株 屎肠球菌生产用菌种 CMCC32901 来源于中国食品药品检定研究院中国医学细菌保藏管理中心。
1.1.2 主要试剂和仪器 2% 含糖牛肉及其琼脂培养基与 10% 牛奶培养基均由中检院三药公司配制;其他试剂均为国产分析纯;DNA 提取试剂盒购自美国 Qiagen 公司;细菌培养箱为中仪国科(北京)科技有限公司产品;VITEK 全自动微生物鉴定仪为梅里埃诊断产品(上海)有限公司产品。
1.2 方法
1.2.1 形态观察与生化鉴定 将生产用菌株CMCC32901 接种于 2% 含糖牛肉培养基复苏,然后再接种至 2% 含糖牛肉琼脂平板与牛奶培养基,观察菌落形态、染色镜检,分析其产酸特性,并使用 VITEK 全自动微生物鉴定仪进行鉴定。
1.2.2 16S rRNA 基因序列分析 取菌株过夜培养物,按照 DNA 提取试剂盒说明书进行细菌全基因组 DNA 提取,并使用通用引物 27F(5' AGAG TTTGATCMTGGCTCAG 3')和 1492R(5' GGYTA CCTTGTTACGACTT 3')进行扩增和测序,并将测序拼接结果提交 NCBI 数据库中进行 BLAST 比对分析。应用 DNASTAR 软件将其与 NCBI 数据库中已发表肠球菌的 16S rRNA 序列进行比较分析。序列比较和聚类分析所引用的菌株及其在NCBI 数据库中的序列号如下:(LT223664)、(AJ301832)、(AJ301827)、(NR041708)、(MF369829)、(MN055952)、(NR115765)、(NR115764)、(AJ420802)、(MN055932)、(AB362590)、(LC097068)、(AJ301836)、(DQ411809)、(AB932524)、(LC097072)和(AF061005)。
1.2.3 全基因组测序与生物信息学分析 将提取的细菌全基因组 DNA 送上海人类基因组研究中心进行测序。使用美国 Illumina 公司的第二代solexa 测序技术进行全基因组测序,即取 1 μg DNA 利用超声仪打断,切胶回收 300 bp 片段,随后纯化的 DNA 利用 TruSeqTMDNA Sample Prep Kit-Set A 制备文库,并利用 TruSeq PE Cluster Kit 进行扩增,最后在illumine 机器(X10)上进行测序反应。应用 velvet V1.2.03 对数据进行拼装,结合软件 glimmer 3.02 进行基因预测;搜寻 NCBI 的 nr 库,KEGG 蛋白数据库以及 SEED 蛋白数据库进行基因功能注释,利用 CDD 数据库进行直系同源簇(COG)分类,通过 KEGG 数据库构建代谢通路;并参考文献[6]应用 PathogenFinder 软件对 CMCC32901 基因组序列进行细菌的致病性预测分析。
2 结果
2.1 屎肠球菌生产用菌种的形态和生化特性
屎肠球菌 CMCC32901 在含糖牛肉琼脂平板上生长良好,菌落表面光滑,中间隆起,边缘整齐。革兰氏染色为阳性球菌,接种牛奶培养基过夜后,明显引起牛奶凝固。VITEK 全自动微生物鉴定仪进行生化鉴定为屎肠球菌。
2.2 16S rRNA 基因序列分析
将 CMCC32901 菌株 16S rRNA 基因测序拼接结果在 NCBI 网站进行 Blast(https://blast.ncbi. nlm.nih.gov/Blast.cgi)分析,发现其与 NCBI 数据库中收录的不同屎肠球菌菌株的 16S rRNA 序列相似度大于 99.5%,而与其他肠球菌菌株之间的 16S rRNA 序列相似度均大于 92.0%。将其与NCBI 收录其他 16 种肠球菌属菌株进行系统发育进化分析,结果显示 CMCC32901 与另一株屎肠球菌以及菌株共属于一个亚分支,CMCC32901 与菌株 16S rRNA 基因序列同源性高达 99.4%,与粪肠球菌的进化稍有不同(图 1)。

图 1 生产用菌株CMCC32901 与NCBI 数据库中不同肠球菌属菌株的16S rRNA 基因序列系统进化分析
Figure 1 Phylogenetic analysis for 16S rRNA gene of production strain CMCC32901 and differentspecies strains from NCBI
2.3 全基因组序列测定与分析
应用 Solexa 测序技术进行全基因组测序分析,共获得 4606666 个读长片段,测序深度达到待测细菌基因组大小的 500倍。采用 velvet V1.2.03 软件对测序数据进行拼装,最终获得40 个基因组 contig 片段,其中最大 contig 片段为 461121 bp,平均每个 contig 片段大小为69 064 bp,N50 大小为 180 554 bp。经拼接比对分析发现屎肠球菌 CMCC32901 染色体全基因组序列全长为2394726 bp(表 1),同时通过软件 glimmer 3.02 进行基因预测,最终在 CMCC32901 菌株中鉴定出 2718 个基因,平均长度为 881 bp,编码基因占整个基因组序列的 86.7%,其平均 GC 含量为 39.1%。基因组中非编码基因间区域大小为
367 870 bp,在基因组序列中占比为 13.3%,在非编码 RNA 中鉴定出40 个 tRNA 与3 个 rRNA 序列。
2.4 全基因组序列基因功能分析
应用 COG 数据库注释上述已获得的全基因组序列,发现屎肠球菌 CMCC32901 菌株中共有 2064 个基因具有明确的生物学功能,COG 功能注释的预测编码蛋白有 2059 个,其中与氨基酸运输、碳水化合物代谢相关的蛋白最多,数量高达 527 个,占总注释基因的25.6%,主要为氨基酸、碳水化合物、无机离子的转运与代谢(图 2A、B 和 M),其次为负责转录的蛋白,为 184 个。此外,仍有 12.1%(250 个)的未知功能蛋白无法获得功能注释(图 2J),有待后续进一步的分析。

表 1 屎肠球菌CMCC32901 菌株的全基因组特征
通过 KEGG 数据库构建代谢通路,分析表明屎肠球菌 CMCC32901 菌株中 1402 个基因参与 KEGG 代谢通路的信息。KEGG 功能分类主要包括对细胞内过程、环境信息处理过程、遗传信息处理过程、代谢和生物系统(图 3)。其中代谢类基因数量最多,占 KEGG注释总基因数的 51.7%;其次为环境信息处理过程基因数量,具体包括膜转运和信号转导通路,占比分别为 11.8% 和 4.6%。

图 2 屎肠球菌CMCC32901 菌株基因组的COG 聚类分析
Figure 2 COG clustering analysis of the genome ofCMCC32901
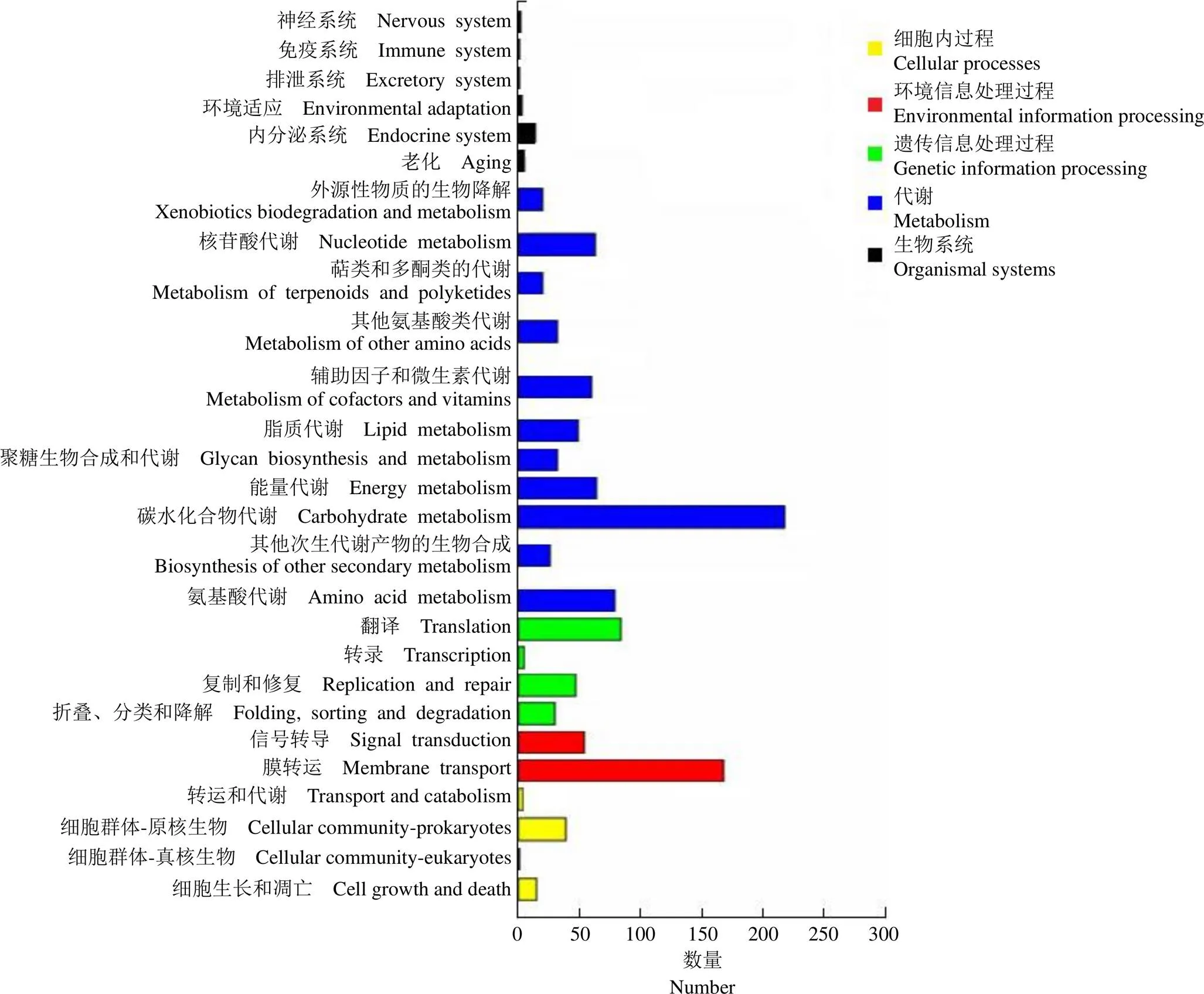
图 3 屎肠球菌CMCC32901 菌株基因组的KEGG 代谢通路分类
Figure 3 Classification of KEGG metabolic pathways in the genome ofCMCC32901
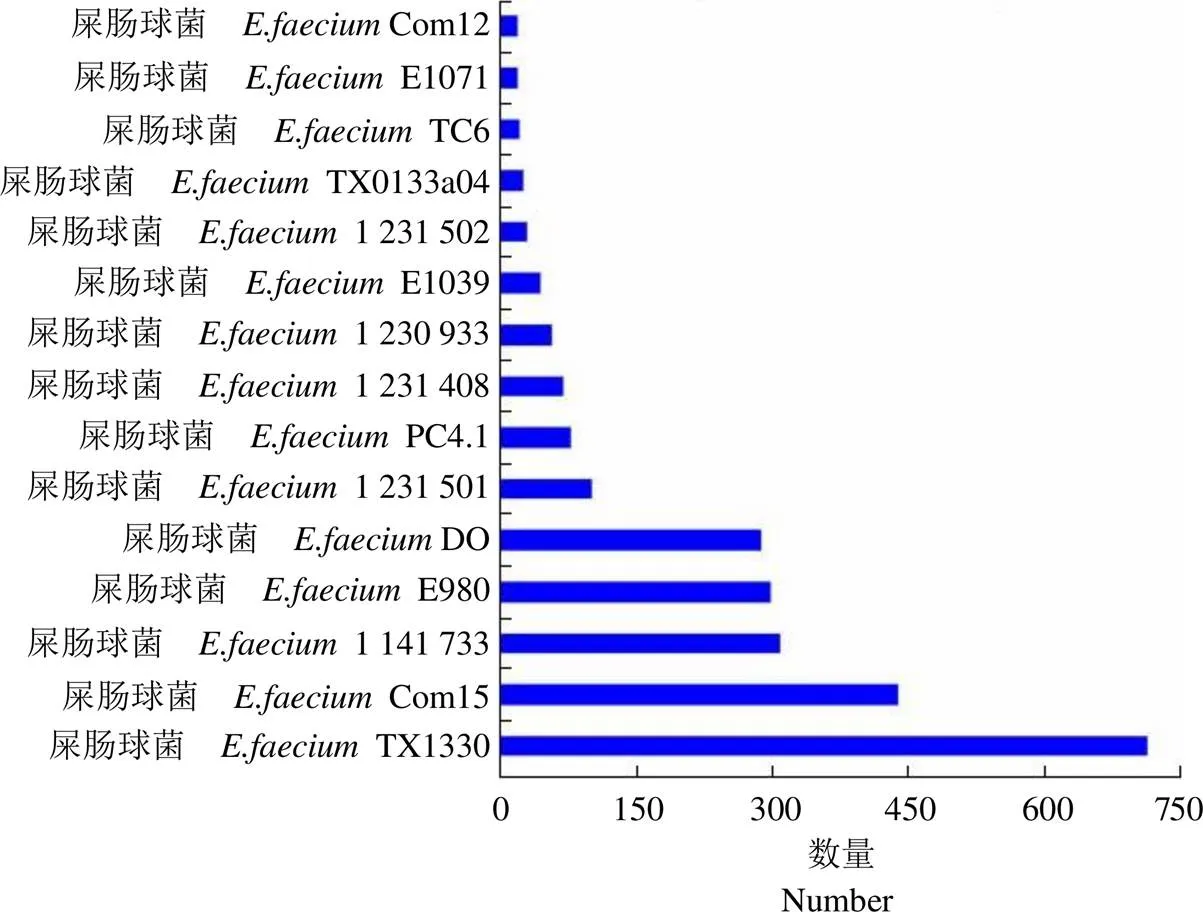
图 4 生产用菌种CMCC32901 菌株与其他不同屎肠球菌菌株的同源蛋白分析
Figure 4 Homologous protein analysis of production strain CMCC32901 and other differentstrains

表 2 屎肠球菌CMCC32901 菌株致病性预测分析
同时,将屎肠球菌 CMCC32901 菌株基因组的全部基因预测蛋白进行 blast 同源蛋白分析匹配,发现同源蛋白来源于 73 个物种,但其中以屎肠球菌菌属特异性同源蛋白最高,为 2589 个,进一步分析发现 CMCC32901 菌株与屎肠球菌 TX1330 菌株比例同源性蛋白比率最高(达 27.5%),为 713 个(图 4)。
2.5 细菌的致病性预测分析
应用 PathogenFinder 软件进行细菌的致病性预测分析,在 CMCC32901菌株中发现 5 种对人致病性相关蛋白,获得 5 种匹配蛋白详细的种属来源以及蛋白相似度(表 2),并提示屎肠球菌 CMCC32901 具有潜在的对人致病性。
3 讨论
乳酶生由于疗效可靠、副作用小,使用方便,在临床上广泛用于调理胃肠道不适。但该药品长期以来都作为生化药品管理,在其生产和质量控制上也一直按照化学药品的要求规定进行[7]。但理论上,乳酶生作为活菌制剂,按照其产品特性应属于微生态活菌制品[5]。为了加强乳酶生质量控制,在 2015 版《中国药典》二部首次收录乳酶生,并在制法要求中,明确规定生产过程不仅要遵守《药品生产质量管理规范》,而且还应符合现行版《中国药典》三部中微生态活菌制品总论的要求。因此,对于乳酶生的质量控制,尤其生产用菌种的要求越来越高[3-4]。
随着高通量测序技术的发展,已经广泛用在生产用菌种的全基因组水平研究及揭示其基因特征和功能,更为后续研究提供基础[8]。本研究应用高通量测序技术首次对我国乳酶生的生产用菌种屎肠球菌 CMCC32901 进行分析,获得该菌种的全基因组特征,并通过该菌基因组 COG 聚类分析发现,氨基酸、碳水化合物、无机离子的转运与代谢等都是高度富集的,推测可能与屎肠球菌能大量分解糖类产酸的特性相关。分析菌株基因组 KEGG 通路注释也证实,其中与新陈代谢途径相关的基因最多,进一步支持 COG 数据库注释的分析结果。这些研究结果也为优化乳酶生的生产工艺,提高其产量奠定基础。
众所周知,要成为一株生产用菌种,前期需要大量的有效性和安全性的验证试验[2],屎肠球菌 CMCC32901 自 20 世纪 50 年代起一直用于国内乳酶生的生产,中间也历经多次命名的变化,生产用菌种的质量控制能达到菌株水平就显得尤为重要[5]。由于传统的质控方法只能鉴定到屎肠球菌,而并不能提供每株屎肠球菌的特定信息。正如以前研究发现,16S rRNA 基因序列分析也仅能鉴定屎肠球菌[5],本研究结果也发现不同屎肠球菌菌株之间16S rRNA 基因序列几乎完全相同,与其他肠球菌之间的同源性较高,系统发育进化分析也显示各自的遗传进化关系。在此基础之上,我们获得该生产用菌种的全基因组信息,通过同源性分析发现,绝对多数基因组中预测编码蛋白仅与 NCBI 数据库中收录的屎肠球菌蛋白高度同源,并首次从全基因组水平进一步证实乳酶生 CMCC32901 生产用菌种为屎肠球菌,获得的屎肠球菌特异性同源蛋白为未来实现生产用菌种的菌株水平鉴定与质量控制提供数据支持。同时,从全基因组水平进行生产用菌种基因特征分析,也为其他微生态类药品生产用菌种的研究提供范例。
多数肠球菌常栖居在宿主的肠道和女性生殖道,属于人类的正常菌群之一。但是近年来,涉及致病肠球菌的院内感染事件不断增多,其安全性问题引起了人们的关注。这些临床分离的致病菌株大多数属于粪肠球菌和屎肠球菌,主要引起尿路感染、腹腔感染、菌血症和心内膜炎等[9-10]。安全性是微生态活菌制品的最基本要求,在现行版《中国药典》三部中微生态活菌制品总论中明确规定应通过动物试验检测其生产用菌种是否存在不安全因素,以保证人体用药安全[4]。尽管本研究所用屎肠球菌 CMCC32901 为批准上市多年的乳酶生生产用菌种,安全性已在临床上得到证实[1, 5],但随着生物技术的发展,人们对微生态制剂有了更加全面的了解,为进一步加强其生产用菌株的质量控制,FAO/WHO 工作组专家推荐应结合体内和体外试验,全面评估生产用菌株的安全性,包括菌株的耐药谱分析、特定代谢产物与已知致病因子的检测等[11]。本研究通过分析屎肠球菌CMCC32901 的全基因组序列,全面了解了该生产用菌种的编码基因与代谢通路特征,并预测发现其基因组中存在5 种潜在致病相关蛋白,但这些预测蛋白是否能表达,表达后是否会产生致病性等均有待于研究,这些研究结果将推动微生态制剂生产用菌种安全性评价体外试验的研究。
综上所述,本研究首次获得了我国乳酶生生产用菌种——屎肠球菌 CMCC32901 全基因组的基本特征,并以此分析了基因功能、同源性及致病性,不仅为后续鉴定生产用菌种到菌株水平的质量控制提供数据支持,也为未来乳酶生的生产工艺优化奠定基础。
志谢 感谢上海人类基因组研究中心郑华军、朱永强和金磊给予的帮助。
[1] Zhao L, Zhu YJ, Zhao ZJ. Clinical effect of domperidone combined with lactase on functional dyspepsia in children. Clin Med, 2019, 39(11):95-97. (in Chinese)
赵亮, 朱永杰, 赵振江. 多潘立酮联合乳酶生治疗小儿功能性消化不良的临床效果. 临床医学, 2019, 39(11):95-97.
[2] Wang JZ. Vaccine quality control and evaluation. Beijing: People's Medical Publishing House, 2013:79-88. (in Chinese)
王军志. 疫苗的质量控制与评价. 北京: 人民卫生出版社, 2013: 79-88.
[3] Chinese Pharmacopoeia Commission. Pharmacopoeia of the People’s Republic of China. Volume 2, 2020. Beijing: China Medical Science Press, 2020:830-831. (in Chinese)
国家药典委员会. 中华人民共和国药典. 2020年版二部. 北京: 中国医药科技出版社, 2020:830-831.
[4] Chinese Pharmacopoeia Commission. Pharmacopoeia of the People’s Republic of China. Volume 3, 2020. Beijing: China Medical Science Press, 2020:62-68. (in Chinese)
国家药典委员会. 中华人民共和国药典. 2020年版三部. 北京: 中国医药科技出版社, 2020:62-68.
[5] Tian WH, Yu G, Zhao ZJ, et al. Identification and drug-resistance spectrum of a lactasin-producing strain. Chin J Biol, 2013, 26(9): 1285-1289. (in Chinese)
田万红, 喻钢, 赵志晶, 等. 乳酶生生产菌株的鉴定及耐药谱的分析. 中国生物制品学杂志, 2013, 26(9):1285-1289.
[6] Cosentino S, Voldby Larsen M, Møller Aarestrup F, et al. PathogenFinder--distinguishing friend from foe using bacterial whole genome sequence data. PLoS One, 2013, 8(10):e77302.
[7] Zhu B, Liu T, Wei T, et al. Revision on the quality standards of lactasin and its preparations. Drug Stand China, 2011, 12(2):167-168. (in Chinese)
朱斌, 刘涛, 韦涛, 等. 乳酶生及制剂质量标准存在问题及修订建议. 中国药品标准, 2011, 12(2):167-168.
[8] Borgers K, Ou JY, Zheng PX, et al. Reference genome and comparative genome analysis for the WHO reference strain for Mycobacterium bovis BCG Danish, the present tuberculosis vaccine. BMC Genomics, 2019, 20(1):561.
[9] Arias CA, Murray BE. The rise of the Enterococcus: beyond vancomycin resistance. Nat Rev Microbiol, 2012, 10(4):266-278.
[10] Wang S, Guo Y, Lv J, et al. Characteristic of Enterococcus faecium clinical isolates with quinupristin/dalfopristin resistance in China. BMC Microbiol, 2016, 16(1):246.
[11] Food and Agriculture Organization of the United Nations, World Health Organization. Guidelines for the evaluation of the probiotics in food. London Ontario, Canada: 2002 [2020-08-11]. http://www.who. int/foodsafety/fs_management/en/probiotic_guidelines.pdf.
Whole genome sequence analysis for lactasin production strain
WANG Shan-shan, SHI Ji-chun, DU Zong-li, SHI Gang, LONG Xin-xing, XU Ying-hua, YE Qiang
To understand the molecular characteristics of the whole genome of production strain for lactasin in China.
After culturingCMCC32901 for the production of lactasin, the morphological observation, biochemical identification and 16S rRNA gene sequence analysis were performed. The high-throughput sequencing technology was used to determine the whole genome sequence of production strain CMCC32901. The basic characteristics, gene function, homology and pathogenicity of the genome were analyzed.
The 16S rRNA gene sequence of CMCC32901 had greater than 99.5% similarity with differentstrains included in the NCBI database, and greater than 92.0% similarity for otherstrains. The whole genome sequence ofCMCC32901 chromosome was 2 394 726 bp with an average GC content of 39.1%, and contained a total of 2718 genes including 2059 COG functions and 1402 KEGG metabolic pathways. Homology analysis showed that 2589 predicted coding proteins were specific for. Predictive analysis for the bacteria pathogenicity showed that five proteins related to human pathogenicity were found in the genome of CMCC32901 strain.
It is the first time for obtaining the basic characteristics of the whole genome of the lactasin production strainCMCC32901 in China, which lays the foundation for the subsequent optimization of the production process of lactasin production and the quality control of the production strains.
Lactasin;; Production strain; Whole genome; Quality control
s: YE Qiang, Email: qiangyee@nifdc.org.cn; XU Ying-hua, Email: xuyh@nifdc.org.cn
10.3969/j.issn.1673-713X.2020.06.004
Author Affiliation: Key Laboratory of the Ministry of Health for Research on Quality and Standardization of Biotech Products, National Institute for Food and Drug Control, Beijing 102629, China
国家重点研发计划(2018YFC1603900);医学微生物资源子平台运行与服务(NIMR-2019)
叶强,Email:qiangyee@nifdc.org.cn;徐颖华,Email:xuyh@nifdc.org.cn
2020-07-13
*同为第一作者
