里氏木霉与黏绿木霉中非核糖体肽合成酶的生物信息学分析
2020-12-09祝友朋韩长志
祝友朋 韩长志

摘要:为了更好地解析里氏木霉和黏绿木霉在次生代谢产物关键基因方面的差异,以非核糖体肽合成酶(NRPS)为例,通过antiSMASH预测网站对上述2种木霉开展次生代谢产物基因找寻。结果表明,里氏木霉中含有87个NRPS蛋白,而黏绿木霉中含有214个NRPS蛋白,后者明显高于前者;同时,利用ProtComp v9.0、PHD等生物信息学预测网站对上述木霉中NRPS蛋白进行亚细胞定位、理化性质以及二级结构等特征分析,明确2种木霉中NRPS蛋白具有以下共同特征:理论等电点多集中在5.01至7.00之间,不稳定性系数多集中在30.01至50.00之间,高达75%以上的蛋白质属于亲水性蛋白质,而二级结构组成特征则以α螺旋为主,转运肽情况尚未明确,保守结构域较多,亚细胞定位多集中在线粒体等。
关键词:里氏木霉;黏绿木霉;非核糖体肽合成酶(NRPS);理化性质;生物信息学分析
中图分类号:S435.1文献标识码:A文章编号:1000-4440(2020)05-1119-07
Abstract:In order to better analyze the differences of the key genes of secondary metabolites between Trichoderma reesei and Trichoderma virens, the secondary metabolite genes of T. reesei and T. virens were searched through prediction website of antiSMASH, taking non-ribosomal peptide synthetase (NRPS) as example. The results showed that there were 87 NRPS proteins in T. reesei and 214 NRPS proteins in T. virens, the number of NPRS proteins in T. virens was significantly higher than that in T. reesei. Meanwhile, ProtComp v9.0, PHD and other websites for bioinformatics prediction were used to analyze the characteristics of NRPS proteins in T. reesei and T. virens, including subcellular localization, physicochemical properties and secondary structure, etc. The following common characteristics of NRPS proteins in T. reesei and T. virens are clear: the theoretical isoelectric points are mostly concentrated between 5.01 and 7.00, the instability indices are mostly concentrated between 30.01 and 50.00, more than 75% proteins are hydrophilic, while the compositional characteristics of secondary structures are dominated by α-helix. Besides, the information of transit peptides is not clear, there are many conserved domains and the subcellular localizations are mostly concentrated in mitochondria and so on.
Key words:Trichoderma reesei;Trichoderma virens;non-ribosomal peptide synthetase(NRPS);physical and chemical properties;bioinformatics analysis
里氏木霉(Trichoderma reesei)与黏绿木霉(Trichoderma virens)均属于木霉属真菌中的重要种类,分别在工业、农业生产方面发挥着重要作用[1-2]。里氏木霉QM6a作为重要的工业菌株,在实现植物组织降解及利用方面发挥着重要作用,黏绿木霉GV29-8作为木霉属中重要的生防菌株之一,其在抑制植物病原菌生长发育方面具有重要的作用。2003年,国外学者首次通过转录组学研究对里氏木霉野生型QM6a开展了全局性分析[3],之后里氏木霉QM6a以及黏绿木霉GV29-8的全基因组序列得以释放[2,4],为开展深入分析提供了重要的数据支撑。前人研究发现黏绿木霉对立枯丝核菌(Rhizoctonia solani)[5]、终极腐霉(Pythium ultimum)[6]等植物病原菌均有較好的抑制作用。针对里氏木霉的突变菌株开展了大量研究工作[4,7-8],目前,里氏木霉己经成为世界上研究和应用最广泛的纤维素酶工业生产菌种,世界各国科学家最初采用物理化学诱变的方法进行了大量的菌株改良工作,研发出数十种的工业菌株[9]。
非核糖体肽合成酶(NRPS)作为真菌合成次生代谢物的关键酶,是由多个催化结构域组成的高度模块化的肽合成酶,包括腺苷酰化结构域(A结构域)、巯基化结构域(T结构域)和缩合结构域(C结构域)等[10]。在非核糖体肽(Non-ribosomal peptides, NRP)合成过程中,首先A结构域筛选和激活底物氨基酸,其次底物氨基酸被转运并连接到T结构域,然后经过C结构域催化形成肽键,最后经过多次循环延伸形成多肽链,在硫酯酶结构域(TE结构域)催化下通过水解的方式从NRPS中释放出来。NRP种类繁多、结构复杂,在真菌生长发育、信号传导及致病等过程中都发挥着重要的作用[11]。前人研究发现,由苹果斑点落叶病病菌(Altenaria alternata)产生的AM-毒素[12]、玉米圆斑病病菌(Cochliobolus carbonum)产生的HC-毒素[13],以及玉米小斑病病菌(Cochliobolus heterostrophus)[14]、水稻胡麻斑病病菌(Cochliobolus miyabeanus)[15]、拟南芥黑斑病病菌(Alternaria brassicicola)[15]、柑橘褐斑病病菌(Alternaria alternata)[16]和禾谷镰刀菌(Fusarium graminearum)中的NPS6基因、苹果树腐烂病病菌(Valsa mali)的NPS12基因[11]均与上述病原真菌的致病性有关[17];同时,前人对于硬枝树花(Ramalina conduplicans)地衣型真菌中的NRPS基因开展了克隆和鉴定工作[18]。然而,学术界尚未见有关里氏木霉和黏绿木霉中NRPS的数量和理化性质的报道,也未见有关具有不同功能的木霉中NRPS与其功能关系的研究报道。
本研究通过SMURF网站预测里氏木霉和黏绿木霉的次生代谢基因分布情况,获得编码聚酮化合物合酶(PKS)、非核糖体肽合成酶以及萜烯合酶(TPS)等基因簇情况,进一步利用antiSMASH对其开展基因组数据分析,鉴定和注释潜在的编码PKS、NRPS以及TPS的基因,从而得到上述木霉中NRPS蛋白,同时,通过ProtComp v9.0、expasy、PHD等软件对上述NPRS蛋白进行生物信息学分析,明确木霉中NRPS的亚细胞定位、理化性质以及二级结构等特征,为今后深入研究里氏木霉和黏绿木霉NRPS的功能及其作用机制提供理论依据。
1材料与方法
1.1木霉中NRPS蛋白找寻
首先,通过SMURF(Secondary Metabolite Unknown Regions Finder)[19](网址为http://smurf.jcvi.org/upload.php)预测里氏木霉QM6a(全基因组序列来源于http://genome.jgi-psf.org/Trire2/Trire2.home.html)和黏绿木霉GV29-8(全基因组序列来源于http://genome.jgi.doe.gov/TriviGv29_8_2/TriviGv29_8_2.home.html)的次生代谢基因分布情况,获得编码PKS、NRPS、TPS等基因簇情况,然后利用antiSMASH(antibiotics & Secondary Metabolite Analysis SHell)[20](网址为https://fungismash.secondarymetabolites.org/#!/start)对上述2种木霉进行基因组数据分析,鉴定和注释潜在的PKS、NRPS、TPS等基因。
1.2NRPS蛋白理化性质分析
运用蛋白质数据库(网址为http://www.expasy.ch/tools/protparam.html)分析NRPS蛋白的理化性质,包括理论等电点、不稳定性系数、总平均亲水性等,采用Protscale(网址为http://web.expasy.org/protscale/)分析NRPS蛋白的疏水性[21]。
1.3NRPS蛋白二级结构分析
采用PHD(网址为http://www.sbg.bio.ic.ac.uk/phyre2/html/page.cgi?id=index)对2种木霉中的NRPS蛋白开展二级结构预测及分析[22]。
1.4NRPS蛋白转运肽分析
采用TargetP 1.1 Server(网址为http://www.cbs.dtu.dk/services/TargetP-1.1/index.php)分析NRPS蛋白的转运肽[23]。
1.5NRPS蛋白保守结构域分析
采用SMART(网址为http://smart.embl-heidelberg.de/)分析NRPS蛋白的保守结构域[24]。
1.6NRPS蛋白亚细胞定位分析
采用ProtComp v9.0(网址为http://phobius.sbc.su.se/)分析NRPS蛋白的亚细胞定位情况,并对分析数据进行整理归纳。
2结果与分析
2.1黏绿木霉与里氏木霉NRPS蛋白数量差异
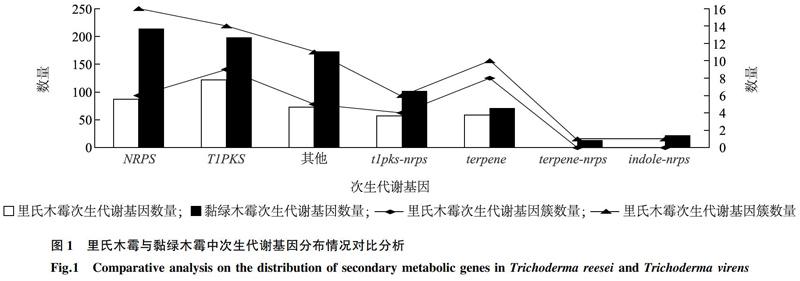
通过SMURF及antiSMASH预测和基因注释,发现里氏木霉QM6a中含有32个次生代谢基因簇,基因簇的大小为6~21个基因,共涉及398个基因;而黏绿木霉GV29-8中则含有59个次生代谢基因簇,基因簇的大小为4~35个基因,共涉及793个基因。里氏木霉含有87个NRPS蛋白,黏绿木霉含有214个NRPS蛋白(图1)。此外,里氏木霉和黏绿木霉中数量较多的次生代谢合成基因都为T1PKS和NRPS,但黏绿木霉含有terpene-nrps、indole-nrps。
2.2NRPS蛋白的理论等电点
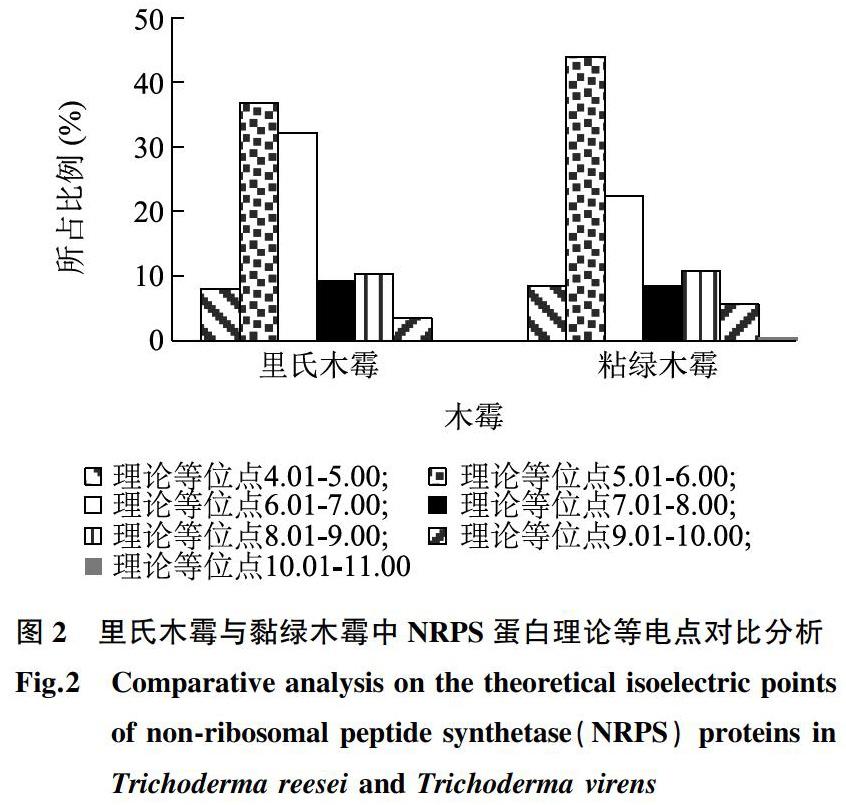
对2种木霉中全部NRPS蛋白的理论等电点进行对比分析,结果表明,数值范围为4.25~10.43,其中,里氏木霉中蛋白ID为XP_006968101.1的NPRS蛋白的理论等电点为4.25,黏绿木霉中蛋白ID为XP_013954358.1的NPRS蛋白的理论等电点为10.43。进一步以理论等电點间隔单位为1.00进行整理分析,发现里氏木霉与黏绿木霉中NRPS蛋白理论等电点多集中在5.01~7.00,其数量分别为60、142个,所占比例分别为68.97%、66.36%(图2)。同时,对属于酸性蛋白质(理论等电点小于6.01)的NRPS蛋白进行分析,里氏木霉与黏绿木霉中NRPS蛋白数量分别为39、112个,所占比例分别为44.83%、52.34%;对属于中性蛋白质(理论等电点位于6.01~8.00)的NRPS蛋白进行分析,里氏木霉与黏绿木霉中中性NRPS蛋白数量分别为36、66个,所占比例分别为41.38%、30.84%;对属于碱性蛋白(理论等电点大于8.00)的NRPS蛋白进行分析,里氏木霉与黏绿木霉中碱性NRPS蛋白数量分别为12、36个,所占比例分别为13.79%、16.82%(图2)。由此可见,两种木霉中NRPS蛋白多属于酸性蛋白质。
2.3NRPS蛋白的不稳定性系数
对2种木霉中全部NRPS蛋白的不稳定性系数进行统计分析,结果表明,里氏木霉与黏绿木霉中NRPS蛋白的不稳定性系数集中在30.01~50.00,其数量分别为65、169个,所占比例分别为74.71%、78.97%。同时,对NRPS蛋白中不稳定性系数小于40.01(稳定蛋白质)的分泌蛋白质进行分析,里氏木霉与黏绿木霉中稳定NRPS蛋白数量分别为35、93个,所占比例分别为40.23%、43.46%;其次对不稳定性系数大于40.01(不稳定蛋白质)的分泌蛋白质进行分析,里氏木霉与黏绿木霉中不稳定NRPS蛋白数量分别为52、121个,所占比例分别为59.77%、56.54%(图3)。上述结果表明,两种木霉中的NRPS蛋白多属于不稳定性蛋白质。
2.4NRPS蛋白的亲水性
进一步对2种木霉中的全部NRPS蛋白进行亲水性分析,结果表明,NRPS蛋白多属于亲水性蛋白质(亲水性值小于0),里氏木霉与黏绿木霉中亲水性NRPS蛋白数量分别为69、166个,所占比例分别为79.31%、77.57%;同时,对疏水性蛋白质(亲水性值大于0)进行统计,里氏木霉与黏绿木霉中疏水性NRPS蛋白数量分别为18、48个,所占比例分别为20.69%、22.43%。上述结果表明,里氏木霉和黏绿木霉中NRPS蛋白多属于亲水性蛋白质。
此外,进一步对全部NRPS蛋白亲水性最强氨基酸残基、疏水性最强氨基酸残基种类进行统计分析,结果显示里氏木霉中NRPS蛋白亲水性最强氨基酸残基有18种,缺少C(半胱氨酸)、M(甲硫氨酸),而黏绿木霉中NRPS蛋白亲水性最强氨基酸残基有19种,缺少C,存在较多的亲水性最强氨基酸残基都为E(谷氨酸)、D(天冬氨酸)、R(精氨酸)、K(赖氨酸)、N(天冬酰胺);里氏木霉中NRPS蛋白疏水性最强氨基酸残基有18种,缺少E、Y(酪氨酸),而黏绿木霉中NRPS蛋白疏水性最强氨基酸残基有20种,存在较多的疏水性最强氨基酸残基都为A(丙氨酸)、L(亮氨酸)、V(缬氨酸)、G(甘氨酸)、I(异亮氨酸)(图4)。
2.5NRPS蛋白的二级结构组成特征
对里氏木霉与黏绿木霉中NRPS蛋白进行二级结构预测,结果表明,2种木霉中NRPS蛋白均含有无规则卷曲、α螺旋、β折叠、跨膜螺旋(TM)等二级结构,其中,里氏木霉NRPS蛋白中上述二级结构所占比例分别是22.59%、44.36%、12.55%、9.34%,而黏绿木霉NRPS蛋白中所占比例分别21.74%、44.16%、13.22%、9.45%(图5)。
2.6里氏木霉和黏绿木霉NRPS蛋白的转运肽预测
对里氏木霉和黏绿木霉中NRPS蛋白的转运肽进行预测分析,以明确2种木霉中NRPS蛋白的定位情况和预测可靠性。在定位情况中,里氏木霉NRPS蛋白定位在分泌通路、线粒体及其他位置的数量分别为11、8、67个,尚有1个NPRS蛋白未能实现预测;而黏绿木霉NRPS蛋白定位在分泌通路、线粒体及其他位置的数量分别为35、19、152个,尚有8个NPRS蛋白未能实现预测(图6)。
2.7NRPS蛋白中的保守结构域
里氏木霉NRPS蛋白中有79个具有保守结构域,占比为90.80%;黏绿木霉NRPS蛋白中有184个具有保守结构域,占比为85.98%(图7)。
2.8NRPS蛋白的亚细胞定位
里氏木霉和黏绿木霉中NRPS蛋白亚细胞定位主要集中在线粒体、细胞质膜和细胞质等,里氏木霉中在上述细胞器中所占比例分别是33.33%、19.54%、16.09%,黏绿木霉中在上述细胞器中所占比例分别是26.64%、21.96%、18.69%(图8)。
3讨论
前期对里氏木霉QM6a和黏绿木霉GV29-8中的分泌蛋白质及CAZymes蛋白开展了预测分析,研究结果表明,前者含有分泌蛋白的数量为356个,占全部蛋白质数量的3.89%,而CAZymes蛋白数量为201个,占全部蛋白质数量的2.20%[4, 25];黏綠木霉GV29-8中分泌蛋白质数量为377个,占全部蛋白质数量的3.03%,而CAZymes蛋白数量为185个,占全部蛋白质数量的1.49%[2,26]。由此可见,里氏木霉QM6a在分泌蛋白质及CAZymes蛋白占全部蛋白质的比例方面均高于黏绿木霉GV29-8,然而,前者中的NRPS数量为87个,占全部蛋白质数量的0.95%,而黏绿木霉GV29-8中NRPS数量高达214个,占全部蛋白质数量的1.72%,前者NRPS蛋白所占比例明显低于后者。基于上述研究结果,根据里氏木霉QM6a作为重要的纤维素降解工业菌株以及黏绿木霉GV29-8作为重要的生防菌株的判断,我们推断,分泌蛋白质及CAZymes蛋白在两种木霉中均对实现其破坏植物细胞壁组织方面发挥着重要作用,而NPRS则在实现其破坏植物纤维组织方面发挥着更加重要的作用,terpene-nrps和indole-nrps在破坏植物病原真菌的细胞壁组织方面发挥着重要作用。当然,是否存在着时间、空间方面的差异性,有待于进一步通过生物学试验进行验证。
同时,本研究通过次生代谢基因预测和注释还明确了里氏木霉QM6a中含有32个次生代谢基因簇,而黏绿木霉GV29-8中则有59个次生代谢基因簇。在这些次生代谢基因簇中不仅编码NPRS蛋白,还涉及T1PKS、terpene-nrps、indole-nrps等蛋白质。上述蛋白质在实现利用植物纤维方面的作用有待进一步明确,具有不同功能的木霉与次生代谢基因簇之间的关系也有待进一步明确。然而,尽管2种木霉在NPRS蛋白数量及次生代谢基因簇数量方面均存在着明显不同,但是其NPRS在理化性质、亚细胞定位、二级结构等方面存在着较多相同特征。基于此,我们推断,2种木霉中NPRS蛋白在功能方面具有同质性,而木霉功能的差异性发挥则表现在其数量上。
本研究通过SMURF及antiSMASH预测网站对里氏木霉QM6a和黏绿木霉GV29-8进行了次生代谢产物基因找寻及NPRS的生物信息学分析,结果表明,前者含有87个NRPS蛋白,而后者含有214个NRPS蛋白,后者所含有的NPRS蛋白数量明显高于前者;同时,明确2种木霉中NRPS蛋白具有以下共同特征:理论等电点多集中在5.01~7.00,不稳定性系数多集中在30.01~50.00,高达75%以上的蛋白质属于亲水性蛋白质,而二级结构组成特征则以α螺旋为主,转运肽情况尚未明确,保守结构域较多,亚细胞定位多集中在线粒体等。就理化性质而言,上述木霉中NRPS蛋白的理化性质以酸性、不稳定性和亲水性为主,亲水性最强氨基酸主要有E、D、R、K、N等,疏水性最强氨基酸主要有A、L、V、G、I等。同时,明确里氏木霉NRPS蛋白相比于黏绿木霉亲水性最强氨基酸中缺少M,而疏水性最强氨基酸中则缺少E、Y。研究结果为进一步开展里氏木霉QM6a和黏绿木霉GV29-8中次生代谢产物基因利用和改造,以及进一步开展不同木霉之间NPRS功能差异化研究提供了基础。
[18]原晓龙,赵能,陈剑,等. 硬枝树花中非核糖体多肽合成酶NRPS基因的克隆与鉴定[J]. 基因组学与应用生物学,2016, 35 (10):2834-2841.
[19]KHALDI N,SEIFUDDIN F T,TURNER G, et al. SMURF: Genomic mapping of fungal secondary metabolite clusters[J]. Fungal Genet Biol, 2010, 47 (9):736-741.
[20]BLIN K, WOLF T, CHEVRETTE M G, et al. antiSMASH 4.0-improvements in chemistry prediction and gene cluster boundary identification[J]. Nucleic Acids Res,2017, 45 (W1):36-41.
[21]GASTEIGER E,GATTIKER A, HOOGLAND C, et al. ExPASy: The proteomics server for in-depth protein knowledge and analysis[J]. Nucleic Acids Research,2003, 31 (13):3784-3788.
[22]LAWRENCE A K, STEFANS M, CHRISTOPHER M Y, et al. The Phyre2 web portal for protein modeling, prediction and analysis[J]. Nat Protocols,2015, 10 (6): 845-858.
[23]OLOF E, SREN B, GUNNAR V H, et al. Locating proteins in the cell using TargetP, SignalP and related tools[J]. Nature Protocols,2007, 2 (4):953-971.
[24]IVICA L, TOBIAS D, PEER B. SMART 7: recent updates to the protein domain annotation resource[J]. Nucleic Acids Research,2012, 40 (D1):302-305.
[25]韓长志,王娟. 基于全基因组序列预测里氏木霉QM6a的分泌蛋白[J]. 华中农业大学学报,2017, 36 (2):28-32.
[26]韩长志. 基于全基因组测序的粘绿木霉Gv29-8中分泌蛋白预测[J]. 河南师范大学学报(自然科学版), 2016, 44 (6):145-148.
(责任编辑:张震林)
