药用资源植物山莨菪的转录组信息分析
2020-06-08夏铭泽张发起
张 雨 夏铭泽 张发起
(1.中国科学院高原生物适应与进化重点实验室,中国科学院西北高原生物研究所,西宁 810001; 2.中国科学院大学,北京 100039)
山莨菪(Anisodustanguticus)隶属茄科(Solanaceae)山莨菪属(Anisodus),是多年生宿根草本植物,产于青海、甘肃、西藏东部、云南西北部等地区,海拔在2 800~4 200 m,生于草坡阳面。山莨菪是重要的资源植物,其根可供药用;莨菪烷类生物碱可以从山莨菪中提取,在医学上具有麻醉、镇痛和解毒等功效;其地上部分可以掺入牛饲料中,有催膘作用[1~2]。山莨菪具有重要的药用价值和经济价值,是一种重要的资源植物。但是由于山莨菪分布生境狭窄,种植量有限,需求量又较高,故被大量挖掘,其资源的可持续利用遭到了严重威胁,在我国《国家重点保护野生植物名录》中已被列为国家Ⅱ级保护植物。目前,对山莨菪的研究主要集中在对山莨菪碱的药理学、病理学和临床应用研究方面[3~4],部分研究集中在山莨菪的光合作用、生长特征、元素含量及传粉生物学方面[5~8],只有少数人对山莨菪进行分子生物学方面的研究,分子标记开发较为落后,且转录组和基因组信息相对缺乏[9~10]。
转录组是组织或细胞转录产物的总和,通过转录组分析,可以进一步对基因的结构和功能进行研究,便于了解基因组的组成和功能,深入探索生物体表达状况、发育的机理及调控网络机制等[11~13]。近年来,高通量测序技术的迅速发展降低了测序成本,有利于转录组分析的发展[14],目前已有多种资源植物完成了转录组测序,如李东[15]对热胁迫下丹参(Salviamiltiorrhiza)的次生代谢比较转录组进行分析,发现了热胁迫对丹参转录组、对迷迭香酸途径关键酶表达及萜类合成途径关键酶表达的影响,为解释热胁迫与有效成分合成与积累过程提供了依据;李依民等[16]通过高通量测序技术对黄三七(Soulieavaginata)的根茎转录组数据进行分析,获得了丰富的转录组数据,为黄三七代谢途径解析、调控机制研究及基因功能鉴定奠定了基础;李太强等[17]对长梗杜鹃(Rhododendroncalvescens)进行转录组分析,发现了176条和人类疾病相关的Unigenes,其中与内分泌及代谢疾病相关的Unigenes有167条,与抗药性有关的Unigenes有9条,为杜鹃属的抗性机理研究及相关病理学研究提供了重要参考;另外还有许多研究人员[18~20]对黑果枸杞(Lyciumruthenicum)、余干子(Phyllanthusemblica)、陆地棉(Gossypiumhirsutum)等资源植物进行了转录组分析,获得了大量数据信息。
鉴于此,本研究利用高通量测序技术,对山莨菪进行转录组测序分析,得到大量测序数据,结合生物信息学方法,拼接、组装和注释测序序列,对山莨菪的基因结构、表达水平和差异、基因富集进行分析,以期为山莨菪转录组和基因组水平的研究提供数据,同时也对山莨菪的保护及合理开发利用奠定分子生物学基础。
1 材料与方法
1.1 材料
本研究的植物材料山莨菪采于青海省同仁县(地理坐标N35°13′58.30″,E101°51′05.50″;海拔3 532 m),采集一年生幼叶,用纯水清洗去除污渍、75%酒精杀菌消毒后迅速置于液氮中处理,再转移到-80℃的超低温冰箱保存备用。凭证标本(标本号:Zhang2018050)存于中国科学院西北高原生物研究所青藏高原生物标本馆(HNWP)。
1.2 方法
1.2.1 山莨菪RNA提取与测序
提取山莨菪的RNA,在对其RNA进行纯化和质量分析之后,构建cDNA文库,采用Illumina HiseqTM测序平台进行测序。
1.2.2 序列的拼接与组装
通过FastQC进行数据评估和数据质控,通过Trimmomatic进行质量剪切,在质量剪切的过程中,需要去除以下情况的序列:①N碱基序列;②reads中的接头序列及Q值低于20的低质量碱基;③长度不高于35nt的reads及配对序列;④被污染的序列等。最终,得到相对准确的有效数据,使用Trinity将有效数据从头组装成转录本,大致过程如下:
①运行seqtk-trinity程序,将fq数据转换成fa数据,并生成both.fa;
②运行Jellyfish程序,生成K-mer目录;
③运行Inchworm程序,组装reads初步得到contigs;
④运行Chrysalis程序,将上一步生成的contigs进行聚类分析,对每个类构建Bruijn图;
⑤运行Butterfly程序,处理上一步得到的Bruijn图,根据图中reads和成对的reads寻找途径,得到最终的isoform序列;
⑥得到Trinity.fasta文件,其haeder信息包含了转录本名称、长度、De Brujin图重构途径等;
⑦对拼接组装得到的转录本去冗余。
在此基础上,统计所得转录本的各项信息,如GC含量、长度分布状况、所含isoform数目分布状况等,为后续分析做准备。
1.2.3 基因功能注释
通过Blast将获得的Unigenes与多个数据库进行比对,取evalue小于等于1e-10,相似度<90%且覆盖度<80%的比对结果,再对山莨菪进行基因功能的注释及分类分析。可进行对比参考的数据库有NT(核酸序列数据库)、NR(非冗余蛋白质序列数据库)、COG(原核生物基因直系同源关系注释系统)、KOG(真核生物基因直系同源关系注释系统)、Swiss~Prot(最常用且全面的蛋白质数据库)、TrEMBL(蛋白质数据库,Swiss~Prot的增补本)、PFAM(最全面蛋白结构域注释分类系统)、CDD(保守区域结构数据库)、GO(国际化基因功能分类体系)、KEGG(有关生物系统的较完善数据库)等。通过与NR、Swiss~Prot、TrEMBL数据库进行对比,可以对CDS序列进行预测;山莨菪的转录本序列与相近物种的近似情况,以及山莨菪同源序列的功能信息可以通过与NR库对比得到;对山莨菪的基因进行GO分类后,可以便于统计基因在生物过程、细胞成分、分子功能下的GO term;另外,对基因做KO注释之后,可以根据KO与pathway的联系对山莨菪进行KEGG代谢通路的分类分析[21~23]。
2 结果与分析
2.1 转录本组装与CDS预测分析
从头测序组装之后得到158 378个Transcripts,得到71 463个Unigenes,对编码序列进行预测之后得到47 685个CDS(见表1)。对Transcripts而言(图1A),分布在200~300 bp的数量是最多的,其次是300~400 bp(42 231条)、大于等于2 000 bp(19 215条)和300~400 bp(14 864条)。对Unigenes而言(图1B),按分布范围内数量排序的话,依次是200~300 bp(29 326条)、300~400 bp(11 003条)、400~500 bp(5 955条)、大于等于2 000 bp(4 146条);对CDS而言,长度在100~200 nt内的序列最多,有12 928条,占27.11%,其次为200~300 nt(11 259条)、300~400 nt(4 838条)、400~500 nt(2 980条)。无论是Transcripts、Unigenes还是CDS,长度最短的序列数量最多,而随着序列长度增加,所获得的拼接数量就越少(大于等于2 000 bp的序列除外)。Unigenes的平均长度为651.1 bp,最短的Unigene长度为201 bp,最长为8 526 bp,N50长度为1 115 bp,总长度为46 529 443 bp。
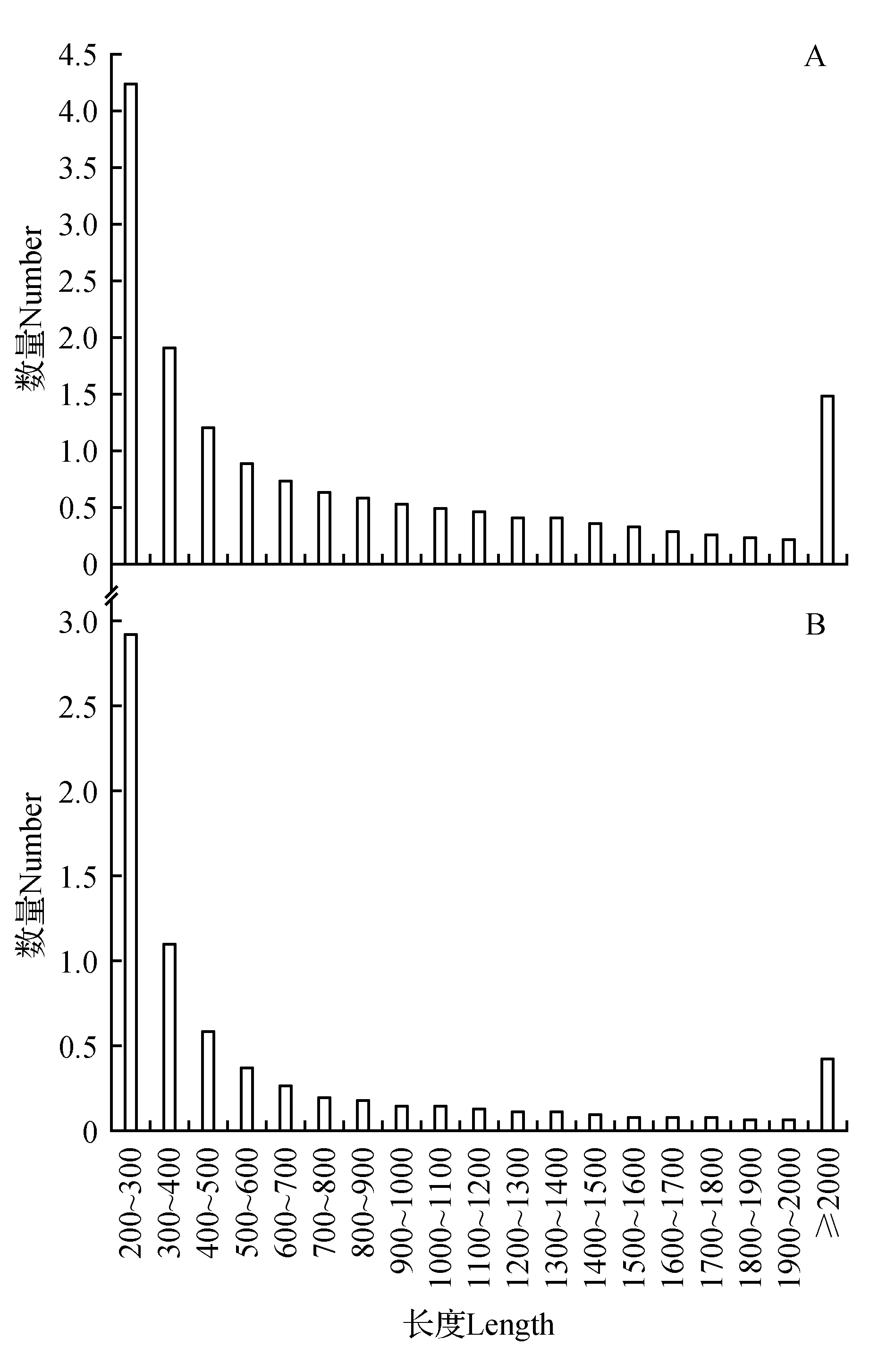
图1 山莨菪Transcript与Unigenes的长度分布图Fig.1 Distribution of transcript and unigene length for A.tanguticus

表1 拼接结果统计
2.2 功能注释及COG分类
以CDD、KOG、NR、NT、GO、Swissprot、PFAM、KEGG、TrEMBL这9个数据库作为参考,将de novo组装得到的71 463个基因序列与之进行比对和注释(见表2)。注释到CDD、KOG、NR、NT、PFAM、Swissprot、TrEMBL、GO、KEGG数据库种的Unigenes分别有19 526、13 110、39 621、38 769、14 393、24 163、39 653、29 309和3 679条,注释到CDD数据库种的Unigenes最多,占比27.32%,而注释到KEGG数据库的Unigenes最少,占5.15%。在9个数据库中至少1个数据库注释成功的Unigenes为47 624条,占总Unigenes数的66.64%;共有2 415个Unigenes与9个数据库都能匹配成功,占总Unigenes数的3.38%(见表2);另外,尚未注释成功的Unigenes数量较多,有23 839条,约占1/3。
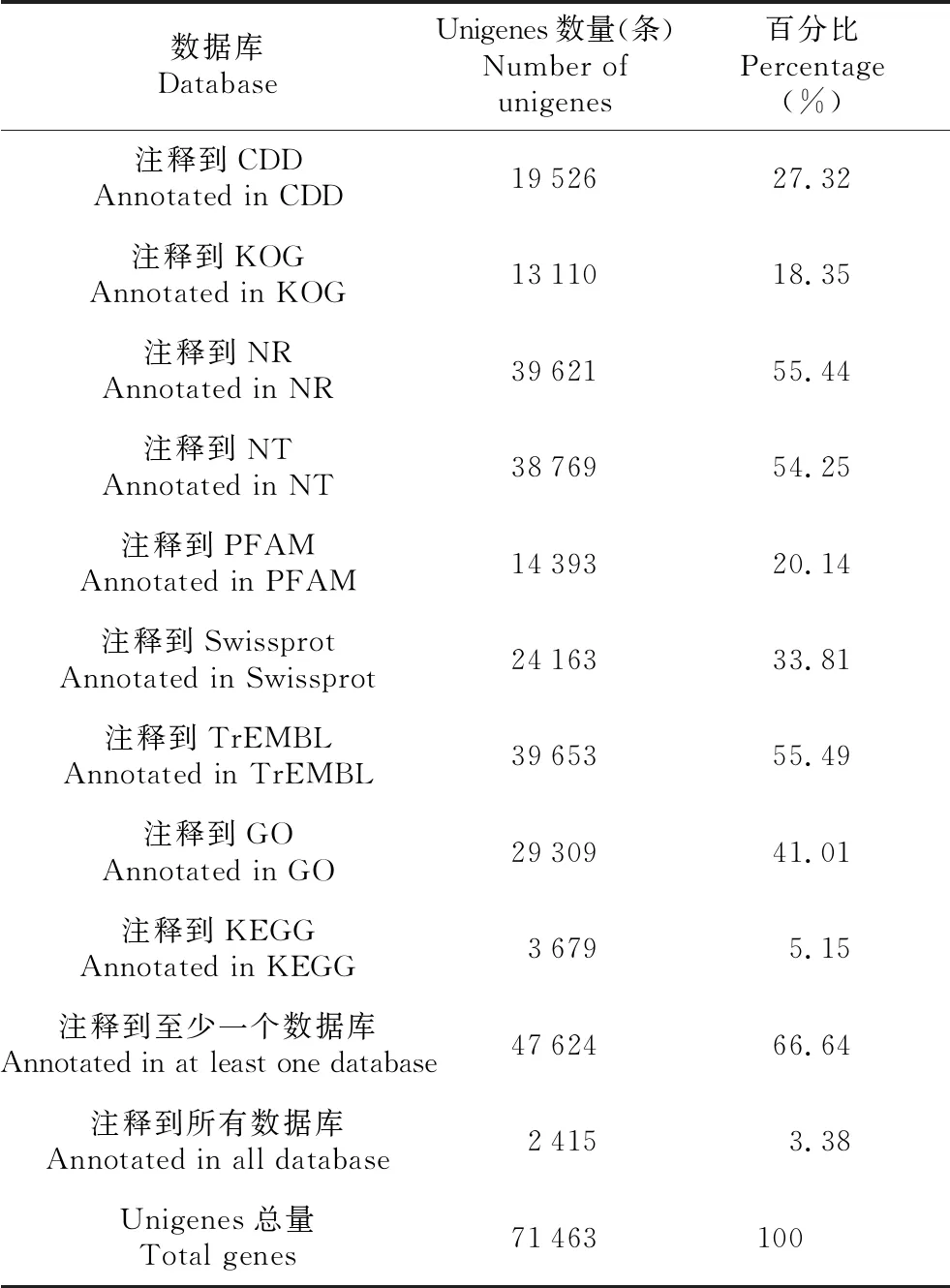
表2 Unigenes注释结果
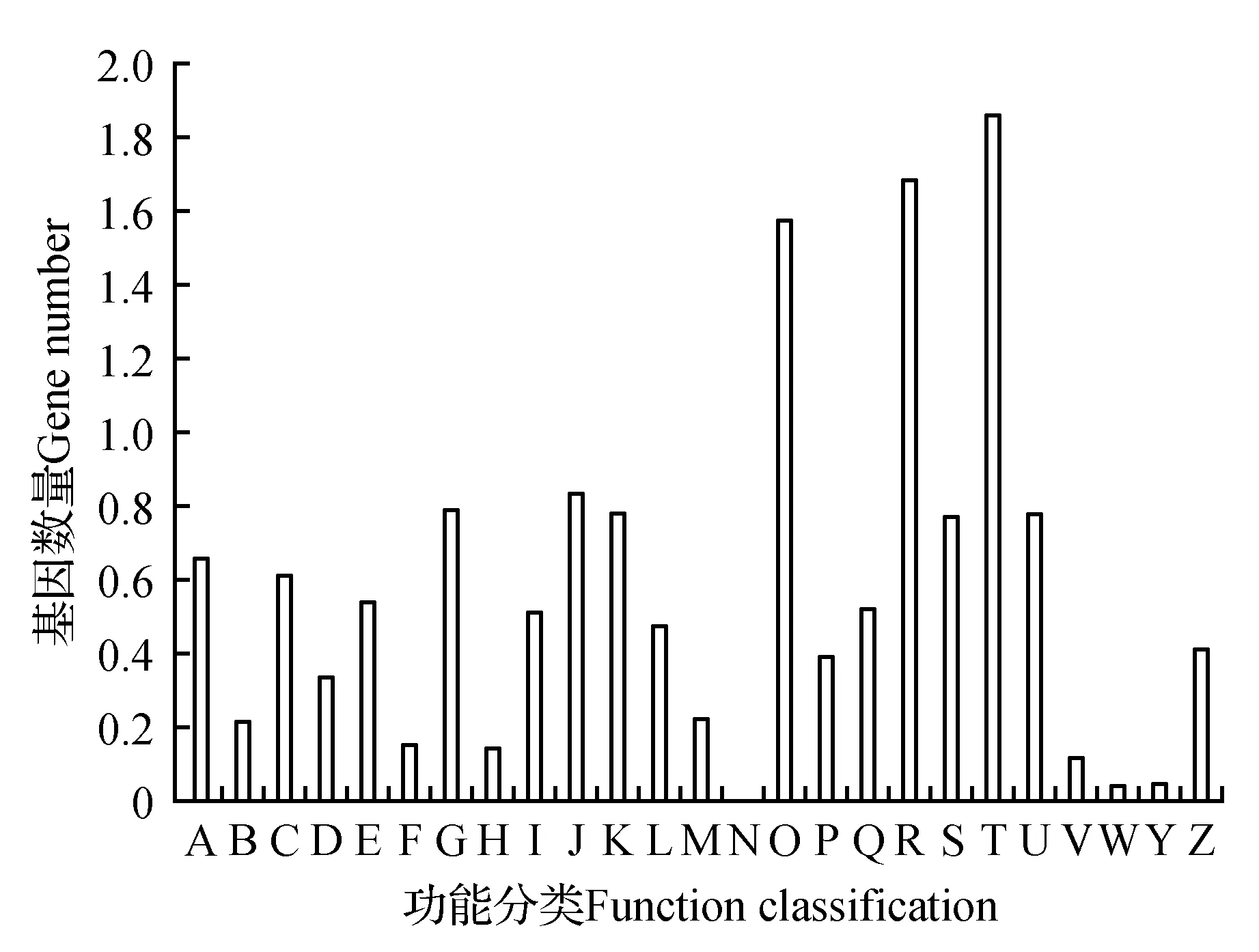
图2 山莨菪转录组Unigenes的KOG功能分布图 A.RNA加工和修饰RNA;B.染色体结构和动力学;C.能源生产与转化;D.细胞周期调控、细胞分裂、染色体分离;E.氨基酸转运和代谢;F.核酸转运和代谢;G.碳水化合物转运和代谢;H.辅酶转运和代谢;I.脂类转运和代谢;J.翻译、核糖体结构和生物发生;K.转录;L.复制、重组和修饰;M.细胞壁/细胞膜生物发生;N.细胞活性;O.翻译后修饰、蛋白转运;P.无机离子转运和代谢;Q.次生代谢物合成、转运和代谢;R.只有一般功能预测;S.未知功能;T.信号传递机制;U.细胞间运输、分泌物和囊泡运动;V.防御机制;W.细胞外结构;Y.核结构;Z.细胞骨架Fig.2 KOG functional annotation distribution of unigenes of transcriptome for A.tanguticus A.RNA processing and modification; B.Chromatin structure and dynamic; C.Energy production and conversion; D.Cell cycle control,cell division,chromosome partitioning; E.Amina acid transport and metabolism; F.Nucleotide transport and metabolism; G.Carbohydrate transport and metabolism; H.Coenzyme transport and metabolism; I.Lipid transport and metabolism; J.Translation,ribosomal structure and biogenesis; K.Transcription; L.Replication,recombination and repair; M.Cell wall/membrane/envelope biogenesis; N.Cell motility; O.Posttranslational modification,protein turnover,chaperones; P.Inorganic ion transport and meabolism; Q.Secodary metabolites biosynthesis,transport and catabolism; R.General function prediction only; S.Function unknown; T.Signal transduction mechanisms; U.Intracelluar trafficking,secretion,and vesicular transport; V.Defense mechanisms; W.Extracellular structures; Y.Nuclear structure; Z.Cytoskeleton
2.3 KOG功能注释
将Unigenes与KOG蛋白质库进行比对,有13 110条Unigenes获得注释,占总Unigenes的18.35%,Unigenes根据其功能被分为了26个类别,都与山莨菪最基本的生命活动相关(见图2)。其中,在已经明确生物学功能的Unigenes中(占94.65%),信号传递机制最多,有1 873条Unigenes,占比为12.76%,其次是只有一般功能预测和翻译后修饰,分别为1 703(11.60%)和1 580(10.76%)。与转录、翻译、化合物运输、能量生产转化、代谢等功能相关的Unigenes处于相对较多的数量,数量在500~900条,而注释出来与染色体结构、核结构、细胞骨架、细胞周期等相关序列是较少的,有的甚至只有不到10条。
2.4 NR功能注释
通过与NR库(evalue<0.000 01)进行比对,得到39 621个被注释的Unigenes,未获得注释的基因较少,仅有22个(0.056%)。与山莨菪能比对上有8个同科(茄科)植物,这8个同科植物主要是茄属、辣椒属和烟草属的植物,彼此之间具有较近的亲缘关系,另外还有两种非茄科的植物,分别是豆科和藜科植物,所占比例较小,有3 422(8.64%)的Unigenes零星分布于其他392个物种中。在所有的注释中,注释为与阳芋(Solanumtuberosum)相关的Unigenes最多,有8 598条(21.70%),其次为辣椒(Capsicumannuum)和烟草(Nicotianatabacum),分别占16.24%和13.45%,比例所占较大的前几种同科植物均可说明与山莨菪具有较高的序列同源性。
2.5 GO功能注释及分类
为全面描述山莨菪基因和基因产物的属性,了解山莨菪表达基因的功能分布状况及其所代表的生物学意义,将Unigenes通过与Swissprot、TrEMBL数据库的比对,得到GO功能注释信息,注释后得到29 309个Unigenes,对其进行统计分析后(表3),可分为分子功能、生物学过程和细胞组分3个大类,分别有16、24、22个子类,共有62个子类,其中结合、催化活性、细胞过程、代谢过程、细胞、细胞部分及细胞器等获得注释较多,而注释为金属伴活动分子功能调节器、结构分子活性、传译调治活性、细胞、膜封闭腔、拟核的Unigenes基本是最少的。
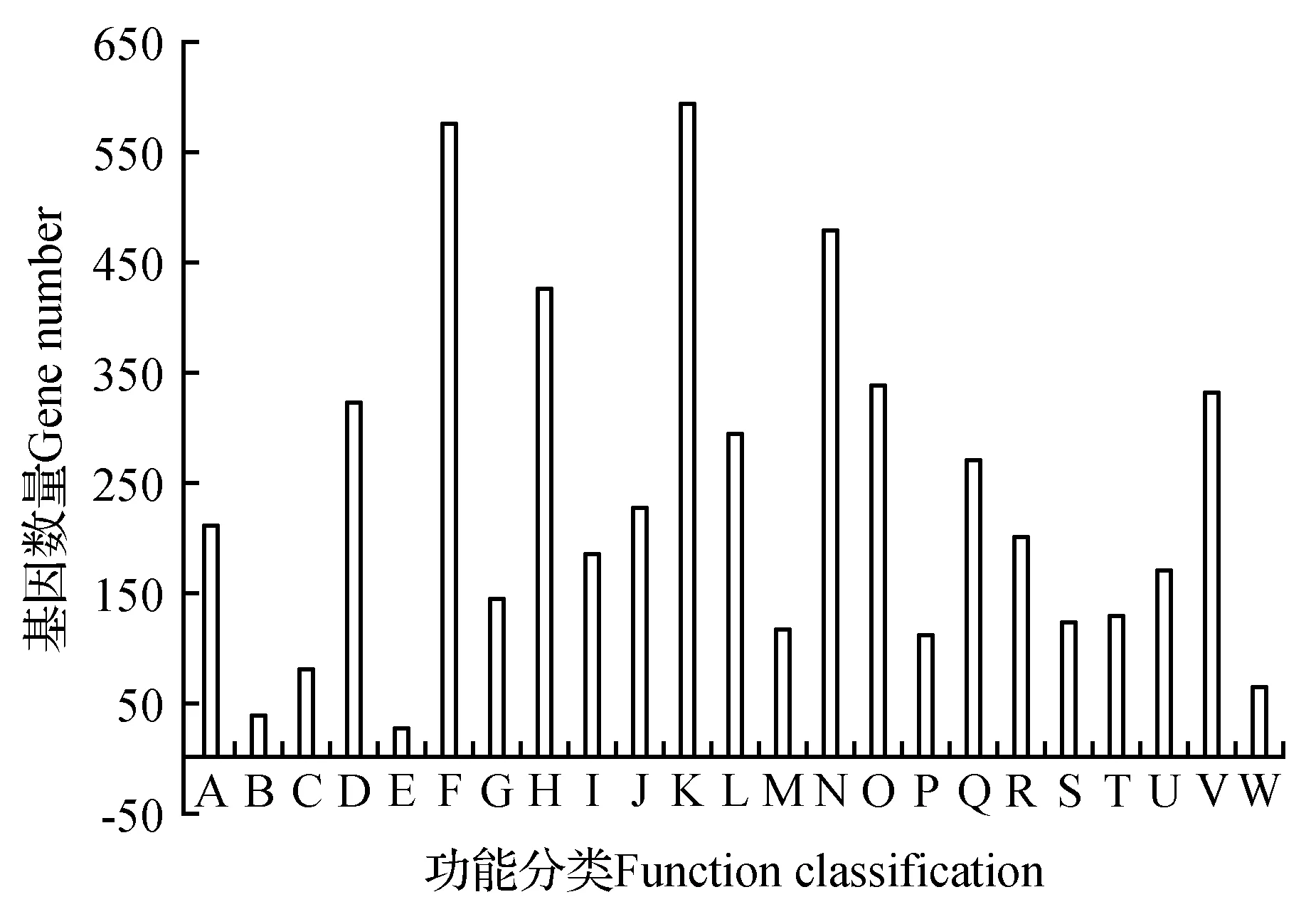
图3 山莨菪转录组Unigenes的KEGG功能注释分布统计图 A.细胞生长和死亡;B.细胞运动;C.细胞群体;D.运输和分解代谢;E.膜运输;F.信号转导;G.信号分子和互作作用;H.折叠排序与退化;I.复制和修复;J.转录;K.翻译;L.氨基酸代谢;M.其他次级代谢产物合成;N.碳水化合物代谢;O.能量代谢;P.多糖合成与代谢;Q.类脂(化合)物代谢作用;R.辅助因子和维生素的代谢;S.其他氨基酸的代谢;T.萜类化合物和聚酮化合物的代谢;U.Nucleotide metabolism;V.概观;W.异生素生物降解和新陈代谢Fig.3 KEGG functional annotation distribution of unigenes of transcriptome for A.tanguticus A.Cell growth and death; B.Cell motility; C.Cellular community; D.Transcript and catabolism; E.Membrane transport; F.Signal transduction; G.Signaling molecules and interaction; H.Folding sorting and degradation; I.Replication and repair; J.Transcription; K.Translation; L.Amino acid metabolism; M.Biosynthesis of other secondary metabolites; N.Carbohydrate metaboliam; O.Energy metabolism; P.Glycan biosynthesis and metabolism; Q.Lipid metabolism; R.Metabolism of cofactros and vitamins; S.Metabolism of other amino acids; T.Metabolism of terpenoids and polyketides; U.Nucleotied metabolism; V.Overview; W.Xenobiotics biodegradation and metabolism
2.6 KEGG代谢通路分析
以KEGG代谢库(evalue<0.000 01)为参考,对代谢通路进行统计和分类分析。有3 679条Unigenes被注释,这3 679条被注释的基因根据涉及到的代谢通路分为4大类,23个子类(见图3)。在4种代谢大类中,代谢相关的通路获得注释最多,为2 640个,占比49.45%,其次分别是遗传信息处理、细胞过程、环境信息处理,分别获得了1 438(26.93%)、656(12.29%)、605(11.33%)个注释。23个亚类中,与代谢相关的通路最多,有12条,包括氨基酸代谢、碳水化合物代谢、其他次生物质代谢、能量代谢、糖生物合成与代谢、脂类物质代谢、辅助因子和维生素代谢、其他氨基酸代谢、萜类化合物和聚酮化合物代谢、核苷酸代谢、辅助因子代谢、概观;与遗传信息处理、细胞过程、环境信息处理相关的通路较少,分别有4条、4条、3条。在这23个子类代谢途径中,翻译获得的注释最多,为595条,占11.14%,其次为信号转导、碳水化合物代谢,折叠排序和退化,分别为576条(10.79%)、479条(8.97%)和428条(8.01%)。

表3 山莨菪转录组Unigenes的GO功能分类统计
有7 075条Unigenes归入到209条代谢途径中,按照基因获得的注释数量从高到低排列,将前13个代谢通路列于表4中,其中以核糖体代谢通路最多,占3.92%,其次为碳代谢和植物激素信号转导,分别占2.64%和2.45%。
2.7 山莨菪药用活性成分分析
山莨菪植株体内含有生物碱、萜类、苯丙素类、黄酮类、糖苷类、醌类、聚酮类、有机酸及酚类等药效成分(表5),其中生物碱、萜类、黄酮类和糖苷类成分的代谢通路分别有5条(以异喹啉生物碱生物合成途径,二苯乙烯类、二芳基庚烷类和姜辣素生物合成途径为主)、4条(以萜类骨架生物合成途径为主)、3条(以类黄酮生物合成为主)、2条(以泛醌和其他萜类化合物—醌生物合成途径为主),其余药效成分目前基本均只有1条代谢途径。在所有药效成分中,萜类和苯丙素类所对应的unigenes是最多的,均有67条,生物碱、醌类及黄酮类所对应的unigenes数量也较多,分别有30、28、22条,而糖苷类、聚酮类、有机酸及酚类对应的unigenes是相对较少的,分别只有2,2和11条。对山莨菪不同药效成分的代谢通路、所对应unigenes的数量及类型进行分析,将便于复杂基因调控网络的建立,有助于为人工合成所需药用成分提供研究基础和思路。
表4 山莨菪Unigenes数量最多的13个代谢通路
Table 4 Top thirteen metabolic pathways involved inA.tanguticusunigenes
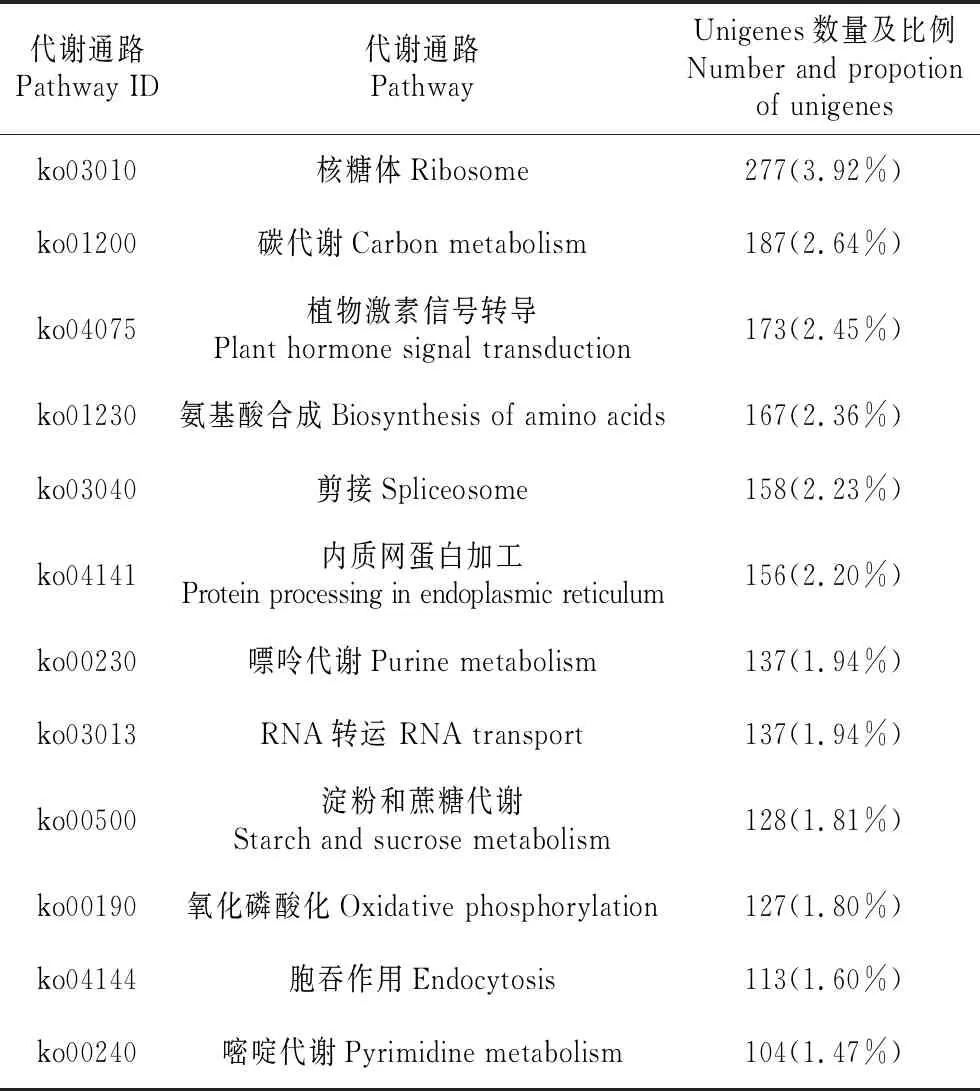
代谢通路Pathway ID代谢通路PathwayUnigenes数量及比例Number and propotion of unigenesko03010核糖体Ribosome277(3.92%)ko01200碳代谢Carbon metabolism187(2.64%)ko04075植物激素信号转导Plant hormone signal transduction173(2.45%)ko01230氨基酸合成Biosynthesis of amino acids167(2.36%)ko03040剪接Spliceosome158(2.23%)ko04141内质网蛋白加工Protein processing in endoplasmic reticulum156(2.20%)ko00230嘌呤代谢Purine metabolism137(1.94%)ko03013RNA转运 RNA transport137(1.94%)ko00500淀粉和蔗糖代谢Starch and sucrose metabolism128(1.81%)ko00190氧化磷酸化Oxidative phosphorylation127(1.80%)ko04144胞吞作用Endocytosis113(1.60%)ko00240嘧啶代谢Pyrimidine metabolism104(1.47%)
2.8 SSR与SNP检测分析
使用BCFtools根据Mapping结果找出可能的SNP位点(单核苷酸位点多态性),最终一共获得31 382个SNP位点,3 675个InDel,分析发现山莨菪Unigenes序列上SNP分布不均匀,转换突变类型的SNP数量比颠换突变的2倍略多(见图4)。其中转换突变类型(A→G、C→T、G→A、T→C)有20 168个,占64.26%,颠换突变类型(A→C、A→T、C→A、C→G、G→C、G→T、T→A、T→G)有11 219个,占35.74%。采用MISA基于拼接所得转录本序列信息进行SSR(简单序列重复标记)分析,图5的SSR密度分布图显示,有6种SSR重复类型,其中单碱基重复、二碱基重复和三碱基重复类型占绝大多数,每百万碱基中出现的单碱基重复的SSR个数有56.52个,占45.30%,二碱基重复和三碱基重复的个数分别为28.22和37.29,分别占22.62%、29.89%,而四碱基重复、五碱基重复和六碱基重复所占比例仅占2.19%。丰富的SSR和SNP位点对山莨菪遗传图谱构建、遗传多样性分析和亲缘关系研究具有重要的意义和价值。
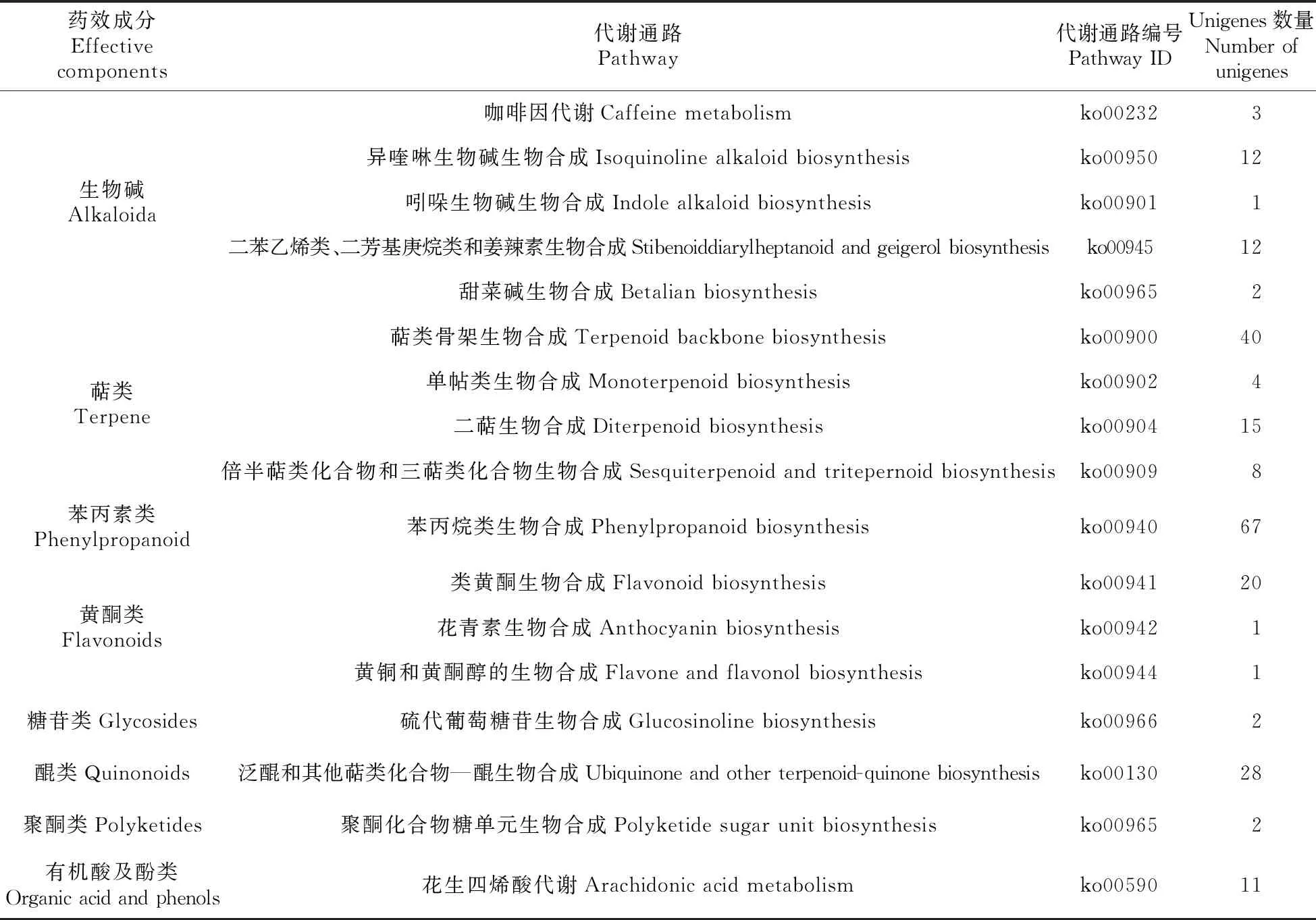
表5 山莨菪药用活性成分代谢通路及基因统计
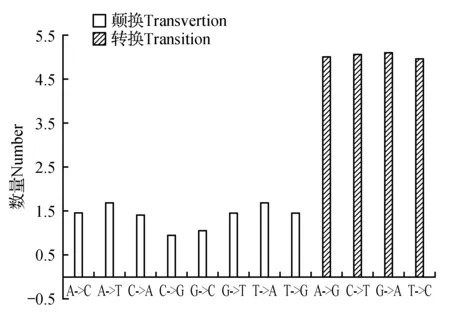
图4 山莨菪转录组Unigenes的突变谱系图Fig.4 Mutation pedigree of unigenes of transcriptome for A.tanguticus
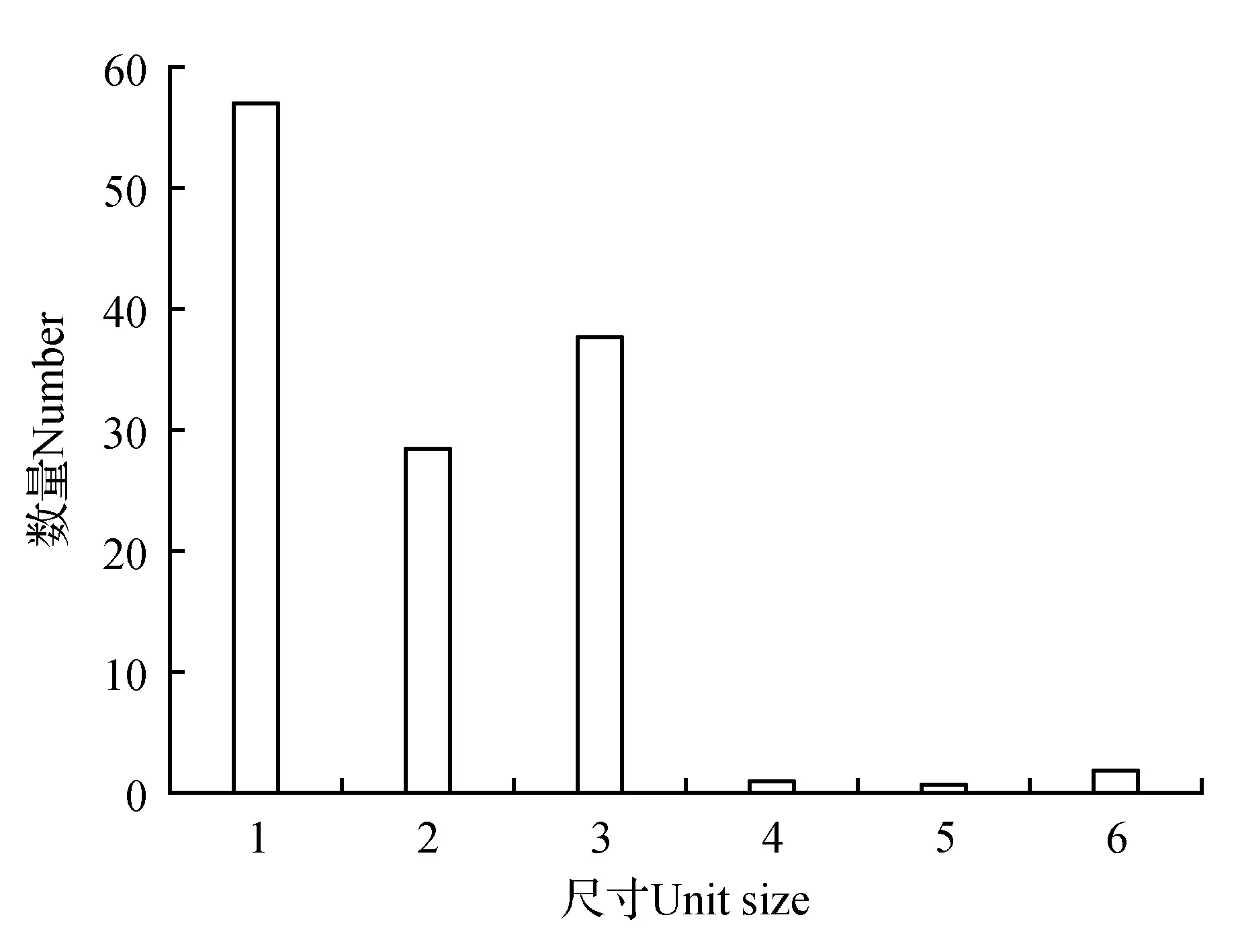
图5 山莨菪转录组Unigenes的SSR密度分布图Fig.5 SSR Density Distribution Map of unigenes of transcriptome for A.tanguticus
3 讨论
本研究采用Illumina测序平台对山莨菪进行高通量测序、从头组装、拼接、去冗余,结合多个数据库对基因进行注释和分析,一共获得了158 378条Transcripts,71 463条Unigenes,对编码序列进行预测后获得,47 685条CDS。最长的Unigenes为8 526 bp,最短的为201 bp,N50为1 115 bp,平均长度是651.1 bp,结合一些分析较为成熟的常见资源植物的拼接结果,如芝麻Sesamumindicum(N50为1 006 bp,平均长度683 bp)[24]、樟树Cinnamomumbodinieri(N50为1 023 bp,平均长度584 bp)[25]、百合Liliumbrownii(平均长度673 bp)[26]、铁皮石斛Dendrobiumofficinale(平均长度660 bp)[27],可知山莨菪的序列拼接质量与上述植物相近,而仿刺参Apostichopusjaponicus与山莨菪虽然在基因组和转录组发展方面具有类似的情况,但其拼接后序列的平均长度却仅为344 bp[28],因此,在山莨菪分子生物学方面的研究较少、转录组和基因组信息相对缺乏的情况下,还能获得与转录组分析较为成熟的植物比较相近的拼接结果,可知其拼接质量是较高的。
将Unigenes与9大数据库(CDD、KOG、NR、NT、GO、Swissprot、PFAM、KEGG、TrEMBL)进行对比,注释成功的Unigenes有47 624(66.64%)条,但仍有23 839(33.36%)条序列未能获得注释,数量较多,究其原因,可能有以下3点:①一般而言,在测序质量较高的前提下,序列片段的长度越长,注释的成功率和可靠性就越高,因而山莨菪的拼接质量较优,但结果却有33.36%的Unigenes无法获得注释,这可能是因为其序列本身长度就较短,因此很难在同源性比对时获得结果;②目前山莨菪的分子生物学研究仍处于初级阶段,基因组学研究的匮乏导致基因功能注释信息的缺失,因此会有部分序列很难得到注释信息;③山莨菪本身转录组可能具有一定的特异性,由于之前尚未有山莨菪转录组方面的研究作为参考,相关功能基因的研究也较少,所以可能使得部分特异性基因得不到识别和注释。因此,测序技术及质量的提高,基础功能数据库的不断完善,以及对山莨菪基因组学方面研究的进一步探索,很大程度上可有效减少未被注释序列的数量。
成功注释到KOG数据库中的Unigenes有131 100条,根据表1中Unigenes的注释数量及比例可知,注释到该库的比例与注释到NR数据库的55.44%、注释到NT数据库的54.25%、注释到GO的41.01%以及注释到TrEMBL的55.49%相比而言,注释的比例相对较低,仅有18.35%,排除这几个数据库本身注释难易程度差异的影响,这可能还与数据库内部数据的丰富程度、山莨菪本身转录组信息是否完善等因素有关。从KOG功能注释图中可以了解到,在获得注释的26类Unigenes中,信号传递、转录翻译、能量生产转化、代谢等相关基因被注释的数量较多,而与细胞结构、周期相关的基因数量却较少,由被注释功能基因的数量可以推测不同基因表达丰富度的差异,从而判断不同生命活动对山莨菪生长发育的重要程度。
在与NR数据库比对之后,注释为阳芋同源种的Unigenes有8 598条,远高于其他几个被注释物种,出现这种情况,可能是由于山莨菪与阳芋的进化史和生活史较为接近,可以说明山莨菪与阳芋的亲缘关系更紧密一点,而与其他物种的亲缘关系相对较远。对拼接组装获得的Unigenes进行了代谢途径分析和功能分类,共获得3 679条Unigenes注释,注释可细分为4个代谢通路大类,其中定位到代谢通路相关的基因最多,占49.45%,证明山莨菪具有较强的代谢活动能力。进一步对山莨菪的药用活性成分(物碱、萜类、苯丙素类、黄酮类、糖苷类、醌类、聚酮类、有机酸及酚类等)的代谢通路及相关Unigenes数量和类型进行统计分析,发现与生物碱相关的代谢通路最多,而萜类和苯丙素类所对应的Unigenes数量最多,该分析将有助于推进山莨菪药用成分合成机理的探索,为所需成分的提取和复合成分的合成提供研究基础。
转录组测序分析后,共检测出31 382个SNP多态位点,两种突变类型中以转换突变为主,占2/3;在SSR分析中,获得六种SSR重复类型,其中以单碱基重复、二碱基重复和三碱基重复为主。通过SNP分析与SSR分析,可以构建较高分辨率的山莨菪遗传图谱,有助于提高目的基因定位的准确性,同时促进山莨菪群体遗传学、比较基因组学和分子系统学的发展。
