猪基因表达调控数据库(GereDB)的构建
2020-06-04石博妹姚敏余平黄廷华
石博妹 姚敏 余平 黄廷华

摘要:【目的】构建猪基因表达调控数据库(GereDB),为从基因水平解释猪的生长发育规律、遗传育种和疾病防控等提供科学依据。【方法】从NCBI下载小鼠和猪的RNA序列原始数据进行序列比对,根据序列同源性将小鼠的基因表达调控信息转移给猪,并建立猪基因表达调控信息网络,整理加工后根据区域结构,以Linux为操作系统、Apache为Web服务器、MySQL为数据库、Python为服务器端脚本解释器构建猪GereDB数据库。【结果】从NCBI下载的Fast数据共包含291182条猪核苷酸序列,通过序列比对和手工整理,注释筛选出67000多条猪核苷酸序列;将小鼠的基因表达调控信息转移给猪,获得的猪基因表达调控关系链接有67027条,构建了豬GereDB数据库(http://www.thua45.cn/geredb-wp/),并开发GEREA生物信息学分析工具以发现猪基因表达调控因子。在猪GereDB数据库中有116个调控因子可调控100多个基因,说明其在猪转录组调控中发挥重要作用。GEREA生物信息学分析工具在已发表的猪乳腺组织数据集上进行测试,结果显示,与母猪分娩前14 d相比,分娩后1 d母猪乳腺中26个调控因子的靶基因显著差异表达(FDR<0.05),其中FGF2调控因子在母猪泌乳方面发挥重要作用。【结论】猪GereDB数据库能提供猪基因表达和调控间关系的信息,且能使用GEREA生物信息学分析工具发掘猪基因表达调控数据,有助于揭示调控因子对高通量测序差异表达基因的调控机制,为从基因水平探究猪的生长发育及疾病防控提供数据信息。
关键词: 猪;基因表达;调控因子;GereDB数据库
Abstract:【Objective】To construct the pig gene expression and regulation database(GereDB) for providing a scientific basis to explain the growth and development, genetic breeding and disease treatment of pigs at the gene level. 【Me-thod】Original RNA sequence data of mouse and pig were downloaded for sequence alignment from NCBI and transferred gene expression and regulation information from mouse to pig according to the sequence homology, analyzed pig gene expression and regulation data to establish the pig gene expression and regulation information network. According to the regional structure after processing, GereDB database of pig was established with Linux operating system, Apache Web server, MySQL database management system,Python for server-side script interpreter. 【Result】A total of 291182 pig nucleotide sequences were contained in Fast data downloaded form NCBI. The mouse gene expression regulation information was transferred to pig, 67027 relationship links in regulating gene expression of pig were obtained , and the pig GereDB database(http://www.thua45.cn/geredb-wp/) was built, GEREA bioinformatics analysis tools were developed to find gene expression regulators of pig. There were 116 regulators could regulate more than 100 genes in pig GereDB database, indica-ting that they played an important role in the transcriptome regulation of pig. The GEREA bioinformatics analysis tool was tested on a published data set of pig breast tissue and the result showed that 26 target genes of regulatory factors appeared significantly differential expression(FDR<0.05) on the sow 1 d after delivery compared with the 14 d before delivery. Moreover, FGF2 was as an vital regulatory factor for the milking of sows. 【Conclusion】Pig GereDB database can provide relationships between pig gene expression and regulation, and GEREA bioinformatics tool can explore pig gene expression regulation data. The database is useful for exploring how differentially expressed genes detected by high throughput experiments are regulated by certain regulator genes and can provide valid data to explore the growth and development, disease control and prevention at gene level.
0 引言
【研究意义】基因表达调控是一个复杂而又重要的过程,在过去的几十年里已取得重大进展(Wachter,2014;Jones,2015),但针对猪的研究相对滞后,尚未形成基因表达调控数据库(GereDB)。GereDB数据库能实现将同一物种转录因子—靶基因相互作用的关系信息进行整理归纳,有助于科技人员快速准确地提取与目的基因相关的基因或蛋白资料,因此,以小鼠GereDB数据为基础,利用序列同源性比对构建猪GereDB数据库,可为掌握猪的生长发育规律和疾病防控机理等提供便捷的分析工具。【前人研究进展】近年来,在人类和小鼠的基因表达调控系统,包括先天免疫系统、代谢系统和信號转导等方面已取得突破性进展(Wachter,2014;Jones,2015),许多重要的基因表达调控因子如TNF、IFNG和MAPK也被发现(Arthur and Ley,2013;Croft and Siegel,2017;Green et al.,2017),但仍有许多问题亟待进一步探究,包括关键调节因子对特定刺激的响应方式及有效发现关键调节因子的方法等(Subramanian et al.,2005;Shojaie and Michailidis,2010)。基因表达调控涉及到复杂的网络,且受多个重要的正、负效应因子控制,而这些效应因子可影响转录组的组成(Huang et al.,2011;Zhao et al.,2016)。为此,数个先进的数据库应运而生,包括TFactS(Essaghir et al.,2010)、HTRIdb(Bovolenta et al.,2012)和TRRUST(Han et al.,2018)。Bovolenta等(2012)建立的HTRIdb数据库整理了人类转录因子—靶基因相互作用关系的信息,研究者可通过此数据库快速准确地提取与研究基因相关的基因或蛋白资料。Essaghir和Demoulin(2012)利用TfactS数据库对已发表的恶性肿瘤基因表达数据及癌症基因组图谱进行分析,筛选出TFRC、MET和VEGFA基因可作为恶性肿瘤标志物候选基因,因此检测患者血清中TFRC、MET和VEGFA基因的表达量对恶性肿瘤确诊有重要意义。Han等(2018)将人源TRRUST数据库升级为TRRUST v2数据库,升级后的TRRUST数据库不仅包含800个人类转录因子调控的8444多个靶基因,还增加了828个小鼠转录因子调控的6552个靶基因,该数据库存储了大量人类和小鼠基因表达调控信息,为筛选调控人类疾病发生过程的关键转录因子提供了参考依据。【本研究切入点】TfactS、HTRIdb和TRRUST数据库是专门针对人类或小鼠的基因表达调控信息,因此,亟需构建猪GereDB数据库以使有效发掘猪基因表达调控的关键调节因子。【拟解决的关键问题】将猪的RNA序列与小鼠的RNA序列进行比对,利用序列同源性将小鼠基因表达调控信息转移给猪,构建猪GereDB数据库,为从基因水平解释猪的生长发育规律、遗传育种和疾病防控等提供科学依据。
1 材料与方法
1. 1 猪和小鼠基因序列获取及比对分析
小鼠RNA序列和猪RNA序列的原始数据(Fasta格式)从美国国立生物技术信息中心(National Center for Biotechnology Information,NCBI)下载;本地化的BLAST软件也从NCBI网页下载,然后在本地安装配置并运行,建立本地化的网页BLAST比对服务。打开命令提示符,切换至保存核苷酸序列的文件夹,同时运行以下命令建立数据库文件:makeblastdb -in db.fasta -dbtype prot -parse_seqids -out dbnam,运行以下命令将猪RNA序列与小鼠RNA序列进行比对:blastp -query seq.fasta-out seq.blast-db dbname-outfmt 6-evalue 1e-5-num_descriptions 10-num_ threads 8,查看比对结果,根据相似性和P确定猪与小鼠间的同源基因,即比对结果序列相似性越高,期望值越小,其同源性越高。本研究最终选择猪和小鼠中的最佳匹配基因对,其最佳匹配原则为P小于10-6,且相似性大于70%。
1. 2 猪基因功能注释
根据猪与小鼠的基因序列比对结果,筛选出序列同源性高的基因序列,将小鼠的基因表达调控信息转移给猪并建立猪基因表达调控信息网络,小鼠GereDB数据库已由GereDB团队开发并发布使用(Huang et al.,2019)。根据小鼠基因表达调控信息(三列数据)在猪基因里查找对应的基因,建立猪基因表达调控信息(三列数据)。小鼠的A序列与猪的a序列相似,小鼠的A1序列与猪的a1序列相似,由于小鼠的A序列正调控A1序列,故推测猪中的a序列和a1序列也存在正调控关系。
1. 3 GereDB数据库系统形成与发布
1. 3. 1 GereDB数据库构建 手工提取的猪基因表达调控相互关系包含3个要素,即基因表达调控因子、靶基因及调控因子与靶基因的直接连接。GereDB数据库以Linux为操作系统,Apache为Web服务器,MySQL为数据库,Python为服务器端脚本解释器,并由这4个软件组建一个稳定、免费的网站系统。
1. 3. 2 GEREA生物信息学分析工具开发 对猪基因表达调控信息网络进行整理加工,并进一步收集整理相关的数据资源,开发GEREA生物信息学分析工具以发现基因表达调控因子,进而对猪基因表达数据进行分析。本研究将活性调控因子定义为一个基因,该基因具有发生在不同表达基因列表中的目标,其频率显著高于预期基因表达,基因表达调控因子—靶基因链接建成后,基因表达分析数据加载于调控因子的靶基因上(图1-A)。基于网络数据可被组织为一个3×3列联表,分析结果中大写字母所代表的含义见图1-B,通过Fisher精确性检验分析可得到一个全局的显著性检验P0、上调表达显著性检验P1及下调表达显著性检验P2(Sun and Yu,2016),然后采用本杰明和霍克伯格方法(FDR)进行校正(Gui et al.,2015)。若P1<0.05说明靶基因的表达变化由该调控因子上调表达引起,若P2<0.05说明靶基因的表达变化是由该调控因子下调表达引起。
1. 3. 3 采用真实猪基因表达数据集测试GEREA生物信息学分析工具 GEREA生物信息学分析工具在已发表的猪乳腺组织数据集上进行测试。母猪乳腺组织受分娩的刺激(Essaghir and Demoulin,2012),在母猪分娩前后分别取其活体乳腺组织进行高通量测序分析。原始转录组数据来自NCBI的GEO数据库,登录号GSE101983。采用Bioconductor中的分位数法对数据进行归一化处理(Bolstad et al.,2003),分析分娩刺激对乳腺中基因调控因子调节靶基因的表达情况。
2 结果与分析
2. 1 猪和小鼠基因序列的获取及比对分析结果
从NCBI genome FTP下载的Fast数据共包含291182条猪核苷酸序列,通过序列比对和手工整理,注释筛选出67000多条猪核苷酸序列。表1列出猪和小鼠前50个相似性最高的基因序列,根据最佳匹配原则,可判定表中数据显著性良好。
2. 2 猪基因表达调控信息网络构建
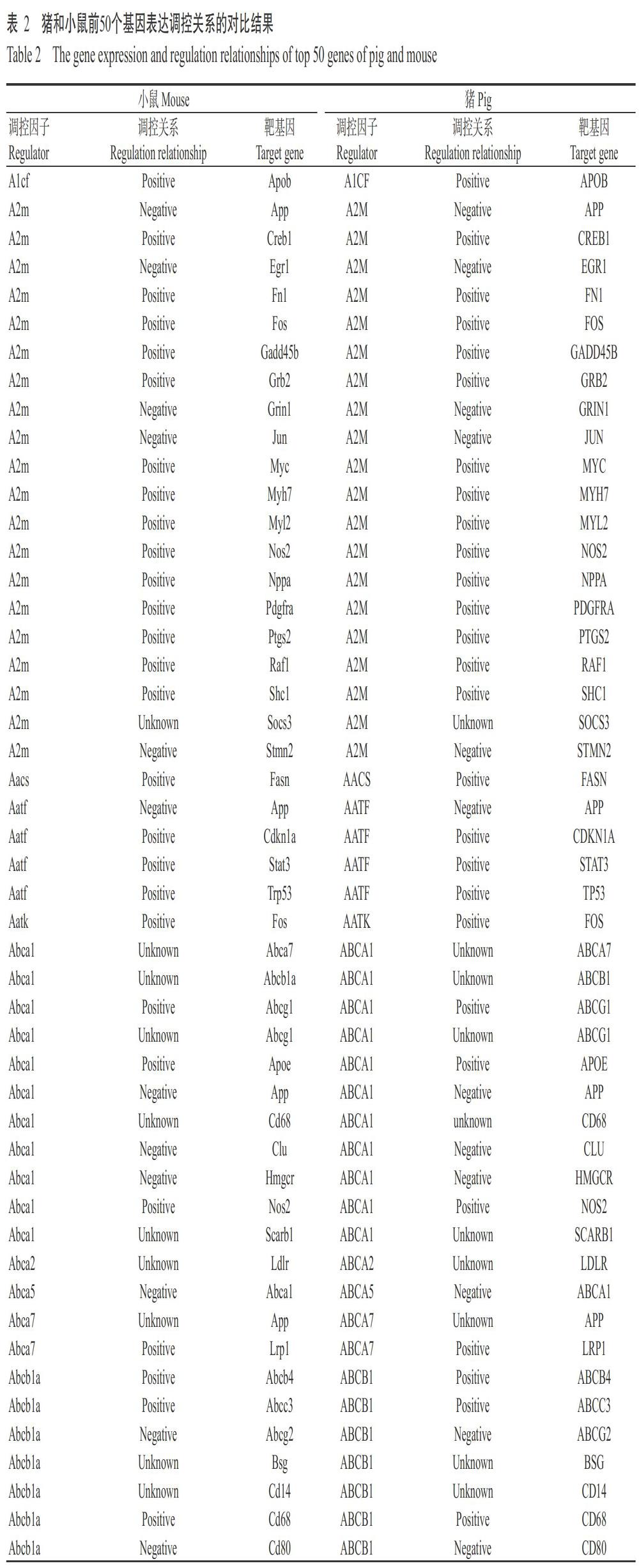
通过将小鼠的基因表达调控信息转移给猪,建立与猪基因表达调控信息相关的数据库,猪的基因表达调控关系链接有67027条,其中猪和小鼠前50个基因表达调控关系的比对结果见表2。
2. 3 GereDB数据库的形成与发布
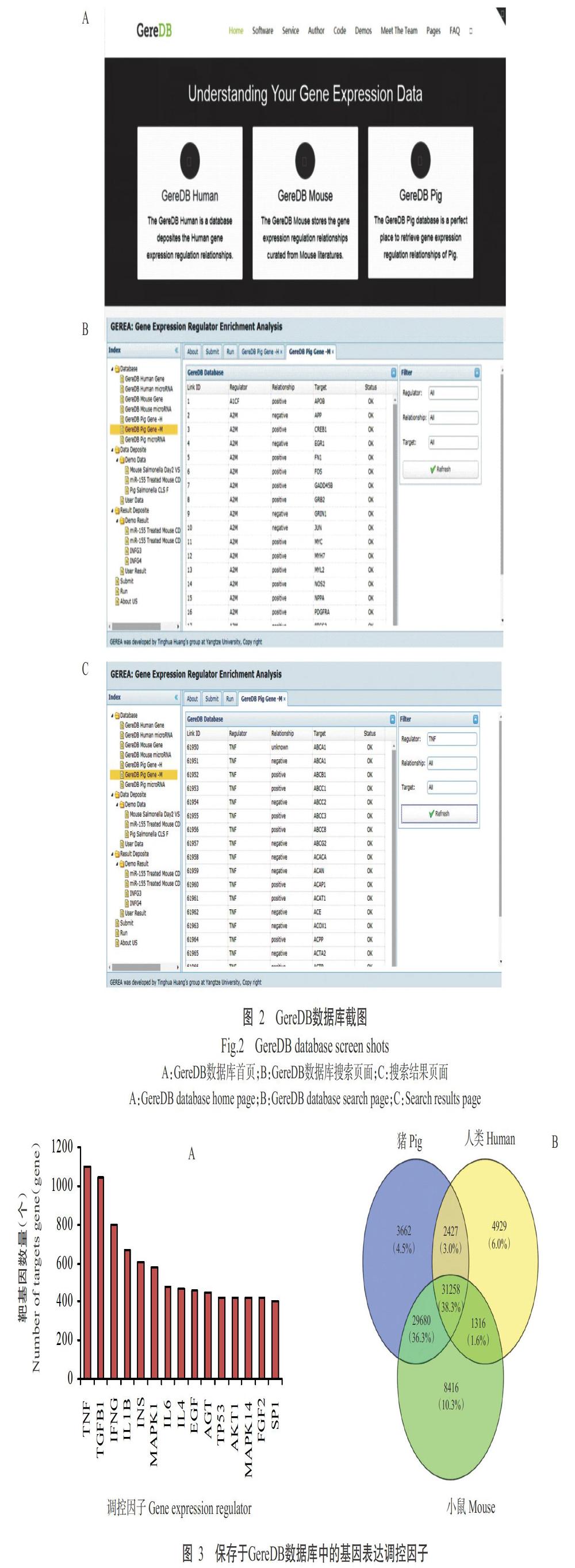
在GereDB数据库首页(http://www.thua45.cn/geredb-wp/)上可看到猪的基因表达数据在最右侧(图2-A),点击进去后进入GereDB搜索页面,可选择搜索Regulator、Relationship或Target链接(图2-B)。GereDB數据库中的数据是根据基因组织,其链接搜索允许使用NCBI官方基因符号(基因名)检索感兴趣的数据。从Links搜索结果页面(图2-C)可获得与基因表达调控因子相关的基因,包括调控基因和靶基因的官方基因符号,以及调控基因对每个靶基因的影响。
2. 4 GereDB数据库是一种独特的基因表达调控资源
通过对GereDB数据库中的调控关系进行分析,发现大量基因表达调控子网络构成了基因表达调控网络体系(Boyle et al.,2014)。图3-A显示靶基因数量最多的前15个网络体系调控因子,若将基因表达调控网络体系分解成单个子网络时,即可查找调控基因表达的最主要调控因子。TNF可调控GereDB数据库中的1103个基因,而TGFB1可调控1042个基因。GereDB数据库中有116个调控因子可调控100多个基因,说明其在猪转录组调控中发挥重要作用。GereDB团队也曾对小鼠和人类的基因表达调控关系进行整理,在GereDB数据库中储存的猪、小鼠及人类基因调控链接数如图3-B所示。小鼠的基因调控链接数为70670条,人类的基因调控链接数为39930条,猪的基因调控链接数为67027条。其中,猪与小鼠的重叠数有60938条(重叠率为74.6%),与人类的重叠数有33685条(重叠率为41.3%),三者的重叠数有31258条,重叠率为38.3%。
2. 5 真实猪基因表达数据集测试GEREA生物信息学分析工具的结果
GEREA分析生物信息学工具运行70 s,结果(表3)显示,与母猪分娩前14 d相比,分娩后1 d母猪乳腺中26个调控因子的靶基因显著差异表达(FDR<0.05)。其中,乳腺组织中FGF2调控的靶基因有19个正调控上调表达、3个负调控上调表达,另有6个正调控下调表达、7个负调控下调表达,提示FGF2调控因子在母猪泌乳方面发挥重要作用。
3 讨论
在人类生物医学研究中,模式生物小鼠的研究在过去十几年间已非常全面深入,尤其一些与疾病发生、生长繁殖及代谢有关的基因在小鼠中已得到深入研究(Vemula et al.,2019;Yamamoto et al.,2019;Yin et al.,2019)。为此,本研究借鉴和参照小鼠已获得的基因表达调控信息,通过序列比对方式,研究相关基因在猪基因表达调控中的特性和功能,并对其进行详细注释,最后将比对结果及相关注释信息储存到专门的GereDB数据库中,建立猪基因表达调控二级数据库,为解释猪的生长发育规律、遗传育种和疾病防控等提供科学依据。
获取基因功能信息的一种有效途径是通过不同物种间序列相似性比对,利用相似性检索分析对基因序列进行分析,推测其代表的基因结构和功能。该方法已成为一种常用的功能基因组研究方法,尤其在新的物种完成基因组测序后,利用其他物种中的已知基因功能信息对新测序物种中的基因进行注释和分析,具有方便快捷且准确有效的特点,获得的功能注释信息可为后续的研究验证提供重要线索(Vallenet et al.,2019)。本研究从NCBI上获取猪和小鼠的基因组序列,通过序列相似性比对分析,以小鼠的基因表达调控信息为原材料,成功获取猪基因表达调控关系数据。本研究构建的猪GereDB数据库是猪专门化本地二级数据库,基于其本地化BLAST检索系统是通过整合由NCBI开发的数据库搜索程序BLAST工具为用户提供序列比对功能,通过一种局部的算法来获得2个基因序列中的相似性序列,其分析过程与通过NCBI在线BLAST分析一致,参数选择及最终结果也与NCBI在线分析结果的格式类似,但检索速度和准确性得到明显提高,是一套能迅速与大型公开数据库(主要是蛋白数据库或DNA数据库)进行相似性序列比对,寻找相同或相似序列且相对准确而高效的分析工具。BLAST能接受用户输入的序列,通过在本地数据库中进行捜索,然后将用户输入序列的同源序列返回给用户,实现本地核苷酸与蛋白的同源性检索。
GereDB数据库是一个独特、有效、方便的资源,尤其对于人类基因表达数据研究者来说,提供了一个可获得较全面关系信息的平台。对于猪基因的表达和调控,由于缺乏从文献中提取相关数据的有效方法,通常难以获得猪基因表达调控关系数据,而本研究构建的猪GereDB数据库有助于探索高通量测序检测到的差异表达基因是如何被某些调控因子所调控。此外,利用存储于GereDB数据库中猪基因表达与调控间的关系,以及GEREA的调控基因发现算法,能发现编排特定转录谱的调控因子。
4 結论
猪GereDB数据库能提供猪基因表达和调控间关系的信息,且能利用GEREA生物信息学分析工具发掘猪基因表达调控数据,有助于揭示调控因子对高通量测序差异表达基因的调控机制,为从基因水平探究猪的生长发育及疾病防控提供数据信息。
参考文献:
Arthur J S,Ley S C. 2013. Mitogen-activated protein kinases in innate immunity[J]. Nature Reviews. Immunology,13(9):679-692.
Bolstad B M,Irizarry R A,Astrand M,Speed T P. 2003. A comparison of normalization methods for high density oligonucleotide array data based on variance and bias[J]. Bioinformatics,19(2):185-193.
Bovolenta L A,Acencio M L,Lemke N. 2012. HTRIdb:An open-access database for experimentally verified human transcriptional regulation interactions[J]. BMC Genomics,13:405. doi: 10.1186/1471-2164-13-405.
Boyle A P,Araya C L,Brdlik C,Cayting P,Cheng C,Cheng Y,Gardner K,Hillier L W,Janette J,Jiang L,Kasper D,Kawli T,Kheradpour P,Kundaje A,Li J J,Ma L,Niu W,Rehm E J,Rozowsky J,Slattery M,Spokony R,Terrell R,Vafeados D,Wang D,Weisdepp P,Wu Y C,Xie D,Yan K K,Feingold E A,Good P J,Pazin M J,Huang H,Bickel P J,Brenner S E,Reinke V,Waterston R H,Gerstein M,White K P,Kellis M,Snyder M. 2014. Comparative analysis of regulatory information and circuits across distant species[J]. Nature,512(7515):453-456.
Croft M,Siegel R M. 2017. Beyond TNF:TNF superfamily cytokines as targets for the treatment of rheumatic disea-ses[J]. Nature Reviews. Rheumatology,13(4):217-233.
Essaghir A,Demoulin J B. 2012. A minimal connected network of transcription factors regulated in human tumors and its application to the quest for universal cancer biomarkers[J]. PLoS One,7(6):e39666.
Essaghir A,Toffalini F,Knoops L,Kallin A,van Helden J,Demoulin J B. 2010. Transcription factor regulation can be accurately predicted from the presence of target gene signatures in microarray gene expression data[J]. Nucleic Acids Research,38(11):e120.
Green D S,Young H A,Valencia J C. 2017. Current prospects of type II interferon gamma signaling and autoimmunity[J]. The Journal of Biological Chemistry,292(34):13925-13933.
Gui J,Greene C S,Sullivan C,Taylor W,Moore J H,Kim C. 2015. Testing multiple hypotheses through IMP weighted FDR based on a genetic functional network with application to a new zebrafish transcriptome study[J]. BioData Mining,8:17. doi: 10.1186/s13040-015-0050-8.
Han H,Cho J W,Lee S,Yun A,Kim H,Bae D,Yang S,Kim C Y,Lee M,Kim E,Lee S,Kang B,Jeong D,Kim Y,Jeon H N,Jung H,Nam S,Chung M,Kim J H,Lee I. 2018. TRRUST v2:An expanded reference database of human and mouse transcriptional regulatory interactions [J]. Nucleic Acids Research,46(D1):D380-D386.
Huang T,Huang X,Shi B,Yao M. 2019. GEREDB:Gene expression regulation database curated by mining abstracts from literature[J]. Journal of Bioinformatics and Computational Biology,17(4):1950024.
Huang T H,Uthe J J,Bearson S M,Demirkale C Y,Nettleton D,Knetter S,Christian C,Ramer-Tait A E,Wannemuehler M J,Tuggle C K. 2011. Distinct peripheral blood RNA responses to Salmonella in pigs differing in Salmonella shedding levels:Intersection of IFNG,TLR and miRNA pathways[J]. PLoS One,6(12):e28768.
Jones B. 2015. Gene expression:Layers of gene regulation[J]. Nature Reviews. Genetics,16(3):128-129.
Shojaie A,Michailidis G. 2010. Network enrichment analysis in complex experiments[J]. Statistical Applications in Genetics and Molecular Biology,9:Article22. doi: 10.2202/1544-6115.1483.
Subramanian A,Tamayo P,Mootha V K,Mukherjee S,Ebert B L,Gillette M A,Paulovich A,Pomeroy S L,Golub T R,Lander E S,Mesirov J P. 2005. Gene set enrichment analysis:A knowledge-based approach for interpreting genome-wide expression profiles[J]. Proceedings of the National Academy of Sciences of the United States of Ame-rica,102(43):15545-15550.
Sun S,Yu X. 2016. HMM-Fisher:Identifying differential methylation using a hidden Markov model and Fisher?s exact test[J]. Statistical Applications in Genetics and Molecular Biology,15(1):55-67.
Vallenet D,Calteau A,Dubois M,Amours P,Bazin A,Beuvin M,Burlot L,Bussell X,Fouteau S,Gautreau G,Lajus A,Langlois J,Planel R,Roche D,Rollin J,Rouy Z,Sabatet V,Médigue C. 2019. MicroScope:An integrated platform for the annotation and exploration of microbial gene functions through genomic,pangenomic and metabolic comparative analysis[J]. Nucleic Acids Research,48(D1):D579-D589.
Vemula P,Jing Y,Zhang H,Hunt J B Jr,Sandusky-Beltran L A,Lee D C,Liu P. 2019. Altered brain arginine metabolism in a mouse model of tauopathy[J]. Amino Acids,51(3):513-528.
Wachter A. 2014. Gene regulation by structured mRNA elements[J]. Trends Genetics,30(5):172-181.
Yamamoto Y,Hirose N,Kamimura S,Wakayama S,Ito J,Ooga M,Wakayama T. 2019. Production of mouse offspring from inactivated spermatozoa using horse PLCζ mRNA[J]. The Journal of Reproduction and Development,66(1):67-73.
Yin C,Liu B,Wang P,Li X,Li Y,Zheng X,Tai Y,Wang C,Liu B. 2019. Eucalyptol alleviates inflammation and pain responses in a mouse model of gout arthritis[J]. British Journal of Pharmacology,1779(9):2042-2057.
Zhao Y,Chen J,Freudenberg J M,Meng Q,Rajpal D K,Yang X. 2016. Network-based identification and prioritization of key regulators of coronary artery disease loci[J]. Arteriosclerosis,Thrombosis,and Vascular Biology,36(5):928-941.
(責任编辑 兰宗宝)
