云南黄毛草莓的nrDNAITS和cpDNApsbA-trnH序列分子鉴定及进化特征分析
2020-06-04杨俊誉魏世杰苏代发陈杉艳罗志伟沈雪梅ArslanJamil童江云崔晓龙
杨俊誉 魏世杰 苏代发 陈杉艳 罗志伟 沈雪梅 ArslanJamil 童江云 崔晓龙

摘要:【目的】利用核糖體DNA(nrDNA)ITS序列和叶绿体DNA(cpDNA)psbA-trnH序列对云南不同地理区域的黄毛草莓进行分子鉴定,并分析不同地理居群间的分子进化及地理分布特征,为黄毛草莓种质鉴定、保护及开发利用提供理论依据。【方法】以云南省12个不同地理区域的黄毛草莓居群样品为材料,PCR扩增其ITS和psbA-trnH序列,并进行双向测序及序列合并,分别基于ITS和psbA-trnH序列及二者合并序列构建系统发育进化树。最后利用DnaSP 5.10对ITS和psbA-trnH序列进行核苷酸多样性(π)及单倍型数目、类型和多样性(Hd)分析,并对黄毛草莓居群进行中性检验及分子进化特征分析。【结果】基于ITS和psbA-trnH序列的聚类分析结果均显示,12个不同地理区域的黄毛草莓居群样品均与GenBank数据库中下载的黄毛草莓聚在一个分支上,分支自展值均大于阈值(75%),表明这两种序列均可用于黄毛草莓种质的分子鉴定。基于二者合并序列的聚类分析结果与上述结果基本一致,但分支自展值达99%,表明该聚类分析结果更可靠。不同地理区域的黄毛草莓ITS序列间的遗传距离为0~0.014,排序为迪庆州>昆明市>文山州,psbA-trnH序列间的遗传距离为0~0.025,排序为昆明市>迪庆州>文山州,推测ITS和psbA-trnH序列均能显示黄毛草莓居群遗传分化与地理分布格局的相关性。ITS序列长度为668 bp,变异位点百分率为1.8%,共有8种单倍型,Hd和π分别为0.894±0.078和0.006,以昆明市黄毛草莓样品的单倍型最多,为4种;psbA-trnH序列有203 bp,变异位点百分率为2.5%,psbA-trnH序列共有5种单倍型,Hd和π分别为0.788±0.090和0.009,以昆明市黄毛草莓样品单倍型最多,为3种,表明两种序列的单倍型均呈现地理分布格局。黄毛草莓ITS和psbA-trnH序列的中性检验Tajimas D值分别为-0.673和0.227(P>0.1),表明云南省12个不同地理区域的黄毛草莓居群保持稳定状态,在截至目前的历史时间内不存在扩张。【结论】从云南不同地理区域采集的样品均为黄毛草莓。ITS序列和psbA-trnH序列均可作为黄毛草莓的DNA条形码,二者的合并序列更能准确鉴定黄毛草莓种,适用于黄毛草莓分子谱系地理学研究。
关键词: 黄毛草莓;ITS序列;psbA-trnH序列;分子鉴定;进化特征
Abstract:【Objective】The molecular identification of Fragaria nilgerrensis samples in different geographical regions of Yunnan was performed by using ribosomal DNA(nrDNA) ITS sequences and chloroplast DNA(cpDNA) psbA-trnH sequences. At the same time,the characteristics of molecular evolution and geographical distribution in different geographi-cal populations were analyzed,which provided with a theoretical basis on molecular identification,protection,and exploitation and utilization of F. nilgerrensis germplasm resources. 【Method】Taking samples of F. nilgerrensis populations from 12 different geographical regions in Yunnan as the research object. Subsequently, PCR was used to amplify the ITS and psbA-trnH sequences from genomic DNAs of these samples,and performed bidirectional sequencing and sequence combination. Then phylogenetic trees were constructed based on ITS and psbA-trnH sequences and their combined sequences,respectively. Finally, the DnaSP ver5.10 software was used to analyze the nucleotide diversity (π) and the number/type/diversity (Hd) of haplotype for ITS and psbA-trnH sequences. At the same time, the neutrality test and the molecular evolution characteristics of F. nilgerrensis populations were analyzed. 【Result】The clustering analysis results based on ITS and psbA-trnH sequences showed that all samples of the F. nilgerrensis populations from 12 different geographical regions were clustered on one branch with the samples from GenBank database,and bootstrap value in this branch was higher than the threshold of 75%,indicating that both sequences could be used for molecular identification of this species germplasm (resources). The results of the cluster analysis based on the combination sequence of above DNA markers were consistent with the above results,the bootstrap value reached 99%,indicating that the cluster analysis results were more scientific and accurate. The genetic distance between ITS sequences of the F. nilgerrensis populations from different geographical regions was 0-0.014,the order of genetic distance in different regions was Diqing>Kunming>Wenshan. The genetic distance between psbA-trnH sequences was 0-0.025,the order of genetic distance in different regions was Kunming>Diqing>Wenshan. Therefore, it was speculated that both the ITS and psbA-trnH sequences could show the correlation between the genetic differentiation and geographical distribution pattern of F. nilgerrensis populations. The length of the ITS sequence was 668 bp,the mutation rate was 1.8%,the number of haplotypes was eight. Furthermore, the values of Hd and π were 0.894±0.078 and 0.006,respectively. Kunming had the most haplotypes from ITS sequences, with four haplotypes;while the psbA-trnH sequence was 203 bp,the mutation rate was 2.5%,these sequences have five haplotypes,the values of Hd and π were 0.788±0.090 and 0.009,respectively. Kunming had the most haplotypes from psbA-trnH sequences, with three haplotypes. Therefore, the results showed that the haplotypes of both ITS and psbA-trnH sequences presented a specific correlation between evolutionary characteristics and geographical distribution of F. nilgerrensis. The Tajimas D value (neutral test) of the ITS and psbA-trnH sequences were -0.673 and 0.227(P>0.1),respectively. It showed that the populations of F. nilgerrensis from 12 different geographical regions in Yunnan Province were stable and did not experience a recent population expansion event. 【Conclusion】All samples collected from different geographical regions of Yunnan are the species of F. nilgerrensis. The ITS sequence and the psbA-trnH sequence can be used as the DNA barcode,their combined sequence of these DNA barcodes can more accurately identify this species, and both of them are suitable for the molecular phylogeographic study of F. nilgerrensis.
0 引言
【研究意义】黄毛草莓(Fragaria nilgerrensis Schltdl. ex J. Gay)为蔷薇科草莓属多年生草本植物,具有清热解毒、消炎、祛风止咳等功效,可用于口腔破溃、痢疾、跌打损伤、毒蛇咬伤等症,是我国自然条件下分布最广的野生二倍体草莓(邓明琴和雷家军,2005;江纪武,2005;雷家军等,2006),其中,云南省黄毛草莓种质资源极其丰富(中国科学院昆明植物研究所,2006;赵密珍等,2010;杨雷等,2018)。目前,对草莓属种质鉴定主要依据植株的形态学分类,但形态学分类通常易受主观因素影响,导致无法准确鉴定,从而影响其多样性保护及利用(邓明琴和雷家军,2005;任保青和陈之端,2010;刘宇婧等,2011)。从分子水平能更加准确地对黄毛草莓进行种质鉴定及不同地理区域的居群遗传变异分析,对其药用价值和种质资源开发、利用及保护具有重要意义。【前人研究进展】随着分子技术的发展,国际生物条形码联盟(CBOL)的植物工作小组建议使用叶绿体DNA(cpDNA)和核糖体DNA(nrDNA)ITS序列作为植物研究的DNA条形码(宁淑萍等,2008;China Plant BOL Group et al.,2011),其中ITS或ITS2序列被中国DNA条形码植物工作组确定为种子植物的核心条形码(China Plant BOL Group et al.,2011),同时多种序列片段组合使用也被广泛用于植物物种的鉴定(Fazekas et al.,2008;Hollingsworth et al.,2011)。2015年版的《中国药典》将ITS2和cpDNA psbA-trnH序列作为植物类中药材物种鉴定的DNA条形码(林爽等,2017),已广泛用于何首乌属(白明明等,2012)、石斛属(彭小凤等,2015)和黄芩属(吕泽良等,2017)等中药材的分子鉴定。目前,国内外关于黄毛草莓的研究主要集中在组织培养(余桂红等,2002;王燕等,2012)、植株形态学鉴定(邓明琴和雷家军,2005;李洪雯等,2012)、核型分析(赵密珍等,2010;陈丙义等,2015)、植株胁迫(Na et al.,2014)、营养成分(李桂香等,2017)和生理生化(徐苏婷等,2018)等方面,对其进行分子鉴定和遗传进化分析的文献较少。Kress等(2005)利用ITS和psbA-trnH的合并序列对有花植物进行分子鉴定,结果发现利用这2种序列可有效识别和鉴定有花植物;Njuguna(2010)使用ITS和psbA-trnH序列对野生草莓进行分子鉴定,结果发现psbA-trnH序列对黄毛草莓具有很好的鉴别能力(Whitaker,2011)。【本研究切入点】目前,鲜见利用ITS和psbA-trnH序列对云南黄毛草莓种质进行分子鉴定及进化特征分析研究的相关报道。【拟解决的关键问题】以云南省不同地理区域的黄毛草莓居群样品为研究对象,PCR扩增其ITS和psbA-trnH序列,并進行双向测序及序列合并,分别基于ITS和psbA-trnH序列及二者合并序列构建系统发育进化树,最后对黄毛草莓居群进行中性检验及分子进化特征分析,为黄毛草莓种质的鉴定、保护及开发利用提供理论依据。
1 材料与方法
1. 1 试验材料
供试材料为云南省12个不同地理区域的黄毛草莓居群样品(表1)。主要试剂:E.Z.N.A.TM HP Plant DNA Kit(Omega,美国)植物基因组提取试剂盒购自昆明硕阳科技有限公司;Green Taq Mix购自昆明赞纳生物科技有限公司。主要仪器设备:MyCycler普通PCR仪(Bio-Rad,美国)、Vortex-Genie? 2 Vortex振荡仪(MoBio,美国)、小型高速离心机(Thermo,美国)、Syngene G:BOX全自动凝胶成像系统(Syngene,英国)和DYY-12型电泳仪(北京市六一仪器厂)。
1. 2 样品采集
采集样品时,从每个地理区域随机选取10株不同植株个体,将新鲜植株叶片装于50 mL灭菌管中,4 ℃保存并迅速运回实验室,于-80 ℃保存备用。
1. 3 DNA提取
利用液氮和研钵对供试材料进行研磨,并利用E.Z.N.A.TM HP Plant DNA Kit植物基因组提取试剂盒提取其DNA,经1.5%琼脂糖凝胶电泳检测后于 -20 ℃保存备用。
1. 4 PCR扩增
ITS序列的扩增引物为ITS5a(5'-CCTTATCAT TTAGAGGAAGGAG-3')和ITS4(5'-TCCTCCGCT TATTGATATGC-3')(Kress et al.,2005),psbA-trnH序列的扩增引物为psbA3'f(5'-GTTATGCATGAAC GTAATGCTC-3')和trnHf(5'-CGCGCATGGTGGAT TCACAATCC-3')(Hollingsworth et al.,2011)。PCR反应体系:Green Taq Mix 25.0 μL,DNA模板10 ng,10 μmol/L正、反向引物各2.0 μL,ddH2O补足至50.0 μL。扩增程序:94 ℃预变性3 min;94 ℃ 30 s,50 ℃ 45 s,72 ℃ 1 min,进行35个循环;72 ℃延伸7 min。PCR产物用1.5%琼脂糖凝胶电泳检测后送至北京擎科新业生物技术有限公司进行双向测序(正、反向测通)。
1. 5 统计分析
使用DNAstar对测序峰图进行手工校对,去除两端不可靠序列,并将正向和反向序列进行拼接。利用MEGA 6.0对拼接好的序列进行核苷酸组成分析,并基于K2P算法模型和GenBank中已知的野生草莓种序列,通过相邻算法(NJ)分别构建ITS和psbA-trnH序列的系统发育进化树,并对其进行1000次重复的自展(Bootstrap)检验评价(宁淑萍等,2008;姜玉素等,2019);应用SequenceMatrix 1.78将ITS和psbA-trnH序列进行合并,并利用PAUP 4.0对合并序列进行同质性检验(Vaidya et al.,2011),最后利用MEGA 6.0构建基于合并序列的系统发育进化树。此外,利用DnaSP 5.10对ITS和psbA-trnH序列进行核苷酸多样性(π)及单倍型(Haplotype,Hap)数目、类型和多样性(Hd)分析,并对黄毛草莓居群进行中性检验及分子进化特点分析(Librado and Rozas,2009)。
2 结果与分析
2. 1 ITS和psbA-trnH序列的聚类分析结果
2. 1. 1 基于ITS序列的聚类分析结果 对12个不同地理区域的黄毛草莓居群样品ITS序列进行PCR扩增及测序,将ITS序列上传NCBI获取登录号,并从GenBank数据库中下载已发表的10条野生草莓种的有效ITS序列进行聚类分析,构建的系统发育进化树如图1所示。12个不同地理区域的黄毛草莓居群样品ITS序列均与GenBank数据库中下載的黄毛草莓ITS序列聚在一个分支上,该分支的自展值为90%,远大于阈值(75%),说明基于ITS序列的聚类分析结果可靠,本研究采集的样品均为黄毛草莓,与植株形态分类的结果一致。
不同地理区域的黄毛草莓居群样品ITS序列间的遗传距离如表2所示。T45与T17-23、T45与T17-46-2间的遗传距离最远,均为0.014,T17与T18、T19和T20间的遗传距离最近,均为0。采自昆明市的4个样品ITS序列(T1、T5、T6和T7)间的遗传距离为0.004±0.002,采自文山州的4个样品ITS序列(T17、T18、T19和T21)间的遗传距离为0,采自迪庆州的4个样品ITS序列(T45、T46、T17-23和T17-46-2)间的遗传距离为0.009±0.003,说明迪庆州黄毛草莓ITS序列间的遗传距离高于昆明市和文山州黄毛草莓ITS序列间的遗传距离,推测ITS序列能阐明黄毛草莓居群的遗传分化与地理分布格局的相关性。
2. 1. 2 基于psbA-trnH序列的聚类分析结果 将psbA-trnH序列上传NCBI获取登录号,并从GenBank数据库中下载已发表的10条野生草莓种的有效psbA-trnH序列(其所对应的野生草莓种的类型和数量与选取的10条有效ITS序列一致)进行聚类分析,结果如图2所示。12个不同地理区域的黄毛草莓居群样品psbA-trnH序列均能与GenBank数据库中下载的黄毛草莓psbA-trnH序列聚在一个分支上,该分支的自展值为85%,远大于阈值(75%),说明基于psbA-trnH序列的聚类分析结果可靠,且基于psbA-trnH序列的聚类结果与基于ITS序列的聚类结果一致,均将采集的样品鉴定为黄毛草莓,与植株形态分类的结果一致。
不同地理区域的黄毛草莓居群样品psbA-trnH序列间的遗传距离如表3所示。P1与P17、P1与P19、P1与P17-23、P1与P17-46-2间的遗传距离最远,均为0.025;而P5与P6、P46与P17-23、P46与P17-46-2、P17-23与P17-46-2间的遗传距离最近,均为0。采自昆明市的4个样品psbA-trnH序列(P1、P5、P6和P7)间的遗传距离为0.010±0.005,采自文山州的4个样品psbA-trnH序列(P17、P18、P19和P21)间的遗传距离为0.003±0.003,采自迪庆州的4个样品psbA-trnH序列(P45、P46、P17-23和P17-46-2)间的遗传距离为0.005±0.004,说明不同地理区域的黄毛草莓psbA-trnH序列间遗传距离排序为昆明市>迪庆州>文山州。与ITS序列间的遗传距离相比,两种序列均反映出文山州的黄毛草莓样品间遗传距离最小,且psbA-trnH序列也可较好地反映文山州的黄毛草莓样品遗传差异。因此,两种序列共同使用能更好地解释黄毛草莓居群的遗传分化与地理分布格局的相关性。
2. 2 基于ITS与psbA-trnH的合并序列的聚类分析结果
利用SequenceMatrix 1.78分别将12个不同地理区域的黄毛草莓居群样品ITS与psbA-trnH序列及从GenBank数据库中下载的10条野生草莓种的ITS与psbA-trnH序列进行合并,利用PAUP 4.0对合并序列进行同质性检验,结果发现合并序列的同质性检验(P=0.01)满足合并阈值,说明所有序列能进行合并分析。基于合并序列使用MEGA 6.0构建系统发育进化树,结果如图3所示。将ITS和psbA-trnH序列合并后,12个不同地理区域的黄毛草莓居群样品均与GenBank数据库中下载的黄毛草莓聚在同一分支,该分支的自展值为99%,远大于阈值(75%)及基于ITS(自展值为90%)和psbA-trnH(自展值为85%)分别构建的系统发育进化树自展值,且聚类结果与分别基于ITS和psbA-trnH序列的聚类分析结果一致。因此,今后可使用多种序列组合的DNA条形码,并结合植株形态学特征进行草莓种质鉴定,以保证鉴定的准确性。
2. 3 ITS序列特征及单倍型分析结果
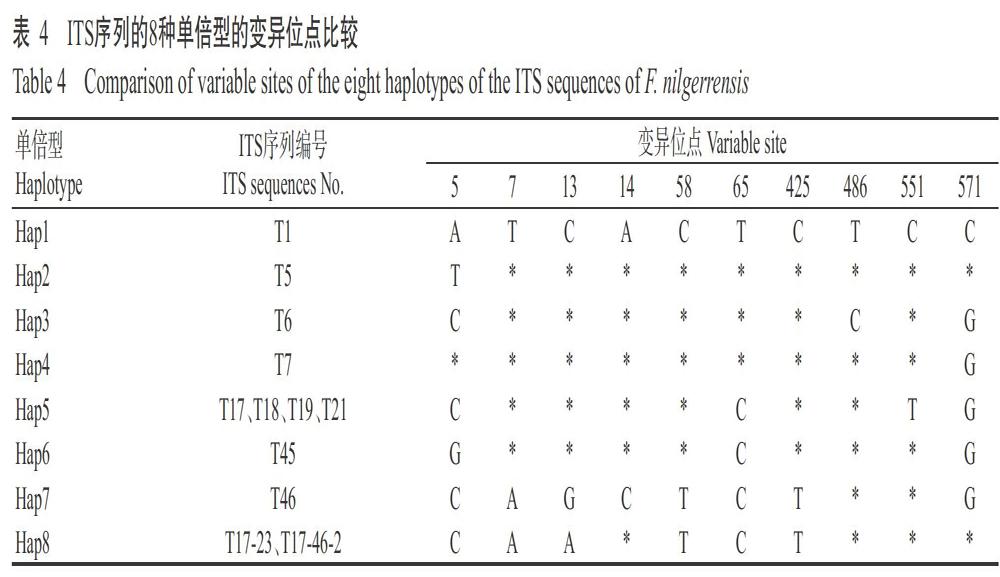
利用MEGA 6.0对所有的黄毛草莓ITS序列进行比对,对位排列后得到668 bp的恒定序列,其中包括649个保守位点、10个变异位点、7个简约信息位点和9处插入/缺失位点,变异位点百分率为1.80%,G+C含量为63.2%~63.8%。其中,9处插入/缺失位点分别位于ITS序列的2、5、6、8、12、13、14、20和665 bp位点。ITS序列共有8种单倍型(表4),Hd为0.894±0.078,π为0.006。8种单倍型中,Hap5分布于文山州广南县(T17)、文山州富宁县(T18)、文山州砚山县(T19)和文山州马关县(T21)的黄毛草莓样品中,Hap8分布于迪庆州维西县保和镇(T17-23)和迪庆州维西县白济汛乡(T17-46-2)的黄毛草莓样品中,其余单倍型均为私有单倍型。Hap1只存在于昆明市嵩明县的黄毛草莓样品(T1)中,Hap2只分布于昆明市盘龙区的黄毛草莓样品(T2)中,Hap3只分布于昆明市富民县的黄毛草莓样品(T3)中,Hap4只分布于昆明市禄劝县的黄毛草莓样品(T4)中,Hap6只分布于迪庆州德钦县的黄毛草莓样品(T45)中,Hap7只分布于迪庆州香格里拉市的黄毛草莓样品(T46)中(表4)。综上所述,昆明市黄毛草莓样品存在4种单倍型(Hap1、Hap2、Hap3和Hap4),文山州黄毛草莓样品存在1种单倍型(Hap5),迪庆州黄毛草莓样品存在3种单倍型(Hap6、Hap7和Hap8)。
2. 4 psbA-trnH序列特征及单倍型分析结果
利用MEGA 6.0对所有的黄毛草莓psbA-trnH序列进行比对,对位排列后得到203 bp的恒定序列,其中包括198个保守位点、5个变异位点和3个简约信息位点,无插入/缺失位点,变异位点百分率为2.5%,G+C含量为18.7%~19.7%。psbA-trnH序列共有5种单倍型(表5),Hd为0.788±0.090,π为0.009。Hap4分布于文山州广南县(P17)、文山州砚山县(P19)、迪庆州维西县保和镇(P17-23)、迪庆州香格里拉县(P46)和迪庆州维西县白济汛乡(P17-46-2)的黄毛草莓样品;Hap3分布于昆明市禄劝县(P7)、文山州富宁县(P18)和文山州马关县(P21)的黄毛草莓样品;Hap2分布于昆明市盘龙区(P5)和昆明市富民县(P6)的黄毛草莓样品;Hap1仅分布于昆明市嵩明县(P1)的黄毛草莓样品,Hap5仅分布于迪庆州德钦县(P45)的黄毛草莓样品(表5)。综上所述,psbA-trnH序列的5种单倍型中,单倍型Hap4的黄毛草莓样品数量最多,昆明市黄毛草莓样品存在3种单倍型(Hap1、Hap2和Hap3),文山州黄毛草莓样品存在2种单倍型(Hap3和Hap4),迪庆州黄毛草莓样品存在2种单倍型(Hap4和Hap5)。
2. 5 ITS序列和psbA-trnH序列的进化特征分析结果
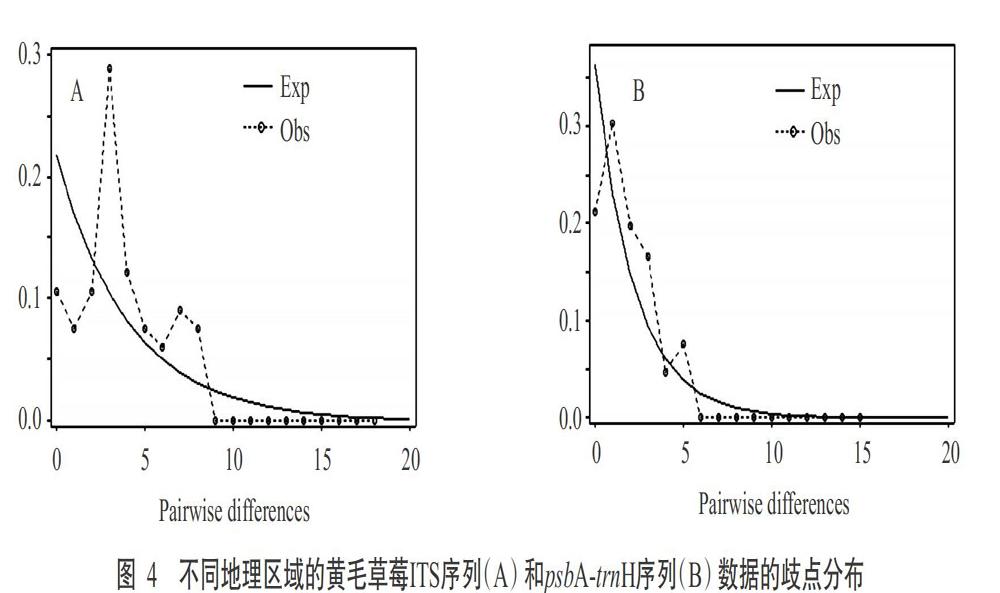
运用DnaSP 5.10分别对黄毛草莓ITS和psbA-trnH序列进行中性检验(Tajimas D),检测不同地理区域的黄毛草莓居群稳定性,并分析序列中分离位点数目与核苷酸多样性之间的关系。其中,黄毛草莓ITS序列的Tajimas D值为-0.673(P>0.1),表明12个不同地理区域的黄毛草莓居群保持稳定状态,在截至目前的历史时间内不存在扩张。黄毛草莓ITS序列的歧点分布情况如图4-A所示。与期望值(Exp)相比,观测值(Obs)曲线呈现多峰,说明居群在截至目前的历史时间内不存在扩张或持续增长,与中性检验结论一致。
黄毛草莓psbA-trnH序列的Tajimas D值为0.227(P>0.1),推测12个不同地理区域的黄毛草莓居群保持稳定状态,在截至目前的历史时间内不存在扩张。黄毛草莓psbA-trnH序列的歧点分布情况如图4-B所示。与期望值(Exp)相比,观测值(Obs)曲线呈现多峰,得到的曲线呈现多峰,说明居群在历史时间内不存在扩张或持续增长,与中性检验结论一致。
可见,云南省不同地理区域的黄毛草莓居群保持稳定状态,在截至目前的历史时间内不存在扩张。
3 讨论
3. 1 黄毛草莓的分子鉴定及DNA分子条形码
目前,国内学者主要依据传统植物形态学方法,从植株茎、叶、花和果等组织器官形态特征对野生草莓进行种质鉴定(代汉萍等,2007;赵密珍等2010)及种质资源收集(苏代发等,2018)。由于部分野生草莓种质的植株具有相似的形态学特征(雷家军等,2008)或采集时形态学指标收集不全(德庆措姆等,2012),导致无法准确地分类鉴定。本课题组在对云南省野生草莓进行收集时,也发现大部分野生草莓植株在没有花和果实的情况下很难通过植株形态学特征准确鉴定到种的水平。近年来,随着植物DNA分子技术的发展,传统植株形态学分类的瓶颈逐渐被攻克,不仅可快捷、准确地实现植物物种鉴定及分类,还可从分子水平探究植物的亲缘关系、系统发育进化及遗传多样性等。国外对野生草莓分子水平的研究起步较早,已有学者利用ITS和psbA-trnH序列对草莓属的栽培品种和野生种进行分子鉴定和系统发育研究(Eriksson et al.,1998;Potter et al.,2002;Njuguna,2010)。本研究首次使用ITS和psbA-trnH序列对云南省自然分布的野生黄毛草莓种质资源进行分子鉴定,并将供试样品的ITS和psbA-trnH序列进行合并,构建了系统发育进化树,结果显示,12个不同地理区域的黄毛草莓样品均与GenBank数据库中下载的黄毛草莓聚在同一分支,该分支的自展值为99%,远大于基于ITS(自展值为90%)和psbA-trnH(自展值为85%)分别构建的系统发育进化树自展值,表明两种序列合并可提高黄毛草莓分子鉴定结果的可信度。因此,ITS和psbA-trnH可作为黄毛草莓的DNA分子条形码。该结果与Potter等(2000)、Njuguna(2010)的研究结论相似。虽然本研究黄毛草莓分子鉴定结果与形态学分类鉴定结果一致,但分子鉴定更加快捷,不易受主观因素影响。
3. 2 黄毛草莓ITS和psbA-trnH序列的进化特征
韩柏明等(2012)、Meng等(2015)、徐苏婷(2017)分别利用SSR分子标记对草莓属不同种之间的亲缘关系和遗传多样性进行分析。随着分子测序技术的完善,现多用nrDNA和cpDNA的序列片段进行相关研究。本研究中12个不同地理区域的黄毛草莓样品ITS序列间遗传距离为0~0.014,从大到小排序为迪庆州(0.009±0.003)>昆明市(0.004±0.002)>文山州(0);12个不同地理区域的黄毛草莓居群样品psbA-trnH序列间的遗传距离为0~0.025,从大到小排序为昆明市(0.010±0.005)>迪庆州(0.005±0.004)>文山州(0.003±0.003),说明不同地理區域的黄毛草莓遗传特征存在差异,推测云南省黄毛草莓ITS和psbA-trnH序列的遗传特性与其地理分布具有一定的相关性。白明明等(2012)、许子欣等(2018)基于ITS序列和psbA-trnH序列分别对不同地理区域的巴戟天和何首乌进行遗传特性分析,结果也发现二者的遗传变异与地理分布存在一定的相关性。
12个不同地理区域的黄毛草莓居群样品ITS序列共有8个单倍型,其中昆明市存在4种单倍型(Hap1、Hap2、Hap3和Hap4),文山州仅存在1种单倍型(Hap5),迪庆州存在3种单倍型(Hap6、Hap7和Hap8);psbA-trnH序列共有5种单倍型,其中Hap4分布最广,昆明市存在3种单倍型(Hap1、Hap2和Hap3),文山州存在2种单倍型(Hap3和Hap4),迪庆州存在2种单倍型(Hap4和Hap5)。依据本研究两种序列的单倍型分布来看,云南省黄毛草莓区域性分布特征明显,不同地理区域具有自己独特的单倍型。该结果与柏国清等(2017)、林爽等(2017)和刘振等(2018)分别对秦巴山区漆树、破布叶和湖南茶树资源的研究结论一致。此外,本研究黄毛草莓ITS和psbA-trnH序列的歧点分布分析结果均表明,云南省12个不同地理区域的黄毛草莓居群保持稳定状态,在截至目前的历史时间内不存在扩张。推测其原因是由于云南省不同地理区域的地形地貌复杂,海拔差异显著、气候和环境因子异质性等特征为黄毛草莓提供了不同的生境条件,维持了黄毛草莓各自的遗传特性。也正因为云南省独特的地理气候资源,致使不同地理区域的黄毛草莓ITS序列和psbA-trnH序列存在丰富的遗传多样性。Njuguna等(2013)、Qiao等(2016)分别利用转录组学和基因组学技术研究发现草莓属植物可能起源于东亚地区,而本研究中使用的DNA条形码能较好地探讨黄毛草莓地理分布格局。因此,在今后的研究中,应加大各居群采样量,并使用多种分子标记共同探讨黄毛草莓谱系地理学的相关问题,为解释草莓属的分子进化和地理分布格局提供理论依据。
4 结论
从云南不同地理区域采集的样品均为黄毛草莓。ITS序列和psbA-trnH序列均可作为黄毛草莓的DNA条形码,二者合并序列更能准确鉴定黄毛草莓种,适用于黄毛草莓分子谱系地理学研究。
参考文献:
白明明,孙小芹,郭建林,李密密,杭悦宇. 2012. 基于psbA-trnH分析的何首乌野生居群遗传多样性[J]. 植物资源与环境学报,21(2):36-44. [Bai M M,Sun X Q,Guo J L,Li M M,Hang Y Y. 2012. Genetic diversity of wild populations of Faliopia multiflora based on psbA-trnH analysis[J]. Journal of Plant Resources and Environment,21(2):36-44.]
柏国清,李为民,陈昊,黎斌,李思锋. 2017. 秦巴山区漆树nrDNAITS和cpDNA序列分子进化特点的分析[J]. 植物研究,37(4):579-586. [Bai G Q,Li W M,Chen H,Li B,Li S F. 2017. Characteristics of molecular evolution of Toxicodendron vernicifluum in Qinba mountains by nrDNA ITS and cpDNA sequence[J]. Bulletin of Botanical Research,37(4):579-586.]
陈丙义,李金凤,霍恒志,万春雁,章镇,乔玉山,糜林. 2015. 6种野生草莓基因组大小估算[J]. 果树学报,32(1):51-56. [Chen B Y,Li J F,Huo H Z,Wan C Y,Zhang Z,Qiao Y S,Mi L. 2015. Estimation of genome size in six wild strawberry species[J]. Journal of Fruit Science,32(1):51-56.]
代汉萍,雷家军,邓明琴. 2007. 长白山野生草莓资源的调查与分类研究[J]. 园艺学报,34(1):63-66. [Dai H P,Lei J J,Deng M Q. 2007. Investigation and studies on classification of wild Fragaria spp. distributed in Changbai Mountains[J]. Acta Horticulturae Sinica,34(1):63-66.]
德庆措姆,格桑,薛莉,雷家军. 2012. 西藏野生草莓资源的地理分布初报[J]. 中国果树,(4):22-24. [De Q C M,Ge S,Xue L,Lei J J. 2012. Preliminary report on geographical distribution of wild strawberry resources in Tibet[J]. China Fruit,(4):22-24.]
邓明琴,雷家军. 2005. 中国果树志·草莓卷[M]. 北京:中国林业出版社. [Deng M Q,Lei J J. 2005. Chinese fruit tree records·Strawberry[M]. Beijing:China Forestry Publi-shing House.]
韩柏明,赵密珍,王静,于红梅. 2012. 草莓属种质资源亲缘关系的SSR标记分析[J]. 园艺学报,39(12):2352-2360. [Han B M,Zhao M Z,Wang J,Yu H M. 2012. Phylogenetic relationships among Fragaria germplasm by SSR markers[J]. Acta Horticulturae Sinica,39(12):2352-2360.]
江纪武. 2005. 药用植物辞典[M]. 天津:天津科学技术出版社. [Jiang J W. 2005. Dictionary of medicinal plants[M]. Tianjin:Tianjin Science and Technology Press.]
姜玉素,李珍,王庆莲,赵密珍,宋志忠. 2019. 草莓KEA家族基因的克隆、鑒定及表达分析[J]. 江苏农业学报,35(2):391-399. [Jiang Y S,Li Z,Wang Q L,Zhao M Z,Song Z Z. 2019. Cloning,characterization and expression analy-sis of KEA family genes in strawberry[J]. Jiangsu Journal of Agricultural Sciences,35(2):391-399.]
雷家军,代汉萍,谭昌华,邓明琴,赵密珍,钱亚明. 2006. 中国草莓属(Fragaria)植物的分类研究[J]. 园艺学报,33(1):1-5. [Lei J J,Dai H P,Tan C H,Deng M Q,Zhao M Z,Qian Y M. 2006. Studies on the taxonomy of the strawberry(Fragaria) species distributed in China[J]. Acta Horticulturae Sinica,33(1):1-5.]
雷家军,代汉萍,赵密珍,吴伟民. 2008. 中国分布四倍体野生草莓的调查研究[J]. 果树学报,25(3):358-361. [Lei J J,Dai H P,Zhao M Z,Wu W M. 2008. Study on the te-traploid Fragaria species distributed in China[J]. Journal of Fruit Science,25(3):358-361.]
李桂香,吕建召,肖湘滇,沈发春. 2017. 云南野生黄毛草莓鲜果的营养成分分析[J]. 大理大学学报,2(6):64-67. [Li G X,Lü J Z,Xiao X D,Shen F C. 2017. Analysis of nutritional compositions of Yunnan wild Fragaria nilgerrensis Schlecht fresh fruit[J]. Journal of Dali University,2(6):64-67.]
李洪雯,刘建军,何健,关斌,王建辉,李旭锋,陈克玲. 2012. 四川及其周边地区野生草莓资源调查、收集与评价[J]. 植物遗传资源学报,13(6):946-951. [Li H W,Liu J J,He J,Guan B,Wang J H,Li X F,Chen K L. 2012. Investigation,collection and evaluation on wild strawberry resources in Sichuan and neighboring regions[J]. Journal of Plant Genetic Resources,13(6):946-951.]
林爽,吴海燕,张宏意,李坤平. 2017. 不同地理居群破布叶的psbA-trnH和ITS2序列及其聚类分析[J]. 中草药,48(7):1403-1408. [Lin S,Wu H Y,Zhang H Y,Li K P. 2017. Cluster analysis on psbA-trnH and ITS2 sequences of Microcos paniculata from different geographical populations[J]. Chinese Traditional and Herbal Drugs,48(7):1403-1408.]
刘宇婧,刘越,黄耀江,龙春林. 2011. 植物DNA条形码技术的发展及应用[J]. 植物资源与环境学报,20(1):74-82. [Liu Y J,Liu Y,Huang Y J,Long C L. 2011. Progress and application of DNA barcoding technique in plants[J]. Journal of Plant Resources and Environment,20(1):74-82.]
刘振,成杨,赵洋,杨培迪,杨阳. 2018. 基于叶绿体rbcL和trnH-psbA序列的湖南茶树资源遗传多样性与亲缘关系研究[J]. 热带作物学报,39(1):40-45. [Liu Z,Cheng Y,Zhao Y,Yang P D,Yang Y. 2018. Genetic diversity and relationship study of Hunan tea germplasm resources based on chloroplast rbcL and trnH-psbA sequence[J]. Chinese Journal of Tropical Crops,39(1):40-45.]
吕泽良,张永刚,韩梅,刘翠晶. 2017. 不同产地黄芩ITS2和psbA-trnH条形码序列分析[J]. 吉林农业大学学报,39(1):42-48. [Lü Z L,Zhang Y G,Han M,Liu C J. 2017. Sequence analysis of ITS2 and psbA-trnH DNA barcode of wild populations of Scutellaria baicalensis Georgi from different producing areas[J]. Journal of Jilin Agricultural University,39(1):42-48.]
宁淑萍,颜海飞,郝刚,葛学军. 2008. 植物DNA条形码研究进展[J]. 生物多样性,16(5):417-425. [Ning S P,Yan H F,Hao G,Ge X J. 2008. Current advances of DNA barcoding study in plants[J]. Biodiversity Science,16(5):417-425.]
彭小凤,何涛,淳泽,胡亚东,万闰兰. 2015. 基于叶绿体psbA-trnH和核糖体5S rRNA基因间隔区序列的石斛种间和种内鉴别[J]. 应用与环境生物学报,21(5):887-896. [Peng X F,He T,Chun Z,Hu Y D,Wan R L. 2015. Interspecific and intraspecific identification of Dendrobium based on the psbA-trnH intergenic region sequences and the 5S rRNA gene spacer sequences[J]. Chinese Journal of Applied & Environmental Biology,21(5):887-896.]
任保青,陈之端. 2010. 植物DNA条形码技术[J]. 植物学报,45(1):1-12. [Ren B Q,Chen Z D. 2010. DNA Barco-ding Plant Life[J]. Bulletin of Botany,45(1):1-12.]
苏代发,童江云,杨俊誉,陈杉艳,罗志伟,沈雪梅,赖泳红,Arslan Jamil,魏世杰,崔晓龙. 2018. 中国草莓属植物种质资源的研究、开发与利用進展[J]. 云南大学学报(自然科学版),40(6):1261-1276. [Su D F,Tong J Y,Yang J Y,Chen S Y,Luo Z W,Shen X M,Lai Y H,Arslan J,Wei S J,Cui X L. 2018. Advances in research,exploitation and utilization of Fragaria spp. germplasm resources in China[J]. Journal of Yunnan University(Natural Scien-ces Edition),40(6):1261-1276.]
王燕,陈丙义,章镇,高志红,乔玉山. 2012. 黄毛草莓组织培养与快繁技术研究[J]. 西南农业学报,25(1):252-256. [Wang Y,Chen B Y,Zhang Z,Gao Z H,Qiao Y S. 2012. Study on tissue culture and rapid propagation technique of Fragaria nilgerrensis[J]. Southwest China Journal of Agricultural Sciences,25(1):252-256.]
徐苏婷. 2017. 不同果色五叶草莓遗传结构的形成及机制[D]. 上海:上海师范大学. [Xu S T. 2017. Formation and mechanism of genetic structure of Fragaria pentaphylla with different fruit color[D]. Shanghai:Shanghai Normal University.]
徐苏婷,陈露茜,李钧敏. 2018. 二倍体与四倍体黄毛草莓的光合特性[J]. 江苏农业科学,46(5):110-112. [Xu S T,Chen L Q,Li J M. 2018. Study on photosynthetic characteristics of diploid and tetraploid Fragarianil gerrensis[J]. Jiangsu Agricultural Sciences,46(5):110-112.]
许子欣,冉志芳,杨小彤,郝庆秀,余意,周洁,郭兰萍. 2018. 基于psbA-trnH和rDNA ITS序列的不同地理居群巴戟天聚类分析[J]. 南方农业学报,49(12):2364-2370. [Xu Z X,Ran Z F,Yang X T,Hao Q X,Yu Y,Zhou J,Guo L P. 2018. Cluster analysis of Morinda officinalis How in di-fferent geographical populations based on psbA-trnH and rDNA ITS sequences[J]. Journal of Southern Agriculture,49(12):2364-2370.]
杨雷,李莉,董辉,季文章,杨莉. 2018. 应用灰色关联分析法筛选优良草莓新品种(系)[J]. 江西农业学报,30(12):47-50. [Yang L,Li L,Dong H,Ji W Z,Yang L. 2018. Screening of excellent strawberry new varieties(strains) by using grey relevant analysis method[J]. Acta Agriculturae Jiangxi,30(12):47-50.]
余桂红,马鸿翔,吴伟民,李晓刚,孟宪凤. 2002. 草莓屬野生种质资源的离体培养[J]. 湖北农学院学报,22(4):316-318. [Yu G H,Ma H X,Wu W M,Li X G,Meng X F. 2002. The in vitro culture of wild germplasm resources of strawberries[J]. Journal of Hubei Agricultural College,22(4):316-318.]
赵密珍,王壮伟,陶磅,钱亚明,王静,仇明华. 2010. 滇西北野生草莓资源考察研究初报[C]//第四届全国果树种质资源研究与开发利用学术研讨会论文汇编. 北京:园艺学会,136-138. [Zhao M Z,Wang Z W,Tao P,Qian Y M,Wang J,Qiu M H. 2010. Preliminary study on wild strawberry resources in northwest Yunnan[C]//Collection of papers of the 4th national symposium on research,development and utilization of fruit germplasm resources. Beijing:Horticultural Society,136-138.]
中国科学院昆明植物研究所. 2006. 云南植物志[M]. 第十二卷. 北京:科学出版社. [Kunming Institute of Botany,Chinese Academy of Sciences. 2006. Flora of Yunnan. Volume 12[M]. Beijing:Science Press.]
China Plant BOL Group,Li D Z,Gao L M,Li H T,Wang H,Ge X J,Liu J Q,Chen Z D,Zhou S L,Chen S L,Yang J B,Fu C X,Zeng C X,Yan H F,Zhu Y J,Sun Y S,Chen S Y,Lei Z,Wang K,Yang T,Duan G W. 2011. Comparative analysis of a large dataset indicates that internal transcribed spacer(ITS) should be incorporated into the core barcode for seed plants[J]. Proceedings of the National Academy of Sciences,108(49):19641-19646.
Eriksson T,Donoghue M J,Hibbs M S. 1998. Phylogenetic analysis of Potentilla using DNA sequences of nuclear ribosomal internal transcribed spacers(ITS),and implications for the classification of Rosoideae(Rosaceae)[J]. Plant Systematics and Evolution,211(3-4):155-179.
Fazekas A J,Burgess K S,Kesanakurti P R,Graham S W,Newmaster S G,Husband B C,Percy D M,Hajibabaei M,Barrett S C. 2008. Multiple multilocus DNA barcodes from the plastid genome discriminate plant species equa-lly well[J]. PloS One,3(7):e2802.
Hollingsworth P M,Graham S W,Little D P. 2011. Choosing and using a plant DNA barcode[J]. PloS One,6(5):e19254.
Kress W J,Wurdack K J,Zimmer E A,Weigt L A,Janzen D H. 2005. Use of DNA barcodes to identify flowering plants[J]. Proceedings of the National Academy of Scien-ces,102(23):8369-8374.
Librado P,Rozas J. 2009. DnaSP v5:A software for comprehensive analysis of DNA polymorphism data[J]. Bioinformatics,25(11):1451-1452.
Meng F,Liu L,Peng M,Wang Z,Wang C,Zhao Y. 2015. Genetic diversity and population structure analysis in wild strawberry(Fragaria nubicola L.) from Motuo in Tibet Plateau based on simple sequence repeats(SSRs)[J]. Biochemical Systematics and Ecology,63:113-118.
Na Y W,Jeong H J,Lee S Y,Choi H G,Kim S H,Rho I R. 2014. Chlorophyll fluorescence as a diagnostic tool for abiotic stress tolerance in wild and cultivated strawberry species[J]. Horticulture,Environment and Biotechnology,55(4):280-286.
Njuguna W. 2010. Development and use of molecular tools in Fragaria[D]. Corvallis:Oregon State University.
Njuguna W,Liston A,Cronn R,Ashman T L,Bassil N. 2013. Insights into phylogeny,sex function and age of Fraga-ria based on whole chloroplast genome sequencing[J]. Molecular Phylogenetics and Evolution,66(1):17-29.
Potter D,Gao F,Bortiri P E,Oh S H,Baggett S. 2002. Phylogenetic relationships in Rosaceae inferred from chloroplast matK and trnL-trnF nucleotide sequence data[J]. Plant Systematics and Evolution,231(1-4):77-89.
Potter D,Luby J J,Harrison R E. 2000. Phylogenetic relationships among species of Fragaria(Rosaceae) inferred from non-coding nuclear and chloroplast DNA sequences[J]. Systematic Botany,25(2):337-349.
Qiao Q,Xue L,Wang Q,Sun H,Zhong Y,Huang J J,Lei J J,Zhang T C. 2016. Comparative transcriptomics of strawberries(Fragaria spp.) provides insights into evolutio-nary patterns[J]. Frontiers in Plant Science,7:1839. doi: 10.3389/fpls.2016.01839. eCollection 2016.
Vaidya G,Lohman D J,Meier R. 2011. SequenceMatrix:Concatenation software for the fast assembly of multi-gene datasets with character set and codon information[J]. Cladistics,27(2):171-180.
Whitaker V M. 2011. Applications of molecular markers in strawberry[J]. Journal of Berry Research,1(3):115-127.
(責任编辑 陈 燕)
