志贺氏菌CRISPR位点的比较基因组学及与质粒数量的相关性分析
2020-05-24曲道峰陆诗铫陈跃文黄东萍龚俏玲易松强韩剑众
曲道峰 陆诗铫 陈跃文 黄东萍 龚俏玲 易松强 韩剑众*
(1 浙江工商大学食品与生物工程学院 杭州310018 2 江西省畜牧技术推广站 南昌330046
成簇的规律间隔的短回文重复序列(CRISPR)广泛分布于细菌和古生菌的基因组中,它是由RNA 介导的、可遗传的获得性免疫系统,与细菌水平基因转移关系密切,可以识别并剪切加工质粒等外源遗传物质,进而降低基因的水平转移。CRISPR 位点由重复序列和间隔序列组成,与前导序列、Cas 蛋白基因共同构成了CRISPR 系统[1]。重复序列保守性高,具有回文结构,能转录并形成RNA 二级结构,可以形成茎环结构[2]。间隔序列是高度可变的,研究发现噬菌体、大质粒等外源性可移动遗传原件是主要来源。前导序列和重复序列相互连接,当细菌体内产生新的间隔序列时,可以识别该新序列并启动转录产生前crRNA(CRISPR RNAs-)。在CRISPR位点附近存在CRISPR 相关蛋白质(CRISPR-associated,Cas)基因,可以编码Cas 蛋白,CRISPR/Cas 系统有3 个类型,即CRISPR/Cas 系统类型Ⅰ、Ⅱ、Ⅲ,依据Cas蛋白基因的差别每个类型可细分为几个亚类[3]。Haft 等根据cas 基因的相似程度将其分为45 个家族,在每个CRISPR 位点中都存在cas1 和cas2家族基因,可用这两个家族基因作为分子标记来鉴定CRISPR 系统[4]。
肠杆菌科经常是医学和科学界关注的重点和研究的热点,作为影响公共健康的一个重要标志,在公共卫生领域中对细菌食源性病原体进行深入研究意义颇深[5]。志贺菌属于肠杆菌科,是细菌性痢疾的病原体,同时也是一种最古老的人类特异性病原菌,有研究发现志贺菌属在35 000-170 000年之前就已经进化,与人类的起源和发展关系密切[4]。志贺氏菌根据O 抗原可分为4 种类型,分别为痢疾志贺氏菌、福氏志贺氏菌、鲍氏志贺氏菌和宋内志贺氏菌[6]。在我国,福氏志贺氏菌比较常见,而基于CRISPR 系统特点的研究还比较少,且研究结果不一致[7]。本文主要比较和分析了40 株基因测序的志贺氏菌的CRISPR 位点,研究在不同志贺氏菌种中CRISPR 位点结构上的差异,对间隔序列与插入序列的同源性进行比较,判断CRISPR 位点与RNA 二级结构稳定性的关系[8]。
1 材料和方法
1.1 材料
从NCBI genome 数据库中获取40 株志贺氏菌的遗传信息和基因组全序列(http://www.ncbi.nlm.nih.gov/genome),其中包括6 株鲍氏志贺氏菌(Shigellaboydii),5株痢疾志贺氏菌(Shigelladysenteriae)、18 株福氏志贺氏菌(Shigellaflexneri)以及11 株宋内氏志贺氏菌(Shigellasonnie)。基因组序列及其GenBank 编号见表1。
表1 40 株志贺氏菌基因组序列信息汇总Table1 The information of the genome sequence of 40 strains of shigella
1.2 志贺氏菌中CRISPR 位点分析
在CRISPRs Database(http://crispr.i2bc.parissaclay.fr/crispr/)网站上查找并获取40 株志贺氏菌CRISPR 的重复序列(repeat)和间隔序列(spacer),分析其CRISPR 位点的结构,同时在CRISPR Finder(http://crispr.i2bc.paris-saclay.fr/Server/)和CRT(CRISPR Recognition Tool)软件中进行查找预测[9]。
1.3 CRISPR 结构的核酸序列分析
将志贺氏菌的CRISPR 序列进行BLAST(https://blast.ncbi.nlm.nih.gov/Blast.cgi)线上比对,利用Clustal X 软件进行多序列比对,通过CRISPR BLAST 和CRISPR Finder 对志贺氏菌的CRISPR 位点的重复间隔序列进行对比分析,再利用WebLogo 将CRISPR1~3,CRISPR-Q1~Q4 的重复序列进行可视化。
1.4 间隔序列的同源序列查找
将各菌种的间隔区提交到IS Finder 和INTEGRALL 网站进行在线比对,通过CRISPRs Database 数据库比对,寻找已有的间隔序列中的外源同源序列,以及新发现的间隔序列,分析间隔区序列的同源序列是否来源于外源DNA。使用BLAST 和CRISPRTarget 来分析间隔序列。
1.5 CRISPR 位点与RNA 二级结构稳定性的相关性分析
预测CRISPR 重复序列的RNA 二级结构。通过RNAfold 网站对CRISPR 位点的重复序列进行RNA 二级结构预测,并研究重复间隔序列的基数差异CRISPR1~3,CRISPR Q1~Q4 是否直接影响RNA 二级结构的茎环稳定性。
1.6 CRISPR 位点的质粒相关性分析
在NCBI 网站上查询并下载相关菌株质粒的完整序列,菌株CRISPR 位点数量与菌株的质粒数量之间的关系采用非参数检验Wilcoxon rank sum test 进行比较分析。由Origin 8.5 软件完成相关的数据统计分析。
2 结果
2.1 志贺氏菌中CRISPR 结构位点的基因组分布情况
研究了40 株志贺氏菌并查找其CRISPR 位点,共发现CRISPR 位点241 个,其中可疑位点(Possible CRISPR)235 个,可确定位点(Confirmed CRISPR)6 个,且发现的绝大多数CRISPR 位点位于染色体上,只有2 个CRISPR 位点位于质粒上。经统计发现这40 株菌的平均CRISPR 个数是6个,其中6 株鲍氏志贺氏菌的平均CRISPR 位点个数是2.5 个,5 株痢疾志贺氏菌的平均个数是13.8 个,18 株福氏志贺氏菌的平均个数是5.5 个,以及11 株宋内氏志贺氏菌的平均个数是5.3 个。统计过程中还发现,痢疾志贺氏菌1617 这一菌株中的CRISPR 位点个数高达50 个,其中可疑位点49 个。而鲍氏志贺氏菌4444-74、痢疾志贺氏菌WRSD3、宋内氏志贺氏菌786_SSON 等菌株中均只含有一个CRISPR 位点。
成簇规律间隔的短回文重复序列和它的辅助蛋白(CRISPR-associated,Cas)构成了CRISPR/Cas 系统。cas 基因有45 个家族,对40 株志贺氏菌的CRISPR/Cas 系统进行分析,结果表明:该菌CRISPR/Cas 系统均属于Ⅰ-E 型,未发现Ⅱ类和Ⅲ类的Cas 系统。通过对241 个CRISPR 位点的结构研究,根据位点的相似性,该细菌的CRISPR 可分为8 类:CRISPR1,CRISPR2,CRISPR3,CRISPRQ1,CRISPR -Q2,CRISPR -Q3,CRISPR -Q4 和CRISPR-Q5。CRISPR1 位于核心基因cysD/iapcysH 和cysD/iap-cysH 之间,距离CRISPR2 约20 kb。CRISPR1-3 的Cas 结构具有较强的保守性。总的来说,在5 个 可疑CRISPR 中,CRISPR-Q1,CRISPR-Q2,CRISPR-Q4,CRISPR-Q5在大多数志贺氏菌中广泛分布。这5 个可疑CRISPR 序列包含一些独特的分隔符,暗示他们是保守的CRISPR。此外,还发现大多数的CRISPR 位点仅显示一个重复,CRISPR-Q5 在菌株中出现的次数并不多,故本文不作研究。
为了鉴定志贺氏菌中的cas 基因,从40 株志贺氏菌中选取9 株具有代表性的菌株,用CRISPR位点图来研究CRISPR1~3 的相关信息。结果发现这9 株菌都有CRISPR3 结构,除福氏志贺氏菌外其它3 类的CRISPR 位点都比较复杂,含有很多cas 基因和其它基因片段,也包含很多插入序列。由图1表明,cas 基因的排列顺序具有高度的一致性,其排列顺序为cas2-cas1-cas6-cas5-cas7-cse2-cse1-cas3。
2.2 重复序列与Cas 蛋白的进化分析
由表2可知,即使在同种细菌中的不同种类的菌株之间,CRISPR 位点重复序列的碱基数量和分布也有显著的区别,而且同类细菌中重复序列数也不存在规律性。比如鲍氏志贺氏菌中的15 个CRISPR 位点中共含有重复区域个数60 个,且仅有1 次重复。痢疾志贺氏菌的69 个CRISPR 位点中共包含重复序列个数157 个,其中重复数最高可达35 个。18 株福氏志贺氏菌的99 个CRISPR位点中共含有202 个重复序列个数,平均每株重复个数在11~15 个之间。11 株宋内志贺氏菌的58个CRISPR 位点中共含有137 个重复序列个数,重复个数16 个的菌株较多。
对40 株志贺氏菌的重复序列进行遗传聚类分析(图2a),发现不能对不同种细菌进行区分。9株菌株的CRISPR/Cas 系统中均含有cas3 基因,且是广泛分布的,其表达“R-环解旋酶-退火酶”。对9 株志贺氏菌进行遗传聚类分析(图2b),与重复序列相似,cas3 基因的保守性较强,不可区分同种细菌的不同菌株。

图1 9 株志贺氏菌CRISPR1~3 位点的分布Fig.1 The distribution of CRISPR1~3 of 9 Shigella strains

表2 不同CRISPR 位点重复序列的分析Table2 Analysis of the repeat sequence of different CRISPR loci
2.3 前导序列和间隔序列的特征分析
研究发现 40 株志贺氏菌的CRISPR 位点中存在516 条间隔序列,其序列长度呈高度统一,29 bp 长度的重复序列与32 bp 长度的间隔序列相匹配,27 bp 长度的重复序列与69 bp 长度的间隔序列相匹配,39 bp 长度的重复序列与49 bp 长度的间隔序列相匹配。将一个重复序列和一个与之相邻的间隔序列称为一个重复单元,统计表明40 株志贺氏菌中重复单元序列长度基本在100 bp 以下,因此大胆假设在CRISPR 位点附近存在这样的基因——它拥有能够严格调控重复单元长度的功能。
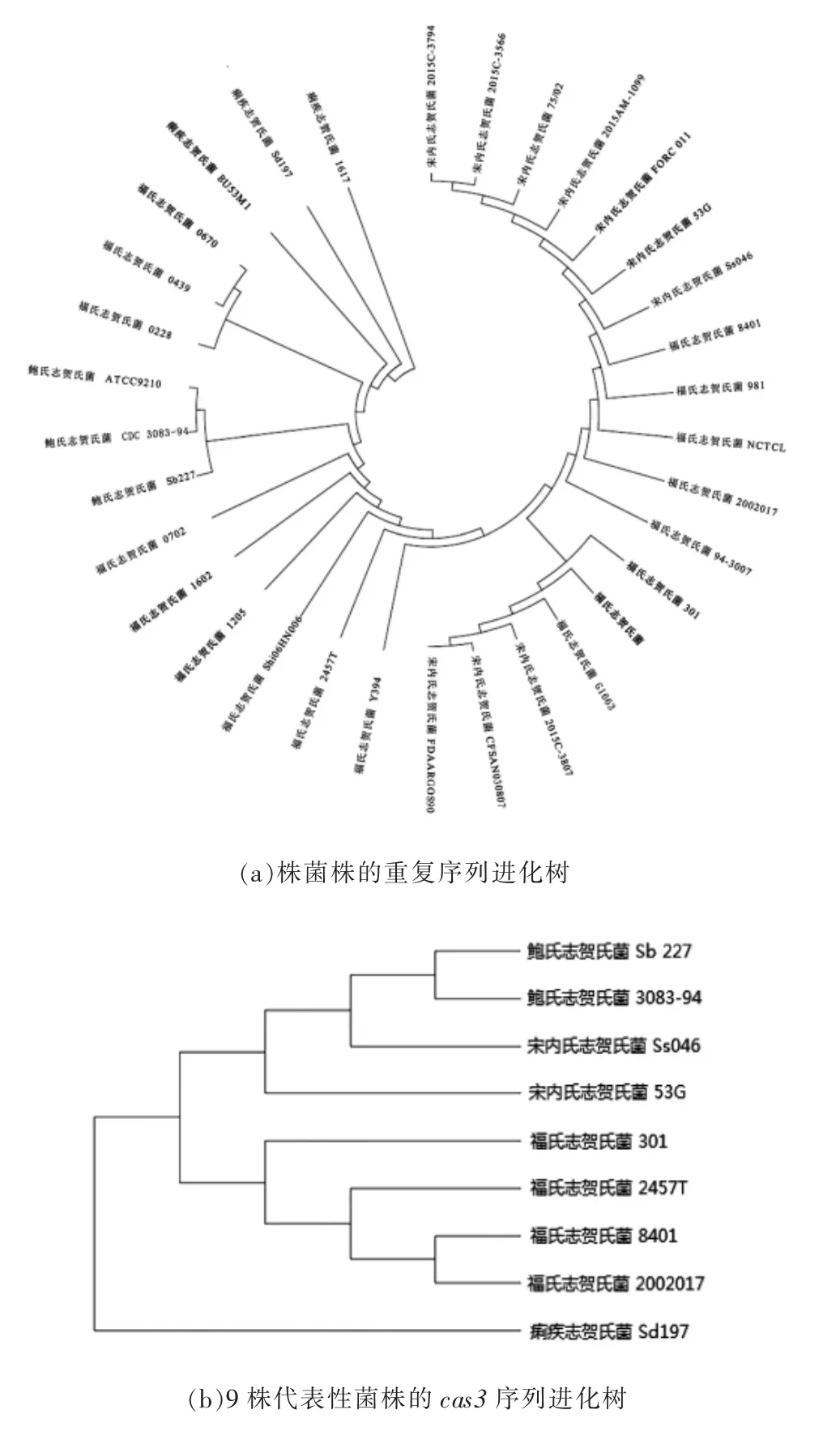
图2 志贺氏菌相关序列进化树Fig.2 The termination-associated sequences of repeats
在INTEGRALL 上对这些间隔序列比对分析,无法查找到完全对应的可移动遗传元件,而大部分间隔序列中存在长度为12~15 bp 的序列,可以发现与其相对应的来源于其它菌株的基因片段序列,比如质粒、转座子、整合子、插入序列、耐药基因等可移动基因元件[1]。在鲍氏志贺氏菌Sb227的CRISPR 中的间隔序列就有一段15 bp 的基因序列与克雷白氏肺炎杆菌中的质粒pRBDHA|pspB 有一定的同源性,在痢疾志贺氏菌BU53M1 的CRISPR 结构中的第2 个间隔序列就和硫碱弧菌属HL-EbGR7 的整合酶基因IntI 24 有一定的同源基因,在鲍氏志贺氏CDC 3083-94 的CRISPR 结构中的第3 段间隔序列中有13bp 的基因序列与大肠杆菌的转座酶基因TnpR 具有一定的同源性。仍然存在某些间隔序列无法查找出与其同源的基因,也许是当前INTEGRALL 数据库中可移动遗传原件的相关数据还不是十分完整,也有可能是外源基因进入该菌株后发生了碱基突变。
上述试验证明间隔序列中的一部分序列与其它菌种中的一些可移动基因原件有一定的同源性,对同种细菌的不同株细菌而言,其间隔序列可能不存在同源性,这可能与同种细菌的生长环境不同有关。生长环境的不同决定了其CRISPR 位点结构之间的差异。志贺氏菌和大肠杆菌之间关于CRISPR 位点的相同性和特异性尚待探索。一些间隔区与几种已知的质粒和细菌序列相关,而其它的甚至没有已知的序列,这大概反映了未识别的噬菌体,质粒或细菌序列。相关研究发现,在CRISPR 位点的上游端也许含有一段前导序列,其长度约在300~500 bp 之间。通过多重序列对比分析,发现在CRISPR1-3,CRISPR-Q1~Q4 结构的5'侧翼区的1 000 bp 序列中出现较多的AAAAA 和TTTT 结构,同时碱基突变较多,表明此区域基因移动频繁,与基因的启动调控有关[10]。
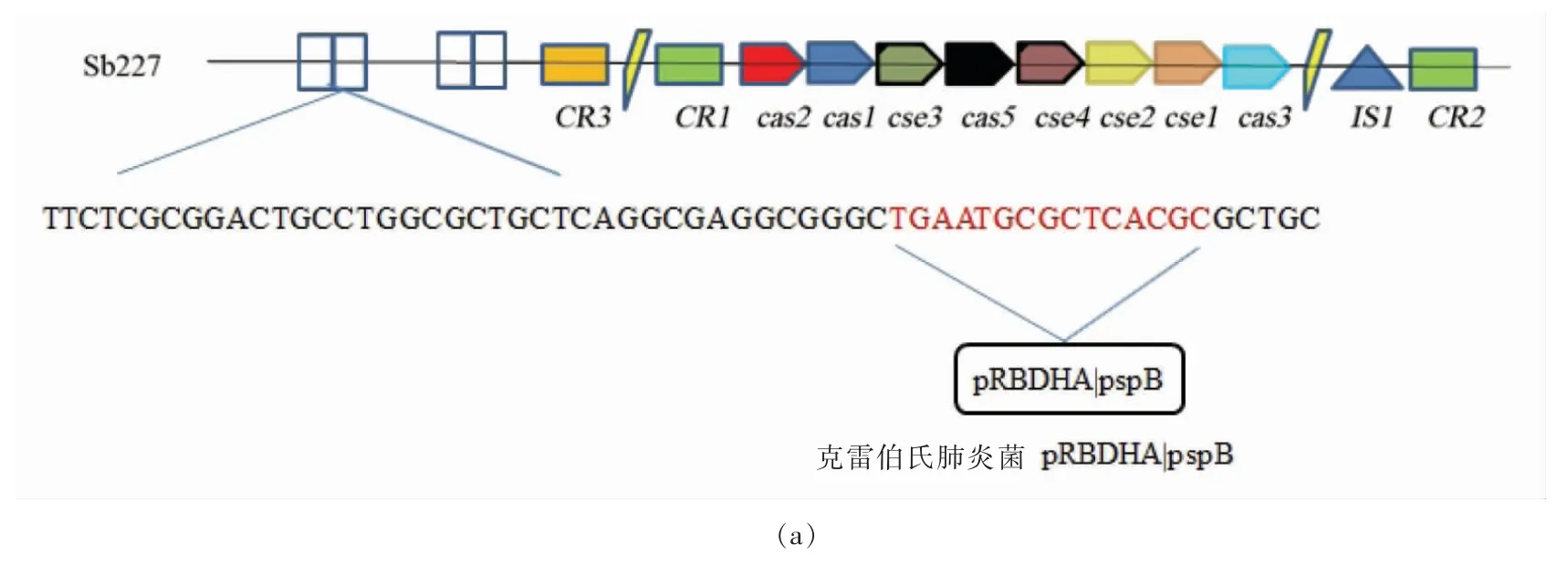

图3 鲍氏志贺氏菌227 和痢疾志贺氏菌197 CRISPR 位点中间隔序列同源性序列分析Fig.3 The spacer homologous sequence analysis of Shigella boydii str.227 and Shigelladysenteriae str.197
2.4 CRISPR 位点与RNA 二级结构稳定性的相关性分析
之前研究表明CRISPR 重复序列可能形成稳定的发夹形状的RNA 二级结构,部分具有回文性质。通过RNAFold Web 检测每个CRISPR 位点重复序列的RNA 二级结构和热力学最小自由能(MFE)。

图4 重复序列的Weblogo 和其RNA 二级结构Fig.4 The Weblogo and secondary structure of repeats
由图4a 可知,CRISPR1 和CRISPR2 位点的热力学系最小自由能△G=-14.45 kcal/mol,CRISPR3位点的热力学最小自由能△G=-8.71 kcal/mol。由图4b 可得,CRISPR-Q1 中第1 个和末端重复序列之间的差异是明显的,CRISPR-Q2,CRISPR-Q3和CRISPR-Q4 中的第1 个和末端重复之间没有差异。热力学集合的自由能△G=-14.75,-21.33,-9.76 和-17.92 kcal/mol。RNA 二级结构在每个末端由环组成,茎在6~12 bp 之间,其中CRISPR-Q2的MFE(△G = -21.330 kcal/mol)大于其它CRISPR(P <0.05),而CRISPR2 的MFE(△G =-8.71 kcal/mol)是7 个CRISPR 位点中最小的,表明由于茎中碱基对数目较多,其二级结构更稳定。
2.5 重复间隔序列数量与质粒数量的相关性分析
如表3所示,对40 株志贺氏菌的重复和间隔序列数以及平均质粒数进行统计,发现共含有67个质粒,平均含有1.7 个质粒,其中有的菌株不含质粒,有的菌株中高达7 个质粒。6 株鲍氏志贺氏菌的平均质粒个数为1 个,5 株痢疾志贺氏菌的平均质粒数为0.6 个,18 株福氏志贺氏菌平均有1.33 个质粒,11 株宋内氏志贺氏菌的平均质粒数为3.2 个,且均具有结合转移区(Conjugation Transfer)。分析志贺氏菌CRISPR 位点和质粒的相关性后,未发现质粒数与其有很大的关联度,重复间隔序列多的菌株质粒数并不一定多,反之亦成立。从相关性分析上无法得到间隔序列越多质粒越少或越多的结论。通过其它文献了解到质粒是一个复杂的可移动基因元件,需要通过绘制质粒图谱做进一步的研究,这也是今后需要研究的方向。

表3 4 类志贺氏菌的重复间隔序列数与平均质粒数Table3 The number of repeat interval and the average number of plasmid of 4 kinds of Shigella
3 讨论
志贺氏菌是人类患病的重要病原体,细菌性痢疾是由志贺氏菌引起的一种急性肠道传染病,在世界各地仍是一个非常严重的公共卫生和社会问题,造成了极大的经济上的重负,特别是在发展中国家已经成为一个重大的健康威胁[5,11]。
CRISPR 簇是一个特殊DNA 重复序列家族,存在于细菌和古生菌基因组中,可作为防御外源遗传物质的“基因武器”[12]。在40%的已测序细菌和90%的已测序古生菌中存在CRISPR。CRISPR序列是由许多的重复序列区(repeat)和间隔序列区(spacer)组成,重复序列区保守,存在回文结构,能够形成发卡结构[13],而间隔区是被细菌俘获的外源DNA 序列。前导区域(leader)位于序列上游,作为CRISPR 序列的启动子[14]。在CRISPR 上游还有一个多态性的家族基因,该基因编码的蛋白和CRISPR 序列区域一起发挥作用,被命名为CRISPR 关联基因(CRISPR associated,cas)。当前人们研究发现了cas1-cas10 等多种类型的cas 基因。cas 基因与CRISPR 序列共同进化,在细菌中形成了高度保守的CRISPR/Cas 系统。
CRISPR 系统是细菌用以抵御外源移动基因原件侵袭的免疫系统,其中CRISPR/Cas9 是一个新兴的极具研究价值的基因编辑工具[15]。CRISPR系统可以抑制携带耐药基因的质粒、转座子、整合子等外源基因的入侵,进而防止耐药基因在细菌种间的水平传播,特别是致病细菌之间的传播,对临床治疗和畜牧业意义重大。本研究通过生物信息学方法对志贺氏菌的CRISPR 位点进行分析,掌握志贺氏菌中CRISPR 位点的分布情况及结构等信息,分析推测CRISPR 和外源质粒之间的联系,结果显示间隔序列与许多耐药基因和整合子、转座子、质粒间的同源性程度不同。
目前已确认一些志贺氏菌的CRISPR 位点的结构。本研究中对志贺氏菌CRISPR 结构的生物信息学分析可能为阐明志贺氏菌CRISPR 结构的功能提供信息。根据在CRISPRs Database 上得到的有效信息,志贺氏菌中的CRISPR 包括确定位点和可疑位点,以及独特的间隔区域。间隔物形成的机制将为分析间隔物功能提供线索。在第1 个和最后1 个重复之间的基数差异CRISPR1~3 将直接影响RNA 二级结构的稳定性[3]。
志贺氏菌中的一些CRISPR 位点高度保守,除CRISPR-Q1 外,可疑的CRISPR 仅包含两个及两个以下的间隔序列,这些间隔序列具有90%的相似性[10]。CRISPR-Q1 不太活跃,被认为是古生菌株的象征。这些研究数据并没有显示CRISPR 作为志贺氏菌属免疫系统的组成成分之一,而CRISPR-Q1 中的间隔序列的数目和是否存在CRISPR1 和CRISPR2 可能是与间隔物的其它特征相关联。CRISPR-Q1 中的多个间隔区域在一些区域中表明CRISPR-Q1 可能参与CRISPR1 或CRISPR2 的功能[4]。
根据对cas 基因的分析,发现40 株志贺氏菌的cas 基因排列顺序统一性极高,cas3 基因与重复序列一样也具有相似的保守性,这和先前文献[16]报道相一致。这表明在进化过程中志贺氏菌也面临着相似的选择压力。前导序列在同种细菌中保守性不是很强,碱基突变或碱基缺失现象易发生,说明前导序列基因比较活跃,推测其与启动CRISPR 基因表达有关[17]。对间隔区序列进行分析,虽然在Integrall 数据库中不能找到与间隔序列完全匹配的序列,但能找到相关序列与间隔序列中部分序列完全匹配,同时这些序列多数来源于质粒、噬菌体、耐药基因、整合子和转座子[18]。这说明志贺氏菌的CRISPR 系统能抵御外源基因的侵袭,尚未发现CRISPR 位点数量与质粒数量以及间隔序列数量与质粒数量之间具有统计学意义的相关性[19]。作者预测该情况是因质粒类型的不同,也许是因一些质粒具有抵抗CRISPR 结构的能力所致,这需要对质粒结构进行更深入的研究。
