最大团问题的可编程的DNA分子系统计算模型①
2020-05-18严洋洋殷志祥
严洋洋, 殷志祥
(安徽理工大学数学与大数据学院,安徽 淮南 232001)
0 引 言
自从电子自计算机问世以来,为人类社会带来了巨大的便利,渐渐已经成为社会发展中不可或缺的一部分。可随着科技的发展变迁,对电子计算机的各种要求越来越高,尤其是在智能化,运算速度,存储等方面,一些问题也难以得到很好的解决,如NP—问题,使得人们不得不寻找新的计算方法。经过一系列的研究,DNA分子开始进入研究员的视野。DNA分子具有高并行性,高存储,高特异性,易操作等特性。伴随着生物化学与分子生物学技术的不断发展,以DNA分子这些良好的特性为桥梁的新型计算方法,使得DNA计算成为了研究员们的热点研究领域[1]。
DNA计算是一种在分子层面借助生物分子技术进行计算的新方法,具有高容量,高度并行性,微小性等特点,为解决非线性问题提供了一条新的道路。Adelman借助DNA编码解决了7顶点Hamilton路径问题,开创了DNA计算的首例。[2]目前,在DNA计算领域已经取得了巨大的成就,极大的促进了计算机科学、生物科学、化学、数学等学科的的发展。DNA自组装理论的建立,是DNA计算的亮点之一,由此,研究员利用其特性解决最大团问题[3-4]、二部图完全匹配问题[5]等问题。文献[6]利用DNA计算计算求解可满足性问题以及文献[7]中逻辑门模型的构建。由于DNA链置换反应具有很强的灵敏性、高准确性、自发性等特点,使得DNA链置换技术成为DNA自组装发展中最具特色的一部分。[8-9]
在这里,设计了一种基于可编程的DNA分子系统[10]求解最大团问题的模型,可以对该分子系统进行编程,编程程序是由一系列DNA指令组成定义反应顺序的链。在该分子系统中,设计了在环部分具有识别区域的茎环结构,将起始双链体作为引发剂,与化学发夹和指令发夹发生链置换反应,形成多聚化学链。首先,借助图的顶点排列组合出所有可能性组合,生成所有的数据池。然后,借助分子系统进行筛选,得到满足团定义的所有可能性组合。接着,再检测满足条件的多聚化学链上低聚物,判断和读取最大团。
1 可编程的DNA分子系统
该DNA分子系统的的汇编程序是由一系列DNA指令组成定义反应顺序的链,合成具有确定序列的低聚物。只需要更改指令集就可重新编程,以进行组合合成。
起始双链体(图1a)具有诱发作用,它是由一条3′端带有增长的低聚物(带颜色的小圆圈表示)的货物(Cargo)单链与单链I形成的双链体结构,并且在链I的3′端带有确定的脚趾。化学发夹与指令发夹,上半部分是其环区域,并且有确定的碱基对序列,在交错反应中,能够与下方的延伸出的脚趾区互补配对。在其径区域化学发夹与指令发夹的碱基对序列是相同的,同样也与起始双链体的茎区域的碱基对序列相同。编程的顺序最主要依赖于指令发夹的组装。指令发夹的方向是确定的,如:指令发夹I>A,识别链I,并将其链接到A上,指令发夹A>B,识别链A,并将其链接到B上,。两个发夹的单链脚趾区域用于启动发生杂交反应,同时控制组装顺序,即组装顺序是通过成对的互补脚趾之间相互作用进行编程。终止发夹是指令发夹的一种,其作用是用来终止链的扩张。
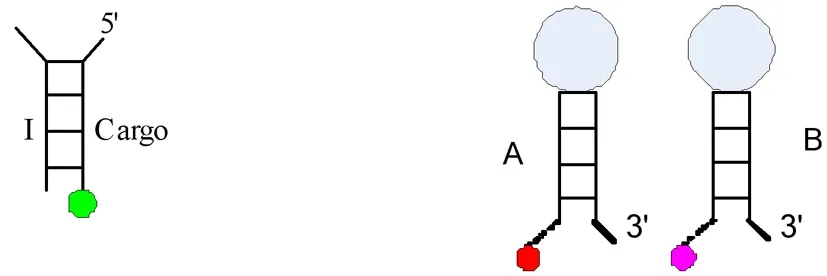
图1a 起始双链体(I:Cargo) 图1b化学发夹

图1c 指令发夹 图1d 终止发夹
该分子系统的组装(图2)是由交错排列的开放的化学发夹与指令发夹通过DNA链置换反应形成的线性双链体,是一维的杂交链反应。起始双链体(I:Cargo),与第一个指令发夹I>A的互补的脚趾进行DNA杂交反应,茎链被打开进行迁移(沿着红色箭头方向),进而导致发夹的环区域开放,形成了四臂分支结构。环开放后,激活脚趾区,该区域指定下一步要添加化学发夹A。这时化学发夹A的没有携带货物的脚趾区域插入,完成链接,形成四臂分支结构。当每个发夹添加到链中时,其环开放,然后通过脚趾区域进行链杂交,进而启动下一阶段链的增长,这说明了通过交替添加指令发夹与化学发夹进行链的扩张。形成的双链体的每条链都是不连续的,一条链由化学发夹组成,一条有指令发夹组成,组装的顺序是通过成对的互补脚趾之间相互作用形成进行编程的。当每个发夹添加到链中时,货物通过迁移,链接到新添加的发夹上,整个链的扩张由Ti>Tend,终止。
2 最大团问题的可编程的DNA计算模型
2.1 最大团的基本定义
设简单图G=(V,E),其中非空集合V是顶点集,E是边集。给定一个图,其任意顶点子集都有可能是G的团。设U是G的一个顶点子集,对于顶点子集U中任意两顶点u,v,均与E中的边邻接,则U是G的团。若图G中的任意团都有|U′||U|,则U是G的最大团。
2.2 最大团基本算法
对于一个n顶点的简单图G=(V,E),其中V={V1,V2,…,Vn},若G的任意顶点子集都不是G的团,则该图的任意两顶点都不是邻接的。
Step1 每个顶点用一个化学发夹来表示,利用DNA分子系统对图G的顶点进行设计、编码。排列组合出所有的可能性组合,对于n顶点的简单图的顶点子集共有2n个,即生成数据池。
Step2 为满足团的定义对数据池进行检测。借助可编程的DNA分子系统对图中任意一个顶点子集进行筛选。不失一般性,设顶点集Vi={Vi1,Vi2,…,Vik},其中k≥2。若是Vi在图G的一个团中,则Vi中任意两点都是邻接的。即若两顶点在同一个团中,则这两顶点必定邻接。开始,加入起始双链体(I:Cargo),与指令发夹进行匹配,激活脚趾区,然后在脚趾区进行杂交反应,进而开放发夹的环区域,根据指定的顺序,加入化学发夹。于是化学发夹由具有识别作用的脚趾区开始插入,进行杂交反应,使得增长性的低聚物发生迁移。然后继续加入指令发夹,进入下一阶段的链的扩张,最后添加的终止发夹停止,形成线性双链体。对k(n≥k≥2),从大到小逐次检验,在检验过程中,需要多次对图的顶点进行编码。删除低聚物不能连接在一起的线性双链体,剩下的都满足团的定义。
Step3 观察最后的线性双链体中低聚物的个数,通过DNA测序,读出图的最大团及其顶点。
3 实例应用
以图3,5顶点简单图为例,顶点集V={1,2,3,4,5},边集E={(1,2),(2,3),(3,4),(4,5),(2,5)(2,4)(3,5)},利用该DNA分子系统进行最大团问题的求解。

图2 DNA分子系统组装机制

图3 5顶点的简单图
Step1 将这五个顶点用分别用五个化学发夹表示,排列出这些顶点的所有可能性组合,生成数据池,顶点子集个数共有32个。
Step2结合2.2中的Step2,检测数据池,各个顶点子集进行判断。本例中k的最大值为k=5。对顶点进行编码,用符号表示为V1:A,V2:B,V3:C,V4:D,V5:Ti,加入起始双链体(I:Cargo),开始杂交反应,此后按照I>A→A→I>B→B→B>C→C→C>D→D→D>T5→T5→T5>Tend的顺序逐次加入。最后加入的T5>Tend用于终止反应,形成了线性双链体。
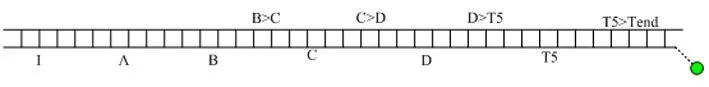
图4 判断顶点集V=(1,2,3,4,5)
仅有起始双链体货物的在链上进行了迁移,其它代表图的各个顶点的发夹上的货物没有完成迁移。因此,该图本身不是是团。
下面逐次对k(4≥k≥2)进行检验,其中一个最大的顶点子集V=(2,3,4,5),判断是否为图的团。对图的各个顶点重新编码,用符号表示为:V2:B',V3:C′,V4:D′,V5:T5′依照上述步骤,按照I>B′→B′→B′>C′→C′→C′>D′→D′→D′>T5′→T5′>Tend的顺序逐次加入进行操作。最后可以的得到如图5所示的线性双链体。链上的货物发生迁移,增长的低聚物连接在一起,可以判定顶点集V=(2,3,4,5)可以构成图的一个团。
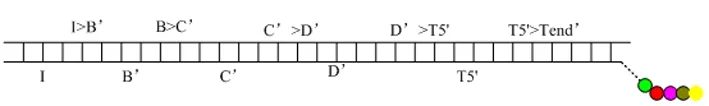
图5 判断顶点子集V=(2,3,4,5)
将低聚物不能连接到一起的线性双链体移除,剩下的都满足团定义。
Step3,最后观察,满足团定义的线性双链体,读出最大团。经过验证,该实例的最大团由顶点集V=(2,3,4,5)构成。
4 结 论
借助DNA计算求解NP完全问题,可编程性、自主、高并行性,是十分重要的追求。在前文中,主要借助可编程的DNA分子系统求解最大团问题。通过起始双链体的诱发,由化学发夹和指令发夹杂交反应交错排列构成线性双链体,通过链的迁移将可增长的低聚物转移到每个发夹上,最终检测低聚物的个数来读取最大团。在理论上,对于图的规模没有限制,技术上也已经成熟,操作简单、便利。相信,随着生物技术的发展,DNA计算将会用更广阔的舞台,DNA分子自组装在数据存储、智能机器也将展现它的巨大潜力。
