2017年~2018年四川省猪δ冠状病毒的检测与遗传进化分析
2019-11-22殷鑫欢杨晓宇鲁令华徐志文
冯 宇,殷鑫欢,徐 雷,杨晓宇,鲁令华,徐志文,2,朱 玲,2
(1.四川农业大学动物生物技术中心,四川 成都 611130;2.四川动物疫病与人类健康四川省重点实验室,四川 成都 611130)
猪 δ 冠状病毒(Porcinedelta coronavirus,PDCoV)是新发现的冠状病毒,属于δ 冠状病毒属,为单股正链RNA 病毒。同α 冠状病毒属的成员—猪流行性腹泻病毒(Porcine epidemic diarrhea virus,PEDV)和猪传染性胃肠炎病毒(Transmissible gastroenteritis virus,TGEV)一样,PDCoV 也可导致仔猪出现严重的腹泻或呕吐,并进一步引起仔猪脱水死亡,对养猪业造成了一定的经济损失。PDCoV 的基因组全长约25.4 kb,是冠状病毒中基因组长度最小的病毒[1]。PDCoV基因组结构与PEDV相似,为5'UTRORF1ab-S-E-M-NS6-N-NS7-3'UTR.
2012年香港学者首先报道了PDCoV,随后于2014年在美国多个州的猪场暴发由该病引起的疫病。目前已在美国、韩国、中国、日本、东南亚(泰国、越南)等多个国家报道在发病猪群中检测出PDCoV[2-5]。在中国,多个省份也报道了该病毒引起的疫病,但各省份该病的流行率差异较大。在江西省的流行率高达33.71 %[6],在广东省稍低为22 %[7],但在江苏和湖北仅有4.94 %和6.45 %的流行率[8],鉴于研究发现PDCoV 在不断的变异进化,并出现病毒重组现象[9],推测出现该现象的原因可能是各省PDCoV 流行株的基因差异导致了病毒毒力的不同进而影响了疫病的流行率。本研究对2017年~2018年四川地区PDCoV 的流行情况做了调查,同时对两株四川病毒株进行全基因组序列分析,为揭示PDCoV 在中国的流行情况及其基因的多样性,以及后续对该疫病的防控提供参考依据。
1 材料与方法
1.1 病料样品与主要试剂 141 份样品为2017年~2018年采集自四川省遂宁市、眉山市、绵阳市以及南充市等各地区大中型猪场有腹泻症状的仔猪粪便与小肠组织(2017年共64 份样品,2018年共77份样品)、TRIzol、TaqPCR Master Mix (2×,blue dye)、pMD19-T 载体、DL2000 DNA Marker、DH5α大肠埃希菌感受态细胞、质粒DNA 小量纯化试剂盒、DNA 胶回收试剂盒均购自生工生物工程(上海)有限公司;反转录试剂盒购自TaKaRa 公司。
1.2 引物合成 根据现有文献的报道[10-11],合成检测PDCoV、PEDV、TGEV 和猪轮状病毒(Porcine rotavirus,PoRV)的引物,同时合成16 对扩增PDCoV全基因组的引物(引物序列省略,读者需要可提供),引物均由上海生工生物工程技术服务有限公司合成。
1.3 临床样品的PCR 检测 按常规方法处理小肠组织样品、粪便样品,采用TRIzol 法提取样品中的总RNA,并反转录为cDNA,以相应引物PCR 检测PDCoV、PEDV、TGEV 和PoRV,并设不含模板的阴性对照组。PCR 扩增产物经2 %琼脂糖凝胶电泳检测,并经测序鉴定。
1.4 PDCoV 全基因组测序与序列分析 从1.3 中随机挑选两份测序鉴定为PDCoV 阳性的病料样品,提取其总RNA,利用16 对引物分段扩增PDCoV 全基因组序列,获得的PCR 产物连接至pMD19-T 载体,并由上海生工生物工程有限公司测序,将所测得的16 个分段序列利用SeqMan 拼接,得到PDCoV的全基因组序列。利用Editseq 和Megalign 软件对该两株PDCoV 与该病毒参考株的全基因组序列进行比对、分析。采用MEGA 6 软件中的最大似然法构建PDCoV 全基因组与S 基因的系统发育树,分析四川PDCoV 株和参考株之间的亲缘关系。
2 结果与讨论
2.1 四川地区PDCoV 的检测结果 对141 份仔猪的腹泻病料样本进行PCR 检测,具体结果详见表1。表明四川地区PDCoV 的感染率较低,而PEDV仍是引起仔猪腹泻的主要病原,同时存在两种病毒混合感染引起仔猪腹泻的情况。
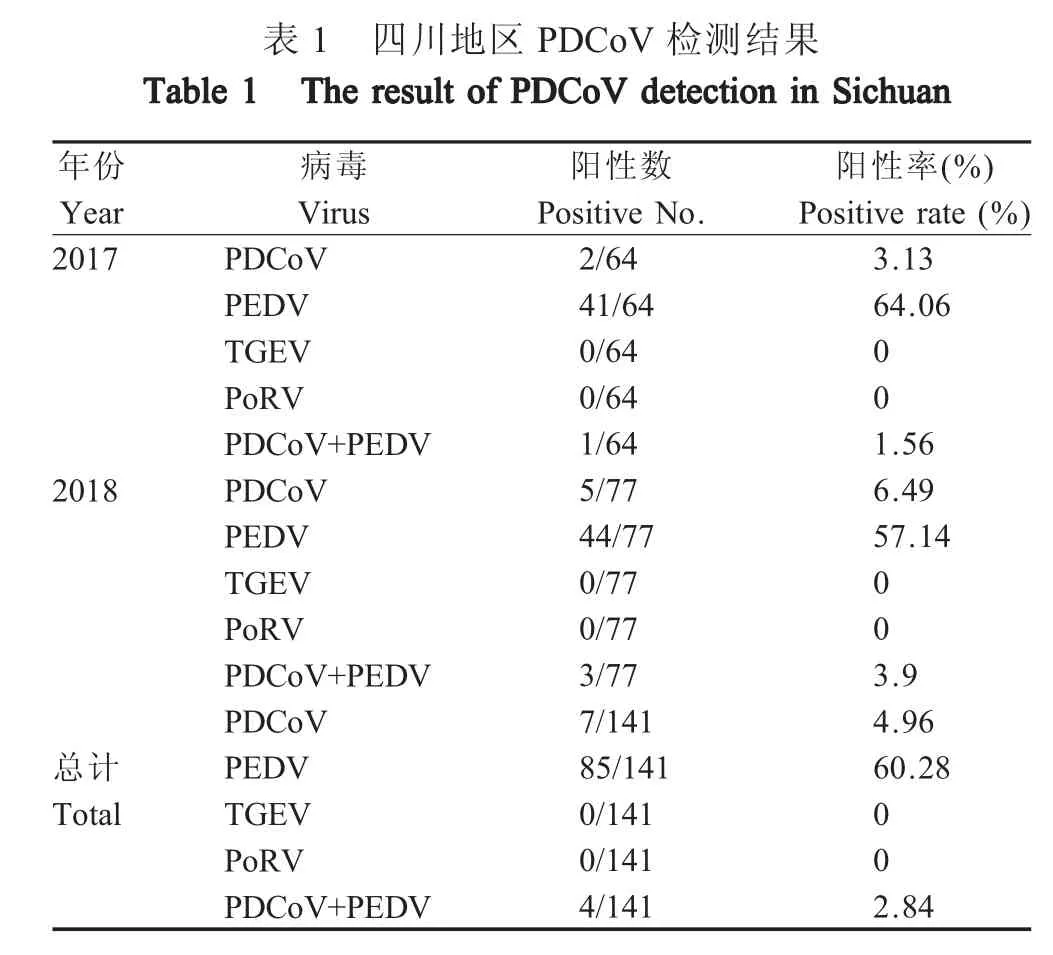
表1 四川地区PDCoV 检测结果年Year 2017 Virus PDCoV PEDV TGEV PoRV PDCoV+PEDV PDCoV PEDV TGEV PoRV PDCoV+PEDV PDCoV PEDV TGEV PoRV PDCoV+PEDV Positive No.2/64 41/64 0/64 0/64 1/64 5/77 44/77 0/77 0/77 3/77 7/141 85/141 0/141 0/141 4/141 Positive rate (%)3.13 64.06 00 2018 1.56 6.49 57.14 00总计Total 3.9 4.96 60.28 00 2.84
2.2 两株PDCoV 的全基因组扩增及序列分析结果以随机选取的两份鉴定为PDCoV 阳性的病料样品提取RNA,反转录为cDNA 作为模板,经分段PCR扩增获得16 段PCR 产物,分别将这些PCR 产物克隆至pMD19-T 载体后测序,利用SeqMan 对测序结果拼接,得到两株PDCoV 的全基因组序列,分别命名为CHN/Sichuan/2017 和 CHN/Sichuan/2018 (序列号分别为MK330604、MK330605)。对两株PDCoV全基因组序列分析,结果显示除去3' 端的polyA 结构,两株病毒的基因组全长分别为25 402 nt 和25 413 nt。其基因组结构相似,除了在3'UTR 端有11 nt 的缺失外,无其它碱基的缺失或插入。通过Editseq 和 Megalign 软件分析两株病毒之间的同源性,结果显示两株病毒间同源性为99.3 %,将其同时与PDCoV 参考株比对,显示与参考株之间的同源性97.6 %~99.0 %。表明所测序的两株病毒确实是PDCoV。
同时,两株病毒均与CHN/SHJS/SL/2016 株同源性最高,为99.0 %;与越南和泰国株同源性最低。然而先前报道的四川分离株CH/Sichuan/S27/2012 却与东南亚病毒株(越南和泰国)同源性高达98.8 %[12],提示四川地区PDCoV 流行株发生了变化。另外,利用Megalign 软件对两株病毒与参考株进行序列比对分析,结果显示其与最早发现的病毒株(HKU-155和HKU-44)、两个四川病毒株、东南亚病毒株和CH/Sichuan/S27/2012 株以及其它 4 个中国病毒株(CH-HuNan、 SHJS/SJ、 CHN-HeB1、 CHN-SD) 在ORF1a 区域均有 6 nt (1739TTTGAA1744)缺失(图1)。与CH/Sichuan/S27/2012 株比较,两个病毒株在ORF1a区域有9 nt (2810CCGGTTGGT2818)的插入。其次,与其他国家的PDCoV 株比较,两株病毒在S 基因有3nt(19476TAA19478)的缺失,并且该部分缺失出现在大多数中国PDCoV 株中(图1)。研究显示S 蛋白在与细胞受体相互作用中起重要作用[13],提示该部分的缺失可能会影响PDCoV 与细胞受体的结合。另外,本研究显示 CHN/Sichuan/2017 在 3'UTR 区域有 11 nt(25 042 nt~25 052 nt)的缺失(图1),鉴于 3'UTR 区域对β 冠状病毒的RNA 复制有重要作用[14],推测该部分的缺失可能会影响CHN/Sichuan/2017 株的RNA 复制。上述结果表明,四川地区PDCoV 在不断地变化,且该地区目前流行株与中国大陆PDCoV株基因特征相似,同源性较高,而与东南亚病毒株同源性相对较低。

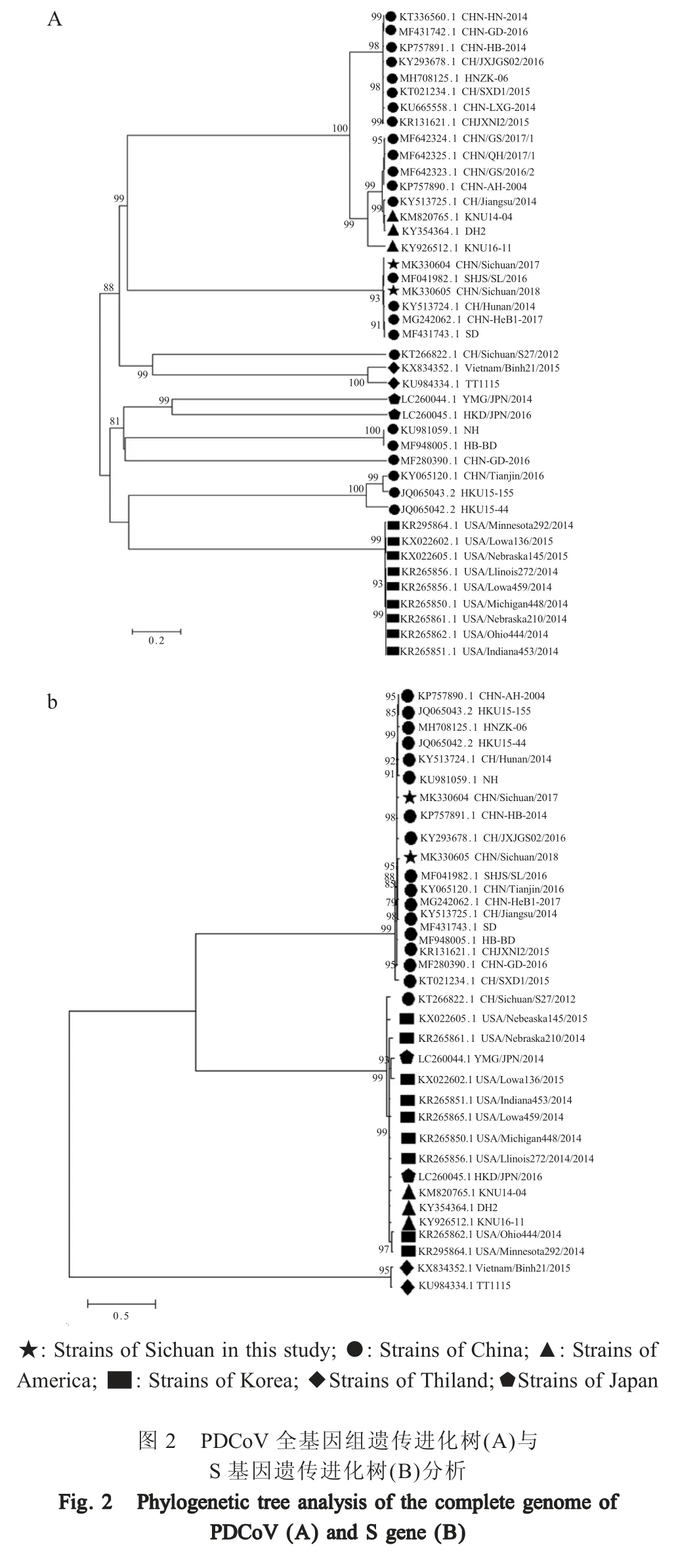
2.3 两株PDCoV 的遗传进化分析 将两株PDCoV的全基因组和S 基因进行系统发育分析结果显示,所有PDCoV 株呈现出明显的地域特征,与以往报道结果一致[15]。同时两株PDCoV 全基因组进化树分析显示,中国PDCoV 呈现4 个小分支,该两株病毒与中国内地病毒株组成一个小分支,而CH/Sichuan/S27/2012 株与东南亚病毒株相似,单独形成一个分支,提示CH/Sichuan/S27 与东南亚病毒株的进化来源可能相同(图2A)。另外,基于S 基因的进化树结果显示CH/Sichuan/S27 与美国、韩国和日本的病毒株组成一个大分支,而四川地区两株病毒与中国内地PDCoV 株聚集成群(图2B)。进一步表明四川地区PDCoV 株不断变异进化,而且该病毒具有基因多样性特征。
PDCoV 作为新发现的引起猪腹泻的冠状病毒,在中国多省均有报道由该病毒引起疫病的流行病学调查,而对于养猪大省四川关于该病的详细调查较少,所以本研究结果为了解四川地区PDCoV 的分子流行情况,以及中国PDCoV 的分子流行特征和遗传多样性提供了实验数据,为后续PDCoV 病毒学研究提供参考。
