昌黎产区酒类酒球菌(Oenococcus oeni) 的遗传多样性及系统发育分析
2019-09-06余东亮刘树文
余东亮,石 侃,孟 强,刘树文,3,4,*,何 玲
(1.西北农林科技大学葡萄酒学院,陕西 杨凌 712100;2.秦皇岛金士国际葡萄酒庄有限公司,河北 昌黎 066600;3.陕西省葡萄与葡萄酒工程技术研究中心,陕西 杨凌 712100;4.陕西省合阳葡萄试验示范站,陕西 渭南 715300;5.西北农林科技大学园艺学院,陕西 杨凌 712100)
苹果酸-乳酸发酵(malolactic fermentation,MLF)是优质葡萄酒酿造中必不可缺的工艺技术[1]。MLF对葡萄酒具有降酸、改善口感、增加风味复杂性及微生物稳定性的重要作用[2]。事实上,由于乙醇发酵结束后,葡萄酒高乙醇含量、低pH值和一定浓度的SO2等严苛条件,常引起MLF的延迟或停滞,加之不同种类葡萄酒乳酸菌的存在,增加了代谢风险物质如组胺等的产生,MLF成为相对于乙醇发酵更为难以控制的发酵过程[3-6]。
研究发现,酒类酒球菌(Oenococcus oeni)是营养缺陷型细菌,却能更好地适应葡萄酒的严苛生境,是推动MLF的主导菌及葡萄酒最终质量风格的塑造者,且它们的MLF发酵特性存在菌株水平的特异性[7-11]。此外,不同的葡萄酒生境中O. oeni资源存在多样性和特异性,赋予产品地域特色[12-14]。因此,筛选本土优良O. oeni菌株不仅能推动MLF顺利进行,而且能满足市场对产品多样化及产品典型性的需求[15]。Ruiz等[16]从西班牙卡斯蒂利亚-拉曼恰(Castilla La Mancha)产区丹魄(Tempranillo)葡萄酒中,筛选出本土的O. oeni优良菌株C22L9,证实该菌株赋予葡萄酒独特的产区感官特色。目前,西班牙、意大利、阿根廷和智利等发达的葡萄酒生产国愈加重视并开展本土O. oeni优良菌株的研究和开发[17-21]。因此,在菌株水平对我国葡萄酒产区O. oeni进行遗传多样性及其系统发育研究,为开发利用我国本土O. oeni种质资源,推动我国葡萄酒产业发展具有重要意义。O. oeni缺乏错误修配基因(mutS和mutL,它们具有纠正多种碱基对的错配,防止产生过多突变),被认为具有超突变性[22];而一些研究证实O. oeni不同菌株间存在高度的保守性[23-25]。由于O. oeni这种高突变性和高保守性的矛盾,因此,在对O. oeni进行菌株水平的遗传研究中,强分辨能力的研究方法十分必要[26]。荧光标记扩增片段长度多态性(amplified fragment length polymorphism,AFLP)技术具有操作便捷、分辨率高、重现性好等优点,为研究O. oeni的自然种群与典型的地理环境间的遗传关系提供了有效途径[27-29]。
近年来,在我国的葡萄酒酿造过程中,启动MLF的O. oeni商业制剂几乎全部来自国外,不利于呈现葡萄酒产品的区域典型性和风格,导致产品同质化[30],制约着我国特色葡萄酒产业的发展。昌黎产区是我国政府首批“葡萄酒原产地保护区”。该产区在葡萄酒酿造过程中,启动MLF的O. oeni商业制剂几乎全部来自国外的LAFFORT公司和LALLEMAND公司,且型号单一,致使葡萄酒产品同质化严重。因此,本研究采用荧光标记AFLP技术,对筛选自昌黎产区不同酒庄的222 株O. oeni进行遗传多样性和发育规律的研究,以期为开发利用我国不同产区内本土O. oeni种质资源,助力我国特色葡萄酒产业发展,提供方法和理论支持。
1 材料与方法
1.1 材料与试剂
226 株分离株筛选自昌黎产区5 家具有代表性酒庄,见表1。O. oeni菌株SD-2a和31-DH作为模式菌株,分别来自西北农林科技大学葡萄酒学院微生物实验室和中国食品发酵工业研究院。采用葡萄酒生境中分离的短乳杆菌(Lactobacillus brevis)xja1和xj20,植物乳杆菌(L.plantarum)xj14、xj25和xja2,马里乳杆菌(L.mali)xja7和xja8作为参照菌株,均来自西北农林科技大学葡萄酒学院微生物实验室[31]。
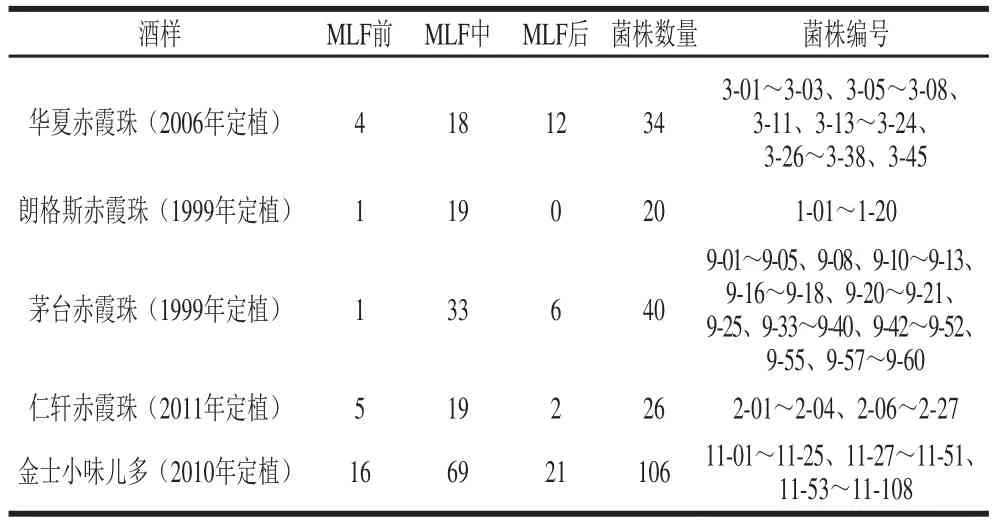
表1 分离菌株及来源Table 1 Isolated strains and their origins
苹果酸监测采用爱尔兰Megazyme L-Malic Acid(LMALATE)试剂盒;RNase、DNA polymerase、内切酶EcoRI和MseI、T4 DNA Ligase 日本TaKaRa公司;引物及荧光标记引物均由生工生物工程(上海)股份有限公司合成。
ATB培养基:1 L含蛋白胨10 g,酵母浸出物5 g,葡萄糖10 g,硫酸镁0.2 g,硫酸锰0.05 g,盐酸半胱氨酸0.5 g,番茄汁250 g,调节pH值至4.8。
ATB固体培养基:每1 L ATB液体培养基加入20 g的琼脂粉。
1.2 仪器与设备
C1000基因扩增仪 新加坡Bio-Rad公司;Veriti梯度扩增仪 美国基因有限公司;Nanodrop ND-1000核酸蛋白仪 英国BioDrop公司;ChampGel 500 plus全自动凝胶成像系统 北京赛智创业科技有限公司。
1.3 方法
1.3.1 酒样的选取
据昌黎产区地理分布特点,选取5 家具有代表性的酒庄,于2016年榨期葡萄酒乙醇发酵结束后,立即采用消毒容器从葡萄酒发酵罐(其中金士小味儿多为5 t发酵罐,其他均为20 t发酵罐)中直接取样,避免接种O. oeni商业制剂的污染。酒样基本理化指标见表2。
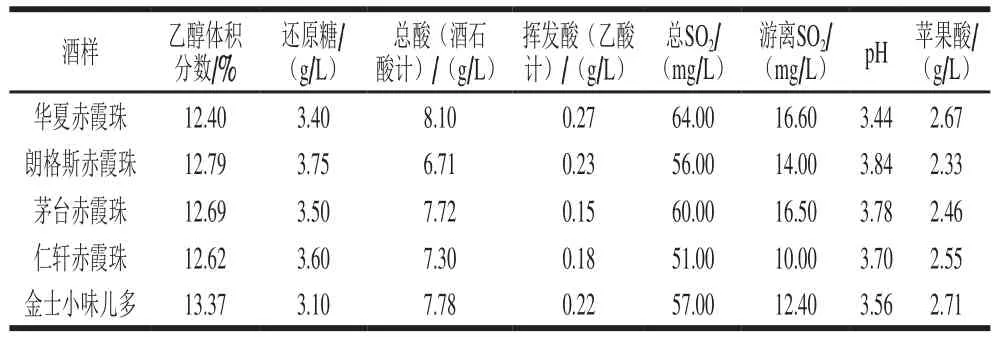
表2 酒样基本理化指标Table 2 Physical and chemical indexes of wine samples
1.3.2 细菌的分离
酒样取回后,分装于消毒后500 mL长颈烧瓶内封好,放置20 ℃恒温培养箱中,监控自发MLF,一式三份。监测酒样中苹果酸含量消耗变化,每个酒样分MLF前期、中期(苹果酸消耗50%~70%)、后期(剩余苹果酸<20%)取样。分别取0.1 mL酒样,使用0.85%生理盐水(灭菌)稀释10-1~10-6梯度,采用O. oeni的ATB分离培养基涂平板[32],培养皿封严后,于26 ℃恒温厌氧培养3~8 d。待平板长出菌落后,按表型差异(生长时间和菌落大小等),挑选单菌落。随后,采用灭菌ATB培养基进行单菌落培养和划线分离,反复纯化2 次后,用20%灭菌甘油保存于-20 ℃备用。
1.3.3 细菌总DNA的提取
参考Cappello等[27]方法有改动。分离株和模式菌株于ATB培养基培养,参照菌株于MRS培养基培养,均至对数生长期,提取DNA。提取过程中,添加RNase,提高DNA纯度。提取模板DNA用0.8%琼脂糖凝胶电泳,检测质量。浓度由Nanodrop ND-1000核酸蛋白仪检测。
1.3.4O. oeniSpecies-specific聚合酶链式反应(polymerase chain reaction,PCR)
O. oeni特异性引物参照Zapparoli等[33]方法。Forward:5’-TAATGTGGTTCTTGAGGAGAAAAT-3’;Reverse:5’-ATCATCGTCAAACAAGAGGCCTT-3’。选用反应总体积25 μL:2.5 μL 10× PCR Buffer,20 ng DNA模板,5 mmol/L dNTP mixture,上下游引物各500 mmol/L和1 U的DNA polymerase。PCR程序为:94 ℃预变性300 s,1 个循环;94 ℃变性30 s,55 ℃退火30 s,72 ℃延伸60 s,30个循环;72 ℃延伸600 s,1 个循环。反应结束后,PCR产物经过1.5%的琼脂糖凝胶电泳检测,在凝胶系统仪上观察并记录结果。
O. oeni引物特异性验证:采用模式菌株和参照菌株验证扩增特异性。
1.3.5 荧光标记AFLP分析[34]
1.3.5.1 双酶切和连接反应
选取内切酶EcoRI和MseI组合。反应总体积20 μL:约200 ng DNA模板,10 UEcoRI于37 ℃保温酶切4 h。结束后直接添加5 UMseI在60 ℃温育3 h。酶切结束后80 ℃保温20 min灭活。然后,添加3 μL的T4 DNA Ligase、EcoRI和MseI接头,在30 μL总反应中,于16 ℃保温过夜连接。连接反应结束后,于65 ℃保温10 min,灭活T4 DNA Ligase。
1.3.5.2 连接产物的预扩增和选择性扩增
预扩增体系25 μL:2.5 μL 10× PCR Buffer,1 μL DNA模板(双酶切连接产物),5 mmol/L dNTP mixture,上下游引物各500 mmol/L和1 U的DNA polymerase。PCR程序为:94 ℃变性120 s,1 个循环;94 ℃变性30 s,56 ℃退火60 s,72 ℃延伸60 s,30 个循环;72 ℃延伸420 s。
选择性扩增反应体系为25 μL,选择性扩增引物采用已筛选的E1-M1 FAM组合:2.5 μL 10× PCR Buffer,3 μL DNA模板(预扩增产物稀释10 倍),5 mmol/L dNTP mixture,上下游引物各500 mmol/L和1 U的DNA polymerase。PCR程序为:94 ℃变性120 s,1 个循环;94 ℃变性30 s,66 ℃退火30 s(每个循环下降1 ℃),72 ℃延伸120 s,10 个循环;然后,94 ℃变性30 s,56 ℃退火30 s,72 ℃延伸60 s,20 个循环;最后,60 ℃延伸30 min,于12 ℃保存。
以上反应结束后,均取5 μL扩增产物于1.5%琼脂糖凝胶电泳检测扩增效果。电泳检验合格,选择性扩增产物送生工生物工程(上海)股份有限公司进行毛细管电泳检测。
1.4 数据分析
经ABI377自动测序仪检测数据,并进行统计分析。其中峰高阈值设定为100,将AFLP检测指纹图谱,按照等位基因出现条带记为1(峰高阈值≥100),未出现条带记为0(峰高阈值<100),构建二进制矩阵,采用NTSYS 2.10软件,基于非加权配对算术平均法(Unweighted Pair-Group Method with Arithmetic means,UPGMA)进行聚类分析,构建系统发育树。
2 结果与分析
2.1 O. oeni Species-specific PCR鉴定
2.1.1 O. oeni引物特异性验证
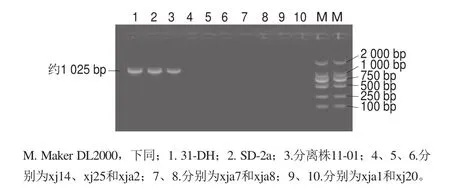
图1 O. oeni的引物特异性验证PCR电泳结果Fig. 1 Electrophoresis profiles of PCR amplification with O. oeni-specific primers
如图1所示,O. oeni模式菌株31-DH和SD-2a均可扩增出唯一、清晰的条带(泳道1、2),约1 025 bp;分离自葡萄酒生境的短乳杆菌、植物乳杆菌和马里乳杆菌等参照菌株均未能扩增出条带(泳道4~10),证实Zapparoli等[33]设计的O. oeni Species-specific PCR引物具有典型的特异性。其中,泳道3为分离株11-01,能扩增出和模式菌株相同的单一条带,证实为O. oeni菌株。随后,对分离株均进行Species-specific PCR鉴定。
2.1.2 分离株的Species-specific PCR鉴定
来自昌黎产区5 个代表性酒庄的酒样,经自发MLF,按前、中和后期取样,采用ATB分离培养基,得到具有表型差异的226 株分离株。226 株分离株均提取有效的DNA,后采用已验证的O. oeni特异性引物进行Species-specific PCR鉴定。经Species-specific PCR电泳检测,222 株分离株被确认为O. oeni菌株。4 株分离株1-03、2-26、3-29和3-32经电泳检测,均没有特异性条带出现,确认为非O. oeni菌株。图2为部分分离株的Species-specific PCR电泳检测图,其中分离株2-26(泳道22)没有特异性条带,其他22 株分离株均呈现约1 025 bp的唯一特异性条带,因此,确认2-26为非O. oeni菌株,其他均为O. oeni菌株。
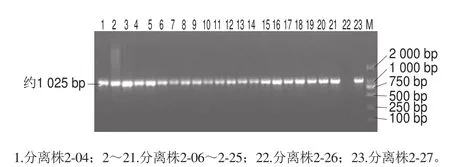
图2 部分分离株的Species-specific PCR鉴定电泳结果Fig. 2 Electrophoresis profiles of selected isolates identified by species-specific PCR
2.2 222 株O. oeni的荧光标记AFLP分析
2.2.1 分离株的荧光标记AFLP多态性分析
运用荧光标记AFLP技术,采用已筛选的带有荧光标记选择性引物E1-M1 FAM组合,在222 株分离株中检测出287 个位点条带,其中呈多态性位点条带数为285 条,多态性位点比率高达99.30%。金刚等[35]认为O. oeni基因组的G+C含量在40%左右,建立了基于HindIII和MseI组合的AFLP分析体系。基于HindIII和MseI内切酶组合优化的选择性扩增引物组合,其多态性位点比率在10%~52.90%之间,远低于本实验选择性引物E1-M1 FAM组合的99.30%。随高通量测序技术的发展,发现O. oeni基因组的G+C含量在37%左右,在多态性上,内切酶EcoRI和MseI的组合属于AFLP分析的经典双酶切组合[36],优于HindIII和MseI的双酶切组合。
2.2.2 分离菌株的遗传多样性分析
如图3所示,选择性荧光标记引物E1-M1 FAM组合,可以将分离自昌黎产区5 个具有代表性酒庄分离的222 株具有表型差异的O. oeni,区分为221 个AFLP遗传型,相似性系数为74%~98%。分离株1-02和9-51分别来自朗格斯赤霞珠和茅台赤霞珠,与其他分离株的相似性系数为74%,是最低的。分离株3-07分离自华夏赤霞珠,与其他菌株的相似性系数为75%,居其次。分离自金士小味儿多的106 株O. oeni,有105 个AFLP遗传型,它们的最小相似性系数为78%,而菌株11-105和11-106的亲缘关系最近,其相似性系数为98%。相对而言,分离自仁轩赤霞珠的25 株O. oeni的最小相似性系数为82.5%,高于分离自其他酒庄分离株之间的最小相似性系数。结果表明,分离自5 个不同酒庄的222 株O. oeni展现了菌株间丰富的遗传多样性。
由此可见,荧光标记AFLP技术对分离自不同酒庄的O. oeni菌株具有强大的遗传分型能力,为筛选不同酒庄内O. oeni优良菌株提供了技术方法。脉冲场凝胶电泳之前被认为是O. oeni最为有效的分型技术,然而,该方法对地缘关系较近的菌株不能实现有效区分,且该技术操作要求高,实验结果不够稳定[37-38]。随机扩增片段长度多态性DNA-PCR在O. oeni分型研究中,具有经济、快速和高灵敏性的特点,但是存在低重现性的缺陷[39,5]。数目可变串联重复序列分析技术展示了强有效的遗传分型能力[40-41],然而,由于目前对重复序列的遗传功能的认识还有限,基于重复序列的遗传分析受到微生物学家的质疑[42]。基于RNA水平的差异显示PCR技术受环境影响大,重现性差[26]。本实验中,基于全基因组进行的双酶切荧光标记AFLP指纹图谱技术,精确度高,分辨力强且经济简便,不仅将分离自昌黎产区不同酒庄的O. oeni菌株实现有效的分型,且对来自同一酒庄的O. oeni菌株也可实现分型。

图3 昌黎产区222 株O. oeni的AFLP遗传图谱分析Fig. 3 UPGMA dendrogram derived from AFLP patterns of 222 O. oeni strains from Changli
高通量测序技术使得对O. oeni在全基因组水平实现遗传分型和发掘其遗传进化规律成为可能[43]。然而,酒类酒球菌基因组较小,基于高通量技术积累的丰富数据,如何进行深入挖掘和生物信息学分析,还存在较大的挑战[44-45]。此外,在O. oeni菌株水平上进行遗传分型的研究中,高通量测序还存在成本高的问题。因而,具有经济简便、分辨力强的分子标记技术,如荧光标记AFLP技术在O. oeni的遗传进化分析中仍具有不可取代的作用。
2.2.3 分离菌株的遗传发育分析
基于UPGMA分析方法,对分离自昌黎产区的222 株O. oeni构建的系统发育树见图3。在相似性系数为81.7%处,222 株分离株呈现出清晰的A和B两个主要的进化类群。这与国内外基于序列和全基因组分析得出的O. oeni遗传进化规律是一致的[34,46]。在这2 个主要类群中,A类群菌株全部分离自朗格斯酒庄、仁轩酒庄和茅台酒庄,说明这3 个酒庄的O. oeni菌株具有相近的进化关系;而B类群菌株来自金士酒庄和华夏酒庄,说明它们之间的进化关系较为紧密。事实上,金士酒庄和华夏酒庄同属昌黎产区的碣石山小产区,地理位置和气候特点较其他3 个酒庄更为接近。在A类群中,存在A1、A2、A3、A4和A5共计5 个清晰的簇群结构,A1(8/9)和A5(17/18)簇群菌株主要来自仁轩酒庄,A2(5/5)簇群的菌株分离自朗格斯酒庄,A3(23/26)和A4(8/8)簇群主要分离自茅台酒庄,说明这些进化簇群与它们的分离起源具有清晰的遗传关系,同时,分离自同一酒庄的O. oeni菌株间也存在明显不同的进化发育途径。在B类群种,这种进化发育规律尤为明显,B1(99/102)簇群几乎均分离自自金士酒庄,B2(5/5)和B3(21/21)簇群全部分离自华夏酒庄。在系统进化树上,分离自同一酒庄的O. oeni菌株种群形成独特的簇群结构,揭示了分离自昌黎产区的O. oeni分子遗传发育规律与其分离起源存在极为典型的特异性关系。
此外,分别来自朗格斯赤霞珠和茅台赤霞珠的分离株1-02和9-51处于系统进化的最底层,均来源于17 a树龄的赤霞珠(昌黎产区最早建立的酒庄酒酿酒葡萄基地),或许是昌黎产区O. oeni的系统发育起始菌株。
Knight等[13]通过风味化学分析,验证了具有区域代表性的酿酒微生物直接对本区域葡萄酒风格特色的影响,提出酿酒微生物属于葡萄酒“风土”的概念。El Khoury等[14]提出,酿酒微生物是否属于葡萄酒“风土”组成,从微生物学的角度看,意味着是否存在遗传独特、起源不同的微生物种群结构,对6 个世界知名葡萄酒产区的O. oeni菌株种群进行生物地理学分析,在分子遗传学上,发现不同葡萄酒产区存在独特的O. oeni的种群结构,推断O. oeni和酿酒酵母等酿酒微生物同样属于葡萄酒“风土”的重要组成之一。然而,其研究并未发现O. oeni种群结构在地理起源上存在遗传特异性。本研究分离自昌黎产区不同酒庄的O. oeni种群结构,在分子水平上呈现出清晰的遗传发育和分离起源的特异性关系,进一步证实O. oeni资源是葡萄酒“风土”的重要组成之一。
3 结 论
本研究对分离自昌黎产区5 家具有代表性酒庄的222 株具有表型差异的O. oeni菌株,进行荧光标记AFLP指纹图谱分析,区分为221 个AFLP遗传型,证实荧光标记AFLP技术具有极强的遗传分型能力,揭示了昌黎产区O. oeni丰富的遗传多样性。通过UPGMA分析方法,构建分离的O. oeni系统发育树,可以清晰地看到2 个主要的进化类群,极其重要的是,分离自同一酒庄的O. oeni菌株种群形成了独特的簇群结构,呈现出遗传发育和分离起源的典型特异性关系,证实了O. oeni资源是葡萄酒“风土”的重要组成之一。研究结果证实了昌黎产区存在丰富的O. oeni资源,且不同酒庄的O. oeni资源呈现出典型的遗传特异性,这为开发我国具有地域特色的O. oeni资源提供了技术和理论支持。
