基于高通量测序开发玉米高效KASP分子标记
2019-05-31陆海燕王凤格
陆海燕 周 玲 林 峰 王 蕊 王凤格 赵 涵,*
1江苏省农业科学院 / 江苏省农业生物学重点实验室, 江苏南京 210014; 2北京市农林科学院玉米研究中心, 北京 100097
玉米是重要的粮食及饲料作物, 由于具有较高的遗传多样性已成为重要模式作物[1]。其核苷酸多态性变化是所有作物中较高的, 不同材料基因组之间的差异达到 1.42%, 大于人类与黑猩猩之间的差异(1.34%)[2]。丰富的基因组变异为开发具有高多态性分子标记提供了可能。根据变异序列的长短将基因组变异分为单核苷酸变异(SNP)及插入缺失(Indel 2~50 bp)和>50 bp的序列重排(SV)[2]3种类型。基于Indel和 SV开发的标记不仅检测通量低, 而且成本相对较高[3]。SNP标记具有分布广泛、遗传稳定性好且易于实现自动化等优点, 目前已迅速取代传统的分子标记[4-5]。
高通量测序技术的快速发展, 有效推动玉米基因组结构变异的研究。Jiao等[6]利用重测序技术对6个玉米自交系进行DNA结构变异分析, 发现中国玉米自交系中存在大量SNP位点的变异, 报道揭示了玉米基因组中超过100万个SNP。2018年, Sun等[7]报道了 Mo17的从头基因组装配, 提供了又一个重要的玉米参考基因组序列。这一个参考基因组的产生为广泛比较玉米中种内基因组多样性提供了前所未有的机会, 通过比对B73和Mo17基因组, 发现了980多万个 SNP位点、140多万的插入/缺失, 该研究为开发基于SNP位点的分子标记提供了理论依据和数据支撑。SNP是单核苷酸变异产生的多态性, 无法通过常规的PCR技术与凝胶电泳技术利用长度的差异来区分其多态性[8]。目前尽管开发了多种 SNP基因分型方法, 包括定点测序技术、等位基因特异性PCR (AS-PCR)、裂解扩增多态性序列(CAPS)、温度开关 PC (TS-PCR)和基因重测序[9-11], 但这些方法都存在低通量或高成本等局限性[12]。近些年, 高通量的SNP位点检测几乎都采用芯片技术。赵久然等[13]利用基于 SNP位点的芯片技术揭示中国玉米育种种质的遗传多样性与群体遗传结构。王文斌等[14]利用含有2846个高质量SNP位点的芯片进行了31份骨干自交系群体遗传结构、遗传多样性分析。基于芯片技术的SNP基因分型平台的通量较高, 每张芯片包含几百到数百万的SNP位点[15], 但大多数基于芯片的基因分型平台用于样本量较多的全基因组扫描时成本较高[16]。
KASP (Kompetitive Allele Specific PCR), 即竞争性等位基因特异性PCR技术, 以其高度稳定性、准确性和低成本的特点, 已经被广泛应用于高通量SNP分型, 尤其在样本量大, SNP位点少时, KASP的应用特点最为显著[17]。Rasheed等[12]开发70个小麦 KASP分子标记, 并成功验证了这些标记与品种亲本和双亲本群体的表型相关, 揭示了 KASP分子标记在小麦育种中的潜在应用。国际玉米和小麦改良中心使用 KASP, 每年产生超过一百万个数据点用于作物改良[18]。KASP在小到中等数量的标记应用中显示出了成本效益和可扩展的灵活性, 如在双亲本群体的质量控制分析、标记辅助的轮回选择、辅助回交和QTL精细定位等方面发挥重要作用[17]。KASP分子标记的开发需要综合考虑目的区段DNA的唯一变异, 多态性以及位点特异性等因素, 基于重测序数据可以筛选出目的区段100 bp内只有一个SNP位点的变异, 而通过高密度芯片筛选的位点无法保证唯一性。
本研究利用遗传来源不同的205份玉米自交系的全基因组重测序信息, 筛选出多态性高的二态性SNP位点, 基于这些位点以及侧翼序列信息开发KASP分子标记, 并利用其中46个代表玉米自交系进行验证, 获得了一套在染色体上均匀分布且多态性高的KASP分子标记。这些标记可以为玉米SNP基因分型研究提供重要的基础和分析方法, 同时为种质改良及杂交种组配提供DNA水平上的信息。
1 材料与方法
1.1 材料及全基因组测序数据
选用来自 7个不同类群的 205份玉米自交系,其二代原始序列下载于NCBI (https://www.ncbi.nlm.nih.gov/)的 3个 SRA (Sequence Read Archive)数据库(PRJNA82843、SRP011907和 PRJNA260788)。删除其中低测序深度的材料。利用生物信息学分析技术比对这些材料的序列, 选择最小等位基因频率(MAF)大于 0.05的位点, 删除基因型缺失率超过20%的位点, 以哈迪温伯格平衡显著性阈值(HWE)P<0.001为标准再次过滤, 最终保留多态信息含量(PIC)>0.4并且是二态性的目的SNP位点[19-20]。
从 7个类群中, 分别抽出一定数量的代表自交系(详细信息见表1)为实验材料, 用于KASP验证。
1.2 标记开发和筛选
根据 SNP位点和侧翼序列设计 PCR扩增引物,开发KASP分子标记。每个标记设计2条SNP特异性引物(F1/F2)和一条通用引物(R), F1尾部添加能够与FAM 荧光结合的特异性序列, F2尾部添加能够与HEX荧光结合的特异性序列。KASP引物设计由mIndel软件包[21]完成, 引物由生工生物工程(上海)股份有限公司合成。
对46份代表自交系的叶片进行取样, 按照上海浦迪植物基因组提取试剂盒操作步骤提取基因组DNA。KASP 反应总体系为 5 μL, 包含 2×KASP Master Mix 2.5 μL、KASP Assay Mix (引物混合工作液) 0.07 μL、浓度为 20 ng μL-1的模板 DNA 2.43 μL。KASP 反应程序为, 第一步: 94℃, 15 min; 第二步:94 ℃ , 20 s, 61~55 ℃ , 1 min,每个循环降低0.6℃, 共进行10个循环; 第三步: 94 ℃ , 20 s, 55 ℃ , 1 min,共进行 26个循环, PCR在购买自 LGC公司型号为Hydrocycler 16的水浴PCR仪中进行。通过KASP荧光分析仪(LGC公司型号为PHERAstar plus)扫描分析PCR结果。根据基因型分型效果和在玉米基因组染色体上的分布等信息, 挑选有效的KASP标记。
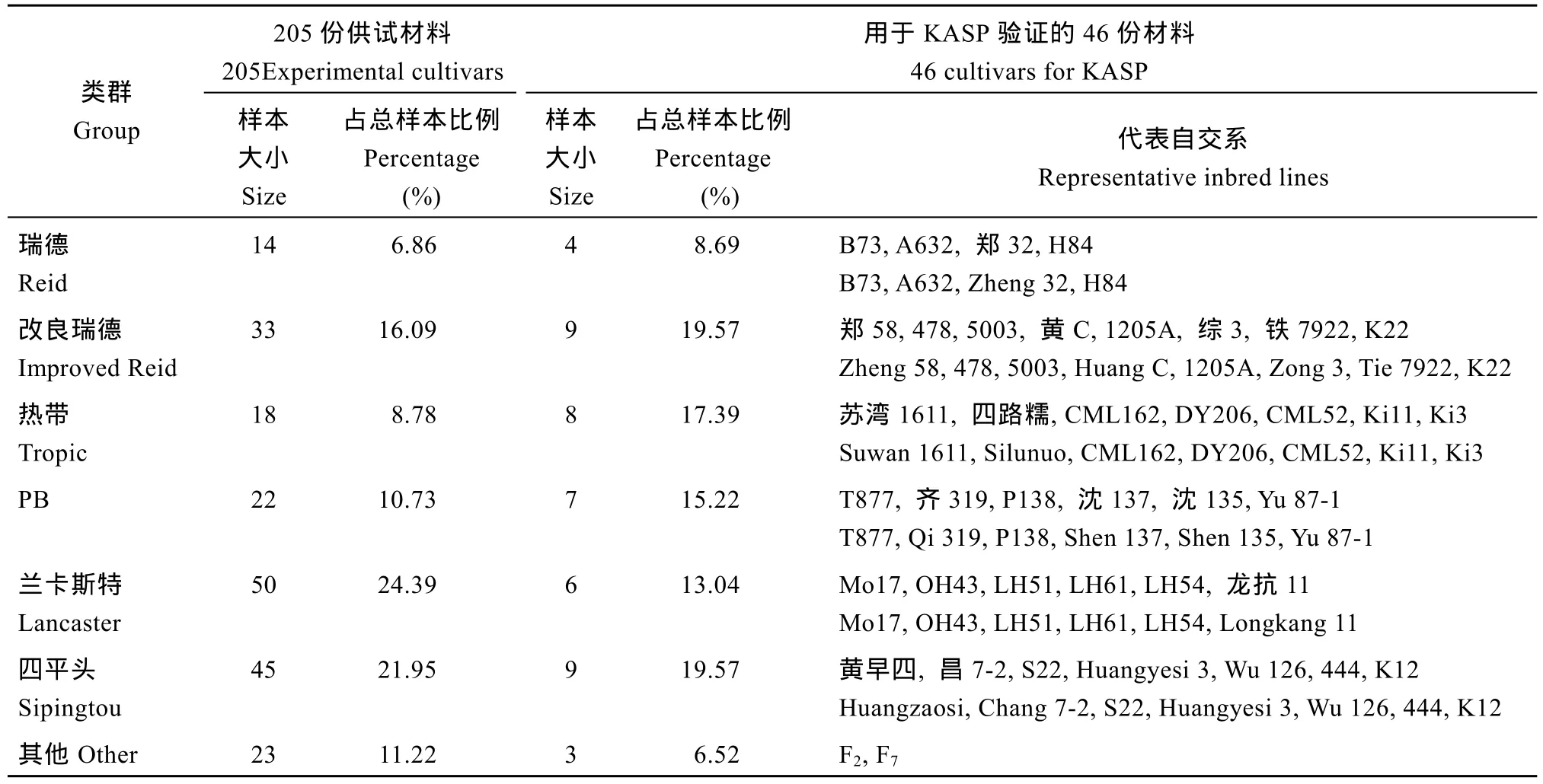
表1 实验材料详细信息Table 1 Details of experimental materials
1.3 数据分析
采用编写的Perl脚本和VCFtools软件计算最小等位基因频率(MAF)、多态信息含量(PIC)[20,22]。利用TASSELV5.0软件的邻接算法(N-J)[23]计算205份玉米自交系之间的遗传距离并构建聚类图。运用 R软件的cor[17]函数包对205份多样性自交系的总SNP位点与实验验证筛选出的KASP标记位点进行遗传相关性分析。
2 结果与分析
2.1 SNP位点的筛选和KASP分子标记的开发
利用 205份玉米自交系全基因组测序数据开发SNP位点, 对总 SNP位点进行过滤(缺失率<20%,MAF>0.05, PIC>0.4), 共筛选出 1,660,336个二态性SNP位点, 这些位点均匀分布在玉米基因组的10条染色体上。采用Perl脚本进一步筛选出SNP位点前后各100 bp没有其他变异位点的SNP, 通过多窗口比对, 选择在不同材料之间具有保守型的SNP位点。经过引物软件设计, 开发出700个KASP分子标记。
根据开发设计的 700对 KASP引物, 对 B73、Mo17、昌7-2、郑58等46份玉米自交系进行基因型分型。获得了近490个高质量的分子标记引物, 这些标记位点可以准确清楚地将相同基因型的自交系聚类(聚类形式如图 1), 同时阴性对照 NTC (No Template Control)始终聚集在一起, 且不产生信号, 成功率达到70%。其他引物被淘汰的原因主要包括可以将相同基因型的样本聚类, 但是聚集略有分散, 或是同样可以分类但在一些基因型中的扩增效率不高等。
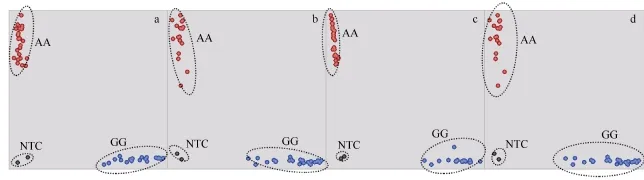
图1 KASP标记基因分型图Fig. 1 Genotyping map of KASP markers
2.2 KASP分子标记的挑选和评价
为了进一步筛选出具有代表性的标记, 对鉴定出的490个标记进行过滤, 主要依据能够将HEX型和 FAM 型的基因型精确分型且 MAF>0.40、PIC>0.45, 以及在染色体上的位置分布均匀等信息, 最终筛选出202个位点(引物信息见附表), 这些位点覆盖在玉米基因组的10条染色体上, 平均间隔为8.74 Mb (图2-A)。利用Perl脚本对这些位点的等位基因频率等方面进行总体评价, 202个SNP位点PIC变化范围为0.458~0.469, 平均值为0.463, MAF变化范围为 0.434~0.467, 平均值为 0.451 (图 2-B), 表明 202个SNP位点具有高特异性和高稳定性。

图2 202个SNP位点遗传多态性Fig. 2 Genetic polymorphisms of 202 SNPs
2.3 KASP分子标记在品种鉴定中的应用
基于实验获得的202个KASP标记位点和重测序数据获得的1,660,336个二态性SNP位点, 分别对205份材料构建了系统发育树。图3显示, 基于两组不同数据构建的进化树均将205份自交系划分为 7大类群, 分别是瑞德群(代表自交系 B73)、改良瑞德群(代表自交系郑 58)、热带群(代表自交系Ki11、Suwan 1611、Four-row)、PB 群(代表自交系T877、Qi 319)、兰卡斯特群(代表自交系 Mo17、OH43)、四平头群(代表自交系黄早四、昌7-2)、其他种质群(代表自交系 F2、F7)。类群划分结果高度一致, 其中201份材料的划群结果相同, 4份材料的划群结果不同。图3-A显示, 205份自交系中用于KASP验证的46份代表自交系, 分别分布于各自的类群中, 46份自交系的类群划分与系谱来源相一致。表明202个KASP标记具有较好的重复性, 能够代替总SNP位点进行类群划分。另外, 利用R语言对 KASP标记位点与重测序数据筛选的总 SNP位点进行了遗传距离相似性分析, 两者之间的遗传距离相似性高达 89.5%, 进一步表明 202个 KASP标记在鉴定玉米种质资源研究方面具有可靠性和代表性。
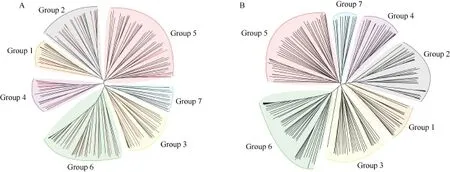
图3 Neighbor-joining (N-J)树分析Fig. 3 Neighbor-joining (N-J) tree analysis
3 讨论
玉米是一种经典的遗传模型, 是世界范围内的重要作物。玉米基因组在不同自交系间具有高度的遗传多样性。SNP标记具备二态性、等位基因性、高密度性、高代表性、高遗传稳定性等优点, 是构建生物高通量基因分型适合的标记。基于测序数据开发KASP标记不仅有大量SNP变异位点作为数据支撑, 而且能够筛选到唯一SNP位点变异的目的片段, 此外通过参数的设置和多窗口比对能够选择在不同材料之间具有保守性的位点。但是基于测序数据开发KASP标记的方法不仅要求基因组信息的准确性, 测序数据的分析和有效SNP位点的筛选等相关技术往往也需要配备生物信息学专业知识较强的科研人员, 因此目前关于高效 KASP标记的开发研究较少。
本研究基于 205份自交系重测序数据, 过滤出1,660,336个SNP位点, 根据位点侧翼序列信息, 将其中700个位点开发为KASP标记。利用KASP平台验证, 获得了较高设计成功率, 主要原因是基于大量的重测序数据, 不适合用来设计 KASP引物的位点在前期已经被过滤, 同时又对位点进行多态性评估, 这些步骤可以极大提高KASP引物设计效率。但是, 有些位点由于结构复杂, 测序深度浅, 或者序列信息存在错误, 可能导致一些引物设计失败。InDel (insertion-deletion)变异是同一位点不同大小片段的插入缺失, 变异结构比 SNP变异复杂, 一些结构复杂的位点将来可以开发成基于 InDel变异的KASP标记。
我们根据490个标记在玉米基因组染色体上的分布位置、多态信息含量等信息, 从中挑选出能对玉米品种特异性和真实性有效鉴定的 202个 KASP标记。205份材料的划群结果显示其中 201份材料划群结果相同, 表明202个位点与总SNP位点的聚类分析结果高度一致, 且 202个 KASP标记的位点与总SNP位点的遗传距离相似性系数达到89.5%。本研究结果证明202个KASP标记位点能够代替总SNP位点应用在玉米种质资源鉴定等方面的研究。
KASP检测基因分型可以应用于分子辅助育种、种质资源鉴定、样本群体分析、性状基因的精细定位、全基因组关联分析等。何中虎等人[24]开发了一套基于 KASP技术检测小麦功能基因的引物; 余四斌及其团队[25]基于KASP技术开发了用于水稻特殊营养物质、水稻产量等基因分型的引物。本研究开发的玉米高效KASP分子标记能够用于玉米群体多样性分析、群体结构分析、玉米种质资源的鉴定等研究, 大大减少了后期田间工作量, 可为提高玉米育种效率提供参考。
4 结论
基于高通量测序开发的 202个 KASP标记, 在染色体上分布均匀, 平均间隔8.74 Mb, PIC和MAF的平均值分别为0.463、0.451, 能成功区分玉米的杂种优势群。KASP标记将在玉米杂种优势群划分、连锁群构建、种质资源遗传背景确定等方面发挥作用。
附表 请见网络版: 1) 本刊网站 http://zwxb.chinacrops.org/; 2) 中国知网 http://www.cnki.net/; 3) 万方数 据 http://c.wanfangdata.com.cn/Periodical-zuowxb.aspx。
