棉花光籽基因n2的精细定位
2019-04-10李思敏左东云程海亮张友平王巧连刘珂冯晓旭耿洪伟宋国立
李思敏,左东云,程海亮,张友平,王巧连,刘珂,冯晓旭,耿洪伟,宋国立*
(1.新疆农业大学,乌鲁木齐830052;2.中国农业科学院棉花研究所/ 棉花生物学国家重点实验室,河南安阳455000)
棉花(Gossypium)优质高产一直是育种的重要目标[1-2]。一般认为纤维细胞是以线性细胞生长模式进行延伸,由胚珠外表皮细胞发育而成[3-4]。棉花纤维的分化和发育包括原始细胞的分化和突起(开花后-3~3 d)、纤维细胞的伸长(开花后2~20 d)、次生壁加厚(开花后15~45 d)和脱水成熟(开花后45~50 d)4 个阶段,各个阶段之间相互重叠,没有明显的界限,纤维细胞的分化时间决定纤维的长度,长纤维在早期分化,而短纤维在晚期分化。棉纤维的品质性状主要包括纤维长度、强度、伸长率、马克隆值等,这些性状主要受基因型的控制,其中纤维的起始与伸长直接影响纤维的数量与长度,而次生壁加厚期纤维素的合成则影响纤维的强度与马克隆值等性状[5]。
棉花纤维发育过程与拟南芥(Arabidopsis)毛状体发育具有相似性。现已证明,MYB 家族蛋白在拟南芥毛状体发育和棉纤维生长发育中都有重要作用,而复合体MYB-bHLH-WD40(MBW)控制拟南芥毛状体的启动,主要包括GL1、GL3和TTG1 这3 个基因[6-10]。控制棉纤维起始的关键转录因子基因(MYB25-like)编码MIXTA 类的MYB 转录因子,与形成MBW 的GL1 属于不同类型,在纤维发育过程中不可缺失;干涉Gh-MYB25-like基因,可导致棉花种子无纤维,其他部分毛状体不受影响[11-14]。目前,棉花中存在多种纤维发育突变体,主要有陆地棉无绒有絮突变体N1和n2,超短纤维突变体Li1、Li2和Li3,无绒无絮突变体MD17、SL1-7-1 和XZ142。从20 世纪初,前人就开始了对陆地棉光籽突变体的遗传研究。Thadania 等研究认为,陆地棉光籽无短绒性状是单基因控制的显性性状[15-16]。Kohel 等分别把显性光籽基因N1和隐性光籽基因n2定位在同源染色体12 和26 号上[17]。Endrizzi 等采用单端体定位的方法,将光籽基因n2定位到染色体D12 上[18]。而Rong 等认为n2位于染色体A12 上[19]。宋丽等在海陆群体中将n2定位在染色体A12 上,在陆陆群体中将n2定位在了D12 上[20]。
本研究以陆地棉光籽突变体n2和海岛棉新海21 为亲本配制杂交组合,收获F1种子,并自交产生F2群体,对该群体纤维发育性状进行调查和遗传分析,完成光籽基因n2的初步定位和精细定位,对候选区间进行基因预测,克隆候选基因,并分析其在亲本中的表达差异,为揭示n2基因控制棉花纤维起始的分子机制提供理论基础。
1 材料与方法
1.1 试验材料与表型数据收集
本试验配制杂交组合所用亲本材料为海岛棉品种新海21(Xinhai 21)和陆地棉光籽突变体n2,由中国农业科学院棉花研究所种质中期库提供,并由本课题在田间进行多年连续自交保存。2个亲本纤维发育性状稳定,新海21 的表型为短绒,n2的表型为光籽。用其配制的F1也经过多年调查,纤维发育表型性状同新海21 一致。
本试验共构建2 个F2群体,群体Ⅰ由554 个单株组成,群体Ⅱ由3 443 个单株组成。表型性状分为有短绒和无短绒(光籽)2 种。选取F2群体各单株上成熟开裂的棉铃,去除长纤维后观察棉籽上短绒的有无,每次均与亲本比对,与新海21 表型一致的标记为有短绒,与n2表型一致的标记为光籽。但在分离群体中,有一些处于中间表型的单株,其种子表面大部分为光籽,仅在珠孔端和合点端有少量短绒,在性状调查时,参照Du 等[21]和丁业掌等[22]的分类方法,将其归为光籽。进行卡方适合性检验,以确定F2群体性状是否符合3∶1 的分离比。
1.2 DNA的提取和聚合酶链式反应(Polymerase chain reaction,PCR)
采用宋国立等[23]改良的CTAB 法从亲本和F2群体的新鲜叶组织中提取基因组DNA。所有引物由苏州金唯智生物科技有限公司和生工生物工程(上海)股份有限公司合成。配制PCR 反应体系所用到的TaqDNA 聚合酶、10×PCR Buffer 以及dNTPs 等试剂均购自北京全式金基因生化科技有限公司。PCR 反应体系中模板DNA 30 ng,2.5 mmol·L-1dNTPs 0.5 μL,10×PCR Buffer 1 μL,5 U·μL-1TaqDNA 聚合酶0.1 μL,10 μmol·L-1正反向引物各0.2 μL,加ddH2O至10 μL。PCR 扩增反应程序:94 ℃5 min;94 ℃30 s,55 ℃30 s,72 ℃1 min,25 个循环;72 ℃5 min。PCR 产物用8%(质量分数)的非变性聚丙烯酰胺凝胶电泳分离。
1.3 分子标记的开发
宋丽等[20]将n2基因定位在A12 上标记NAU0943 和BNL1679 之间的6 cM 区间内。利用软件SciRoKo 34 在A12 序列中查找简单序列重复(Simple sequence repeat,SSR)位点,并将得到的引物于2 个亲本间进行多态性筛选。当n2基因缩小到没有可用的多态性SSR 标记的区间范围后,对任意序列进行扩增,用于加密图谱。所有引物序列均由Primer 5 软件获得。
1.4 基因的精细定位
从群体Ⅰ中挑选200 个单株进行初步定位,其中包括150 株毛籽单株,50 株光籽单株。利用Joinmap 4.0 进行连锁分析。通过作图将n2基因定位到标记NAU0943 和BNL1679 之间的6 cM(centimorgan)区间后,根据SSR 在染色体上的位置提取目标区间的序列,继续设计新的SSR 引物,并筛选出具有多态性的引物,用得到的引物扩增群体Ⅱ所有单株的DNA,根据重组数据,将目标区域锁定在更小的范围内。
1.5 定位区间候选基因预测
根据精细定位的结果得到候选区间,从棉花注释数据库(http://cgp.genomics.org.cn)获得预测的基因序列,并在表达序列标签(Expression sequence tag,EST)数据库中进行BLAST 比对以确认预测基因的准确性,进而获得候选基因。
1.6 候选基因的表达量分析
采集开花-3 d、-1 d、0 d、1 d、3 d、5 d、7 d、10 d 的胚珠,用天恩泽基因科技公司的RNAout 2.0 试剂盒提取总RNA。反转录所用试剂盒PrimeScriptTMⅡ1st strand cDNA Synthesis Kit(TaKaRa)和荧光定量试剂盒TaKaRa SYBR Premix ExTaqⅡ(Perfect Real Time)均购自宝生物(大连)生物公司。实时荧光定量(Quantitative real time,qRT)-PCR 反应体系:模板cDNA 2 μL,10 μmol·L-1正、反向引物各0.8 μL,SYBR®RPremix ExTaqⅡ (2×)10 μL,ROX Reference DyeⅡ(50×)0.4 μL,ddH2O 6 μL。PCR 反应条件:95 ℃30 s;95 ℃5 s,60 ℃30 s,40 个循环;95 ℃15 s,60 ℃1 min,95 ℃15 s(ABIPRISM@7500-Fast Real-Time PCR system)。根据基因序列设计qRT-PCR 引物,内参基因设为ACTIN,引物信息参见表1。表达量分析用2-ΔΔCt法。

表1 qRT-PCR所用基因及引物序列Table 1 Genes and primers used in qRT-PCR verification
2 结果与分析
2.1 性状调查及遗传分析
对2 个F2分离群体的3 997 个单株进行田间性状调查,对表型数据进行统计分析,得到光籽单株690 株,毛籽单株2 994 株,中间性状单株313株(归为光籽类型[21-22])。结果显示,2 个群体的纤维发育性状分离比为3∶1。卡方适合性检验结果显示,分离群体的性状分离比符合孟德尔遗传分离规律(χ2=0.59<χ20.05=3.84),与前人的研究结果[20]一致。
2.2 SSR标记的开发与筛选
结合AD 基因组序列,首先在70~76 Mbp 区间内设计了96 对引物,均分布在A12 上。将各引物在2 个亲本间进行多态性筛选,共得到20 对多态性SSR 标记。利用这20 对引物进行连锁分析,得到13 对与目标基因连锁的标记(表2)。当n2基因缩小到没有可用的多态性SSR 标记区间范围后,对任意序列进行扩增,在亲本间进行多态性筛选,得到10 对与目标基因紧密连锁的SSR 标记(表2)。
2.3 光籽基因n2的初步定位
宋丽等[20]将n2基因定位在A12 上标记NAU0943 和BNL1679 之间的6 cM 区间内。本试验在进一步加密遗传图谱的基础上,验证了该区间的准确性。从群体Ⅰ中挑选200 个极端单株的小群体,其中包括50 株光籽单株,150 株毛籽单株,结合已有的结果和新的SSR 标记在该群体中的交换率,将目标区域锁定在了NAU0943 和Z10之间,物理距离约为615 kbp(图1a)。

表2 与n2基因连锁的标记Table 2 Polymorphic markers linked with the n2 gene
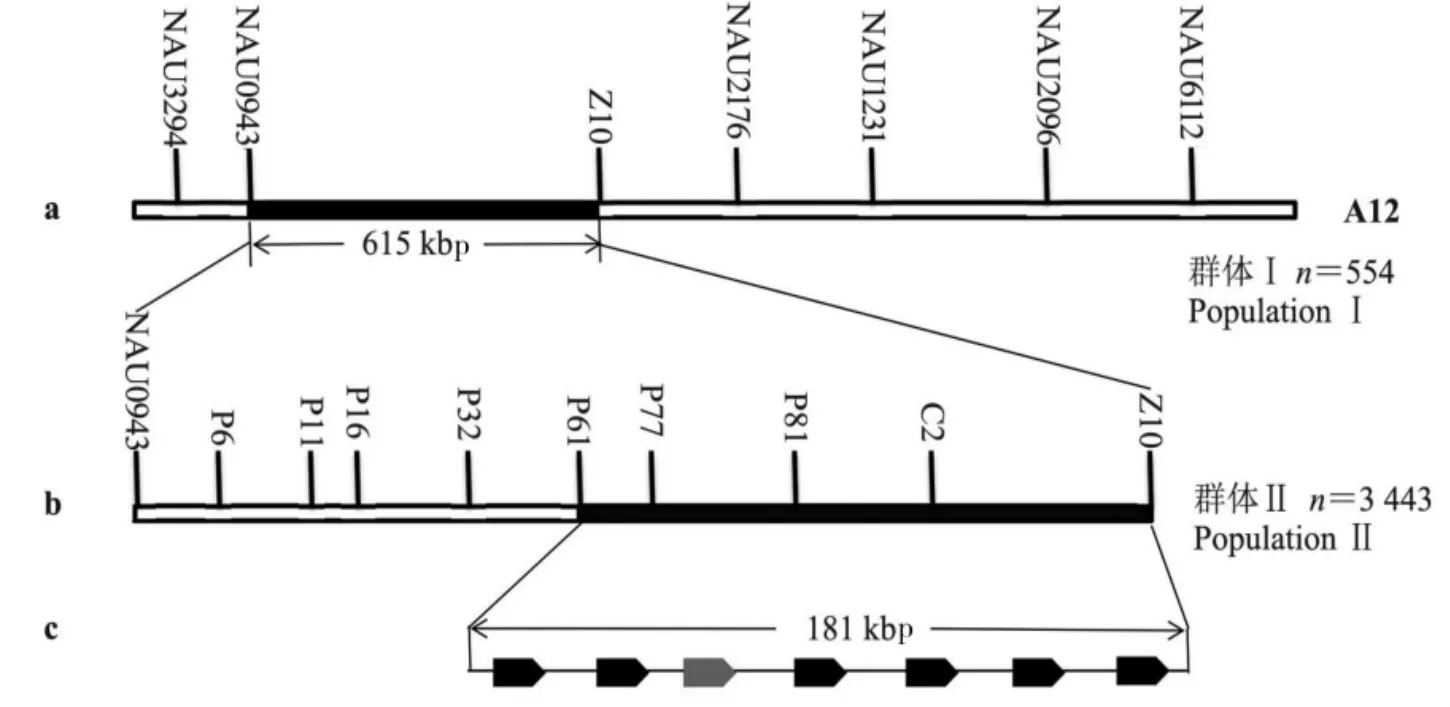
图1 n2基因的定位Fig.1 Mapping of n2 gene
2.4 光籽基因n2的精细定位
初步定位得到10 个与目标基因紧密连锁的SSR 标记,为验证定位区间并进一步缩小n2的范围,以这10 个标记扩增群体Ⅱ所有单株的DNA,根据重组数据,进一步将候选区间缩小至P61 与Z10 之间(图1b,c),物理距离181 kbp。
2.5 候选区间基因预测
以候选区间的基因组参考序列进行基因预测,结合基因组的注释结果,在181 kbp 的序列中存在7 个预测基因,分别为1414、1415、72098、1417、1418、1419、1420(表3)。
2.6 候选基因的表达量分析
候选基因中,72098 在所选时期的2 个亲本中的表达量都有显著差异(图2),而其他6个基因在2 个亲本中的表达量没有显著差异。这说明在棉花胚珠中,该基因表达量的不同可能是造成纤维发育性状差异的原因,它可能在光籽形成中起重要作用。

表3 定位区间内7个候选基因的注释及基因全长Table 3 Annotation and full length of seven candidate genes in mapping interval

图2 候选基因72098在2个亲本中的相对表达量比较Fig.2 Expression analysis of the candidate gene 72098 in the parents
3 讨论与结论
棉花纤维作为天然纺织原料在国民生产中举足轻重,而纤维突变体,尤其是光籽突变体更是棉花遗传育种上重要的种质资源。陆地棉纤维突变体的遗传研究,可以丰富陆地棉种质资源。而棉种上附着的短绒纤维往往可能携带许多病菌,易造成各类病害(如黄萎病等)的发生[24]。如果棉种上有短绒,则会严重阻碍种子的吸水膨胀,从而影响发芽率,使棉花生产受到一定的影响。而光籽突变体材料具有种子不易传播病虫害且吸水快,出苗整齐,纤维强度好,纤维容易从种子上分离等优点,是棉花育种的重要资源。该研究以陆地棉光籽突变体为切入点,对棉纤维材料进行生物学研究,进而阐明棉纤维分化发育的分子机理,对进一步提高棉纤维产量和品质具有重要意义。
陆地棉纤维发育突变体一般是由于1 个或少数几个质量性状基因发生突变造成的。前人研究表明,n2位于 A12 上标记NAU0943 和BNL1679 之间的6 cM 区间内[20]。但该距离对于基因克隆或育种利用来说不够接近。随着棉花基因组序列的发展,利用高分辨率遗传图谱和全基因组序列,大大加快了基因定位和克隆的步伐[25-27]。在本研究中,我们选取陆地棉光籽突变体n2及海岛棉品种新海21 为亲本,为了确保结果的准确性,构建了2 个分离群体,分别用于n2基因的初步定位和精细定位,定位群体达3 997 个单株。根据棉花基因组序列,开发了一批染色体特异的分子标记,当目标区域锁定到较小范围之内后,根据重组单株的个数,确定目标基因的候选区间,并用连锁标记扩增群体Ⅱ所有单株的DNA,以确保结果的准确性。原则上,如果群体数目增大,得到的重组单株数目就越多,定位区间也越小。最后,n2基因被定位在染色体A12 上181 kbp 区间内,定位区间中含有7 个候选基因,并通过检索EST 库得到确认。候选基因的qRT-PCR 分析结果显示,基因72098 在所选时期的2 个亲本中的表达量都有显著差异,而其他6个基因在2 个亲本中的表达量没有显著差异。
基因72098 编码蛋白激酶。据报道,拟南芥中含有多个钙依赖蛋白激酶(Calcium-dependent protein kinases,CDPKs)基因,其中有成员被报道与根毛生长或花粉管通道伸长有关。cDNA 芯片及qRT-PCR 的数据均显示CPK1、CPK32 和CPK5 的表达在纤维伸长期显著升高。利用CDPK 的通用底物检测棉纤维发育不同时期钙离子依赖型蛋白激酶总活性发现,随着纤维的伸长,CDPK 总活性显著增加,在开花后10~15 d达到最高。在没有纤维的棉花突变体中,CDPK总活性一直维持在较低水平,提示CDPK 类激酶在纤维发育过程中扮演重要角色[28-29]。由此我们猜测,基因72098 可能在光籽形成中起重要的作用,且为候选基因的可能性较大。但光籽表型的差异是由于基因序列本身差异还是表达量差异引起的,亦或者是两者共同作用导致的,有待进一步验证。
