江华苦茶不同单株间亲缘关系的cpDNA序列分析
2017-10-10罗军武
成 杨,刘 振,杨 阳,罗军武
(1. 湖南农业大学 园艺园林学院,湖南 长沙 410128;2. 湖南省农业科学院 茶叶研究所,国家茶树改良中心湖南分中心,湖南 长沙 410125)
江华苦茶不同单株间亲缘关系的cpDNA序列分析
成 杨1,2,刘 振2,杨 阳2,罗军武1*
(1. 湖南农业大学 园艺园林学院,湖南 长沙 410128;2. 湖南省农业科学院 茶叶研究所,国家茶树改良中心湖南分中心,湖南 长沙 410125)
DNA序列分析在物种的系统进化、分类和鉴定等方面展示出了强大的生命力,其中cpDNA序列分析因具有基因组小、进化速率适中、母系遗传等特点,已被大量用于系统进化研究。本研究采用4对cpDNA引物对收集来的32份江华苦茶资源进行了亲缘关系研究,其中有3对测序成功。扩增序列长度分别为634(1F-724R)、499(rbcla-rev)、532(rbcla-ajf634)。变异位点数量分别为5(1F-724R)、4(rbcla-rev)、4(rbcla-ajf634)。变异率从高到低分别是rbcla-rev(0.80%)、1F-724R(0.79%)、rbcla-ajf634(0.75%)。将3对引物测序得到的序列拼接,按照MP法构建了分子系统树,将参试的32份茶树资源分为4大类。本研究为江华苦茶亲缘关系研究开辟了一条新途径,为江华苦茶资源的应用和保护研究提供理论参考。
江华苦茶 ;cpDNA;DNA序列分析
茶[Camellia sinensis (L.) O.Kuntze]属于山茶属(Camellia)茶组(Sect.Thea),为多年生常绿木本植物,起源于我国云南地区。由于世代异花授粉和人类的选择性种植,茶树种质资源的变异体繁多,亲缘关系较难确定[1-2]。
江华苦茶作为湖南四大典型地方茶树资源[3],茶多酚物质含量丰富,具有很高的开发利用价值[4]。但由于受交通、经费等因素的制约,江华苦茶入圃保存的资源量仍非常有限。随着人类生产活动的不断扩大,江华苦茶资源的入圃保存已非常必要。但是种质资源保护的前提是要了解各单株的遗传背景,以便以尽可能少的资源量保存最多的遗传多样性。
分子生物技术的发展为资源亲缘关系研究提供了更加便捷可靠的方法,而DNA序列分析是区分个体间遗传差异最直接的方法,可提供基因组特异区的完全遗传信息,为植物鉴定、分类等研究提供快捷的方法[5],并且已经在多个植物当中得到了运用[6-8]。作为DNA序列分析的一个部分,cpDNA序列有着相对保守、基因组较小、多拷贝、母系遗传的特点,使得叶绿体基因组序列被大量用于系统进化研究[9-11]。在茶树方面也已经有了这方面应用的报道,陈春梅[12]采用了3对叶绿体DNA序列进行了山茶属植物的亲缘关系研究。
本研究采用cpDNA序列分析技术对江华苦茶的不同单株进行比较分析,探讨江华苦茶单株间的亲缘关系和遗传多样性水平,以便为江华苦茶这一珍稀资源的保护和选育提供理论依据。
1 材料和方法
1.1 材料
本研究共采集到江华苦茶样品32个,对照为猪婆茶(gene22)。样品来源及编号见表1。所有样品均取一芽二叶新梢,液氮迅速冷冻处理,然后将其保存在-40℃冰箱中备用。

表1 32个参试的样品来源Table 1 Resource of 32 tested tea samples
1.2 方法
1.2.1 D N A的提取
采用试剂盒(天根生化科技有限公司,型号DP305-02)进行DNA提取,提取方法参照试剂盒说明书。
1.2.2 引物合成
参照高连明等[13]的引物序列,共选用4对cpDNA序列(表2),由天根生化科技有限公司进行引物合成。
1.2.3 P C R扩增体系
扩增程序见表2,扩增体系见表3。PCR扩增在Thermo-5020型PCR仪(Thermo Fisher Scienti fi c. Int’l trade Co., Ltd.)上进行,扩增产物通过1.2%琼脂糖凝胶电泳检测,PCR 产物送上海生工生物技术有限公司进行测序。
1.2.4 数据处理
使用dnastar软件进行序列拼接,先剪去序列两端不可靠的碱基序列和引物序列,然后再将正反两个引物的序列进行比对,对序列进行编辑后获得正反引物序列一致的DNA序列。通过MEGA6.0软件中的ClustalW 功能对所选材料的序列进行比对,适当手工校正,去除边缘不准确的部分序列,整理以待分析,计算变异碱基数[14]。采用MP法(most parsimonious tree,MP)构建分子系统树,对分支的可靠性评价使用靴带值(bootstrap,1000次重复)分析,自展数值75% ~ 100%表示强支持率,50% ~ 74%表示弱支持率,小于 50%表示不支持[15]。

表2 4对引物特征Table 2 The characterization of 4 pairs of primer
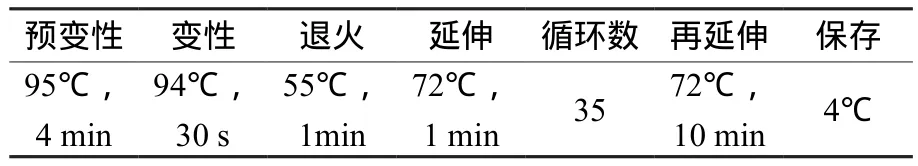
表3 PCR反应程序Table 3 The system of PCR reaction

表4 PCR扩增体系Table 4 The system of PCR ampli fi cation
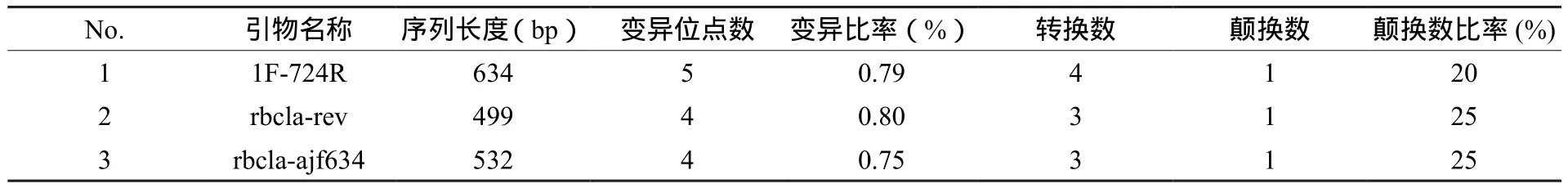
表5 DNA序列特征Table 5 DNA sequence characteristics
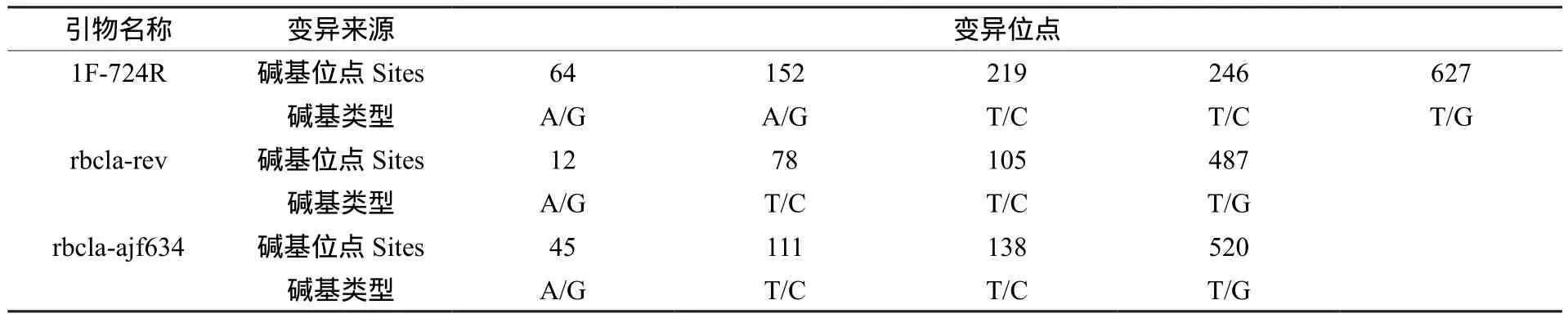
表6 不同引物的变异位点Table 6 Variable sites of different Primers
3 结果分析
3.1 不同序列的特征与差异
本研究共使用cpDNA序列引物4对,其中3对引物测序成功(1F-724R、rbcla-rev和rbclaajf634),trnH2-psbAF序列虽然获得了测序结果,但是由于出现poly结构,正反方向序列比对后多个样品中存在大量正反方向序列不一致的位点。同时该引物在之前的实验当中,出现了测序失败的情况,因此综合判断下,最后放弃这一引物,仅采用其他3对引物进行数据分析。测序成功的3对引物(表5),扩增序列长度分别为634(1F-724R)、499(rbcla-rev)、532(rbcla-ajf634)。变异位点数量分别为5(1F-724R)、4(rbcla-rev)、4(rbcla-ajf634)。变异率从高到低分别是rbcla-rev(0.80%)、1F-724R(0.79%)、rbcla-ajf634(0.75%)。
3.2 参试材料的亲缘关系
按照表5从1到3的引物顺序将测序所得到的3个序列拼接。采用MEGA 6.0软件按照MP法构建分子系统树(图1),将参试的32个样品分成4个大类。类群1包括7个样品,自展值达到97%,该类群与另外三个类群相互之间亲缘关系较远,而与外类群猪婆茶(gene22)靠的比较近。类群2为最大的类群,共有13个样品聚集在一起,自展值达到了88%。其中包括了从湖南省茶叶研究所茶树种质资源圃采集5个样品中的4个(gene23、gene 24、gene 25、gene 26)。类群3只有gene 2和gene 14两个样品,数量最少,自展值达到了96%。最后的类群4则由10个样品组成,包括采集自江华县码市镇的6个样品当中的5个(gene28、gene 29、gene 30、gene 32、gene 33),自展值达到83%。
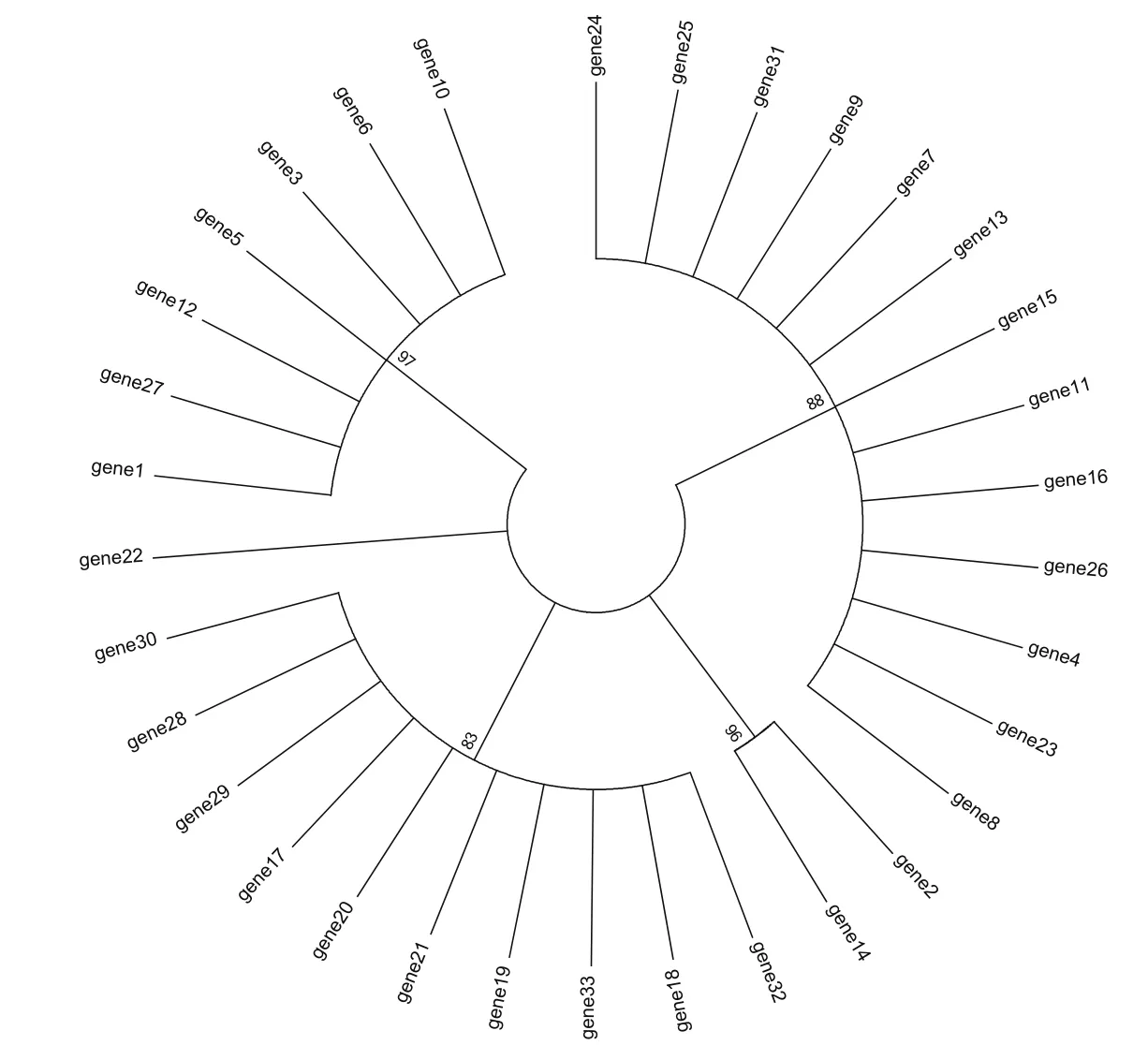
图1 基于3个序列和最大简约法(MP)构建的系统发育树 (图中数字表示抽样检验的自展百分比)Fig. 1 A phylogenetic tree constructed by maximum parsimony method with 3 sequences (The numbers in the fi gure indicate percentage of sampling inspection)
4 讨论
本研究共采用4个cpDNA序列,其中有3个获得可用条带,DNA碱基变异率的平均值为0.78%,小于本实验室前期在16份不同茶树变种间的变异率(4.25%,试验数据未发表)。这一方面说明群体资源的遗传多样性要远小于不同变种间的遗传多样性,本研究采用的试验材料全部是江华苦茶的群体单株,而之前研究中采用的是茶组植物的不同变种材料,另一方面也说明cpDNA变异率对于种间和种内的样品辨识度相当明显。同时,本研究中使用的trnH-psbA序列虽然没有采用进行结果分析,但是与陈春梅的实验[12]以及之前实验结果类似,序列当中有着较高的碱基变异率,在之后的研究中可以更多的关注trnH-psbA区域。
采自湖南省茶叶研究所茶树资源圃的5个品种中,除了苦茶3号(gene27)外,其余4个品种都聚在一个类群中,其中有两个是选育品种(gene23的潇湘红21-3和gene26的未命名品种),该类群可能是将来选育品种的重点考虑对象。同时,来自江华县码市镇的6个样品当中,有4个来自同一村庄的样品聚集在一起,亲缘关系表现出非常强烈的地域性。因此,在江华苦茶资源入圃保护时,应重点考虑同一区域资源亲缘关系相似度高的特点,尽量选择亲缘关系较远的单株。
[1]陈亮, 虞富莲, 杨亚军, 等. 茶树种质资源与遗传改良[M].北京: 中国农业科学技术出版社, 2006.
[2]陈亮, 虞富莲, 童启庆. 关于茶组植物分类与演化的讨论[J].茶叶科学 , 2000, 20(2): 89-94.
[3]沈程文, 黄意欢, 黄建安, 等. 湖南典型茶树地理种群遗传多样性[J]. 农业生物科技学报, 2007, 15(5): 855-860.
[4]刘湘鸣.江华苦茶性状研究[J]. 茶叶通讯, 2006, 33(3): 4-7.
[5]郑小艳, 曹家树, 滕元文. DNA序列分析在植物分子系统学研究中的应用现状——以蔷薇科为例[J]. 园艺学报,2009, 36 (12): 1827-1836.
[6]刘丽, 聂平, 肖炳燚, 等. 基于DNA条形码序列分析的5种菖蒲属药用植物鉴定[J]. 中南药学, 2015, 13(4): 388-392.
[7]刘锋, 李涛, 余乃通, 等. 海南番茄蕨叶型CMV分子检测及其序列分析[J]. 基因组学与应用生物学, 2015, 34(8):1723-1728.
[8]刘红梅, 张宪春, 曾辉. DNA序列在蕨类分子系统学研究中的应用[J]. 植物学报, 2009, 44(2): 143-158.
[9]李毳. 基于cpDNA序列分析不同生境芦苇种群的遗传结构 [J]. 生态环境学报 , 2016, 25(8): 1315-1319.
[10]莫文娟, 李少锋, 邱乾栋, 等. 基于cpDNA rps16序列分析兰考泡桐与白花泡桐和毛泡桐的遗传关系[J]. 林业科学研究 , 2016, 29(3): 377-382.
[11]李欣, 刘占林, 王祎玲, 等. 基于叶绿体DNA序列分析东亚七筋姑的遗传进化[J]. 西北植物学报, 2008, 28(6):1112-1117.
[12]陈春梅. 基于cpDNA 序列的茶树及其近缘植物的亲缘关系研究[M]. 北京: 中国农业科学院, 2014.
[13]高连明, 刘杰, 蔡杰, 等. 关于植物DNA条形码研究技术规范[J]. 植物分类与资源学报, 2012, 34 (6) : 592-606.
[14]Tamura K, Stecher G, Peterson D, et al. MEGA6: Molecular Evolutionary Genetics Analysis version 6.0[J]. Molecular Biology and Evolution, 2013(30): 2725-2729.
[15]朱元娣, 曹敏格, 许正, 等. 基于ITS和matK序列探讨新疆野苹果与中国苹果的系统演化关系[J]. 园艺学报, 2014,41(2): 227-239.
Analysis of cpDNA Sequence of Kinship among Different Strains of Jianghua Kucha
CHENG Yang1,2,LIU Zhen2,YANG Yang2,LUO Jun-wu1*
(1. College of Horticulture and Landscape Architecture, Hunan Agricultural University, Changsha 410128, China; 2. National Tea Improvement Center Hunan sub – center, Tea Research Institute of Hunan Academy of Agricultural Sciences, Changsha 410125, China)
DNA sequence analysis has shown a strong vitality in the phylogenetic evolution, classification and identi fi cation of species. The cpDNA sequence analysis has been widely used in phylogenetic studies because of its small genome, moderate evolution rate and maternal inheritance. In this study, four pairs of cpDNA primers were used to study the genetic relationship among 32 Jianghua Kucha resources. Three of them were successfully sequenced. Ampli fi cation sequence length was 634 (1F-724R), 499 (rbcla-rev), 532 (rbcla-ajf634), respectively. The number of mutations was 5(1F-724R), 4 (rbcla-rev), 4 (rbcla-ajf634), respectively. The variance rates were rbcla-rev (0.80%), 1F-724R (0.79%), rbclaajf634 (0.75%), respectively, from high to low. The sequences of the three pairs of primers were staged, and the molecular system trees were constructed according to the MP method. The 32 tea trees were divided into four groups. This study has opened up a new way for the research on the genetic relationship of Jianghua Kucha, which provides a theoretical reference for the application and protection of bitter tea resources in Jianghua.
Jianghua Kucha,cpDNA,Analysis of DNA sequence
S571.1,R282.5
A
1009-525X(2017)03-22-26
2017-07-31
2017-08-12
国家现代农业产业技术体系建设专项(CARS-23)、湖南省农业科技创新项目(2017JC15)
成杨(1985-),男,江苏如皋人,助理研究员,在读硕士研究生,研究方向:茶树分子育种。
*通讯作者:罗军武(1957-),男,湖南邵阳人,教授,主要从事茶树种质资源与育种、茶树栽培生理方面的研究与教学。E-mail:luojunwu11@sina.com
