牛科物种胰岛素诱导基因1(INSIG1)研究进展
2017-07-18罗建椿邱立华范新阳苗永旺
罗建椿,邱立华,范新阳,苗永旺*
(1.云南农业大学动物科学技术学院,云南 昆明 650201;2.云南省怒江州兰坪县畜禽品种改良站,云南 兰坪 671400)
文献综述
牛科物种胰岛素诱导基因1(INSIG1)研究进展
罗建椿1, 2,邱立华1,范新阳1,苗永旺1*
(1.云南农业大学动物科学技术学院,云南 昆明 650201;2.云南省怒江州兰坪县畜禽品种改良站,云南 兰坪 671400)
胰岛素诱导基因1编码蛋白(INSIG1)定位于内质网膜上,是调控脂代谢的重要因子。INSIG1主要通过调控固醇调控元件结合蛋白(SREBP)和3-羟-3-甲基戊二酰辅酶A还原酶(HMGR)来影响脂质代谢。越来越多的研究结果揭示了INSIG1在生物体内作用的复杂性。本文综述了INSIG1与SREBP和HMGR之间在脂代谢调控过程中的相互作用机制、INSIG1的结构及表达调控、INSIG1对牛科动物泌乳、生长等性状的影响。
胰岛素诱导基因1;固醇调控元件结合蛋白;3-羟-3-甲基戊二酰辅酶A还原酶;表达调控;泌乳性状;基因多态性
胰岛素诱导基因(Insulin induced gene,INSIGs)编码蛋白是近年来发现的一种在脂肪代谢与脂肪细胞分化中发挥调控作用的蛋白。1993年,Diamond等[1]最早从再生肝脏中克隆获得了INSIG1基因。自INSIG1被发现后,其功能一直未知,直到2002年,Yang等[2]发现其能够有效的阻止SREBP家族成员的激活,INSIG1的功能及其作用机制才开始为研究人员所关注。INSIGs基因有INSIG1和INSIG2两种亚型,且INSIG2在哺乳动物中含有两个转录本INSIG2a和INSIG2b,但两者编码蛋白质相同。已经发现,INSIGs基因的表达受胰岛素和摄入的游离脂肪酸、葡萄糖和其它营养素的调控。INSIG1蛋白为镶嵌于细胞内质网(endoplasmic reticulum,ER)膜上的蛋白质,其亲水性的N端和C端延伸段伸入胞浆,其余大部分都是位于跨膜区,共有6个跨膜螺旋锚定在ER膜上,并且含有5-16个氨基酸构成的亲水环[3],其中第3和第4个跨膜区对INSIG1与羟固醇/SCAP复合体的结合比较重要,因此,INSIGs蛋白又被称为羟固醇结合蛋白[4]。Lee等[5]发现,INSIG1蛋白的跨膜区1处有一个甘氨酸残基,为gly-95,这个氨基酸对于INSIG1蛋白调控固醇的功能起重要作用。INSIG1和INSIG2的蛋白序列高度相似,均为ER上的膜蛋白。在人类中INSIG1和INSIG2的氨基酸序列具有59%的相似性,跨膜区相似性更高,达到85%[6-7]。它们具有相似的功能,都参与了与固醇调节因子结合蛋白(Sterol-regulatory element binding proteins,SREBP)及SREBP裂解活化蛋白(SREBP cleavage activating protein, SCAP)的结合。它们以INSIG-SCAP-SREBP复合物的形式固定在内质网上,在细胞内固醇类物质的参与下,调控SREBP的活化,从而反馈调节细胞对外源性胆固醇的摄取[8]。INSIG2与INSIG1相比,INSIG2缺少了INSIG1的N端的50个氨基酸残基,但INSIG1的降解速度较快,这与它们所含的氨基酸残基不同有关[9]。
INSIG1具有多种生物功能,它参与了生物体内的脂质代谢,蛋白调控,与牛科物种的生长发育、泌乳及胴体重等性状有关。
1 INSIG1作用机制
INSIG1主要通过两个路径发挥作用:一个是通过INSIG1-SCAP-SREBP的细胞内稳衡调节,另一个是通过3-羟-3-甲基戊二酰辅酶A还原酶泛素化降解调节(图1)。其中SCAP的固醇敏感结构域(sterol sensing domain,SSD)和还原酶第二跨膜区内的一个四肽序列YIYF是在固醇诱导下与INSIGs结合所必需的[10-12]。在哺乳动物细胞中,INSIG1通过结合SCAP阻止SREBP/SCAP复合体从ER转运到高尔基体[6]。SREBP在哺乳动物中是参与固醇和脂肪酸合成调节的重要转录因子,直接参与调控与胆固醇、甘油三酯、脂肪酸及磷脂的生物合成及摄取过程中相关的30多个基因的表达[13]。固醇和脂肪酸对维持机体基本生理功能和正常代谢十分重要,它们在维持细胞稳定性、流动性等功能方面发挥重要作用。SCAP既是SREBP的护送者,也是固醇感应器之一。Gong等[14]研究发现,当INSIG1蛋白第205位的Asp突变为Ala时,INSIG1将不能与SCAP结合,揭示INSIG1的205位Asp是调控SREBP蛋白所必需的。当细胞内胆固醇水平升高时,INSIG1与SCAP相互作用,以INSIG1-SCAP-SREBP复合物的形式固定在内置网上,这时负责蛋白质从ER运向高尔基体(Golgi)的一种介导蛋白——细胞质被膜复合体II(coat protein complex II,COPII)不能结合到SCAP,因此SCAP-SREBP复合体不能被COPII蛋白转移到高尔基体上;当细胞内胆固醇耗尽时,INSIG1与SCAP的亲和力降低,从复合物中解离,SCAP护送SREBP从ER转运到高尔基体,SREBP前体在高尔基体内被两个特异的蛋白酶依次加工,裂解成为成熟的SREBP活性片段,进入细胞核,与多个靶基因启动子、增强子的类固醇反应元件(steroid hormone response elements, SREs)结合,从而行使其转录调控功能,促进固醇和脂肪酸生物合成中相关基因的表达[15-17]。之后,游离的INSIG1蛋白被迅速泛素化降解,而当细胞内固醇再次积累到一定水平时,INSIG1蛋白泛素化过程停滞,INSIG1蛋白质浓度趋于稳定[18]。这主要是因为无固醇细胞中INSIG1蛋白的降解需要泛素连接酶gp78的参与,这种酶与INSIG1结合的亲和力要高于INSIG2,固醇可以通过从INSIG1上置换出gp78,从而避免INSIG1蛋白泛素化而被降解[19]。Lee等[18]研究发现,INSIG1蛋白的Ser-149位点会促进其泛素化降解。Yang等[2]研究发现,固醇含量较高时,固醇可与SCAP结合使其构象改变,促进INSIG1与SCAP结合,从而阻止SCAP-SREBP由ER到高尔基体的转运,使SREBP裂解成熟过程受阻。
此外,INSIG1是多个SREBPs的作用目标,即它本身也是SREBP作用的靶基因,其mRNA的表达水平受到SREBP的调节。SREBP可以调控INSIG1基因的启动子。研究发现,INSIG1基因的转录起始位点上游380bp发现了固醇应答元件[20]。羟固醇可以下调INSIG1基因的表达,降固醇药物可上调其表达[21],而不饱和脂肪酸可以稳定INSIG1水平,使它不受泛素化的影响[22]。在动物细胞中,低渗休克和内质网应激也可以通过SREBP对胆固醇的抑制降低INSIG1的水平[9]。
另一方面,作为总固醇代谢的最重要的酶之一,3-羟-3-甲基戊二酰辅酶A还原酶(3-hydroxy-3-methylglutaryl CoA reductase,HMGR)是ER上总固醇合成的限速酶[23]。HMGR可以产生甲羟戊酸,甲羟戊酸可以转化成固醇和其它产物。HMGR的表达受到多级复杂的调控,防止生成过多的总固醇,维持体内总固醇的平衡。INSIG1在高固醇水平下通过与HMGR的SSD结合而促进它的降解,而在固醇较低水平时,HMGR降解比较缓慢,HMGR含量和活性增加,促进总固醇合成[11]。Sever等[24]建立了不能表达INSIG1的细胞系,发现固醇抑制SREBP的过程明显变慢,而这个过程和HMGR的降解过程可以通过INSIG1的过表达而恢复,揭示INSIG1是还原酶泛素化降解所必需的。SCAP和HMGR与INSIG1的结合是竞争性的,二者不能同时结合INSIG1蛋白。Kuwabara等[25]通过研究SCAP和HMGR的序列发现,SCAP与HMGR在跨膜区上高度相似,跨膜区有与INSIG1结合的SSD。Lee等[26]将SSD区域基因突变或敲除时,INSIG1不能与SCAP和HMGR结合,导致INSIG1既不能将SREBP固定在ER上,也不能改变HMGR泛素化降解速度。然而,在酵母中,INSIG1通过与SSD结合而抑制HMGR的泛素化降解[27]。HMGR在转录水平上还受SREBP2的调控。当机体需要甲羟戊酸衍生产物增加时,SREBP2会结合到HMGR的启动子上,激活其转录[28]。
近年研究发现,INSIG1的作用也受其它因素的影响。它最早在从肝脏中分离出来时,就被发现具有胰岛素敏感性,它的表达水平随着胰岛素水平的改变而改变。当肝脏的胰岛素水平上升时,INSIG1基因的mRNA表达水平升高,而当胰岛素水平下降时,INSIG1基因的mRNA水平也会下降[1]。此外,葡萄糖水平也会影响INSIG1基因的表达。谢红艳等[29]发现,低浓度的葡萄糖可以上调3T3-L1细胞中INSIG1的mRNA水平,从而抑制脂肪细胞早期脂质合成和分化。INSIG1还可能通过调节 PPARγ的表达,参与糖代谢的调控。这些研究揭示INSIG1基因可能处于调控网络的核心位置,调控多种因素介导细胞脂代谢。
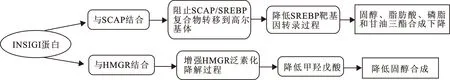
图1 INSIG1功能机制图(引自Dong等[12])
2 INSIG1基因的结构及表达
普通牛的INSIG1基因(AC_000161)全长约13.2kb,包含6个外显子和5个内含子,定位于4号染色体(BAT4)。外显子1的全部和外显子2的前23 bp位于5’UTR,外显子6的后1845bp位于3’UTR,基因结构如图2所示。牛INSIG1基因(NM_001077909)CDS区长831bp,编码276个氨基酸。序列比对分析,牛INSIG1基因与山羊INSIG1基因的相似性最高[30]。INSIG1在水牛和奶牛的乳腺组织中高度表达,且奶牛泌乳期表达量是干奶期的12倍[31, 32]。对人17个组织的基因表达研究发现,INSIG1基因在肝脏中高度表达,而INSIG2则在肌肉、肺和大脑等各组织中广泛表达[33]。
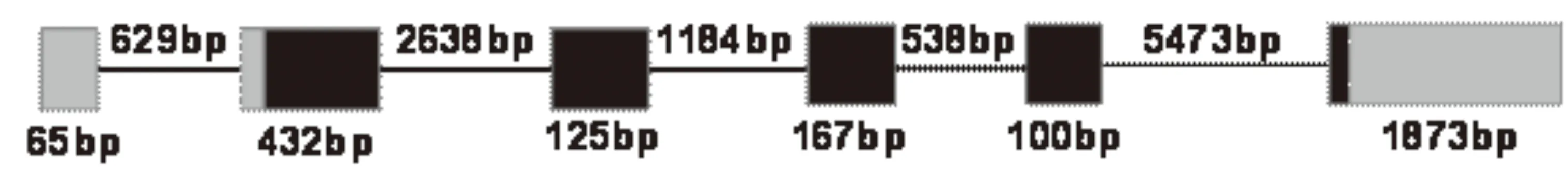
图2 普通牛(Bos taurus)INSIG1基因结构注:方框为外显子,灰色方框为非翻译区,黑色方框为编码区,横线为内含子。
3 INSIG1对牛科物种性状的影响
Bionaz等[31]基于对奶牛多个泌乳时期乳腺中54个与乳脂代谢相关基因的表达水平分析,推测出乳脂代谢的调控网络图,提出SREBP在泌乳过程乳脂代谢网络中处于核心位置。此外,Xu等[34]在奶山羊乳腺上皮细胞中过表达SREBP1后,显著提高了乳脂合成过程中多个重要基因的表达,同时细胞中甘油三酯以及C16:0与C18:0含量升高,揭示了SREBP在乳脂合成调控的关键作用。而INSIG1是调控SREBP的重要蛋白,揭示INSIG1在乳脂合成中发挥了重要作用。王苗等[13]成功构建了INSIG1基因的超表达载体并转染奶山羊乳腺上皮细胞,发现参与脂肪酸从头合成基因(ACCα、FASN)及去饱和基因(SCD1)的mRNA表达量均显著下调,参与甘油三酯合成的关键基因(GPAM、DGAT2)的mRNA表达量显著下降,揭示INSIG1对山羊乳腺脂质代谢具有重要作用。Wu等[32]检测了水牛INSIG1基因在10个组织中的表达水平发现,INSIG1在乳腺中表达量最高,其次是肝,认为INSIG1在乳脂合成的调控中起到重要作用。南雪梅[35]通过荧光定量检测了奶山羊乳腺组织不同泌乳时期INSIG1的表达量,发现在泌乳35天时,表达量达到峰值,揭示INSIG1基因在奶山羊泌乳过程中发挥重要作用。因此,进一步研究INSIG1的遗传变异,分析它们与牛、羊的泌乳性状的关联,具有重要的理论和实践意义。
INSIG1除了对泌乳性状有影响外,还与家畜其他经济性状有关。INSIG1是脂肪和胆固醇合成反馈调节的重要成分,对细胞中的脂质自稳态的维持有重要作用,它可以控制前脂肪细胞分化、成熟等过程,防止脂肪细胞分化过度或分化不良,从而对于脂肪沉积、肥胖的控制可能都有意义[36]。Liu等[37]通过PCR-RFLP和DNA测序的方法,检测了秦川牛INSIG1基因的多态性,发现了4个多态性位点,又与体长、体高、臀宽,出栏体重和胴体重等性状进行了关联分析,发现在A4366G位点是GG基因型的牛个体臀更宽,胴体更重,T4534C位点是CC基因型的牛个体身体更长,臀更宽以及T5001C位点是CC基因型的牛个体身体更长。Sun等[38]通过DNA池、PCR-RFLP、PCR-SSCP和DNA测序的方法,检测了643头南阳牛,共发现了10个多态性位点,其中4个位于编码区,同时将多态性位点与体重进行关联分析,但是没有发现有显著的相关性。
4 INSIG1研究的问题与展望
众多研究结果表明,INSIG1基因在脂质和糖代谢中发挥了重要的生理功能。它与动物的泌乳性状以及其它经济性状存在一定相关性,而且与肥胖、糖尿病等多种疾病有关,但当前在INSIG1基因的研究和应用中仍存在一些问题亟待进一步解决。比如,INSIG1基因是否还有别的亚型;INSIG1与泌乳性状的关联性在水牛及其它牛科物种所做的研究不多;INSIG1影响动物经济性状的研究较少,其分子机制也还不清楚。水牛奶营养全面,其营养价值远高于荷斯坦牛奶,联合国粮食及农业组织(FAO)认为水牛是最具开发潜力和开发价值的家畜。由于该基因与泌乳性状密切相关,因此我们可以针对奶水牛的泌乳性状做关联分析,同时可以利用生物信息学来预测水牛INSIG1基因的遗传变异,为改良和培育具有优良泌乳性能的品种提供参考。此外,有关INSIG1基因编码区多态性的研究以及本实验室通过克隆INSIG1基因,发现其编码区较为保守,因此,我们有必要对其非编码区进行相关研究。
[1] Diamond R H,Du K,Lee V M,et al.Novel delayed-early and highly insulin-induced growth response genes[J].The Journal of Biological Chemistry,1993,268(20):15185-15192.
[2] Yang T,Espenshade P J,Wright M E,etal.Crucial step in cholesterol homeostasis:sterols promote binding of SCAP to INSIG-1,a membrane protein that facilitates retention of SREBPs in ER[J].Cell,2002,110(4):489-500.
[3] Feramisco J D,Goldstein J L,Brown M S.Membrane topology of human Insig-1,a protein regulator of lipid synthesis[J].The Journal of Biological Chemistry,2004,279(9):8487-8496.
[4] Radhakrishnan A,Ikeda Y,Kwon H J,etal.Sterol-regulated transport of SREBPs from endoplasmic reticulum to Golgi:Oxysterols block transport by binding to Insig[J].Proceedings of the National Academy of Sciences,2007,104(16):6511-6518.
[5] Lee P C W,Debose-Boyd R A.Intramembrane glycine mediates multimerization of Insig-2,a requirement for sterol regulation in Chinese hamster ovary cells[J].Journal of Lipid Research,2009,51(1):192-201.
[6] Yabe D,Brown M S,Goldstein J L.Insig-2,a second endoplasmic reticulum protein that binds SCAP and blocks export of sterol regulatory element-binding proteins[J].Proceedings of the National Academy of Sciences,2002,99(20):12753-12758.
[7] Goldstein J L,Debose-Boyd R A,Brown M S.Protein sensors for membrane sterols[J].Cell,2006,124(1):35-46.
[8] Gong Y,Lee J N,Lee P C W,etal.Sterol-regulated ubiquitination and degradation of Insig-1 creates a convergent mechanism for feedback control of cholesterol synthesis and uptake[J].Cell Metabolism,2006,3(1):15-24.
[9] Lee J N,Ye J.Proteolytic activation of sterol regulatory element-binding protein induced by cellular stress through depletion of Insig-1[J].The Journal of Biological Chemistry,2004,279(43):45257-45265.
[10] Brown M S,Goldstein J L.A proteolytic pathway that controls the cholesterol content of membranes,cells,and blood[J].Proceedings of the National Academy Of Sciences,1999,96(20):11041-11048.
[11] Sever N,Yang T,Brown M S,etal.Accelerated degradation of HMG CoA reductase mediated by binding of Insig-1 to its sterol-sensing domain[J].Molecular Cell,2003,11(1):25-33.
[12] Dong X,Tang S.Insulin-induced gene:A new regulator in lipid metabolism[J].Peptides,2010,31(11):2145-2150.
[13] 王苗,罗军,许会芬,等.山羊INSIG1基因超表达对乳腺上皮细胞中脂质合成的影响[J].畜牧兽医学报,2016,47(9):1806-1816.
[14] Gong Y,Lee J N,Brown M S,etal.Juxtamembranous aspartic acid in Insig-1 and Insig-2 is required for cholesterol homeostasis[J].Proceedings Of The National Academy Of Sciences,2006,103(16):6154-6159.
[15] Sun L P,Li L,Goldstein J L,etal.Insig required for sterol-mediated inhibition of Scap/SREBP binding to COPII proteins in vitro[J].The Journal of Biological Chemistry,2005,280(28):26483-26490.
[16] McphersonR,Gauthier A.Molecular regulation of SREBP function:the Insig-SCAP connection and isoform-specific modulation of lipid synthesis[J].Biochemistry and Cell Biology,2004,82(1):201-211.
[17] Espenshade P J,Hughes A L.Regulation of Sterol Synthesis in Eukaryotes[J].Annual Review of Genetics,2007,41(1):401-427.
[18] Lee J N,Gong Y,Zhang X,etal.Proteasomal degradation of ubiquitinated Insig proteins is determined by serine residues flanking ubiquitinated lysines[J].Proceedings of the National Academy of Sciences,2006,103(13):4958-4963.
[19] Lee J N,Song B,Debose-Boyd R A,etal.Sterol-regulated degradation of Insig-1 mediated by the membrane-bound ubiquitin ligase gp78[J].The Journal of Biological Chemistry,2006,281(51):39308-39315.
[20] Kast-Woelbern H R,Dana S L,Cesario R M,etal.Rosiglitazone induction of Insig-1 in white adipose tissue reveals a novel interplay of peroxisome proliferator-activated receptor ? and sterol regulatory element-binding protein in the regulation of adipogenesis[J].The Journal of Biological Chemistry,2004,279(23):23908-23915.
[21] Janowski B A.The hypocholesterolemic agent LY295427 up- regulates INSIG-1,identifying the INSIG-1 protein as a mediator of cholesterol homeostasis through SREBP[J].Proceedings of the National Academy Of Sciences,2002,99(20):12675-12680.
[22] Lee J N,Zhang X,Feramisco J D,etal.Unsaturated fatty acids inhibit proteasomal degradation of Insig-1 at a postubiquitination step[J].The Journal of Biological Chemistry,2008,283(48):33772-33783.
[23] Goldstein J L,Brown M S.Regulation of the Mevalonate Pathway[J].Nature,1990,343(6257):425-430.
[24] Sever N,Lee P C W,Song B L,etal.Isolation of mutant cells lacking Insig-1 through selection with SR-12813,an agent that stimulates degradation of 3-Hydroxy-3-methylglutaryl-Coenzyme A reductase[J].The Journal ofBiological Chemistry,2004,279(41):43136-43147.
[25] Kuwabara P E,Labouesse M.The sterol-sensing domain:multiple families,a unique role?[J].Trends in Genetics,2002,18(4):193-201.
[26] Lee P C W,Nguyen A D,Debose-Boyd R A.Mutations within the membrane domain of HMG-CoA reductase confer resistance to sterol-accelerated degradation[J].Journal of Lipid Research,2007,48(2):318-327.
[27] Burg J S,Powell D W,Chai R,etal.Insig regulates HMG-CoA reductase by controlling enzyme phosphorylation in Fission Yeast[J].Cell Metabolism,2008,8(6):522-531.
[28] Vallett S M,Scanchez H B,Rosenfeld J M,etal.A direct role for sterol regulatory element binding protein in activation of 3-Hydroxy-3-methylglutaryl coenzyme A reductase gene[J].The Journal of Biological Chemistry,1996,271(21):12247-12253.
[29] 谢红艳,莫朝晖,陈科,等.不同浓度葡萄糖对3T3-L1细胞分化及insig-1和insig-2 mRNA表达的影响[J].中南大学学报(医学版),2008,33(3):238-244.
[30] 王苗.奶山羊INSIG基因对乳腺上皮细胞中脂代谢的影响研究[D].杨凌:西北农林科技大学,2016.
[31] Bionaz M,Loor J J.Gene networks driving bovine milk fat synthesis during the lactation cycle[J].BMC Genomics,2008,9(1):366.
[32] Wu C,Liu L,Huo J,etal.Isolation,sequence characterization,and tissue transcription profiles of two novel buffalo genes:INSIG1 and INSIG2[J].Tropical Animal Health and Production,2014,46(1):33-41.
[33] Krapivner S,Popov S,Chernogubova E,etal.Insulin-induced gene 2 involvement in human adipocyte metabolism and body weight regulation[J].Journal of Clinical Endocrinology & Metabolism,2008,93(5):1995-2001.
[34] Xu H F,Luo J,Zhao W S,etal.Overexpression of SREBP1 (sterol regulatory element binding protein 1) promotes de novo fatty acid synthesis and triacylglycerol accumulation in goat mammary epithelial cells[J].Journal of Dairy Science,2016,99(1):783-795.
[35] 南雪梅.奶山羊乳腺物质代谢研究[D].哈尔滨:东北农业大学,2010.
[36] 王京京,陈斌.胰岛素诱导基因及其在猪育种中应用的研究进展[J].猪业科学,2014(4):92-94.
[37] Liu Y,Liu X L,He H,etal.Four SNPs of insulin-induced gene 1 associated with growth and carcass traits in Qinchuan cattle in China[J].Genetics and Molecular Research,2012,11(2):1209-1216.
[38] Sun J,Gao Y,Liu D,etal.Haplotype combination of the bovine INSIG1 gene sequence variants and association with growth traits in Nanyang cattle[J].Genome,2012,55(6):429-436.
Research Advances of Insulin iInduced Gene 1 (INSIG1) in Bovine Species
LUO Jian-chun1,2,QIU Li-hua1,FAN Xin-yang1,MIAO Yong-wang1*
(1.FacultyofAnimalScienceandTechnology,YunnanAgriculturalUniversity,Kunming,Yunnan650201;2.LivestockandPoultryBreedingStationofLanpingCountyinNujiang,Lanping,Yunnan671400)
INSIG1protein,encoded by insulin induced gene 1(INSIG1),which located in the membrane of endoplasmic reticulum,is an important factorparticipatedin the regulation of lipid metabolism.INSIG1 playroles in lipid metabolism through regulating sterolregulatory element binding proteins(SREBP) and 3-hydroxy-3-methylglutaryl CoA reductase(HMGR).An increasing number of studies have uncovered the complexity of INSIG1 functions in vivo.In this paper,we reviewed the mechanism of the interaction among INSIG1,SREBPand HMGRin the regulation of lipid metabolism,the structure and expression regulation of INSIG1,and the effects of INSIG1 on the lactation and growth traits in bovine species.
INSIG1;sterol regulatory element binding proteins;3-hydroxy-3-methylglutaryl CoA reductase;expressionregulation;lactation traits;gene polymorphism
7-02-22
2017-03-12
国家自然科学基金项目(31460582;30660024);云南省应用基础研究重点项目(2014FA032;2007C0003Z)
罗建椿(1976-),男,畜牧师,主要从事畜禽品种改良工作。
*通讯作者:苗永旺(1964-),男,内蒙古通辽人,教授,博士,主要从事动物遗传学研究。
S813.7
A
1001-9111(2017)03-0031-05
