新疆传统发酵酸奶中酵母菌的分离鉴定及系统发育分析
2017-04-27乔传丽蒋彩虹金丹蒋艾廷卢士玲李王强李宝坤
乔传丽,蒋彩虹,金丹,蒋艾廷,卢士玲,李王强,李宝坤*
(石河子大学食品学院,新疆石河子832000)
新疆传统发酵酸奶中酵母菌的分离鉴定及系统发育分析
乔传丽,蒋彩虹,金丹,蒋艾廷,卢士玲,李王强,李宝坤*
(石河子大学食品学院,新疆石河子832000)
从新疆塔城地区哈萨克族不同牧场家庭中自然发酵牛乳采集18份样品,从中分离筛选得到47株酵母菌,并进行形态鉴定、生理生化鉴定、聚合酶链式反应-变性梯度凝胶电泳(PCR-DGGE),26S rDNA分子生物学鉴定,构建系统发育树。结果表明,47株菌中包括库德毕赤酵母(Pichia kudriavzevii)18株,阿萨希丝孢酵母菌(Trichosporon asahii)10株,酿酒酵母(Saccharomyces cerevisiae)7株,丝孢酵母(Trichosporon coremiiforme)4株,马克斯克鲁维酵母(Kluyveromyces marxianus)4株,东方伊萨酵母(Issatchenkia orientalis)2株,发酵毕赤酵母(Pichia fermentans)1株,近平滑假丝酵母(Candida parapsilosis)1株。鉴定结果对进一步认识并利用哈萨克族传统发酵酸奶中酵母菌资源具有重要意义。
传统发酵酸奶;26S rDNA D1/D2;筛选;分离鉴定;聚合酶链式反应-变性梯度凝胶电泳
传统发酵乳制品是以鲜奶为原料,经乳酸菌、酵母菌等发酵而成,是天然的绿色产品,其微生物资源丰富,营养价值高,并具有较好的益生作用。新疆少数民族多年以来一直保留着传统方法制作发酵乳制品的习惯,为发掘有益微生物提供了宝贵的资源[1]。闫彬[2]的研究中显示酵母菌单独发酵时的乙醇含量明显高于酵母和乳酸菌混菌发酵,可以看出酸乳中的醇香主要由酵母菌产生。张敏等[3]研究发现,经酵母菌发酵后半胱氨酸(Cys)和色氨酸(Ser)在乳清中含量增加,有助于血脂的降低。陈成等[4]研究发现克鲁维酵母在发酵时产生乳糖酶分解乳糖来缓解全球各类人群的乳糖不耐症。但是,目前新疆酸奶中酵母菌资源的开发和利用还存在着不足之处,传统发酵乳制品中对酵母菌的研究相对乳酸菌较少,因此对传统发酵乳制品中酵母菌资源的收集、分离鉴定和生物学特性的研究是其开发利用的基础。
近年来,随着分子生物学技术的快速发展,DGGE技术被越来越多的专家学者应用于分析复杂微生物群落多样性。DGGE技术既可以用于传统发酵食品卫生的菌群结构分析也可以用于纯菌株的分离和筛选,减少分子和生化鉴定的数量,具有快速可靠的特点[5-6]。GKATZIONIS K等[7]利用聚合酶链式反应-变性梯度凝胶电泳(polymerase chain reaction-denaturinggradientgelelectrophoresis,PCR-DGGE)和限制性片段长度多态性(terminalrestrictionfragmentlength polymorphism,TRFLP)技术对斯第尔顿奶酪不同部分酵母菌多样性和活性进行研究。因此,该试验结合传统培养方法和PCR-DGGE技术对传统乳制品中酵母菌多样性进行研究。以新疆塔城牧区哈萨克族普通农户不同家庭自制酸奶为原料,利用传统分离方法初步分离出具有酵母菌特性的菌株,再利用PCR-DGGE法剔除重复菌种及测序比对来分析新疆塔城地区传统发酵酸奶中酵母菌的优势菌群和生物多样性。此研究不仅为新疆塔城地区传统酸奶中酵母菌菌株资源的开发利用奠定基础,也为乳制品的工业化生产培养了优势菌种。
1 材料与方法
1.1 材料与试剂
1.1.1 材料
从新疆塔城地区哈萨克族牧民家庭中采集18份酸奶样品。采集的酸奶装于灭菌的离心管内,密封后放入车载冰箱于4℃运回实验室,即进行后续试验。
1.1.2 试剂
酵母菌基因组DNA提取试剂盒(离心柱型)、2×Taq PCR MasterMix:天根生化科技有限公司;EB核染色剂:北京索莱宝科技有限公司;引物NL1和NL4:上海生物工程有限公司。2%美兰、林格氏碘液、丙烯酰胺/双丙烯酰胺(37.5∶1)浓度为8%、50×TAE缓冲液。
1.1.3 培养基
酵母浸出粉胨葡萄糖(yeast extract peptone dextrose,YPD)培养基:葡萄糖20 g,蛋白胨10 g,酵母浸粉5 g。
碳源培养基:碳源0.5%,硫酸铵0.5%,磷酸二氢钾0.1%,硫酸镁0.05%,氯化钙0.01%,氯化钠0.01%,酵母粉0.02%。
氮源培养基:氮源0.07%,葡萄糖2%,磷酸二氢钾0.1%,硫酸镁0.05%,酵母粉0.02%。
1.2 仪器与设备
DNP-9272电热恒温培养箱:上海精宏实验设备公司;5417R高速冷冻离心机:德国Eppendorf公司;TC-512PCR扩增仪:Techne公司;DM3000荧光电子显微镜:Lecia公司;LD2X-40II电热蒸汽灭菌器:上海申安医疗器械厂;DGGE电泳分析系统:INGENY公司;凝胶成像系统:美国BIO-RAD公司。
1.3 方法
1.3.1 酵母菌的分离纯化
吸取1 mL传统酸奶样品于灭菌的酵母浸出粉胨葡萄糖(YPD)液体培养基中,28℃进行48 h富集培养,连续富集培养2代后吸取1 mL富集培养液于9 mL无菌生理盐水试管中,漩涡均匀。将混合后的样液进行梯度稀释,选取10-4、10-5、10-63个稀释度,用移液器吸取150 μL样液,采用涂布法,将样液均匀涂布于YPD培养基上,置于28℃恒温培养箱中培养24~48h,观察并记录菌落特征并挑取不同形态单个菌落,进行简单染色,观察细胞形态,将类似酵母菌菌株接种于YPD培养基中培养。将酵母菌重复划线几次,进行镜检,接种于YPD液体培养基中,于28℃恒温培养24~48 h后进行菌种保存。其中,保存液与菌液分别为700μL、600μL,存入1.5 mL离心管,标记后放入-20℃保存备用。
1.3.2 酵母菌的生理生化特性鉴定
将分离菌株接种于固体YPD培养基,置28℃恒温培养24~48 h。酵母菌分离菌株主要通过繁殖方式观察、碳源同化试验、氮源同化试验、产酸试验等生理生化特征进行鉴定。
1.3.3 酵母菌的26S rDNA分析
(1)酵母基因组总DNA的提取
吸取1 mL菌悬液,12 000 r/min离心1 min,弃上清,收集菌体按照酵母菌基因组DNA提取试剂盒(离心柱型)说明书上的方法进行提取DNA。将提取到的基因组DNA转移至灭菌的1.5 mL EP管中,做好编号,放于-20℃冰柜保藏备用。
(2)酵母PCR扩增及扩增产物的检测
将制备的基因组DNA作为PCR扩增的模板,PCR扩增目的片段引物序列为:NL1(5'-GCATATCAATAAGCGGA GGAAAAG-3'),NL4(5'-GGTCCGTGTTTCAAGACGG-3')。PCR-DGGE扩增引物,上游引物为NL1GC(5'>GCG GGC CGCGCGACCGCCGGGACGCGCGAGCCGGCGGCG GGC CAT ATC AAT AAG CGG AGG AAA AG<3'),下游引物为LS2(5'>ATT CCC AAA CAA CTC GAC TC<3')引物扩增的目的片段大小在300~700 bp之间,上述引物均由上海生工合成提供。PCR采用50 μL体系:2×Master Mix25μL,NL1和NL4(10μmol/L)各2 μL,DNA模板2 μL,去离子水50 μL。PCR反应条件:95℃预变性5 min,35个循环(94℃变性30 s,55℃退火30 s,72℃延伸1 min),72℃延伸10 min。预先制备的1%的琼脂糖凝胶,扩增反应完毕后,取5 μL的PCR扩增产物点样于琼脂糖凝胶点样孔中进行电泳,电压100 V,电泳液为1×TAE。电泳后用EB染色20 min,紫外灯下观察,目的条带为300~700 bp范围内出现明亮的条带,-20℃冰箱保存备用。
(3)变性梯度凝胶电泳(DGGE)剔除重复菌株
变形梯度凝胶电泳系统分析,变性梯度从30%~50%,试验之前先将DGGE仪200V、60℃预热30min,然后在85V的固定电压下电泳12~14 h。待DGGE完以后,将胶取出放置在超纯水(含0.5 μL/mL EB)中摇床染色30 min,20 min脱色3~4次后在凝胶成像仪上分析。
1.3.4 系统发育及同源性分析
PCR产物经电泳检测合格后,送往上海生工生物工程技术有限公司进行测序,测序得到的26S rDNA基因序列通过NCBI的Blast检索系统(http://www.ncb.nlm.nih. gov/Blast/)进行同源序列搜索,从数据库获得相关种属的26SrDNA基因序列,采用软件Mega5.0中的Neighbor-Joining方法与标准菌株基因序列进行比对,进行1 000次Bootstrap检验后构建系统发育树。
2 结果与分析
2.1 酵母菌分离菌株的菌落形态及细胞形态特征
从新疆塔城地区哈萨克族18份传统发酵酸奶样品中分离得到的47株酵母菌菌株,划线接种于YPD固体培养基培养后,菌落形态见图1,美兰染色结果见图2。
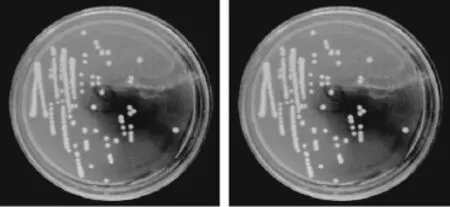
图1 酵母菌菌落形态Fig.1 Colony morphology of yeast strains

图2 酵母菌美兰染色结果(10×100)Fig.2 Methylene blue staining results of yeasts(10×100)
如图1所示,培养皿中有单菌落出现,菌落形态呈圆形,椭圆形,表面光滑湿润,干燥,颜色呈乳白色、米黄色,表面有凸点,边缘整齐。如图2所示,显微镜下细胞染色为蓝色,形态为椭圆、圆、卵圆、腊肠型等,无性繁殖方式为芽殖,一端出芽,细胞形态符合酵母菌特征。
2.2 酵母菌的生理生化鉴定结果
从18个样品中分别分离出47株酵母菌,进行DGGE剔除重复菌株得到8株具有代表性的分离株,菌落形态和生理生化鉴定如表1和表2所示。
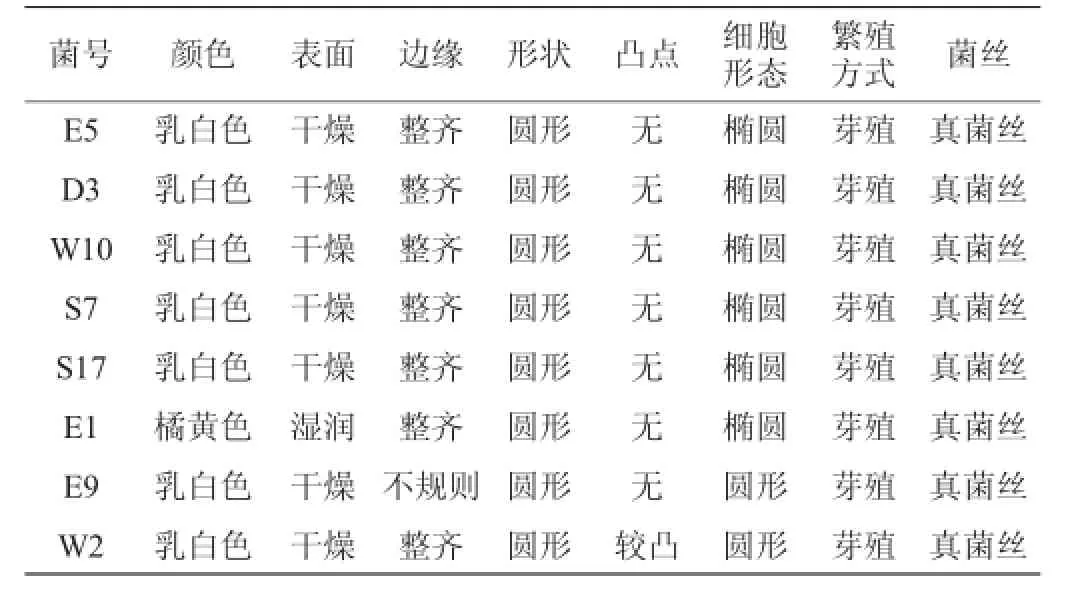
表1 菌株的形态Table 1 Morphology of the strains
结合表1酵母菌的菌落形态和表2生理生化试验鉴定结果,根据《酵母菌的特征与鉴定手册》[9-12]可知,菌株W10被初步鉴定为近平滑假丝酵母(Candida parapsilosis);菌株S3被初步鉴定为发酵毕赤酵母(Pichia fermentansc);菌株S3被初步鉴定为东方伊萨酵母(Issatchenkia orientalis)。其他菌株多为多端出芽的生殖方式,菌落形态差异明显,糖发酵试验有明显差异。另外,在碳源和氮源同化试验中各菌株均表现出不同的生理特性。
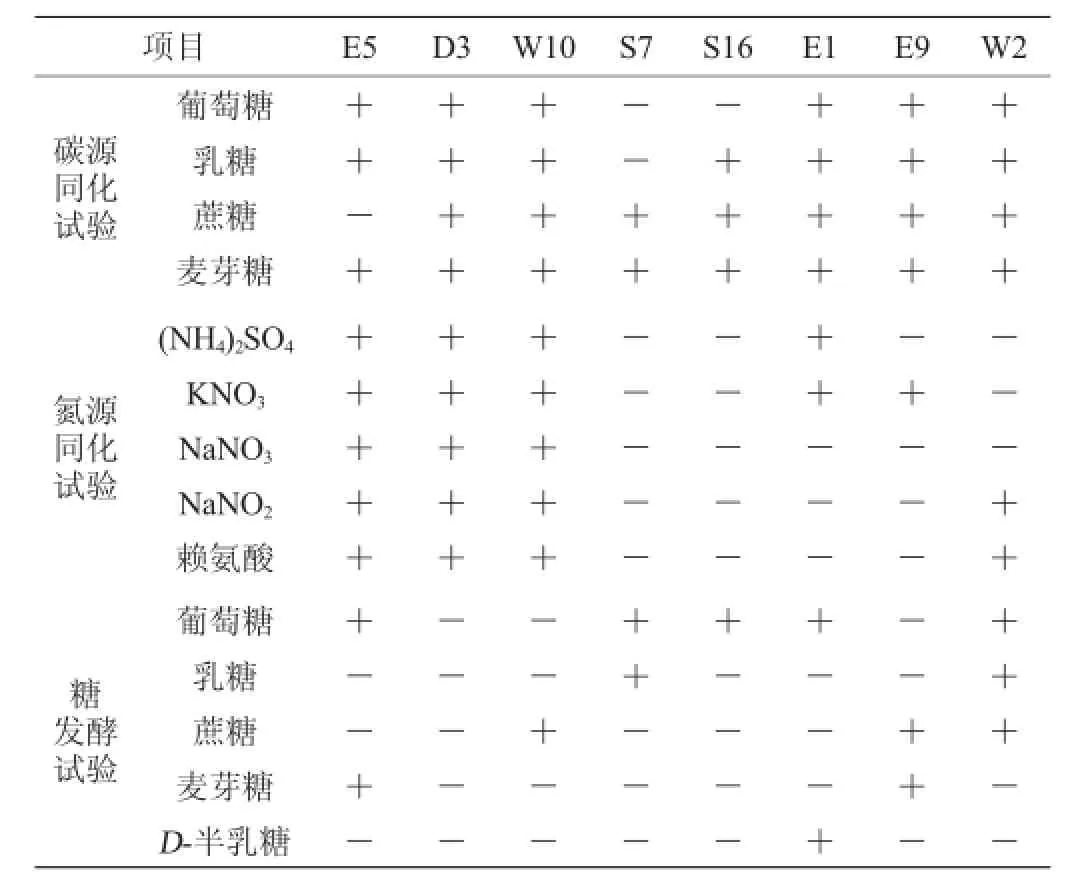
表2 菌株的生理生化鉴定结果Table 2 Physiological and biochemical identification results of strains
2.3 酵母菌的26S rDNA鉴定
2.3.1 酵母菌的PCR扩增结果
将所提取菌株的总DNA用26S rDNA的区引物进行PCR扩增,扩增产物经1.0%琼脂糖凝胶电泳检测,获得约500 bp左右的特异性扩增条带,并且空白组无条带出现,说明未出现污染、无非特异性扩增现象,结果见图3。扩增产物大小符合目的片段要求,且条带清晰,适合进行下一步DGGE试验。

图3 酵母菌PCR扩增产物琼脂糖凝胶电泳图Fig.3 Agarose gel electrophoresis of PCR products from yeasts
2.3.2 DGGE指纹图谱剔除重复菌株
由于生理生化实验和菌落状态检验无法剔除重复菌株,因此进行DGGE试验剔除重复菌株。提取酵母菌DNA,PCR-DGGE扩增引物,进行PCR扩增,产物经1.0%琼脂糖凝胶电泳检测后,通过DGGE剔除重复菌株。DGGE图谱的条带丰富度显示了微生物菌落的多样性,条带越丰富,则多样性越高[8]。菌株PCR-DGGE扩增图谱如图4,从DGGE图谱上找到了不在同一水平线上的8个条带,且条带清晰,可知有不同的菌株8株。不同菌株其DNA不同,经同一引物扩增后产物片段长度相似,但其核苷酸序列不同,因此不同双链DNA片段沿着化学梯度的不同解链行为将在凝胶的不同位置上停止迁移,所以才可以用DGGE剔除重复菌株[12]。图4中不同样品间存在公共条带,说明存在共有的酵母菌类型,而公共条带亮度不同,表明酵母菌在DNA水平上有明显差异。将不同条带的菌株经26S rDNA的区引物进行PCR扩增,扩增产物经1.0%琼脂糖凝胶电泳检测后送往上海生工生物工程技术有限公司进行测序,测序结果与Genbank数据库比对。
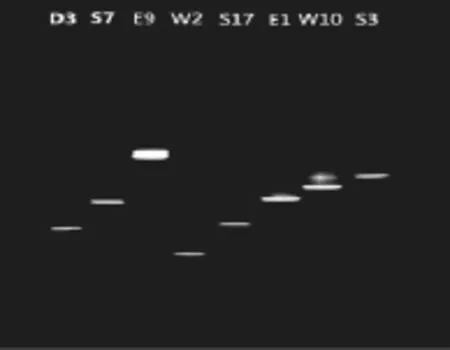
图4 菌株PCR-DGGE指纹图谱Fig.4 PCR-DGGE fingerprint chromatogram of strains
2.4 系统发育分析结果
47株酵母菌分离株的分子生物学鉴定结果如表3所示。
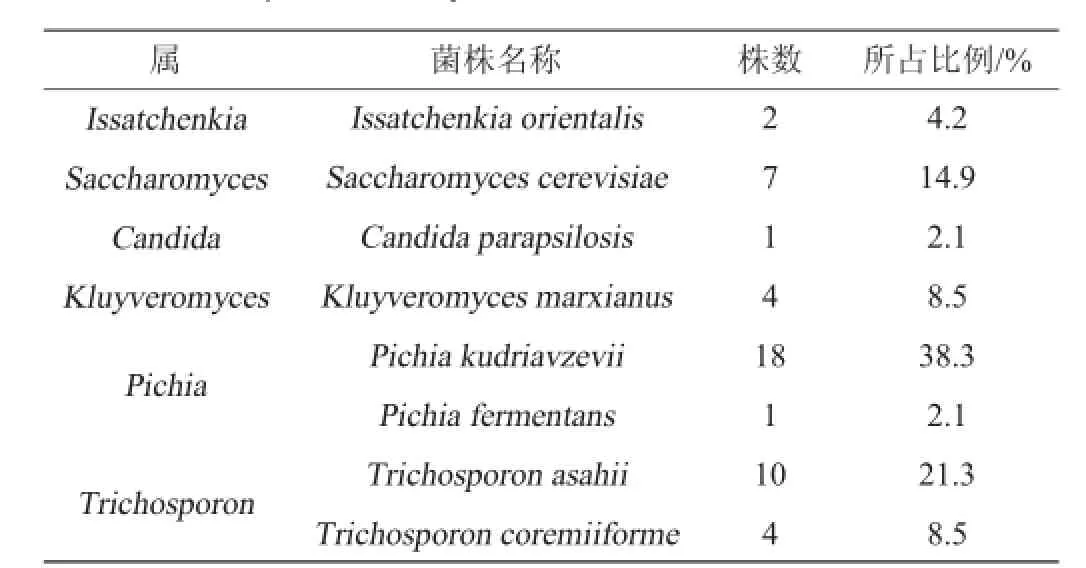
表326S rDNA序列鉴定47株酵母菌结果Table 3 Identification results of 47 yeast strains by 26S rDNA sequences analysis
由表3可以看出,18份样品中分离到47株酵母菌其中包括伊萨酵母(Issatchenkia)、酵母属(Saccharomyces)、假丝酵母(Candida)、克鲁维酵母(Kluyveromyces)、毕赤酵母(Pichia)、丝孢酵母(Trichosporon)6个属,东方伊萨酵母(Issatchenkiaorientalis)、酿酒酵母(Saccharomycescerevisiae)、近平滑假丝酵母(Candida parapsilosis)、马克斯克鲁维酵母(Kluyveromyces marxianus)、库德毕赤酵母(Pichia kudri avzevii)、发酵毕赤酵母(Pichia fermentans)、阿氏丝孢酵母(Trichosporon asahii)、丝孢酵母(Trichosporon coremi iforme)8个种。其中库德毕赤酵母(Pichia kudriavzevii)为优势菌株,占分离株的38.3%,其次为阿氏丝孢酵母(Trichosporon asahii)。从18份样品中分离到47株酵母菌通过PCR-DGGE指纹图谱剔除重复菌株后,得到8株具有代表性的分离株(W10、S7、E9、S3、E1、D3、W2、S17)。8株典型的菌株经PCR扩增,扩增产物送往上海生工测序,测序结果在NCBI使用BLAST比对初步确定菌株的属,GenBank数据库中将所得序列与数据库中已知序列进行比较,用Mega5.0进行相似性分析,构建系统发育树如图5所示。
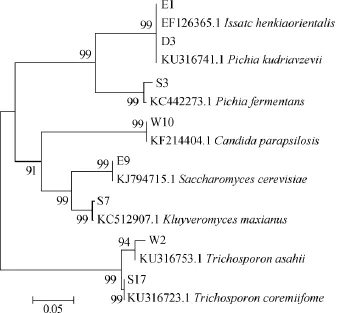
图5 根据26S rDNA D1/D2区域序列生成的8株酵母菌系统发育进化树Fig.5 Phylogenetic tree of 8 yeast strains based on 26S rDNA D1/D2 domain sequences
由图5可知,分离菌株W2与标准菌株的同源性为94%,根据酵母菌种类划分依据,同源相似率低于99%,不能划为同一个种[13],但结合生理生化鉴定结果,可判断此菌株为阿萨希丝孢酵母菌(Trichosporon asahii)。其余所有分离菌株与之对应的标准菌株的同源性均在99%及以上,表明其具有较近的亲缘性。菌株S7为马克斯克鲁维酵母(Kluyveromyces marxianus);菌株E9鉴定为单孢酿酒酵母(Saccharomyces cerevisiae);W10鉴定为近平滑假丝酵母(Candida parapsilosis);S17鉴定为丝孢酵母(Trichosporon coremiiforme)。
LOPANDICA K等[14]发现牛乳中汉逊德巴利酵母(Debaryomyceshansenii),白地霉(Geotrichum candidum),马克斯克鲁维酵母(Kluyveromyces marxianus),解脂耶氏酵母(Yarrowialipolytica)和诞沫假丝酵母(Candidazeylanoides)是最主要的酵母菌属;MUSHTAQ M[15]等研究表明,从酸奶和牛奶中分离出掷孢酵母(Bullera pyricola),假丝酵母(Candida succiphila),卡斯太拉德巴利酵母(Debaryomyces castellii),汉逊德巴利酵母(D.hansenii)和安格斯毕赤酵母(Pichia angusta)。王冠群等[16]对新疆传统发酵乳制品及酵头中酵母菌的分离鉴定,结果表明其中主要的酵母菌为单孢酿酒酵母(Saccharomyces cerevisiae)和东方伊萨酵母(Issatchenkia orientalis)。本试验分离得到的优势菌为库德毕赤酵母(Pichia kudriavzevii)和阿萨希丝孢酵母菌(Trichosporon asahii)。这说明传统发酵酸奶中的酵母菌有其独特的发酵菌种,可能与地缘差异、种类、气候、制作方法等因素有关。
3 结论
本试验对新疆塔城地区哈萨克族传统酸奶18份样品中酵母菌进行分离和鉴定,共得到47株酵母菌,采用传统的分离培养方法、26S rDNA分子生物学鉴定。鉴定结果为库德毕赤酵母(Pichia kudriavzevii)18株,阿萨希丝孢酵母菌(Trichosporon asahii)10株,酿酒酵母(Saccharomyces cerevisiae)7株,丝孢酵母(Trichosporon coremiiforme)4株,马克斯克鲁维酵母(Kluyveromyces marxianus)4株,东方伊萨酵母(Issatchenkia orientalis)2株,发酵毕赤酵母(Pichia fermentans)1株,近平滑假丝酵母(Candidaparapsilosis)1株。本试验将传统培养技术与PCR-DGGE指纹图谱技术相结合,应用于传统乳制品中酵母菌的分离及优势菌群的筛选,并通过基因序列分析使菌种鉴定的准确性、可靠性提高。试验中用PCR-DGGE方法从酸奶样品中获得的菌群结构与传统培养技术获得的有一定的差异,只有将传统分离方法与现代分子生物学方法相结合,才能更全面更真实地反映食品中微生物群落的结构和多样性。本试验用的乳制品样品受环境因素等多方面的影响,样品中优势菌群数量大大降低,给试验中酵母菌的分离带来了一定困难,导致从18份样品中只分离到了47株酵母菌菌株。为进一步开发新疆现有的微生物资源,应确保在无菌条件下采集具有新疆特色传统发酵乳制品的新鲜样品来进行酵母菌的分离筛选;对进一步认识并利用丰富的酵母菌资源具有重要意义;同时也为中国乳制品工业提供优良酵母菌。
[1]张晓旭,葛武鹏,梁秀珍.内蒙古和新疆牧区酸马奶中酵母菌的多样性及其优势菌发酵特性[J].食品科学,2016,37(17):156-162.
[2]闫彬.一组乳酸菌与酵母菌共生关系和风味代谢产物的研究[D].呼和浩特:内蒙古农业大学,2012.
[3]张敏,叶淑红,陈历俊,等.超高效液相色谱分析酵母发酵乳清中的游离氨基酸[J].食品科技,2013,38(8):29-33.
[4]陈成.克鲁维酵母发酵乳清蛋白水解液生产乳清营养酒的研究[J].酿酒,2010,37(2):53-55.
[5]蒋厚阳,陈芝兰,赵国华,等.PCR-DGGE法分析西藏传统发酵乳制品中乳酸菌的多样性[J].食品科学,2014,35(1):167-173.
[6]李蕊婷,卢士玲,李开雄,等.新疆熏马肠中产氨基酸脱羧酶优势细菌的分离及鉴定[J].现代食品科技,2014,30(9):85-91.
[7]GKATZIONIS K,YUNITA D,LINFORTH R S T,et al.Diversity and activities of yeasts from different parts of a Stilton cheese[J].Int J Food Microbiol,2014,177(5):109-116.
[8]吴阳.赛里木酸奶中酵母菌筛选鉴定及发酵特性研究[D].南京:南京农业大学,2012.
[9]ERCOLINI G.PCR-DGGE fingerprinting:novel strategies for detection of microbes in food review[J].J Microbiol Meth,2004,56:297-341.
[10]李静.新疆酸马奶中酵母菌分离鉴定及安全性分析[D].乌鲁木齐:新疆农业大学,2012.
[11]巴尼特JA.酵母菌的特征与鉴定手册[M].胡瑞卿译.青岛:青岛海洋大学出版社,1991:6.
[12]CEUGNIEZ A,DRIDER D,JACQUES P,et al.Yeast diversity in a traditional French cheese"Tomme d'orchies"reveals infrequent and frequent species with associated benefits[J].Food Microbiol,2015,52: 177-184.
[13]ROBNNETT K.Identification and phylogeny of as comycetous yeasts for analysis of nuclear large subunit ribosomal DNA partial sequence[J]. Anton Leeuw,1998,73:331-371.
[14]LOPANDICA K,ZELGERB S,BANSZKYC L K,et al.Identification of yeasts associated with milk products using traditional and molecular techniques[J].Food Microbiol,2006,23:341-350.
[15]MUSHTAQ M,IFTLKHAR F,NAHAR S.Detection of yeast mycoflora from milk and yogurt in Pakistan[J].Pak J Bot,2006,38(3):859-868.
[16]王冠群,韩培杰,杨文菊,等.新疆传统发酵乳制品及酵头中酵母菌的分离鉴定[J].食品与生物技术学报,2015,34(7):691-698.
Isolation,identification and phylogenetic analysis of yeast from traditional Xinjiang fermented yogurt
QIAO Chuanli,JIANG Caihong,JIN Dan,JIANG Aiting,LU Shiling,LI Wangqiang,LI Baokun*
(College of Food Science,Shihezi University,Shihezi 832000,China)
47 yeast strains were isolated from 18 samples of traditional home-made Xinjiang Kazakh naturally fermented yogurt,and then the strains were further identified by morphology analysis,physiological and biochemical identification,PCR-DGGE and 26S rDNA sequencing analysis,finally the phylogenetic tree was constructed.The results showed that the strains were identified as follows:18 strains ofPichia kudriavzevii,10 strains of Trichosporon asahii,7 strains ofSaccharomyces cerevisiae,4 strains ofTrichosporon coremiiforme,4 strains ofKluyveromyces marxianus,2 strains of Issatchenkia orientalis,1 strain ofPichia fermentans,1 strain ofCandida parapsilosis.Therefore,the identification results were significant to recognize and utilize the resource of yeast of traditional Kazakh fermented yogurt.
traditional fermented yogurt;26S rDNA D1/D2;screening;isolation and identification;PCR-DGGE
TS252.54
0254-5071(2017)04-0067-05
10.11882/j.issn.0254-5071.2017.04.015
2016-11-15
国家自然基金地区项目(31460007)
乔传丽(1992-),女,硕士研究生,研究方向为乳制品微生物。
*通讯作者:李宝坤(1979-),男,副教授,博士,研究方向为乳制品微生物。
