氨基化磁性纳米粒子捕获基因组DNA结合PCR快速检测牛奶中金黄色葡萄球菌
2017-04-13白亚龙施春雷王大鹏史贤明
崔 妍,白亚龙,施春雷,王大鹏,史贤明
(上海交通大学农业与生物学院,中美食品安全联合研究中心,微生物代谢国家重点实验室,上海 200240)
氨基化磁性纳米粒子捕获基因组DNA结合PCR快速检测牛奶中金黄色葡萄球菌
崔 妍,白亚龙,施春雷,王大鹏,史贤明*
(上海交通大学农业与生物学院,中美食品安全联合研究中心,微生物代谢国家重点实验室,上海 200240)
本研究对氨基化磁性纳米粒子进行优化,再用优化后的纳米粒子分离牛奶样品中微量的金黄色葡萄球菌基因组DNA,结合PCR检测,可以达到100 CFU/mL的检测限。在此过程中,纳米粒子与DNA的复合物直接加入到PCR体系中进行检测,无需洗脱、纯化等步骤,这样既减少了操作步骤,也减少了DNA损耗,还起到了浓缩的作用。而且,此方法中纳米粒子无需抗体等生物分子标记,成本非常低廉,在生命物质的快速检测中具有广阔的前景与巨大的潜力。
氨基化磁性纳米粒子,金黄色葡萄球菌,PCR,DNA,磁性分离
聚合酶链式反应(PCR)可以在短至几十分钟内将目标DNA片段进行百万倍以上的扩增,目前已经广泛地应用于致病菌检测[1-3]。然而,仅仅应用PCR技术并不能真正实现食源性致病菌快速检测。因为食品成分复杂,目前的DNA纯化技术主要是针对较高浓度的菌液样品,而且有些DNA纯化方法分离得到的产物仍然具有各种PCR抑制物,进而导致扩增失败。因此,一般采取的方法是在PCR检测之前对食品样品中的致病菌进行增菌培养,然而这样检测时间又会被较大幅度的增加。另一种可选的方法是在PCR之前对致病菌(或者致病菌DNA)进行分离与浓缩。
目前比较成功的磁性分离致病菌的方法主要是免疫磁珠法[4-5],它是在磁性纳米粒子上标记了相应的抗致病菌抗体,通过抗原与抗体的特异性结合从而捕获到目标致病菌,然后通过磁性分离将目标菌体分离与富集。这种分离方法完全依赖于抗致病菌抗体的性能,然而,优异的抗致病菌抗体价格昂贵,往往难以获得。致病菌自身极为复杂,菌体表面具有大量的抗原表位,而且有些致病菌血清型复杂,极易出现抗体不能适用于所有菌株的风险。此外,一种抗体对应一种致病菌,多种致病菌检测则需要多种致病菌抗体。因此,本研究旨在将食品经过简单处理,直接从中磁性分离致病菌基因组DNA,从而实现致病菌检测的目的,这样可以使得检测成本显著降低。
目前比较成熟的磁性粒子纯化DNA的方法主要是利用硅包磁性纳米粒子在高盐与低pH条件下将DNA吸附于硅层表面,然后在温和的条件下将目标DNA洗脱下来。但是在这种情况下,除了操作步骤繁琐和操作时间较长之外,既会造成DNA的损耗,又会导致最终DNA溶液体系放大,无法实现浓缩和富集。于是,本研究期望有一种磁性纳米粒子可以直接分离到微量的致病菌基因组DNA,并且可以将得到的微量磁性分离物(磁性纳米粒子与DNA复合物)直接添加到PCR中进行检测,以达到快速且灵敏地检测致病菌的目的。此前,我们发现,氨基化磁性纳米粒子具有这种能力,可以在温和的条件下吸附大量的DNA,经过封闭之后可以直接加入到PCR体系中而不影响PCR扩增[6]。本研究对氨基化磁性纳米粒子氨基化修饰的方法进一步优化以提高分离效率,之后用这种方法分离了牛奶中的金黄色葡萄球菌基因组DNA,再结合PCR技术,从而实现了快速灵敏的分子检测。
1 材料与方法
1.1 材料与仪器
FeSO4·7H2O、FeCl3·6H2O、环己烷、正己醇、氨水等常见化学试剂 分析纯,上海国药集团;正硅酸四乙酯(TEOS)、3-氨丙基三乙氧基硅烷(APTES)、对硝基苯甲醛、溶菌酶、蛋白酶K Sigma-Aldrich公司;TaqDNA聚合酶及其他配套PCR试剂 Thermo-Fisher公司;金黄色葡萄球菌标准菌株ATCC 13565 为本实验室保存菌株。金黄色葡萄球菌特异性检测引物(nuc-f:ATCA TTATTGTAGGTGTATTAGC,nuc-r:CAGGCGTATTC GGTTTC[7]) 由上海生物工程股份有限公司进行合成。培养金黄色葡萄球菌所用培养基为TSB培养基,BD公司生产。
Eppendorf 5415D微量台式离心机离心机、5804R台式高速冷冻离心机、Eppendorf AG PCR仪 德国Eppendorf公司;透射电镜(TEM)H-600 日本日立公司;超声波清洗仪 昆山超声仪器有限公司。
1.2 氨基化硅包磁性纳米粒子的制备
硅包磁性纳米粒子制备参考文献步骤[8],先用共沉淀法制备Fe3O4磁性纳米粒子,然后在反相微乳液中水解TEOS进行包硅,产物用APTES进行氨基化修饰。最终得到的硅包磁性纳米粒子真空干燥后确保无水,分散于三种溶剂(50 mL甲苯、50 mL水、50 mL水∶无水乙醇(1∶4,v/v)溶液)中,超声辅助确保彻底分散,加入2 mL的APTES,持续通氮气,80 ℃回流12 h。用乙醇和水反复洗涤,最终真空干燥。
1.3 分光光度法测定纳米粒子表面氨基数量
以对硝基苯甲醛为偶联试剂,利用分光光度法测定纳米粒子表面的氨基数目[9],具体步骤如下:5 mg氨基化硅包磁性纳米粒子用偶联溶液(1.6 mL冰乙酸添加到无水甲醇中,用无水甲醇定容至200 mL),反复洗涤。磁性分离,在剩余磁性固形物中加入1%的对硝基苯甲醛溶液(溶剂为偶联溶液),混合3 h之后用偶联溶液多次磁性洗涤,除去游离的对硝基苯甲醛,加入1 mL水解溶液(0.4 mL冰乙酸添加到50%的甲醇溶液中,甲醇溶液定容至300 mL),混合1 h后用磁铁将磁珠分离于离心管底部,吸取900 μL上层清液,14000 r/min离心10 min,确保上层清液中没有磁性粒子,吸取上层清液800 μL,测定267 nm处吸光值。同时用梯度稀释的对硝基苯甲醛标准溶液做标准曲线,测定不同样品中的氨基含量。
1.4 氨基化硅包磁性纳米粒子从PCR缓冲溶液中分离DNA性能比较
分别取不同的氨基化硅包磁性纳米粒子配制成1 mg/mL的工作液,取50 μg加入到1 mL金黄色葡萄球菌基因组DNA中(溶剂为1×PCR缓冲液),90 ℃混合10 min,然后磁性分离纳米粒子,分别取分离后与未分离的对照液2 μL作为模板进行qPCR测定,通过计算溶液中减少的DNA量间接比较磁性纳米粒子对DNA的吸附能力。
1.5 人工污染金黄色葡萄球菌的牛奶中基因组DNA的提取方法
取金黄色葡萄球菌过夜培养,用蛋白胨水依次10倍稀释,将不同梯度的金黄色葡萄球菌人工污染牛奶,分别取不同污染量的牛奶10 mL,4 ℃下以转速12000 r/min离心20 min,弃去上层清液以及脂肪层,将收集到的固体沉淀物用0.5 mL含有0.5%曲拉通100的TE缓冲溶液(pH9.0)分散,然后加入50 μL的50 g/L溶菌酶与50 μL的20 g/L蛋白酶K处理30 min。水煮10 min,迅速冷却。用比例为25∶24∶1的苯酚/氯仿/异戊醇进行抽提,所得水层移到一个新的1.5 mL离心管中。加入50 μg磁珠,在90 ℃下混合10 min。外加磁场收集磁性物体,以此作为PCR模板进行扩增。
1.6 qPCR与普通PCR
qPCR体系(25 μL)包含12.5 μL的2×SYBR Green qPCR Mix、10 μmol/L的上下游引物各0.5 μL。PCR热循环步骤:95 ℃保持2 min,每个循环温度为95、60、72 ℃下分别保持10 s,共40个循环。
PCR扩增体系(25 μL)包含1 U聚合酶、浓度为2.5 mmol/L的dNTP1 μL、10 μmol/L的上下游引物各0.5 μL。PCR热循环步骤:94 ℃保持5 min,每个循环温度为94、60、72 ℃下分别保持30 s,共35个循环,最后72 ℃再延伸10 min。扩增产物用2%的琼脂糖凝胶进行电泳,EB染色观察结果。
2 结果与分析
2.1 氨基化磁性纳米粒子表面氨基修饰数量的优化

图2 APTES结合在纳米粒子表面示意图Fig.2 The schematic diagram of binding between APTES and silica shell注:(a)为无水条件下,(b)为有水条件下。
先合成硅包磁性纳米粒子,再通过水解APTES在其表面修饰氨基。在之前的文献中[10-12],很多研究者用甲苯、水、水与乙醇混合物等试剂作为溶剂进行反应,但是哪种方法制备的纳米粒子表面携带更多的氨基并不知晓。本研究分别用甲苯、水、水与乙醇的混合物(体积比1∶4)作为溶剂,将APTES修饰于硅包磁性纳米粒子表面,然后对比各产物表面氨基的数量。本研究中采用对硝基苯甲醛分光光度法测定纳米粒子表面的氨基。对硝基苯甲醛在无水条件下与纳米粒子表面的氨基结合,而在有水条件下会解离下来,通过测定洗脱液中对硝基苯甲醛的量间接获得纳米粒子表面的氨基数目。为了更全面地了解氨基修饰的情况,同时做了在三种溶剂中室温和80 ℃两种不同温度的对比。在测得267 nm处吸光值之后,将数值代入由不同浓度对硝基苯甲醛制备得到的标准曲线(y=9.3913x+0.0134,R2=0.998)。最后的结果如图1所示,与之前报道的用水做溶剂具有较好的氨基修饰效率[13]不同,用甲苯作为溶剂具有更高的氨基修饰率。此外,同样是升高温度,以甲苯为溶剂,升高温度可以增加氨基修饰效率,而以水或水与乙醇混合物为溶剂会降低氨基修饰效率。

图1 不同溶剂制备所得ASMNPs表面携带的氨基数量Fig.1 The amount of amino groups of nanoparticles modified in different solvent 注:水:乙醇混合物体积比为1∶4。
根据文献的报道[13-15],APTES连接到硅表面可以通过两种方式:一种是在无水条件下,APTES与硅表面的羟基直接缩合(图2a);其二,在有水条件下,APTES先水解,然后形成的羟基与硅表面的羟基互相缩合(图2b)。但是,在有水条件下,APTES的水解产物也可以相互缩合交联。之所以得到以上的不同结果,原因可能是:在有水条件下,高温促进了APTES水解产物之间的缩合,而缩合产物和硅层上的羟基并不容易发生结合;在无水条件下,APTES只能与硅表面的羟基缩合,自身之间无法缩合,高温促进了与硅表面羟基的结合。因此,在有水条件下,氨基修饰的数目随温度升高反而降低,在无水条件下随温度升高而增加。根据实验结果来看,用甲苯作为溶剂具有明显的优势。
2.2 表面携带不同氨基数量的纳米粒子对DNA捕获性能的比较分析
根据本课题组之前的研究[6],纳米粒子可能是通过氢键作用及静电作用与DNA结合,氨基在其中充当重要角色,将两种不同氨基携带率的氨基化磁性纳米粒子(MS1:以甲苯为溶剂反应温度为80 ℃条件下得到的氨基化磁性纳米粒子;MS2:以水为溶剂反应温度为80 ℃条件下得到的氨基化磁性纳米粒子)捕获金黄色葡萄球菌基因组DNA的能力进行比较。结果如图3所示,氨基携带率越高的磁性纳米粒子组别Ct值越低,即表面氨基越多,纳米粒子捕获DNA的能力越强。将Ct值代入由不同已知浓度的金黄色葡萄球菌基因组DNA为模板制作的标准曲线(y=-3.458x+33.21,R2=0.9995)中进行计算,MS1与MS2对同样的溶液DNA分离效率分别为97.4%和76.9%。因此,选用以甲苯为溶剂80 ℃下反应的产物进行后续的实验。
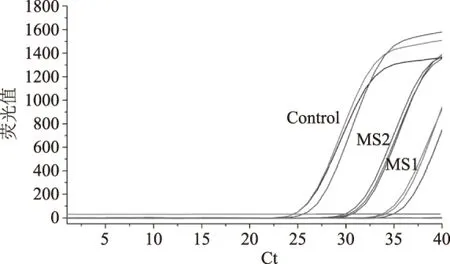
图3 不同氨基携带率的纳米粒子对基因组DNA的吸附能力Fig.3 The DNA adsorption of nanoparticles with different amounts of amino groups
2.3 在人工污染金黄色葡萄球菌牛奶中的应用
将金黄色葡萄球菌过夜培养,并用蛋白胨水稀释成不同的浓度,对购买的牛奶(经过培养法检测不含有金黄色葡萄球菌)进行人工污染,得到不同浓度人工污染金黄色葡萄球菌的牛奶样品,然后按照1.6的方法进行处理,最终用磁性分离得到的吸附有DNA的磁性纳米粒子作为模板进行PCR。PCR产物用2%的琼脂糖凝胶进行电泳,EB染色之后进行观察并拍照。结果如图4所示,当牛奶中金黄色葡萄球菌最低浓度为100 CFU/mL时可以检测到。也就是说,氨基化硅包磁性纳米粒子分离牛奶处理物中致病菌基因组DNA,然后结合普通PCR检测,对于金黄色葡萄球菌检出限为100 CFU/mL。

图4 人工污染金黄色葡萄球菌牛奶的检测灵敏度Fig.4 The sensitivity of this method for detection of Staphylococcus aureus in milk注:NC:阴性对照。
2.4 与其他检测方法的结果对比
如表1所示,为了证明磁性分离在本检测(方法1)中发挥了至关重要的作用,同时对人工污染的牛奶用其他两种DNA提取方法进行提取,然后结合PCR进行检测。这两种方法分别为CTAB法[1](方法2)和试剂盒法(方法3,所用试剂盒为Sangon EZ-10 Spin Column Bacterial Genomic DNA Isolation Kit),这两种方法均无法检出含有105CFU/mL人工污染金黄色葡萄球菌的牛奶样品为阳性。用CTAB法进行提取时,虽然增加了抽提步骤,但是最终得到的DNA提纯物中还是含有大量的不明沉淀物,这些沉淀物的产生可能是因为牛奶中含有大量的蛋白质等杂质,而且多次抽提还会增加DNA的损耗,所以无法检测可能是因为抑制物和损耗两方面的原因。同样地,试剂盒法也可能因为牛奶样品中大量的蛋白质等杂质导致DNA纯化效果不好,导致即使在较高的人工污染浓度下也无法检测到。

表1 几种检测方法灵敏度对比Table 1 The comparison on sensitivities of different detection methods
注:方法1:本方法,即磁性分离DNA结合PCR检测;方法2:CTAB法提取DNA结合PCR检测;方法3:试剂盒提取DNA结合PCR检测,+:有目标条带;-:没有目标条带。
用分子生物学的技术检测食品中致病菌时,很多时候无法做到灵敏的检测,这是因为检测物中含有大量的PCR抑制物,而致病菌在其中的比例非常有限。一般常规的方法无法做到从如此复杂的物质中分离得到微量的致病菌DNA。普通的DNA提取方法主要是针对纯培养物,成分相对简单,只需要简单的离心就可以达到较好的富集和除杂作用,但是对于复杂介质中的微量提取就显得力不从心。磁性分离的优势在这里得以充分展现,一方面,最大程度地去除食品中的PCR抑制物;另一方面,可以将微量的目标物进行有效的分离与浓缩。本研究是用氨基化磁性纳米粒子分离牛奶处理物中的致病菌基因组DNA,这种方法没有特异性,可以应用于任意一种致病菌检测,而且结合多重PCR可以同时检测多种致病菌。这个优势是免疫磁珠无法实现的,一种免疫磁珠只能针对一种特定的致病菌,而且当检测多种致病菌时必须要用到多种抗致病菌抗体。
虽然我们之前发现氨基化硅包磁性纳米粒子直接加入到PCR体系中会对PCR产生抑制[7],这种抑制作用是因为氨基化硅包磁性纳米粒子通过表面吸附了一些PCR成分,但是,我们也发现在磁性捕获DNA之后用类似脱脂奶粉的封闭溶液进行封闭之后再添加进PCR就不会造成明显抑制作用。在本研究中,即使磁性分离后不封闭也不会造成明显抑制,这是因为虽然牛奶经过了多步处理,一些蛋白质类物质依然残留,这些残留物足以将磁性纳米粒子表面进行封闭。
3 结论
用不同的方法在硅包磁性纳米粒子表面修饰氨基基团,经过对比发现用甲苯作为溶剂所制备的氨基化磁性纳米粒子表面携带有更多的氨基。用不同氨基携带量的磁性纳米粒子分离纯化后的金黄色葡萄球菌基因组DNA,结果显示,氨基携带量与DNA分离率相关。将人工污染金黄色葡萄球菌的牛奶样品经过简单处理,然后磁性分离其中的基因组DNA,将磁性纳米粒子与DNA复合物直接加入PCR体系进行检测,灵敏度可以达到100 CFU/mL水平。总的来说,这种方法在检测食品中致病菌污染时有着显著的优势,而且还可以用于病毒检测以及转基因检测等所有涉及到核酸检测的领域。
[1]Yu S J,Chen W Y,Wang D P,et al. Species-specific PCR detection of the food-borne pathogenVibrioparahaemolyticususing the irgB gene identified by comparative genomic analysis[J]. FEMS Microbiology Letters,2010,307(1):65-71.
[2]Suo B,He Y P,Tu S I,et al. A multiplex real-time polymerase chain reaction for simultaneous detection ofSalmonellaspp.,EscherichiacoliO157,andListeriamonocytogenesin meat products[J]. Foodborne Pathogens and Disease,2010,7(6):619-628.
[3]Tang J N,Zhou R,Shi X M,et al. Two thermostable nucleases coexisted inStaphylococcusaureus:evidence from mutagenesis andinvitroexpression[J]. FEMS Microbiology Letters,2008,284(2):176-183.
[4]Panneerseelan L,Muriana P M. An immunomagnetic PCRsignal amplification assay for sensitive detection ofStaphylococcusaureusenterotoxins in foods[J]. Journal of Food Protection®,2009,72(12):2538-2546.
[5]Yang H,Qu L W,Wimbrow A N,et al. Rapid detection ofListeriamonocytogenesby nanoparticle-based immunomagnetic separation and real-time PCR[J]. International Journal of Food Microbiology,2007,118(2):132-138.
[6]Bai Y L,Song M H,Cui Y,et al. A rapid method for the detection of foodborne pathogens by extraction of a trace amount of DNA from raw milk based on amino-modified silica-coated magnetic nanoparticles and polymerase chain reaction[J]. Analytica Chimica Acta,2013,787:93-101.
[7]Tang J N,Shi X M,Shi C L,et al. Characterization of a duplex polymerase chain reaction assay for the detection of enterotoxigenic strains ofStaphylococcusaureus[J]. Journal of Rapid Methods & Automation in Microbiology,2006,14(3):201-217.
[8]Bai Y L,Cui Y,Paoli G C,et al. Nanoparticles affect PCR primarily via surface interactions with PCR components:using amino-modified silica-coated magnetic nanoparticles as a main model[J]. ACS Applied Materials & Interfaces,2015,7(24):13142-13153.
[9]Bruce I J,Sen T. Surface modification of magnetic nanoparticles with alkoxysilanes and their application in magnetic bioseparations[J]. Langmuir,2005,21(15):7029-7035.
[10]Delcampo A D,Sen T,Lellouche J P,et al. Multifunctional magnetite and silica-magnetite nanoparticles:Synthesis,surface activation and applications in life sciences[J]. Journal of Magnetism and Magnetic Materials,2005,293(1):33-40.
[11]Wang Y Y,Su P,Wang S M,et al. Dendrimer modified magnetic nanoparticles for immobilized BSA:a novel chiral magnetic nano-selector for direct separation of racemates[J]. Journal of Materials Chemistry B,2013,1:5028-5035.
[12]Ebrahiminezhad A,Ghasemi Y,Rasoul-amini S,et al. Preparation of novel magnetic fluorescent nanoparticles using amino acids[J]. Colloids and Surfaces B:Biointerfaces,2013,102:534-539.
[13]Van B A,Vrij A. Synthesis and characterization of monodisperse colloidal organo-silica spheres[J]. Journal of Colloid and Interface Science,1993,156(1):1-18.
[14]Vrancken K C,Possemiers K,Van D P,et al. Surface modification of silica gels with aminoorganosilanes[J]. Colloids and Surfaces A:Physicochemical and Engineering Aspects,1995,98(3):235-241.
[15]Vansant E F,Van D P,Vrancken K C. Characterization and chemical modification of the silica surface[J]. Journal of Chromatography-A incl Cumulative Indexes,1996,738(2):313-313.
Development of a rapid detection method forStaphylococcusaureusin milk based on magnetic separation using amino modified silica coated magnetic nanoparticles and polymerase chain reaction
CUI Yan,BAI Ya-long,SHI Chun-lei,WANG Da-peng,SHI Xian-ming*
(MOST-USDA Joint Research Center for Food Safety,School of Agriculture and Biology, and State Key Laboratory of Microbial Metabolism,Shanghai Jiao Tong University,Shanghai 200240,China)
In this study,the synthesis of amino-modified silica-coated magnetic nanoparticles were optimized,the optimal nanoparticles were used to adsorb a trace amount of genetic DNA fromStaphylococcusaureusin milk,and then the complexes were used as DNA templates to be added into PCR. The limit of detection could reach 100 CFU/mL. The DNA and nanoparticle were directly added to PCR system during this process,without any steps of elution and purification. The cost of this magnetic separation method was quite low,for there was no need for nanoparticles to be labelled with biologic molecules such as antibody. This method has a great potential in the rapid detection of living organism.
amino-modified silica-coated magnetic nanoparticles;Staphylococcusaureus;PCR;DNA;magnetic separation
2016-10-21
崔妍(1986-),女,硕士,实验师,研究方向:食品安全与食品生物技术,E-mail:cyan9028@163.com。
*通讯作者:史贤明(1961-),男,博士,教授,研究方向:食品安全与食品生物技术,E-mail:xmshi@sjtu.edu.cn。
国家重点研发计划课题(2016YFD0401102);上海交通大学“Agri-X”基金(AgriX2015005)。
TS207.4
A
1002-0306(2017)07-0295-05
10.13386/j.issn1002-0306.2017.07.049
