利用SSR标记对籼稻品种(系)聚类分析的比较研究
2017-03-31李生强张瑞越周国华黎世龄罗筱平
林 强,李生强,张瑞越,周国华,邹 杰,黎世龄,罗筱平
(宜春学院生命科学与资源环境学院,江西 宜春 336000)
利用SSR标记对籼稻品种(系)聚类分析的比较研究
林 强,李生强,张瑞越,周国华,邹 杰,黎世龄,罗筱平
(宜春学院生命科学与资源环境学院,江西 宜春 336000)
以24个籼稻品种(系)为材料,利用SSR标记技术分别采用NTSYS软件、DPS软件和MAGE软件在4种情况下进行聚类分析。结果表明,无论是利用NTSYS软件通过相关系数,还是利用DPS软件和MAGE软件通过遗传距离进行聚类分析,都基本能将恢复系、三系不育系、短光敏不育系3个类群分开,但由于所选标记及其检测到的等位基因数量不同,聚类的结果也随之不同,特别是水稻恢复系和不育系两大类群下的各亚类的分类结果不同。因此,认为只有首先确定合理的引物数量及其检测到的等位基因数,才能较为准确地进行聚类分析,进而有效地划分杂种优势群。
籼稻;SSR标记;聚类分析;杂种优势群
水稻是世界上最主要的粮食作物之一,全球有50%以上的人以水稻为主食[1]。预计到2050年,全球水稻产量增长1倍才能满足不断增长的人口需求[2]。因此,水稻高产育种一直以来都是育种工作者的重要目标之一。20世纪70年代水稻雄性不育遗传资源的发现和杂种优势的利用,推动了水稻杂交育种的发展,使水稻产量有了突飞猛进的增长。然而,近几十年,中国水稻产量一直没有突破性进展。众所周知,杂交亲本的合理选配是杂种优势有效利用的关键,而适度的遗传差异与杂种优势密切相关。因此,将杂交亲本准确的划分为不同的类群,分析其遗传多样性,可以有效提高杂交水稻强优势组合选育效率。陆作楣等[3]建议以省为单位,将已经推广的杂交稻亲本综合利用分子标记聚类、配合力分析和表型聚类等多种方法,构建水稻杂种优势群,并明确主要的杂交模式。
通过DNA分子标记研究品种间遗传多样性及聚类分析是目前应用广泛的手段之一。研究表明,SSR标记具有操作简单、稳定性好、不受环境和生育期的影响等优点,而且标记多态信息量高,位点已定位在图谱上,适合种质的遗传多样性及聚类分析[4-7]。随着我国玉米遗传多样性及杂种优势群理论的深入研究[8-9],水稻杂交亲本的聚类分析及杂种优势群理论的研究也逐渐受到人们重视。张涛等[10]利用SSR标记研究了三系杂交水稻亲本的遗传多样性;罗小金等[11]用36对SSR引物对籼稻材料进行检测,得到281个等位基因,将58份水稻材料划分为3个类群;姚栋萍等[12]利用19对SSR引物对90份常用水稻品种进行遗传多样性分析,并构建指纹图谱。李爱宏等[13]利用48对SSR标记对20个籼稻品种进行聚类,并分析了杂种优势模式。王胜军等[14]利用72对SSR标记对47个杂交籼稻亲本进行聚类分析,并结合生产实践提出7种杂种优势组合配组模式。
利用分子标记进行聚类分析时,不同研究者选取的标记及检测到的等位基因数量都不相同,但都未说明所选用标记数量的合理性。本研究用94对扩增条带稳定的多态性引物,在24个水稻杂交亲本中检测到272个等位基因,根据PIC值选择不同引物数及相应的等位基因,采用不同的软件进行聚类分析,并对结果进行比较分析,旨在为杂交水稻亲本精确的聚类分析、遗传多样性分析以及杂种优势的高效利用提供理论依据。
1 材料与方法
1.1 试验材料
选用本课题组选配杂交组合的24个籼型水稻品种(系)。包括18个恢复系(93-11、 R148、R140、R0285、R3436、R3253、R1536、R441、R310、D254、R1037、R66、R121、明恢63、R539、R362、R6258和R1113),6个不育系(优IA、五丰A、三A、益丰A、珍汕97A和D38S),其中D38S是宜春学院选育的短光敏核不育系[15-16]。
1.2 SSR分析方法
1.2.1 DNA提取 采取CTAB方法提取水稻基因组DNA[17]。
1.2.2 SSR引物选择 本试验室合成水稻SSR引物近1 000对,根据网上公布数据(http:// www.gramene.org),按照引物在93-11染色体上的位点,所选引物均匀的分布在水稻各染色体上,共选择157对SSR引物。
1.2.3 PCR扩增及产物分离 PCR反应采用20μL体系:模板DNA 2.0μL(25 ng/μL),Taq酶 0.1μL(5.0U/μL),引物2.0μL,dNTP 2.0μL(10 mmol/L),2.0μL 10×Buffer(不含Mg+)MgCl21.0μL(25 mmol/L),用ddH2O补足到20μL。PCR反应程序为:95℃预变性5 min;95℃变性40 s,55~60℃退火(根据引物选择)30s,72℃延伸40 s,35个循环,72℃延伸10 min,扩增产物经3%琼脂糖凝胶分离。
1.3 数据统计分析
SSR扩增产物以0、1统计建立数据矩阵。相同的迁移位置,有带者记为1,无带者记为0。SSR位点的多态性信息量(PIC)按以下公式计算:
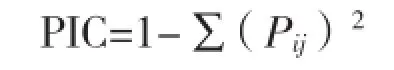
式中,Pij为 i标记第 j个等位基因的频率(Botstein 1980)。根据PIC值选择不同数量引物及相应检测到的等位基因数量,分别利用常用聚类软件NTSYS[18]、唐启义等开发的DPS软件[19]及MEGA软件[20]进行聚类分析,遗传距离计算采用Jicard方法,并对结果进行比较分析。
2 结果与分析
2.1 SSR标记多态性分析
利用157对SSR引物,对24个杂交籼稻亲本品种(品系)进行扩增分析。从中筛选出94对带型清晰稳定的引物分布于水稻12条染色体上。这94对引物共检测出272个等位基因,平均每个位点检测到的等位基因为2.89个,变化范围1~7个。每条染色体上检测出的等位基因范围是8~44个,平均每条染色体检测到等位基因为22.67。所检测到的每个SSR引物位点的多态信息量(PIC)变化范围在0.08~0.89,平均为0.68(表1,图1)。
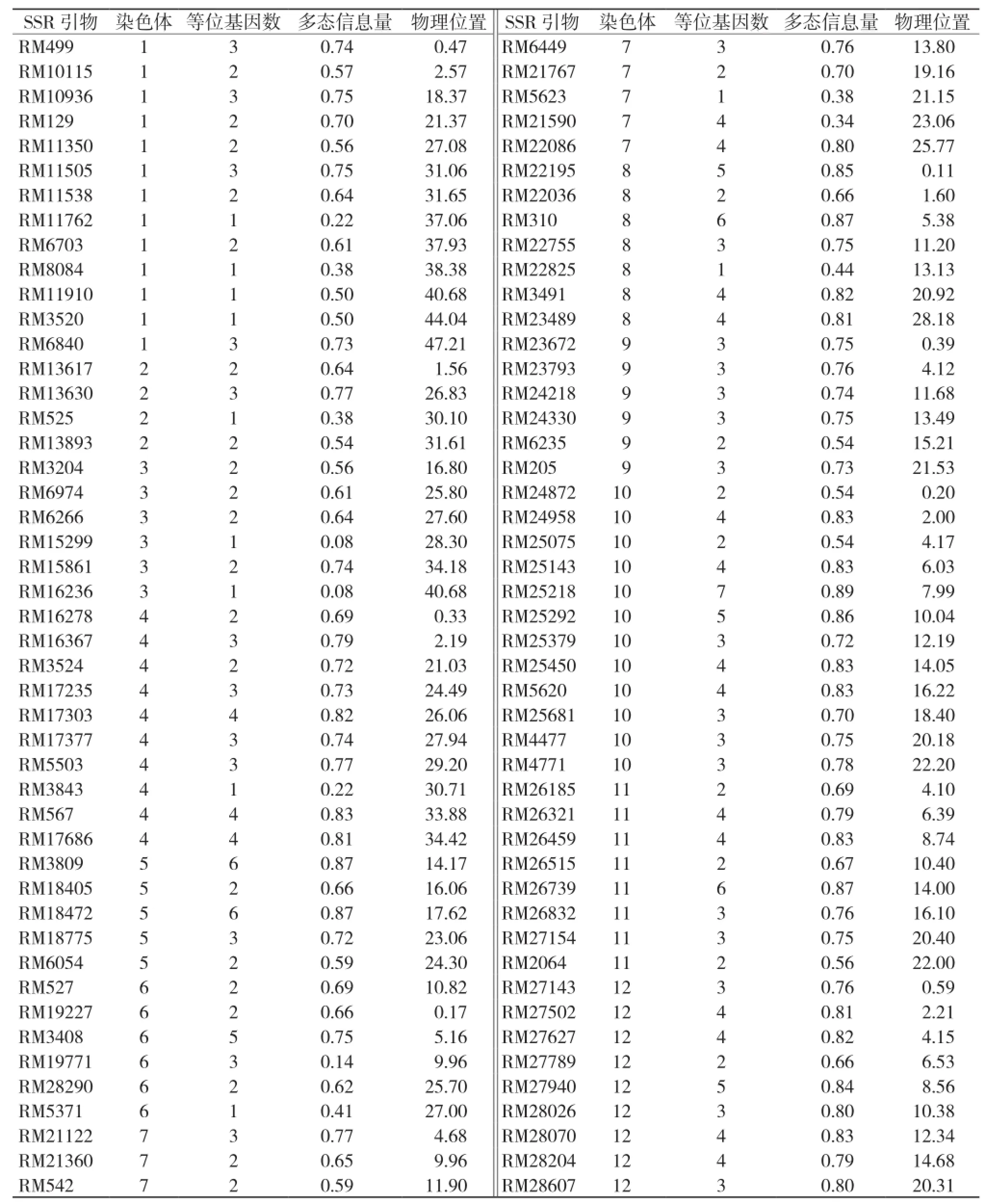
表1 SSR引物的多态信息
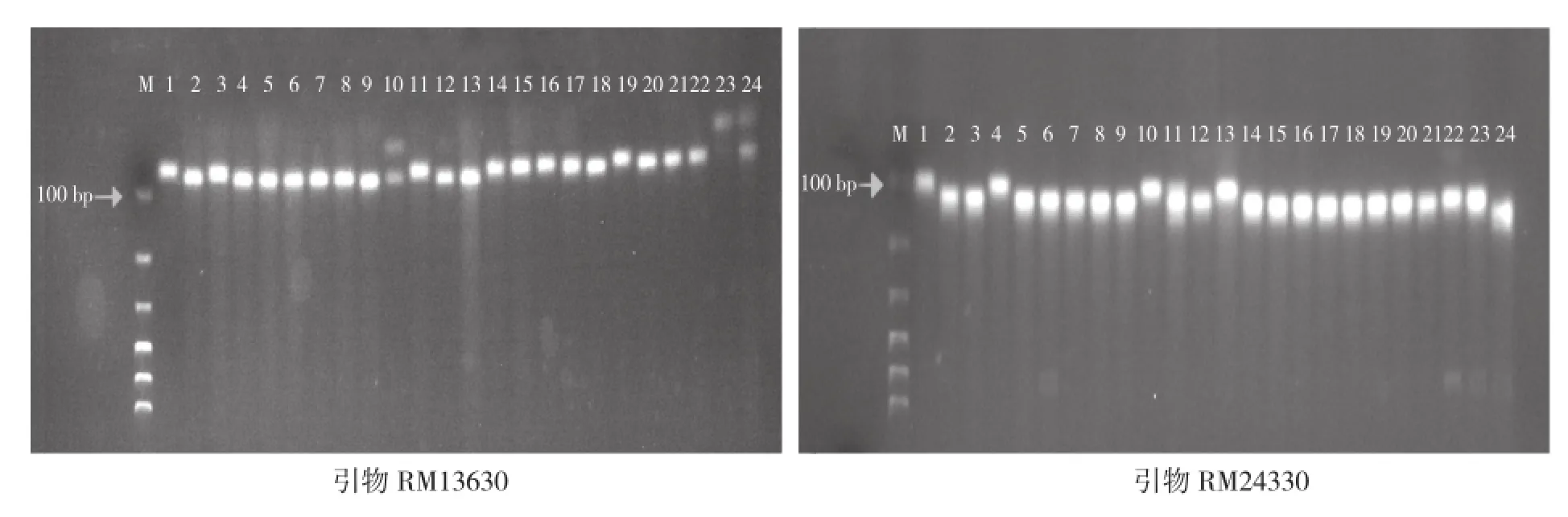
图1 引物RM13630和RM24330在24个籼稻品种(系)扩增情况
2.2 聚类分析比较
2.2.1 利用NTSYS聚类分析 根据PIC值不同,分别选取不同引物及其检测到的等位基因:(1)94对引物检测到272个等位基因;(2)PIC大于0.6时,72对引物检测到234个等位基因;(3)PIC大于0.7时,54对引物检测到200个等位基因;(4)PIC值大于0.75时,37对引物检测到151个等位基因。在这4种情况下利用NTSYS软件分别进行聚类分析,都能将24个水稻品种(系)分为三大类:恢复系类、三系不育系及短光敏不育系D38S。相关系数分别为0.8229、0.8486、0.8019和0.7645。当引物数量少于54对,检测到等位基因少于200的情况下,聚类结果将恢复系R66聚类到三系不育系类群中,并在三系不育系类群下单独聚为一亚类,而三系不育系材料三A聚到恢复系类群中;当引物数量达到72对以上,检测到的等位基因数量达到234以上时,可较为准确地将恢复系和不育系分开,只不过随着当检测到的等位基因数量的增加,相关系数又有减小趋势。同时,在恢复系类群下,不同情况下各亚群聚类结果有所不同(图2)。
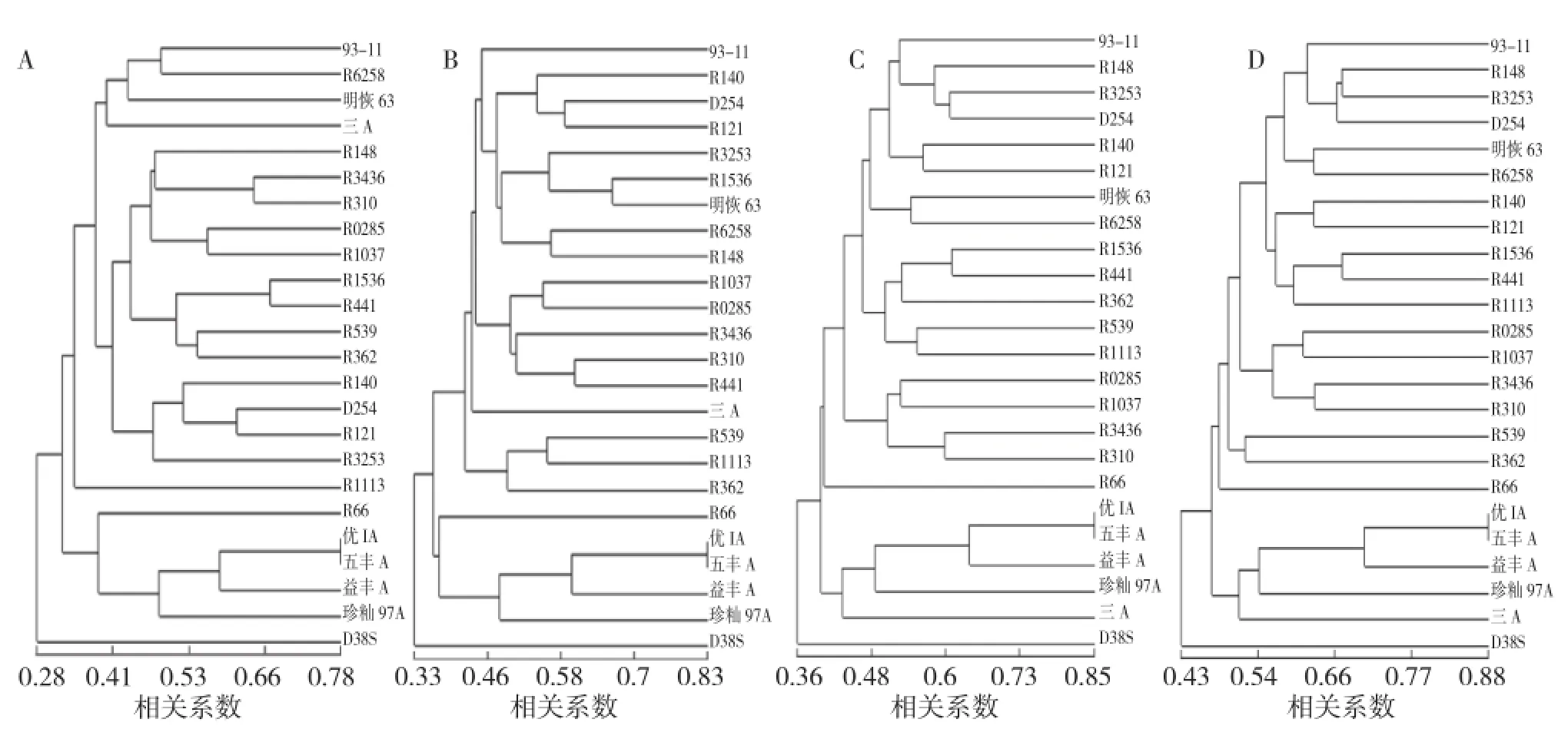
图2 NTSYS软件聚类分析
2.2.2 利用DPS聚类分析 采用相同方法利用DPS软件,在4种情况下分别对24个水稻品种(系)进行聚类分析。图3表明,利用37对引物进行聚类时,结果与NTSYS软件分析类似;而利用54对和72对引物进行聚类分析时,除了恢复系、三系不育系、短光敏不育系三大类群外,明显将恢复系R66单独聚为第4个类群;利用94对引物分析时,将24个水稻品种(系)聚为恢复系、三系不育系和短光敏不育系D38S 3类,这与利用NTSYS软件,在引物数量达到72对以上时的结果相似。

图3 DPS软件聚类分析
2.2.3 利用MEGA聚类分析 根据李亚玲等[17]的分析方法,分别在4种情况下,先使用DPS软件进行0、1数据系统聚类方法获得遗传距离矩阵,然后利用MEGA5.2,采用UPGMA进行聚类分析。当利用37对引物进行聚类分析时,将24个籼稻品种(系)聚为恢复系、三系不育系和短光敏不育系,和前两种软件分析结果相同的是将三A聚在恢复系类群中,不同的是没有将R66聚到三系不育系类群中。当引物数量超过54对时,聚类分析的结果与引物数量达到72对以上,进行NTSYS软件分析时的结果相似,同时也与引物数量达到94对时,进行DPS软件聚类分析结果相似(图4)。
3 结论与讨论
利用SSR标记对水稻品种进行遗传多样性及聚类分析时,不同的研究者选用的标记数及其检测到的等位基因大相径庭。本研究中,利用NTSYS软件通过相似系数进行聚类分析时,所用标记及其检测出的等位基因较少时,将个别不育系聚到恢复系群中,恢复系却聚到不育系群中,随着标记和所检测到的等位基因的增加,相关系数也会随之达到相对最大值,同时将24个水稻品种(系)划分为恢复系、三系不育系和短光敏不育系3类,而当等位基因达到272时,相关系数有减小趋势。而利用DPS和MEGA软件通过遗传距离进行聚类分析,随着标记数量和等位基因的增加,恢复系和三系不育系类群的划分越准确。3种软件都能将24个籼稻品种(系)准确的聚为恢复系、三系不育系和短光敏不育系三大类群,只不过所需要引物数量及检测到等位基因数不同,NTSYS分析需要72对以上的引物,检测到的等位基因数达到234个以上,DPS软件分析需要94对引物检测到272个等位基因数,而MEGA软件分析仅需要54对以上的引物,检测到的等位基因数超过200个时即可。利用分子标记对水稻进行聚类分析是进行水稻遗传多样性研究的有效手段之一,要提高聚类结果的准确性,首先必须根据选用聚类方法分析确定最合理的标记及其检测到的等位基因数量。此外,分子标记在水稻各染色体上的分布情况也会影响到分析结果。

图4 MEGA软件聚类分析
短光敏不育系D38S具有长光可育、短光不育的特性,可实现长日照条件下繁殖、短日照条件下制种,特别是在低纬度地区具有应用前景,是一种新型的两用不育系[16]。在本研究中,不论采用哪种软件,在4种情况下都能将其单独聚为一类,说明D38S与其他三系不育系材料间有较大遗传差异。今后将结合生产上常用两系不育系材料进行对比研究,深入探讨其在生产上潜在的利用价值。
此外,从3种软件易用性方面看,DPS软件具有中文操作界面,且统计与结果分析兼备,在Excel中录入的数据可以直接粘贴至DPS软件进行分析,操作比较简单,但DPS软件不能对生成的聚类图形进行优化编辑。利用DPS软件获得遗传距离矩阵,然后利用MEGA软件进行聚类分析,可以优化编辑聚类图[21],但缺点在于MEGA做聚类分析时,不能识别含有汉字字符的品种(系)名称。而NTSYS软件无法直接粘贴数据,需要经过复杂的操作将数据转换成其可以识别的文件格式,然后才能进行聚类分析。
随着杂交水稻的发展,育种家们不断的培育恢复系与不育系,从中选育具有强杂种优势的组合,一方面杂交亲本间遗传差异逐渐缩小,另一方面工作量不断增大,而选育效率却逐步下降。从玉米杂种优势的成功利用可以清楚地看到,不断提高玉米自交系的一般配合力和组合的特殊配合力是杂交玉米育种工作的主线,而玉米自交系聚类分析及杂种优势群的研究在其中发挥了重要作用[22]。水稻杂种优势利用了几十年,但对杂交亲本的聚类分析及杂种优势群研究相对比较滞后。仅认识到水稻不育系和恢复系是两大优势群是远远不够的,筛选出特殊配合力较高的优势群,是强优势组合高效选育的基础。本研究为水稻杂交亲本精确的聚类分析及杂种优势群研究提供理论依据。
[1] Khush G S. Green revolution:the way forward[J]. Nat Rev Genet,2001,2(10):815-822.
[2] Ray D K,Mueller N D,West P C,et al. Yield trends are insufficient to double global crop production by 2050[J]. PLoS ONE,2013,8:e66428.
[3] 陆作楣,徐保钦. 论杂种优势群理论对杂交稻育种的指导意义[J]. 中国水稻科学,2010,24(1):1-4.
[4] 张羽,陈雪燕,王胜宝,等. 陕西汉中地区主栽水稻品种的SSR多态性分析[J]. 中国农学通报,2011,27(7):34-37.
[5] 束爱萍,刘增兵,余丽琴,等. 水稻SSR标记的遗传多样性研究进展[J]. 植物遗传资源学报,2013,14(5):778-783.
[6] Chen X,Temnykh S,Xu Y,et al. Development of a microsatellite framework map providing genome-wide coverage in rice(Oryza sativa L.)[J]. Theoretical and Applied Genetics,1997,95(4):553-567
[7] 段世华,毛加宁,朱英国. 用微卫星DNA标记对我国杂交水稻主要恢复系遗传差异的检测分析[J]. 遗传学报,2002,29(3):250-254.
[8] 李新海,傅骏骅,张世煌,等. 利用SSR标记研究玉米自交系的遗传变异[J]. 中国农业科学,2000,33(2):1-9.
[9] 李新海,袁力行,李晓辉,等. 利用SSR标记划分70份我国玉米自交系的杂种优势群[J]. 中国农业科学,2003,36(6):622-627.
[10] 张涛,杨蛟,蒋开锋,等. 利用产量功能基因标记分析三系杂交水稻亲本的遗传多样性[J].中国农业科学,2014,47(1):11-23.
[11] 罗小金,贺浩华,付军如,等. 利用SSR分子标记划分籼型水稻杂种优势群[J]. 杂交水稻,2006,21(1):61-64.
[12] 姚栋萍,刘春林,甘玉姿,等. 部分常用水稻品种的指纹图谱构建和遗传多样性分析[J]. 杂交水稻,2016,31(2):47-53.
[13] 李爱宏,戴正元,肖宁,等. 基于SSR标记的不同组群杂交中籼稻亲本配组优势分析[J]. 分子植物育种,2010,8 (1):67-74.
[14] WANG Sheng-Jun,WAN Jian-Min,LU Zuo-Mei. Parental Cluster Analysis in Indica Hybrid Rice(Oryza sativa L1)by SSR Analysis[J]. Acta Agronomica Sinice,2006,32(10):1437-1443.
[15] 黎世龄,高一枝,李会如,等. 短光敏不育水稻宜DS1异交性观察初报[J]. 杂交水稻,1996,(1):32.
[16] 黎世龄,高一枝. 短光敏核不育水稻种质研究[J]. 宜春学院学报,2003,25(6):61-63.
[17] Murray M G,Thompson W F. Rapid isolation of high molecular weight plant DNA[J]. Nucleic Acids Res,1980,8(19):4321-4325.
[18] F James Rohlf. HTSYS-pc:Microcomputer Programs for Numerical Taxonomy and Multivariate Analysis[J]. The American Statistician,1987,41(4):330.
[19] Tang Q Y,Zhang C X. Data Processing System(DPS)software with experimental design,statistical analysis and data mining developed for use in entomological research[J]. Insect Science,2013,20(2):254-260.
[20] Kumar S,Tamura K,Nei M. MEHA3:Integrated software for molecular evolutionary genetics analysis and sequence alignment[J]. Briefings in Bioinformatics,2004,5:150-163.
[21] 李亚玲,韩国民,何沙娥,等. 基于DNA分子标记数据构建系统进化树的新策略[J]. 生物信息学,2008(4):168-170.
[22] 刘亚利,张述宽,苏琪,等. 16个玉米自交系的配合力效应及其聚类分析[J]. 南方农业学报,2011,42(6):578-581.
(责任编辑 杨贤智)
Comparative study on cluster analysis of 24 indica rice varieties(lines)by SSR markers
LIN Qiang,LI Sheng-qiang,ZHANG Rui-yue,ZHOU GUO-hua,ZOU Jie,LI Shi-ling,LUO Xiao-ping
(College of Life Science and Environmental Resources,Yichun University,Yichun 336000,China)
Cluster analysis of 24 indica rice varieties(lines)by 4 different SSR primers using NTSYS,DPS and MAGE software were implemented in this study. The results showed that three groups including restorer lines,cytoplasmic male sterile lines and short photoperiod sensitive sterile lines were distinguished no matter using NTSYS software by the similarity factor,or using DPS and MAGE software clustering analysis based on genetic distance,the results of clustering analysis were different with the number of markers and alleles,especially the sub group under the male sterile lines and restorer lines,which were recognized as dominant group of rice. This study suggests that the number of alleles detected by molecular markers affects the accuracy of clustering results and the effective construction of rice heterosis groups.
indica;SSR markers;cluster analysis;heterotic groups
S511.01
A
1004-874X(2017)01-0001-07
2016-09-25
江西省教育厅科学技术研究项目(151034)
林强(1993-),男,学士,E-mail:linqdeyx@163.com
李生强(1978-),男,博士,讲师,E-mail:lisqyuer@163.com
林强,李生强,张瑞越,等. 利用SSR标记对籼稻品种(系)聚类分析的比较研究[J].广东农业科学,2017,44(1):1-7.
