北宗黄酒麦曲微生物的分离鉴定
2017-03-27任清,侯昌
任 清,侯 昌
(北京工商大学食品学院,食品添加剂与配料北京高校工程研究中心,北京 100048)
北宗黄酒麦曲微生物的分离鉴定
任 清,侯 昌
(北京工商大学食品学院,食品添加剂与配料北京高校工程研究中心,北京 100048)
利用梯度稀释法和划线纯化法对麦曲中的微生物进行分离纯化,得到7 株细菌和5 株真菌。通过形态学特征观察和分子生物学方法进行菌种鉴定,细菌分别为地衣芽孢杆菌(Bacillus licheniformis)、枯草芽孢杆菌(Bacillus subtilis)、短小芽孢杆菌(Bacillus pumilus)、萎缩芽孢杆菌(Bacillus atrophaeus)、克劳氏芽孢杆菌(Bacillus clausii)、根际芽孢杆菌(Bacillus rhizosphaerae)、索诺拉沙漠芽孢杆菌(Bacillus sonorensis)。真菌分别为米曲霉(Aspergillus oryzae)、黑曲霉(Aspergillus niger)、根菌属(Talaromyces radicus)、粗糙脉孢菌(Neurospora crassa)、伞状犁头霉(Absidia corymbifera)。
北宗黄酒;麦曲;微生物;鉴定;分离
黄酒产自中国,是世界上最古老的酒类之一,黄酒酒体柔和、香气优雅、营养丰富,并且具有独特的保健功能[1]。以谷物为原料,“以麦制曲、用曲发酵”的传统酿造工艺成就了我国黄酒独有的品质特征。麦曲是将小麦经轧碎后加水拌匀踏成曲坯,经自然培养而成的一种含多种微生物和酶系的复合糖化发酵剂。酿造过程中,麦曲微生物不仅发挥了糖化发酵作用,而且具有重要的生香增味作用,麦曲微生物的种类和密度对黄酒的出酒率和风味品质有极大的影响[2]。因此,从麦曲中分离鉴定微生物并对其进行生物学研究,对于黄酒生产和品质调控具有重要的意义。
黄酒分南方黄酒和北方黄酒两大系列,南方黄酒以稻米为原料,北方黄酒以粟或黍为原料,所用麦曲也完全不同。近年来,我国黄酒领域的科技工作者已经对南方黄酒麦曲微生物进行了分离鉴定,并对其进行了初步研究分析[3-6],而对北方黄酒麦曲微生物的研究甚少报道,北宗黄酒是我国北方黄酒的典型代表,本研究对北宗黄酒麦曲中的微生物进行分离、纯化和鉴定,以期为北方黄酒生产和品质调控提供理论基础。
1 材料与方法
1.1 材料、试剂与培养基
北宗黄酒麦曲由张家口北宗黄酒酿造有限公司提供;通用引物(27f和1492r、ITS-4和ITS-5)由华大基因公司合成。
2×Utaq PCR Mix、DNA Marker Ⅶ、6×DNA loading buffer 北京庄盟国际生物基因科技有限公司;植物基因组DNA提取试剂盒 天根生化试剂公司。
卵磷脂吐温-80营养琼脂培养基:蛋白胨20 g、牛肉膏粉3 g、氯化钠5 g、卵磷脂1 g、吐温-80 7 g、琼脂15 g,加蒸馏水定容至1 L,加热煮沸溶解,121 ℃高压灭菌20 min。孟加拉红固体(rose bengal agar,RBA)培养基:蛋白胨5 g、磷酸二氢钾1 g、硫酸镁0.5 g、葡萄糖10 g、氯霉素0.1 g、孟加拉红0.033 g、琼脂18.5 g,加蒸馏水定容至1 L,加热煮沸溶解,121 ℃高压灭菌20 min。马铃薯葡萄糖琼脂(potato dextrose agar,PDA)培养基:200 g去皮土豆切成小块放入锅中,加水1 L,煮沸30 min,纱布过滤,加葡萄糖20 g,琼脂粉18 g,加蒸馏水定容至1 L,煮沸溶解,121 ℃高压灭菌20 min;其液体培养基配制时不加琼脂粉。
1.2 仪器与设备
XP基因扩增仪 杭州博日科技有限公司;Axio Image A1显微镜 德国Zeiss公司;DYY-6C型电泳仪北京六一仪器厂;Imager 2200凝胶成像系统 美国Alpha公司;高压灭菌锅 日本三洋公司;恒温培养箱北京天林恒泰科技有限公司。
1.3 方法
1.3.1 菌种的分离纯化
称取10 g粉碎的麦曲,加入装有90 mL无菌水和若干玻璃珠的250 mL三角瓶中,然后置于恒温摇床中,27 ℃条件下200 r/min振荡30 min,至摇散混匀,使麦曲中微生物均匀分散在无菌水中,此样品稀释度为10-1。然后将此样品进行10 倍梯度稀释,并稀释至10-6。取10-3、10-4、10-5的稀释液,分别涂布于卵磷脂吐温-80营养琼脂培养基、RBA培养基、PDA培养基平板,涂布量为100 μL,每种培养基每个稀释倍数各涂布3 个平行,待菌液完全吸收后,倒置于30 ℃恒温培养箱中需氧培养1~5 d。根据菌落生长形态,大小及颜色的不同,分别挑取不同的单菌落接种于相应的固体培养基中进行纯化,直至得到单一微生物。之后进行镜检,确定是单一菌后进行保菌,并放入4 ℃冰箱中保存备用。
1.3.2 菌落及菌体形态观察
将分离得到的细菌在卵磷脂吐温-80营养琼脂上进行划线,于37 ℃条件下需氧培养24 h后观察菌落生长情况及菌落形态。通过革兰氏染色、显微镜观察菌体形态。将分离得到的真菌同时在RBA和PDA固体培养基上需氧培养,25 ℃条件下培养5~7 d后观察菌落生长情况及菌落形态。
1.3.3 细菌的分子生物学鉴定
细菌基因组DNA的提取:将单一菌株接种于LB液体培养基中,37 ℃、200 r/min振荡培养12 h。12 000 r/min离心2 min收集菌体,洗涤,十六烷基三甲基溴化铵(cetyltrimethylammonium ammonium bromide,CTAB)法提取基因组DNA作为PCR的模板[7]。
采用通用引物27f(5′-AGAGTTTGATCCTGGCTCAG-3′)和1492r(5′-TACGGCTACCTTGTTACGACTT-3′)扩增细菌的16S rDNA序列[8]。反应体系(50 μL)为:25 μL 2×Taq PCR MasterMix(含染料)、21 μL ddH2O、2 μL菌悬液、1 μL 27f(10 μmol/L)、1 μL 1492r(10 μmol/L)。PCR扩增程序:94 ℃预变性5 min;94 ℃变性30 s,55 ℃退火30 s,72 ℃延伸90 s,30 个循环;然后72 ℃延伸10 min,4 ℃暂时保存,-20 ℃条件下保存备用。取5 μL的PCR产物用1%琼脂糖凝胶电泳检测。
电泳检测后的PCR产物由华大基因完成纯化和测序,获得16S rRNA基因序列。测序结果在NCBI的GenBank中进行BLAST分析比对,进行同源序列搜索,根据同源序列搜索结果,导找相关模式菌株的16S rRNA基因序列区,与实验菌株一起用MEGA6构建系统发育进化树。
1.3.4 真菌的分子生物学鉴定
挑取真菌平板上的菌丝于20 mL的PDA液体培养基,在25 ℃恒温摇床中200 r/min振荡培养3 d。菌体收集采用2 层无菌纱布过滤菌体,然后用0.85%的无菌生理盐水冲洗2~3 次,最后用无菌滤纸将水分吸尽。将菌体放入无菌研钵中,加入液氮充分碾磨至粉状。然后用植物基因组DNA提取试剂盒提取真菌的全基因组DNA。1%琼脂糖凝胶电泳检测,-20 ℃保存备用。
采用I T S区扩增通用引物I T S-4(5’-TCCTCCGCTTATTGATATGC-3’)和ITS-5(5’-GGAAGTAAAAGTCGTAACAAGG-3’)进行PCR扩增[9]。反应体系(50 μL):25 μL 2×Taq PCR MasterMix(含染料)、20 μL ddH2O、3 μL 模板、1 μL ITS-4(10 μmol/L)、1 μL ITS-5(10 μmol/L)。PCR扩增程序:94 ℃预变性5 min;94 ℃变性30 s,51 ℃退火30 s,72 ℃延伸45 s,30 个循环;然后72 ℃延伸10 min,4 ℃条件下暂时保存,-20 ℃条件下保存备用。取5 μL的PCR产物用1%琼脂糖凝胶电泳检测。
电泳检测后的PCR产物由华大基因完成纯化和测序。测序结果在NCBI的GenBank中进行BLAST分析比对,进行同源序列搜索,根据同源序列搜索结果,寻找相关模式菌株的ITS区基因序列,与实验菌株序列一起用MEGA6构建系统发育进化树。
2 结果与分析
2.1 细菌的菌落形态观察结果
从麦曲中分离,纯化后得到7 株细菌,依次编号为B1、B2、B3、B4、B5、B6和B7(图1)。7 株细菌在卵磷脂吐温-80营养琼脂培养基上需氧培养24 h后,菌落形态上存在一定的差异。B1菌落呈现圆锯齿状,表面湿润有光泽且有大量的黏液,菌落大,直径4~5 mm;B2菌落为浅黄色,较薄,表面黏湿,边缘不整齐,有很多树叶边缘状的突起;B3菌落颜色较淡,菌落边缘不整齐,且不透明;B4菌落表面粗糙不透明,微黄色;B5菌落小,菌落边缘不整齐,微透明;B6菌落微黄色,边缘不整齐,表面黏湿,不凸起,不透明;B7菌落较小,且菌落中心有一白色小点。7 株细菌经革兰氏染色,均为革兰氏阳性菌。
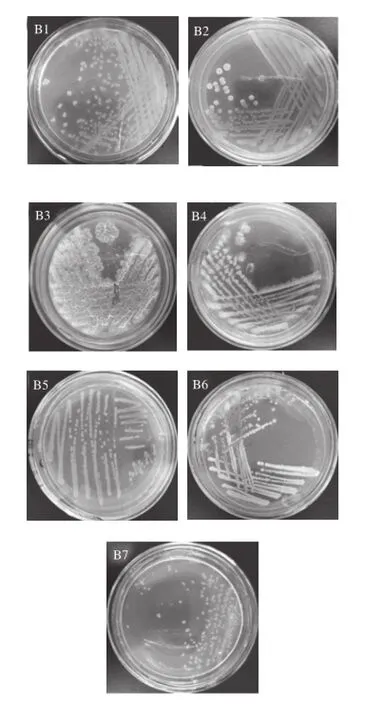
图1 麦曲中7 株细菌在卵磷脂吐温-80营养琼脂及37 ℃条件下的菌落特征Fig.1 Colony morphologies of seven isolated bacterial strains on lecithin Tween 80 nutrient agar (37 ℃)
2.2 细菌16S rRNA基因序列PCR扩增结果
如图2所示,以CTAB提取的7 株细菌的DNA为模板,27f和1492r为引物,PCR扩增后得到的产物经琼脂糖凝胶电泳分析和紫外检测后,所扩增的目的片段电泳条带清晰且特异性强,大小约为1 500 bp,与实验设计吻合。
2.3 细菌16S rRNA基因序列同源性比对与系统发育分析


图3 以16S rRNA基因序列为分子标记的7 株细菌系统发育进化树Fig.3 Phylogentic tree of seven isolated bacterial strains based on their 16S rRNA sequences
将菌株B1~B7的16S rRNA基因序列与GenBank内已知菌株的相应序列进行比对,通过同源性比对结果构建系统发育进化树。如图3所示,菌株B1~B7与多株Bacillus sp.的同源性达99%以上,菌株B1与Bacillus licheniformis NBRC 12200(AB680255.1)在同一分枝上,且通过Bootstrap的验证表明它们具有较高的置信度,支持率可达到100%(图3a),因此可将分离菌株B1确定为地衣芽孢杆菌(Bacillus licheniformis)。菌株B2与Bacillus pumilus ATCC 7061(JQ424887.1)在同一分枝上,且通过Bootstrap的验证表明它们具有较高的置信度,支持率可达到100%(图3b),因此可将分离菌株B2确定为短小芽孢杆菌(Bacillus pumilus)。菌株B3与Bacillus atrophaeus NBRC 15539(AB363731.1)在同一分枝上,且通过Bootstrap的验证表明它们具有较高的置信度,支持率达到99%(图3c),因此可将分离菌株B3确定为萎缩芽孢杆菌(Bacillus atrophaeus)。菌株B4与Bacillus subtilis DSM10 (JQ424889.1)在同一分枝上,且通过Bootstrap的验证表明它们具有较高的置信度,支持率达到100%(图3d),因此可将分离菌株B4确定为枯草芽孢杆菌(Bacillus subtilis)。菌株B5与Bacillus clausii DTM1(KR632489.1)在同一分枝上,且通过Bootstrap的验证表明它们具有较高的置信度,支持率达到100%(图3e),因此可将分离菌株B5确定为克劳氏芽孢杆菌(Bacillus clausii)。菌株B6与Bacillus rhizosphaerae SC-N012(FJ233848.1)在同一分枝上,且通过Bootstrap的验证表明它们具有较高的置信度,支持率达到100%(图3f),因此可将分离菌株B6确定为根际芽孢杆菌(Bacillus rhizosphaerae)。菌株B7与Bacillus sonorensis LG-2(KT725252.1)在同一分枝上,且通过Bootstrap的验证表明它们具有较高的置信度,支持率达到98%(图3g),因此可将分离菌株B7确定为索诺拉沙漠芽孢杆菌(Bacillus sonorensis)。
2.4 真菌的菌落形态观察结果

图4 5 株真菌在PDA和RBA培养基上25 ℃培养7 d的菌落特征Fig.4 Colony morphologies of fi ve isolated fungal strains on PDA and RBA media (25 ℃ for 7 d)
由图4可知,5 株真菌在PDA培养基上生长均较RBA培养基上旺盛。F1在RBA培养基上初期呈现白色,后期呈暗黄色,致密绒毛状,菌落边缘清晰,在PDA培养基上则呈绿色,边缘清晰,孢子密度高;F2在RBA和PDA培养基上菌落形态无太大差异,菌落呈黑褐色,平滑粗糙,菌落边缘清晰,分生孢子头褐黑色放射状,分生孢子梗长短不一,且在PDA上菌落周围有少量白色菌丝;F3在RBA和PDA培养基上菌落形态基本相同,呈灰色菌落,菌落边缘较清晰,无菌丝;F4在RBA和PDA培养基上菌落形态基本相同,生长速率快,菌落边缘不明显,具有疏松的网状长菌丝,有气生菌丝,在显微镜下其菌丝透明,有分枝和分隔,分生孢子呈桔黄色;F5在RBA和PDA培养基上菌落形态基本相同,菌丝无隔,分枝状,广泛蔓延,匍匐菌丝,无定形菌落,边缘不清晰,菌丝初期白色,后期呈淡淡的暗黄色,生长速率较快。
2.5 真菌基因组DNA的提取和PCR扩增验证

图5 5 株真菌菌株ITS区rDNA的电泳检测图Fig.5 PCR analysis of ITS rDNA sequences of fi ve isolated fungal strains
将菌体用液氮充分碾磨至粉状,然后用植物基因组DNA提取试剂盒提取真菌的全基因组作为PCR模板,以ITS4和ITS5为引物,经PCR扩增后得到的产物经琼脂糖凝胶电泳分析和紫外检测后,所扩增的目的片段电泳条带清晰、特异性强,F1~F4条带大小约为600 bp,F5的条带大小约为850 bp,与实验设计吻合(图5)。
2.6 rDNA ITS同源性比对与系统发育分析
将菌株F1~F5的ITS rDNA基因序列与GenBank内已知菌株的相应序列进行比对,通过同源性比对结果构建系统发育进化树。由图6可知,菌株F1与Aspergillus oryzae IFO 5375(AB000533.1)在同一分枝上,且通过Bootstrap的验证表明它们具有较高的置信度,支持率可达到99%(图6A)。菌株F2与Aspergillus niger CBS 139.52(FJ629347.1)在同一分枝上,且通过Bootstrap的验证表明它们具有较高的置信度,支持率达到99%(图6B)。菌株F3与Talaromyces radicus KUC 1679(HM469413.1)在同一分枝上,且通过Bootstrap的验证表明它们具有很高的置信度,支持率达到100%(图6C)。菌株F4与Neurospora crassa(AY681193.1)在同一分枝上,且通过Bootstrap的验证表明它们具有比较高的置信度,支持率达到97%(图6D)。菌株F5与Absidia corymbifera IP 1129.75(DQ118982.1)在同一分枝上,且通过Bootstrap的验证表明它们具有很高的置信度,支持率达到100%(图6E)。

图6 以ITS rDNA基因序列为分子标记的5 株真菌菌株系统发育进化树Fig.6 Phylogentic tree of fi ve isolated fungal strains based on ITS rDNA sequences
结合图4的形态特征与《真菌鉴定手册》[10]中相应的霉菌的描述进行比较发现结果基本一致,因此可以将分离得到的5 株F1~F5菌株可依次确定为米曲霉(Aspergillus oryzae)、黑曲霉(Aspergillus niger)、Talaromyces radicus、粗糙脉孢菌(Neurospora crassa)、伞状犁头霉(Absidia corymbifera)[11]。
3 结论与讨论
通过对麦曲中微生物进行菌落形态观察,镜检和分子生物学鉴定,最终分离纯化得到7 株细菌和5 株霉菌。
绍兴黄酒麦曲中的常见细菌有芽孢杆菌属和葡萄球菌属及少量泛菌属、肠球菌属等的细菌[12]。谭婷婷[13]从黄酒麦曲中分离得到的芽孢杆菌属有:地衣芽孢杆菌(Bacillus licheniformis)、枯草芽孢杆菌(Bacillussubtilis)和蜡样芽孢杆菌(Bacillus cereuss)。本实验从黄酒麦曲中除分离纯化得到地衣芽孢杆菌(Bacillus licheniformis)、枯草芽孢杆菌(Bacillus subtilis)外,还得到了短小芽孢杆菌(Bacillus pumilus)、萎缩芽孢杆菌(Bacillus atrophaeus)、克劳氏芽孢杆菌(Bacillus clausii)、根际芽孢杆菌(Bacillus rhizosphaerae)、索诺拉沙漠芽孢杆菌(Bacillus sonorensis)5 株芽孢杆菌。
实验从黄酒麦曲中除分离纯化得到米曲霉(Aspergillus oryzae)外,还分离纯化得到了黑曲霉(Aspergillus niger)、根菌属真菌(Talaromyces radicus)、粗糙脉孢菌(Neurospora crassa)、伞状犁头霉(Absidia corymbifera)。根据文献报道,粗糙脉孢菌产生的纤维素酶能降解纤维素生成纤维二糖和葡萄糖等小分子物质,作为不能直接利用纤维素的酿酒酵母的碳源底物来进一步发酵生产酒精等[14-16],粗糙脉孢菌还能够发酵葡萄糖和木糖生产乙醇[17-18];伞状犁头霉能产生淀粉分解酶,能够将发酵过程中的糊化、液化、糖化等过程合并为一步直接进行糖化[19];黑曲霉可产生β-葡萄糖苷酶、木聚糖酶、α-鼠李糖苷酶、淀粉降解酶、纤维素酶和半纤维素酶,在发酵糖化过程中能起到较好的作用[20-24];米曲霉是麦曲中主要的霉菌之一,能产生糖化酶、α-淀粉酶、酸性蛋白酶等[25];米曲霉和黑曲霉共同作用,能加快发酵速度,缩短主酵时间,提高黄酒酿造原料利用率,降低黄酒生产成本[26-27]。
[1] 刘月, 祁国栋, 张炳文. 黄酒功能成分的研究进展[J]. 中国酿造, 2015, 34(11): 27-30. DOI:10.11882/j.issn.0254-5071.2015.11.007.
[2] 寿虹志, 凌志勇, 杨旭, 等. 浅析黄酒麦曲中的微生物与黄酒风味的关系[J]. 中国酿造, 2007, 26(8): 55-57. DOI:10.3969/ j.issn.0254-5071.2007.08.017.
[3] 毛青钟. 黄酒机制生麦曲与传统生麦曲的比较探讨[J]. 中国酿造, 2005, 24(5): 42-44. DOI:10.3969/j.issn.0254-5071.2005.05.013.
[4] 方华, 曹钰, 陆健, 等. 黄酒麦曲中主要霉菌的分子鉴定及分类[J].酿酒科技, 2006(3): 45-47. DOI:10.3969/j.issn.1001-9286.2006.03.010.
[5] 陈建尧, 曹钰, 谢广发, 等. 黄酒机械成型麦曲制曲过程中真菌动态变化的研究[J]. 食品与发酵工业, 2008, 34(8): 42-47. DOI:10.13995/ j.cnki.11-1802/ts.2008.08.015.
[6] 曹钰, 陆健, 方华, 等. 绍兴黄酒麦曲中真菌多样性的研究[J]. 食品科学, 2008, 29(3): 277-282. DOI:10.3321/j.issn:1002-6630.2008.03.056.
[7] SHAO B Y, CHEN B, TANG M Y, et al. Optimization of DNA extraction from Salmonella and PCR reaction condition[J]. Food Science, 2007, 28(7): 331-334.
[8] JAMES G. Universal bacterial identification by PCR and DNA sequencing of 16S rRNA gene[M]//PCR for clinical microbiology. Netherlands: Springer, 2010: 209-214. DOI:10.1007/978-90-481-9039-3_28.
[9] CASTAÑEDA-RAMÍREZ G S, de GIVES P M, AGUILARMARCELINO L, et al. Phylogenetic analysis of nucleotide sequences from the ITS region and biological characterization of nematophagous fungi from Morelos, Mexico[J]. Journal of Mycology, 2016, 2016: 1-13. DOI:10.1155/2016/8502629.
[10] 魏景超. 真菌鉴定手册[M]. 上海: 上海科技出版社, 1979: 566-614.
[11] KERK P M, CANNON P F, MINTER D W, et al. Dictionary of the fungi[M]. Oxfordshire: CABI Publishing, 2008: 29-35.
[12] 张中华. 绍兴黄酒麦曲中微生物群落结构的研究[D]. 无锡: 江南大学, 2012: 17-20.
[13] 谭婷婷. 北方黄酒麦曲微生物的分离鉴定及优良菌株特性研究[D].青岛: 青岛科技大学, 2015: 16-19.
[14] XIROS C, TOPAKAS E, KATAPODIS P, et al. Hydrolysis and fermentation of brewer’s spent grain by Neurospora crassa[J]. Bioresource Technology, 2008, 99(13): 5427-5435. DOI:10.1016/ j.biortech.2007.11.010.
[15] 冯炘, 王丹, 辛丽霞. 粗糙脉孢菌产纤维素酶发酵条件研究[J]. 食品科学, 2005, 26(1): 67-70. DOI:10.3321/j.issn:1002-6630.2005.01.010.
[16] DESPANDE V, KESKAR S, MISHRA C, et al. Direct conversion of cellulose/hemicellulose to ethanol by Neurospora crassa[J]. Enzyme & Microbial Technology, 1986, 8(3): 149-152. DOI:10.1016/0141-0229(86)90103-1.
[17] MISHRA C, KESKAR S, RAO M. Production and properties of extracellular endoxylanase from Neurospora crassa[J]. Applied and Environmental Microbiology, 1984, 48(1): 224-228.
[18] 张潇, 朱冬青, 王丹, 等. 粗糙脉孢菌(Neurospora crassa)木糖发酵的研究[J]. 微生物学报, 2003, 43(4): 466-472. DOI:10.3321/ j.issn:0001-6209.2003.04.011.
[19] 秦臻, 蔡素梅, 黄钧, 等. 一株产生淀粉分解酶犁头霉的分离鉴定及其酶学性质[J]. 微生物学通报, 2011, 38(5): 729-735.
[20] 王春丽, 武改红, 陈畅, 等. 黑曲霉原生质体诱变选育β-葡萄糖苷酶高产菌株[J]. 生物工程学报, 2009, 25(12): 1921-1926. DOI:10.3321/ j.issn:1000-3061.2009.12.022.
[21] OMEMU A M, AKPAN I, BANKOLE M O, et al. Hydrolysis of raw tuber starches by amylase of Aspergillus niger AM07 isolated from the soil[J]. African Journal of Biotechnology, 2005, 4(1): 19-25. DOI:10.5897/AJB2005.000-3006.
[22] ZHOU C Y, BAI J Y, DENG S S, et al. Cloning of a xylanase gene from Aspergillus usamii, and its expression in Escherichia coli[J]. Bioresource Technology, 2008, 99(4): 831-838. DOI:10.1016/ j.biortech.2007.01.035.
[23] ŠIMČÍKOVÁ D, KOTIK M, WEIGNEROVÁ L, et al. α-LRhamnosyl-β-D-glucosidase (Rutinosidase) from Aspergillus niger: characterization and synthetic potential of a novel diglycosidase[J]. Advanced Synthesis & Catalysis, 2014, 357(1): 107-117. DOI:10.1002/ adsc.201400566.
[24] 吴升山, 蔡慧农, 苏文金, 等. 黑曲霉DB056发酵α-鼠李糖苷酶和柚苷酶培养基的优化研究[J]. 中国食品学报, 2010, 10(4): 193-201. DOI:10.3969/j.issn.1009-7848.2010.04.030.
[25] MORTEN C, SPOHR A B, JENS N, et al. Morphology and physiology of an alpha-amylase producing strain of Aspergillus oryzae during batch cultivations[J]. Biotechnology & Bioengineering, 2000, 49(3): 266-276. DOI:10.1002/(SICI)1097-0290(19960205)49:3<266::AIDBIT4>3.0.CO;2-I.
[26] 余培斌. 改善绍兴黄酒麦曲品质的初步研究[D]. 无锡: 江南大学, 2013: 37-43.
[27] 余培斌, 陈亮亮, 张波, 等. 双菌种制曲改善黄酒麦曲品质的研究[J].食品与发酵工业, 2012, 38(9): 1-6. DOI:10.13995/j.cnki.11-1802/ ts.2012.09.033.
Isolation and Identif i cation of Microorganisms in Wheat Qu of Beizong Rice Wine
REN Qing, HOU Chang
(Beijing Higher Institution Engineering Research Center of Food Additives and Ingredients, School of Food and Chemical Engineering, Beijing Technology and Business University, Beijing 100048, China)
In this research, we used the gradient dilution and streak plate methods to isolate and purify microorganisms from wheat Qu for Beizong rice wine. A total of seven bacterial strains and fi ve fungal strains were obtained. Through observation of their morphological characteristics and molecular biological characterization, the bacterial strains were identified as Bacillus licheniformis, Bacillus subtilis, Bacillus pumilus, Bacillus atrophaeus, Bacillus clausii, Bacillus rhizosphaerae, and Bacillus sonorensis, and the fungal strains were identif i ed as Aspergillus oryzae, Aspergillus niger, Talaromyces radicus, Neurospora crassa, and Absidia corymbifera.
Beizong rice wine; wheat Qu; microorganism; identif i cation; isolation
10.7506/spkx1002-6630-201704013
TS26;TS201.3
A
1002-6630(2017)04-0077-06
任清, 侯昌. 北宗黄酒麦曲微生物的分离鉴定[J]. 食品科学, 2017, 38(4): 77-82. DOI:10.7506/spkx1002-6630-201704013. http://www.spkx.net.cn
REN Qing, HOU Chang. Isolation and identif i cation of microorganisms in wheat Qu of Beizong rice wine[J]. Food Science, 2017, 38(4): 77-82. (in Chinese with English abstract)
10.7506/spkx1002-6630-201704013. http://www.spkx.net.cn
2016-04-28
公益性行业(农业)科研专项(201303069-07)
任清(1969—),男,副教授,博士,研究方向为食品生物转化和代谢过程中宏基因组学。E-mail:renqing@th.btbu.edu.cn
