利用糖芯片技术检测氨基糖苷类抗生素与RNAs和蛋白质之间的相互作用
2016-11-14王雪玉王晓丽张蓓蕾胡静尹健
王雪玉,王晓丽,张蓓蕾,胡静,尹健
利用糖芯片技术检测氨基糖苷类抗生素与RNAs和蛋白质之间的相互作用
王雪玉1,王晓丽1,张蓓蕾1,胡静2,尹健1
1 江南大学生物工程学院糖化学与生物技术教育部重点实验室,江苏无锡 214122;2 江南大学无锡医学院,江苏无锡 214122
氨基糖苷类抗生素是一类广谱型抗细菌感染药物,其不断增加的细菌耐药性很大程度上限制了它的临床应用,研究和开发新型氨基糖苷类抗生素具有重要意义。将氨基糖苷类抗生素固定到玻璃片基上,制成糖芯片,再分别与荧光标记的RNAs和蛋白质杂交,通过分析杂交后的荧光信号强度检测它们之间的相互作用。结果显示,氨基糖苷类抗生素芯片可以特异性地与rRNA的A位点模拟物、I型核酶和蛋白酶结合。因此糖芯片技术不仅可以检测氨基糖苷类抗生素与rRNAs的特异性结合,而且可以应用于寻找新型RNA结合配体的研究,为快速鉴定和筛选可紧密结合RNA靶标且毒性较低的新型氨基糖苷类抗生素奠定了一定的基础。
氨基糖苷类抗生素芯片,rRNA的A位点模拟物,I型核酶,Klenow DNA聚合酶,磷脂酶C
氨基糖苷类抗生素是一类由氨基糖与氨基环醇通过氧桥连接而成的糖苷类抗生素[1],主要包括来自链霉菌属 (如链霉素)、小单孢菌属 (如西索米星) 的天然氨基糖苷类抗生素,以及半合成氨基糖苷类抗生素 (如阿米卡星)。氨基糖苷类抗生素主要通过与细菌细胞30S核糖体亚单位的16S rRNA解码区的A部位结合来抑制细菌蛋白质的合成[2]。近年来,由于抗生素的滥用,对抗生素的研究也越来越重视。然而,传统的氨基糖苷类抗生素的筛选鉴定方法如质谱法、放射免疫检定法、表面激元共振法等[3-5]需要消耗大量原料。随着糖生物学和糖组学的发展,糖芯片技术正逐步发展成为该领域的新型研究手段。
糖芯片的发展始于21世纪初,通过不同方法将糖分子 (单糖、寡糖、聚糖、氨基糖苷类抗生素等) 固着于固体表面上形成的糖分子点 阵[6-8],可以用于检测三大类物质分子之间的相互作用。糖芯片作为一种生物芯片,目前主要具有以下3个优点:1) 高容量:它能在有限空间的芯片表面上展示大量的糖。2) 高度灵敏性:它所需要糖的点样量远远少于传统检测实验中需要的量,且敏感性远远高于传统分子和免疫实验。3) 长期稳定性:固定在芯片上的糖一般很稳定,无需特殊处理就能长期保存。能够利用少量的配体和分析物平行筛选大量样品的特点使这种技术具备广泛应用的潜力。近年来,生物体内三大类物质之间的相互作用已经可以利用糖芯片技术检测[9-11],但是小分子物质与RNA的结合在芯片技术中研究较少。RNA是药物研发的一个重要靶标,快速筛选一些新的RNA结合配体,可以作为RNA靶标的改进疗法或作为细胞内RNA功能的生化探针。最近,随着RNA生物学功能重要性的不断揭示,人们对RNA的研究兴趣越来越浓厚,研究热点主要有小分子RNA、干扰RNA和控制翻译的RNA等[12-14]。分析鉴定特定RNA的特异性配体可以帮助分析其在细胞内的作用,进而更完整地理解它在细胞进程中的贡献。此外,临床上常用的氨基糖苷类抗生素主要通过结合细菌核糖体RNA,抑制蛋白质合成,发挥其抗菌作用。然而,这些抗生素因为越来越强的细菌耐药性而失去抗菌活性。因此,快速筛选可以特异性与RNA紧密结合但与钝化酶 (一类可以与抗生素特异性结合,增强细菌抗药性的酶)[15]不结合或结合微弱以及毒副性降低的新化合物,将大大促进新型氨基糖苷类抗生素的发现。
在本文中,我们利用糖芯片技术研究氨基糖苷类抗生素与RNAs和蛋白质之间的相互作用。6种氨基糖苷类抗生素 (图1) 被固定到衍生化的片基上,通过氨基糖苷类抗生素芯片与荧光标记的RNA和蛋白质杂交,然后检测氨基糖苷类抗生素与RNAs和蛋白质之间的结合能力。本文中选用的RNAs包括细菌16S rRNA的A位点模拟物、人类18S rRNA的A位点模拟物以及Ⅰ型核酶。这里选用的两个rRNA的A部位模拟物是用来比较它们的治疗效果和副作用的差异性。选用的蛋白质是Klenow DNA聚合酶和磷脂酶C,这两种蛋白酶是检测氨基糖苷类抗生素毒性的模式蛋白[16-18]。通过利用糖芯片技术检测氨基糖苷类抗生素与RNAs和蛋白质之间的相互作用,有助于发现新的RNA治疗靶标,寻找可以特异性与RNA靶标紧密结合但与钝化酶[15]不结合或结合较弱且毒副性较低的新型RNA结合配体。

图1 用来构建氨基糖苷类抗生素芯片的6种氨基糖苷类抗生素
1 材料与方法
1.1 材料
6种氨基糖苷类抗生素以及牛血清蛋白从Sigma-Aldrich购买。四乙二醇琥珀酰基二琥珀酸 (TGDD) 按照原有合成路线合成[19],3-氨基丙基三乙氧基硅烷和,-二琥珀酰亚胺基碳酸酯从上海百灵威化学试剂有限公司购买。实验中所用原料均为商品试剂,未经进一步纯化。实验中所用的水溶液都经0.2 µm膜过滤后使用。
1.2 合成四乙二醇邻苯二甲酰基邻苯二甲酸 (TGPD)
将化合物三缩四乙二醇 (1 g, 5.15 mmol) 溶于二氯甲烷 (DCM, 60 mL) 中,在三乙胺存在的条件下,加入邻苯二甲酸酐 (3.8 g, 25.75 mmol),在0 ℃反应[20],TLC监测原料反应完全后,旋蒸除溶剂,再使用饱和NaHCO3(20 mL) 溶解混合油状物,溶解完全后,用1 mol/L HCl调pH至1,DCM萃取4次 (1×50 mL, 3×40 mL),得到两端带有羧基的中间产物。中间产物在0 ℃加入缩合剂DCC与-羟基琥珀酰亚胺反应[19],TLC监测原料反应完全后,放入–20 ℃冰箱使反应混合物沉淀1 h,抽滤,旋蒸除溶剂,再加入少量DCM溶解油状混合物,放入–20 ℃冰箱,待白色物质沉淀完全,抽滤,旋蒸除溶剂,粗品用硅胶柱层析纯化 (DCM∶MeOH=150∶1→80∶1),得到黄色油状物TGPD。总产率62%。R=0.50 (DCM/MeOH 15∶1)。
1.3 寡核苷酸的合成
两种rRNAs模拟物由宝生物公司合成和标记,为冻干粉状,用DEPC处理水溶解,浓度为100 μmol/L,于–20 ℃保存备用。白色念珠菌Ⅰ型核酶采用Riboprobe®体外转录合成,并在一定条件下复性[21]。
1.4 芯片的设计制作
Gold Seal®的普通玻片经过一系列处理,与3-氨基丙基三乙氧基硅烷反应,修饰成带氨基的片基,检验合格后,分别修饰成三种不同连接臂连接的片基。氨基覆盖的片基被放置在包含 10 mmol/L 的TGDD或TGPD和100 mmol/L,-二异丙胺 (DIPA) 的,-二甲基甲酰胺 (DMF) 溶液中反应过夜,即可得到A、B两种不同连接臂修饰的片基;而氨基覆盖的片基先用,-二琥珀酰亚胺基碳酸酯 (DSC) 处理,再与牛血清蛋白 (BSA) 反应,最后再用DSC处理得到片基C。修饰好的片基真空干燥保存备用。氨基糖苷类抗生素 (10 mmol/L, 25% DMF水溶液) 通过使用Arrayjet自动点样仪按照一定的顺序点样到片基上。点样后,片基置于湿度为70%的环境中,室温孵育过夜。未与片基结合的氨基糖苷类抗生素用水将其从片基上清洗下来。而片基上未结合氨基糖苷类抗生素的琥珀酰亚胺酯用封闭液 (含1% BSA、0.1%吐温20的PBS缓冲液) 室温封闭1 h。最后,片基用PBST和PBS清洗后,真空保存备用。
1.5 芯片与RNAs和蛋白质的杂交
1.5.1 氨基糖苷类抗生素芯片与rRNA杂交
将rRNA A位点模拟物置于含200 mmol/L NaCl和20 mmol/L Hepes的缓冲溶液 (pH 7.4) 中,在60 ℃条件下复性5 min后,自然冷却至室温[22-23]。取100 pmol的rRNA A位点模拟物与芯片杂交,用盖玻片使其在芯片上分散均匀,室温杂交1 h。将芯片置于SSC杂交溶液中清洗10 min将未结合的rRNA A位点模拟物清洗干净,离心干燥。杂交后的芯片用Gene Pix Pro 6芯片荧光扫描仪扫描,并用分子生物学软件分析数据。
1.5.2 氨基糖苷类抗生素芯片与I型核酶杂交
在白色念珠菌Ⅰ型核酶与抗生素芯片的杂交实验中,Ⅰ型核酶加入1×H10Mg缓冲液 (50 mmol/L Hepes, 135 mmol/L KCl, 10 mmol/L MgCl2, pH 7.5),置于55 ℃加热5 min复性,自然冷却至室温[21]。氨基糖苷类抗生素芯片与 10 pmol复性后的Ⅰ型核酶室温杂交1 h。杂交后的芯片置于SYBR green Ⅱ核酸染料 (Molecular probes) 染色约2 min[24]。然后用含0.1%吐温的TBE缓冲液清洗10 min,用DEPC处理水清洗3 min,离心甩干,扫描。
1.5.3 氨基糖苷类抗生素芯片与蛋白酶杂交
磷脂酶C和Klenow DNA聚合酶从Sigma- Aldrich购买。蛋白质都用Pacific BlueTM琥珀酰亚胺酯荧光染料 (Molecular probes) 标记;在 2 mg/mL的蛋白质溶液中加入20 μg Pacific BlueTM琥珀酰亚胺酯荧光探针 (溶解在无水DMF中),在0.1 mol/L NaHCO3缓冲液 (pH 8.8)中室温搅拌反应1 h后,再加入100 μL的1.5 mol/L盐酸羟胺pH 8.5,室温终止反应1 h。反应结束后,反应液经葡聚糖凝胶G 25柱纯化,用PBS缓冲液洗脱。标记过的蛋白质–20 ℃保存备用。
取荧光标记过的蛋白酶2 µL (约 2 ng) 加入杂交缓冲液中 (1% BSA, 100 mmol/L NaCl, 0.1% 吐温20和2 mmol/L盐酸羟胺) 与氨基糖苷类抗生素芯片室温杂交3 h,未结合的蛋白酶用PBST和PBS缓冲液冲洗10 min,离心干燥,扫描。
1.6 数据分析
实验中所有的芯片都用Gene Pix Pro 6芯片荧光扫描仪扫描并用Gene Pix Pro 6软件将荧光信号强度量化,其荧光信号强度由点的归一化处理决定。扫描参数为光强度:70%,PMT:700。每个数据都取自同一批芯片的3组平行的30个点 (3×10,荧光信号强度F中值-背景信号B) 的中值,每组10个点的选择标准为去掉杂交信号明显与其他平行杂交信号相差很大的点,再在其中随机选取。其标准差来自这随机选取的30个数据。并采用Origin Pro 9.0软件分析实验数据,并计算误差值。
2 结果与分析
2.1 衍生化片基的优化选择
6种氨基糖苷类抗生素被固定到3种不同的衍生化的片基上进行片基的优化选择。3种衍生化的片基包括:A) 用四乙二醇琥珀酰基二琥珀酸 (TGDD) 处理过的氨基修饰的片基;B) 用四乙二醇邻苯二甲酰基邻苯二甲酸 (TGPD) 处理过的氨基修饰的片基;C) 氨基修饰的片基经,-二琥珀酰亚胺基碳酸脂 (DSC) 处理后,与牛血清蛋白 (BSA) 反应之后,再用DSC处理的片基。对于每一个固定化过程,我们喷点约 4 nL的氨基糖苷类抗生素溶液 (10 mmol/L,25% DMF水溶液) 到衍生化的片基上,孵育后,用封闭液封闭片基。
之前已有文献报道固定后的氨基糖苷类抗生素仍保留与RNA的结合能力[25],我们将封闭后的片基与100 pmol荧光标记的细菌16S rRNA 的A位点的寡核苷酸模拟物 (图2[25]) 进行杂交,洗去未结合的寡核苷酸,然后进行荧光扫描。结果显示 (图3):氨基糖苷类抗生素与细菌16S rRNA的A位点的结合信号强度均高于每种衍生化的片基的背景信号。覆盖TGDD的片基的荧光信号强度最强;覆盖TGPD的片基的荧光信号强度较低;而经BSA处理过的片基荧光信号最低,背景信号较强。因此,我们选用最优的TGDD片基进行后续的实验。

图2 与氨基糖苷类抗生素芯片杂交的细菌和人rRNA的A位点模拟物(细菌rRNA用TAMRA标记,而人的rRNA用荧光素标记)

图3 三种片基的优化选择
2.2 氨基糖苷类抗生素芯片与rRNA的杂交
利用自动点样仪将6种氨基糖苷类抗生素喷点到TGDD衍生化的片基上,制成氨基糖苷类抗生素芯片,然后检测它们与两种寡核苷酸模拟物 (图2) 的结合能力。氨基糖苷类抗生素与细菌16S rRNA模拟物杂交后的荧光信号强度如图4所示。所有的氨基糖苷类抗生素都能与细菌寡核苷酸模拟物结合,其荧光强度的强弱与其结合能力高低相对应。结果表明,壮观霉素、链霉素、奈替米星和异帕米星与16S rRNA的结合能力均高于阿米卡星;而西索米星与16S rRNA的结合能力低于阿米卡星。氨基糖苷类抗生素与人类18S rRNA模拟物杂交后的荧光信号强度结果如图5所示。受试药物对应的荧光信号强度均明显低于与细菌16S rRNA杂交后的荧光信号强度,表明这些氨基糖苷类抗生素对人类的毒副作用远低于对细菌的抑制作用,符合治疗药物的指标。以上结果表明糖芯片技术可以作为一种氨基糖苷类抗生素的检测手段,快速检测其与不同rRNAs的相互作用。
新型药物需要具备的关键特性之一是其与靶点的结合特异性。目前,针对氨基糖苷类抗生素对某些RNAs的结合特异性的研究已有相关报道[26-28]。而糖芯片技术可以平行检测抗生素与不同RNA序列的结合,通过比较它们的荧光信号强度获得具备结合特异性的RNA序列。在本实验中,奈替米星与16S rRNA杂交的荧光信号强度比与18S rRNA杂交的荧光 信号强度至少高1个数量级;壮观霉素、链 霉素与16S rRNA杂交的荧光信号强度比与18S rRNA杂交的荧光信号强度大约强5倍。相反,西索米星与两种RNA模拟物的杂交具有相似的荧光信号强度。同时,不同的氨基糖苷类抗生素与同一寡核苷酸模拟物杂交的相对荧光信号强度也不同。例如:壮观霉素与16S rRNA杂交的荧光信号强度约是异帕米星、链霉素与16S rRNA杂交的2倍;另一方面,异帕米星与18S rRNA杂交的荧光信号强度约是壮观霉 素与18S rRNA杂交的1.2倍。而西索米星与两种RNA模拟物杂交的荧光信号强度相差不大。因此糖芯片技术不仅可以检测氨基糖苷类抗生素与rRNAs的相互作用,还能检测其结合的特异性。

图4 几种固定化氨基糖苷类抗生素与16S rRNA模拟物杂交(图为每种抗生素与16S rRNA模拟物杂交后的荧光信号强度)
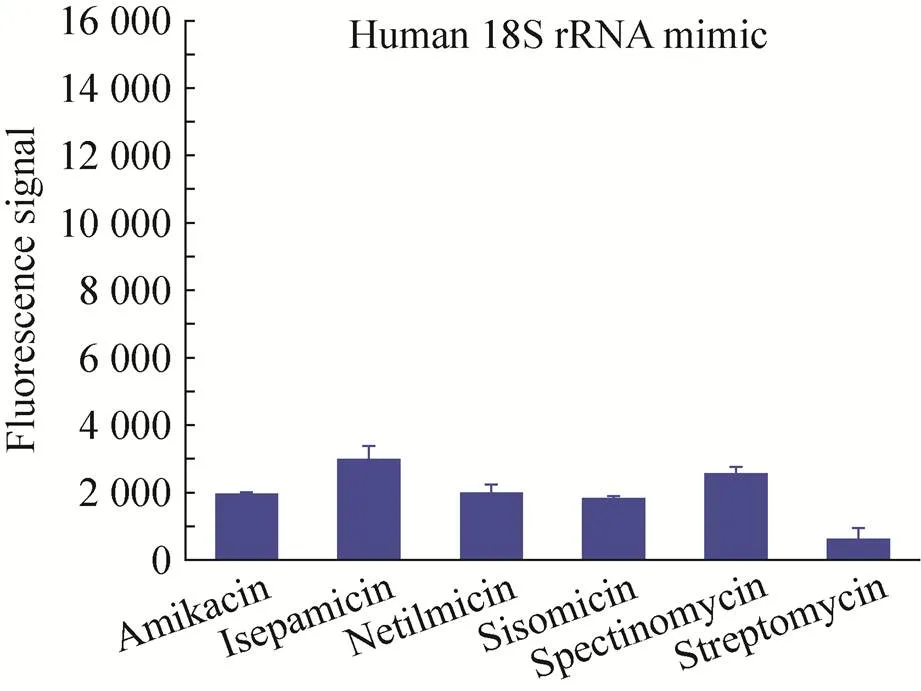
图5 几种固定化氨基糖苷类抗生素与18S rRNA模拟物杂交(图为每种抗生素与18S rRNA模拟物杂交后的荧光信号强度)
2.3 氨基糖苷类抗生素芯片与Ⅰ型核酶的杂交
由于氨基糖苷类抗生素与部分RNA的结合机制尚不明确,因此存在许多未开发的潜在RNA靶标,如人类免疫缺陷病毒 (HIV)、丙型肝炎病毒 (HCV) 以及核酶等[21,29-30]。在检测氨基糖苷类抗生素芯片与短链RNA寡核苷酸模拟物 (长链RNA的一小部分) 的相互作用后,我们尝试利用糖芯片技术直接进行长链RNA与氨基糖苷类抗生素的相互作用研究。本实验选择来源于白色念珠菌Ⅰ型内含子的自我剪切的Ⅰ型核酶,该RNA具有约400个核苷酸的长度,是一种潜在的药物治疗靶标[21],在与氨基糖苷类抗生素结合后能够抑制Ⅰ型内含子的自我剪切[31]。
首先,核酶在其折叠成活性构象的条件下复性 (> 90%)。然后,氨基糖苷类抗生素芯片与10 pmol的I型核酶孵化培养,洗去未结合的RNA,用含核酸染料SYBR GreenⅡ的溶液对糖芯片结合的RNA进行染色。结果如图6所示:壮观霉素的荧光信号强度最强,而阿米卡星的荧光信号强度最低。结果表明,利用糖芯片技术可以直接检测氨基糖苷类抗生素与大分子RNA之间的相互作用,这样可以减少许多现行筛选方法所致的潜在问题,例如使用短链RNA时可能出现的错误折叠。同时该结果再次表明糖芯片技术可以检测氨基糖苷类抗生素与RNA之间的特异性作用。因此,通过改变配体分子和RNA的链长,选用最优的RNA-配体相互作用[32]设计出与各类靶细胞RNA具有高亲和力、高选择性的配体是很有前景的。

图6 几种固定化氨基糖苷类抗生素与Ⅰ型核酶杂交
2.4 氨基糖苷类抗生素芯片与蛋白酶的杂交
氨基糖苷类抗生素可以认为是一种“金属模拟物”,通过与rRNA结合体现其治疗效果,然而,它们也可以因此与蛋白质和脂类物质 (如DNA聚合酶和磷脂酶C) 结合,引发毒副作用,如耳毒性和肾毒性,从而限制其临床应用[17-18, 33]。
我们利用Klenow DNA聚合酶作为模型蛋白[17],通过测试氨基糖苷类抗生素与蛋白质的金属离子结合位点的相互作用,从而检测它们的毒性。结果如图7所示,链霉素的荧光信号强度最强,至少比其他氨基糖苷类抗生素强3倍;其次是西索米星,而奈替米星和壮观霉素具有相似的荧光信号强度;阿米卡星的荧光信号最弱。但是这些结果与对酶的活性抑制不完全对应[31],原因可能是每种抗生素与Klenow DNA聚合酶的结合位点不同。
从蜡样芽孢杆菌分离的磷脂酶C是信号转导的关键酶,可以在不同层次上控制翻译后的修饰[34]。有研究表明,氨基糖苷类抗生素可以激活磷脂酶C的别构调节[18]。磷脂酶C与抗生素芯片的杂交结果显示:链霉素和西索米星的荧光信号强度最强,接下来依次是壮观霉素、阿米卡星、异帕米星,奈替米星的荧光信号强度最低。以上杂交结果显示,链霉素与两种蛋白酶都具有很强的结合,表明链霉素的毒副作用在这几种抗生素中最大,与链霉素本身固有的毒副作用相一致。
综上所述,利用糖芯片技术研究氨基糖苷类抗生素和蛋白质的相互作用,是研究氨基糖苷类抗生素毒副作用的一个简单而快速的方法。同时,氨基糖苷类抗生素作为“金属模拟物”,可以作为先导化合物,促进新型抗生素的研发。
3 结论
本文利用糖芯片检测氨基糖苷类抗生素与RNAs和蛋白质之间的相互作用,得到以下主要结论:首先,糖芯片作为一种检测手段,不仅可以检测氨基糖苷类抗生素与RNAs的相互作用,还能检测其结合的特异性;在应用方面,氨基糖苷类抗生素芯片可以直接与大分子RNA的相互作用,有利于新RNA治疗靶标的发现以及新型抗生素的研发;另外,通过分析氨基糖苷类抗生素芯片与蛋白酶金属结合位点的相互作用,可以促进抗生素的结构改造,提升其药效的同时降低其毒副作用,加速新型抗生素的发现。
REFERENCES:
[1] Kondo S, Hotta K. Semisynthetic aminoglycoside antibiotics: development and enzymatic modifications. J Infect Chemother, 1999, 5(1): 1−9.
[2] Magnet S, Blanchard JS. Molecular insights into aminoglycoside action and resistance. Chem Rev, 2005, 105(2): 477−498.
[3] Ding YL, Hofstadler SA, Swayze EE, et al. Design and synthesis of paromomycin-related heterocycle- substituted aminoglycoside mimetics based on a mass spectrometry RNA-binding assay. Angew Chem Int Ed, 2003, 42(29): 3409−3412.
[4] Shandrick S, Zhao Q, Han Q, et al. Monitoring molecular recognition of the ribosomal decoding site. Angew Chem Int Ed, 2004, 116(24): 3239−3244.
[5] Verhelst SHL, Michiels PJA, van der Marel GA, et al. Surface plasmon resonance evaluation of various aminoglycoside–RNA hairpin interactions reveals low degree of selectivity. ChemBioChem, 2004, 5(7): 937−942.
[6] Wang DN. Carbohydrate microarrays. Proteomics, 2003, 3(11): 2167−2175.
[7] Horlacher T, Seeberger PH. Carbohydrate arrays as tools for research and diagnostics. Chem Soc Rev, 2008, 37(7): 1414−1422.
[8] Park S, Gildersleeve JC, Blixt O, et al. Carbohydrate microarrays. Chem Soc Rev, 2013, 42(10): 4310−4326.
[9] Yang J, Moraillon A, Siriwardena A, et al. Carbohydrate microarray for the detection of glycan-protein interactions using metal-enhanced fluorescence. Anal Chem, 2015, 87(7): 3721−3728.
[10] Childs RA, Palma AS, Wharton S, et al. Receptor-binding specificity of pandemic influenza A (H1N1) 2009 virus determined by carbohydrate microarray. Nat Biotechnol, 2009, 27(9): 797−799.
[11] Rogers CJ, Clark PM, Tully SE, et al. Elucidating glycosaminoglycan-protein-protein interactions using carbohydrate microarray and computational approaches. Proc Natl Acad Sci USA, 2011, 108(24): 9747−9752.
[12] Mendell JT, Olson EN. MicroRNAs in stress signaling and human disease. Cell, 2012, 148(6): 1172−1187.
[13] Wong SC, Klein JJ, Hamilton HL, et al. Co-injection of a targeted, reversibly masked endosomolytic polymer dramatically improves the efficacy of cholesterol-conjugated small interfering RNAs. Nucleic Acid Ther, 2012, 22(6): 380−390.
[14] Piñeiro D, Martinez-Salas E. RNA structural elements of hepatitis C virus controlling viral RNA translation and the implications for viral pathogenesis. Viruses, 2012, 4(10): 2233−2250.
[15] Disney MD, Magnet S, Blanchard JS, et al. Aminoglycoside microarrays to study antibiotic resistance. Angew Chem Int Ed, 2004, 43(12): 1591−1594.
[16] Mingeot-Leclercq MP, Brasseur R, Schanck A. Molecular parameters involved in aminoglycoside nephrotoxicity. J Toxicol Environ Health, 1995, 44(3): 263−300.
[17] Ren YG, Martínez J, Kirsebom LA, et al. Inhibition of klenow DNA polymerase and poly(A)-specific ribonuclease by aminoglycosides. RNA, 2002, 8(11): 1393−1400.
[18] Morris JC, Lei PS, Zhai HX, et al. Phosphatidylinositol phospholipase C is activated allosterically by the aminoglycoside G418. J Biol Chem, 1996, 271(26): 15468−15477.
[19] Dal Pozzo A, Vanini L, Fagnoni M, et al. Preparation and characterization of poly(ethylene glycol)-crosslinked reacetylated chitosans. Carbohydr Polym, 2000, 42(2): 201−206.
[20] Muthusamy S, Gnanaprakasam B, Suresh E. Desymmetrization of cyclic anhydrides using dihydroxy compounds: selective synthesis of macrocyclic tetralactones. Org Lett, 2006, 8(9): 1913−1916.
[21] Disney MD, Haidaris CG, Turner DH. Recognition elements for 5′exon substrate binding to thegroup I intron. Biochemistry, 2001, 40(21): 6507−6519.
[22] Wong CH, Hendrix M, Priestley ES, et al. Specificity of aminoglycoside antibiotics for the A-site of the decoding region of ribosomal RNA. Chem Biol, 1998, 5(7): 397−406.
[23] Wong CH, Hendrix M, Manning DD, et al. A library approach to the discovery of small molecules that recognize RNA: use of a 1, 3-hydroxyamine motif as core. J Am Chem Soc, 1998, 120(33): 8319−8327.
[24] Battaglia C, Salani G, Consolandi C, et al. Analysis of DNA microarrays by non-destructive fluorescent staining using SYBR green II. Biotechniques, 2000, 29(1): 78−81.
[25] Disney MD, Seeberger PH. Aminoglycoside microarrays to explore interactions of antibiotics with RNAs and proteins. Chem A Eur J, 2004, 10(13): 3308−3314.
[26] Griffey RH, Hofstadler SA, Sannes-Lowery KA, et al. Determinants of aminoglycoside-binding specificity for rRNA by using mass spectrometry. Proc Natl Acad Sci USA, 1999, 96(18): 10129−10133.
[27] Ryu, DH, Rando RR. Aminoglycoside binding to human and bacterial A-site rRNA decoding region constructs. Bioorg Med Chem, 2001, 9(10): 2601−2608.
[28] Llano-Sotelo B, Azucena EF, Kotra LP, et al. Aminoglycosides modified by resistance enzymes display diminished binding to the bacterial ribosomal aminoacyl-tRNA site. Chem Biol, 2002, 9(4): 455−463.
[29] Mei HY, Mack DP, Galan AA, et al. Discovery of selective, small-molecule inhibitors of RNA complexes-1. The tat protein/TAR RNA complexes required for HIV-1 transcription. Bioorg Med Chem, 1997, 5(6): 1173−1184.
[30] Otto GA, Puglisi JD. The pathway of HCV IRES-mediated translation initiation. Cell, 2004, 119(3): 369−380.
[31] Michael K, Wang H, Tor Y. Enhanced RNA binding of dimerized aminoglycosides. Bioorg Med Chem, 1999, 7(7): 1361−1371.
[32] Velagapudi SP, Seedhouse SJ, Disney MD. Structure-activity relationships through sequencing (StARTS) defines optimal and suboptimal RNA motif targets for small molecules. Angew Chem Int Ed, 2010, 49(22): 3816−3818.
[33] Mikkelsen NE, Johansson K, Virtanen A, et al. Aminoglycoside binding displaces a divalent metal ion in a tRNA-neomycin B complex. Nat Struct Biol, 2001, 8(6): 510−514.
[34] Mingeot-Leclercq M-P, Tulkens PM. Aminoglycosides: nephrotoxicity. Antimicrob Agents Chemother, 1999, 43(5): 1003−1012.
(本文责编 郝丽芳)
Interactions of aminoglycosides with RNAs and proteins via carbohydrate microarray
Xueyu Wang1, Xiaoli Wang1Beilei Zhang1, Jing Hu2, and Jian Yin1
1,,,214122,,;2,,214122,,
Aminoglycosides are broad-spectrum antibacterials to treat bacterial infections, especially gram-negative bacteria infections. However, aminoglycosides are losing efficacy because of the increase in antibiotic resistance and their inherent toxicity, attracting more interests in developing new aminoglycosides. Several clinically used aminoglycosides are mainly exerted by inhibition of protein synthesis through binding to bacterial rRNA. The bacterial ribosome RNA is the most currently exploited RNA drug target. Identification of new compounds that target RNAs is indispensable to fight with the growing threat that bacteria pose to human safety. In this work, we used carbohydrate microarrays to probe interactions of low molecular weight ligands with RNAs and proteins. Carbohydrate microarrays, comprising hundreds to thousands of different glycan structures on surfaces in a spatially discrete pattern, are sensitive and versatile tools to study the interactions between biological macromolecules. Herein, aminoglycosides have been immobilized onto the modified glass microscope slides and their interactions with RNAs and proteins are then measured through the labeled fluorescence. The results displayed that microarray can be used to detect the binding of aminoglycosides with three types of target molecules, including the small RNA oligonucleotide mimics of aminoglycoside binding sites in the ribosome (rRNA A-site mimics), the large group I ribozyme RNA (approximately 400 nucleotide) and certain proteins (toxicity-causing enzymes, such as DNA polymerase and phospholipase C). For rRNA A-site mimics, the fluorescence intensities of 16S rRNA is stronger than that of 18S rRNA, illustrating that as a screen technique, the microarray method can not only determine the binding affinity to RNA but also detect the specific binding to bacterial rRNA mimic. The ability to screen group I ribozyme RNA can be helpful to the discovery of new RNA therapeutic targets. Binding of immobilized aminoglycosides to toxicity-causing proteins (DNA polymerase and phospholipase C) is a new method to study of aminoglycoside toxicity. These studies lay the foundation for rapid identification of new RNA-binding ligands with strong and specific binding affinity for their desired targets.
aminoglycoside microarrays, rRNA A-site mimics, group I ribozyme, klenow DNA polymerase, phospholipase C
February 22, 2016; Accepted: April 12, 2016
s:Jing Hu. E-mail: hujing@jiangnan.edu.cn Jian Yin. Tel/Fax: +86-510-85328229; E-mail: jianyin@jiangnan.edu.cn
Supported by:National Natural Science Foundation of China (No. 21502071), Natural Science Foundation of Jiangsu Province, China (Nos. BK20140154, BK20150140), the Public Health Research Center at Jiangnan University (No. JUPH201502).
国家自然科学基金(No. 21502071),江苏省自然科学基金(Nos. BK20140154, BK20150140),江南大学公共卫生研究中心项目(No. JUPH201502) 资助。
