猪血液免疫性状的复合基因组选择研究
2016-10-06张巧霞张玲妮刘向东赵书红朱猛进
张巧霞,刘 飞,张玲妮,刘向东,赵书红,2,朱猛进,2*
(1.华中农业大学农业动物遗传育种与繁殖教育部重点实验室,湖北 武汉 430070;2.生猪健康养殖协同创新中心,湖北 武汉 430070)
遗传改良
猪血液免疫性状的复合基因组选择研究
张巧霞1,刘 飞1,张玲妮1,刘向东1,赵书红1,2,朱猛进1,2*
(1.华中农业大学农业动物遗传育种与繁殖教育部重点实验室,湖北 武汉 430070;2.生猪健康养殖协同创新中心,湖北 武汉 430070)
该研究在提出“复合基因组选择(composite genomic selection)”概念的基础上,利用华中农业大学农业动物遗传育种与繁殖教育部重点实验室构建的免疫资源群体数据,通过交叉验证(cross-validation)策略,与标准GBLUP法对照,利用白细胞(WBC)、噬中性粒细胞(NE)等13项血液免疫性状对复合基因组选择的预测效果开展验证。研究结果表明,除血小板(PLT)等3个性状外,所有性状复合基因组选择的准确性均高于标准GBLUP法,分析结果支持复合基因组选择优于基于单一加性遗传组分的GBLUP的结论。同时,还探讨了不同交叉验证参数组合对复合基因组选择准确性的影响,发现最宜交叉验证倍数是性状特异性的,跟性状特性有关。总之,该研究提出了基于全部遗传组分的复合基因组选择法,并得到猪血液免疫性状数据分析结果的初步支持,特别是针对较小规模群体,复合基因组选择可能是提高基因组预测准确性的有效方法。
猪;免疫性状;复合基因组选择;交叉验证;准确性
中国是世界猪肉产量与消费量最大的国家,猪肉占居民肉类消费的63.6%。根据《2015中国猪业发展报告》,猪肉是产值最大的单项农产品,养猪业产值占农业总产值的13%。目前,我国养猪业正处于从传统养猪业向安全、高效的现代养猪业转型的关键发展时期,科技对养猪业发展的重要性愈发突出。根据美国农业部对近50年来各种科技因素的评估报告表明,遗传育种是养殖业科技贡献最大的因素,所以引入最新的育种技术,提升猪遗传育种的科技水平,对于我国养猪业的转型升级具有重要的推动作用。
按历史传承,家畜育种大致经历了表型选择、指数选择、BLUP(best linear unbiased prediction)[1]、分子标记辅助选择(marker-assisted selection,MAS)、 多 分 子 标 记 聚合育种以及基因组选择(genomic selection,GS)等发展阶段。基因组选择最早由Meuwissen等人提出[2],可理解为分子标记辅助选择的高通量化发展,即在全基因组范围内实施分子标记辅助选择的一种新的升级版育种方法。基因组选择代表了最新一代家畜育种技术,具有许多其他育种技术不具备的优点。目前,基因组选择已在奶牛育种中广泛应用,并开始逐渐应用于猪育种实践[3-4]。不过,与具有超大规模育种群的奶牛育种不同,猪育种主要是分散于各个育种公司,公司间种猪群的遗传交流不多,这使得猪实际育种的基础群规模通常要比奶牛育种群小很多。由于受育种基础群规模的限制,猪育种需要针对较小群体规模、准确性更高的基因组选择方法。
为提高较小规模群体基因组选择的准确性,我们提出复合基因组选择(composite genomic selection,CGS)概念。所谓复合基因组选择(亦称复合基因组预测),是指利用全部遗传组分信息开展基因组育种值预测。复合基因组选择同时利用基因组分子标记的加性、显性和上位效应,甚至包括转录组、蛋白组等其他组学信息,通过多种来源信息的整合,以此提升较小规模群体的基因组育种值估计的准确性。该研究以猪血液免疫性状为研究对象,利用交叉验证策略,通过与基于单一加性遗传组分的标准GBLUP法对照,实际验证同时利用加性、显性和上位效应的复合基因组选择的效果。
1 材料与方法
1.1 试验猪群
用于验证该研究的试验猪群来自我室构建的免疫资源群体[5]。该资源群体由杜洛克猪和二花脸猪构建,采用远交F2代设计,F0代由8头无血缘关系的杜洛克公猪和18头二花脸母猪组成,F1代13头公猪与38头母猪避免近亲(全同胞或半同胞)交配,总共获得394头个体的资源群体。
1.2 血液免疫性状
经前腔静脉采血1 mL,维生素K3和EDTA抗凝,采用日本光电MEK-8222K 22五分类流式激光法全自动血液分析仪测定各血液免疫性状,保留表型缺失低于10%的性状,最后纳入分析的性状包括白细胞(WBC)、嗜中性粒细胞(NE)、嗜中性粒细胞百分比(NE%)、淋巴细胞(LY)、淋巴细胞百分比(LY%)、单核细胞(MO)、单核细胞百分比(MO%)、嗜酸性粒细胞(EO)、红细胞(RBC)、血红蛋白(HGB)、平均红细胞体积(MCV)、血小板(PLT)、红细胞分布宽度(RDW)等13项血液免疫指标。
1.3 复合基因组选择模型
该研究所用猪群基因型数据由Illumina公司的猪SNP芯片数据(Illumina PorcineSNP60 Genotyping Beadchip)测定,该芯片总共包括62 163个SNP位点。试验猪群基因组SNP检测由商业公司完成,获得原始数据后,开展检出率或杂交阳性率(call rate)、最小等位基因频率(MAF)、哈迪-温伯格平衡定律(HWE)等常规质控分析,并对缺失基因型进行填充(imputation)。基因型数据的数字化转换及缺失数据的填充用R程序包synbreed完成。复合基因组选择模型组分包括加性关系矩阵(A)、显性关系矩阵(D)、上位关系矩阵(AA、DD和AD)。复合基因组选择由多组分GBLUP完成,所用混合线性模型如下:
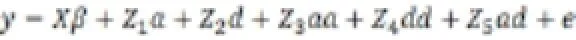
其中,y为性状表型值向量,X为固定效应设计矩阵,Z1、Z2、Z3、Z4、Z5分别为加性遗传组分、显性遗传组分、加性×加性互作组分、显性×显性互作组分、加性×显性互作组分的设计矩阵,β为固定效应向量,a为加性效应向量、d为显性效应向量、aa为加性×加性互作效应向量,dd为显性×显性互作效应向量,ad为加性×显性互作效应向量,e为残差向量。
加性关系矩阵(A)、显性关系矩阵(D)、上位关系矩阵(AA、DD和AD)的构建及复合基因组选择的计算全部由R完成,其中基因组育种值预测采用“再生核希尔伯特空间”(Reproducing Kernel Hilbert Space,RKHS)算法。考虑到纳入全部遗传关系矩阵后的计算量,利用3组参数组合开展复合基因组预测:1)10-倍交叉验证,重复5次;2)5-倍交叉验证,重复10次;3)20-倍交叉验证,重复5次。其中,为简化计算,该研究的基因组预测准确性由预测值和真实表型值之间的相关系数值揭示。
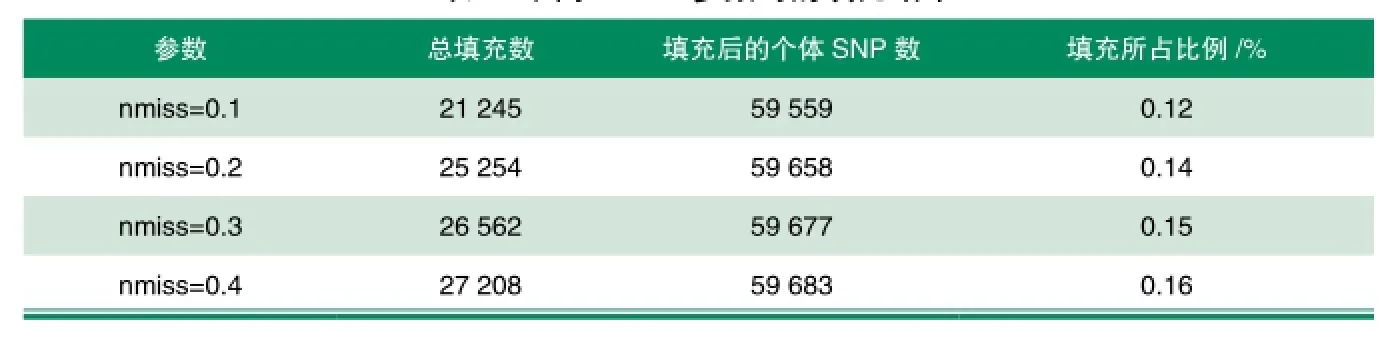
表1 不同nmiss参数时的填充结果
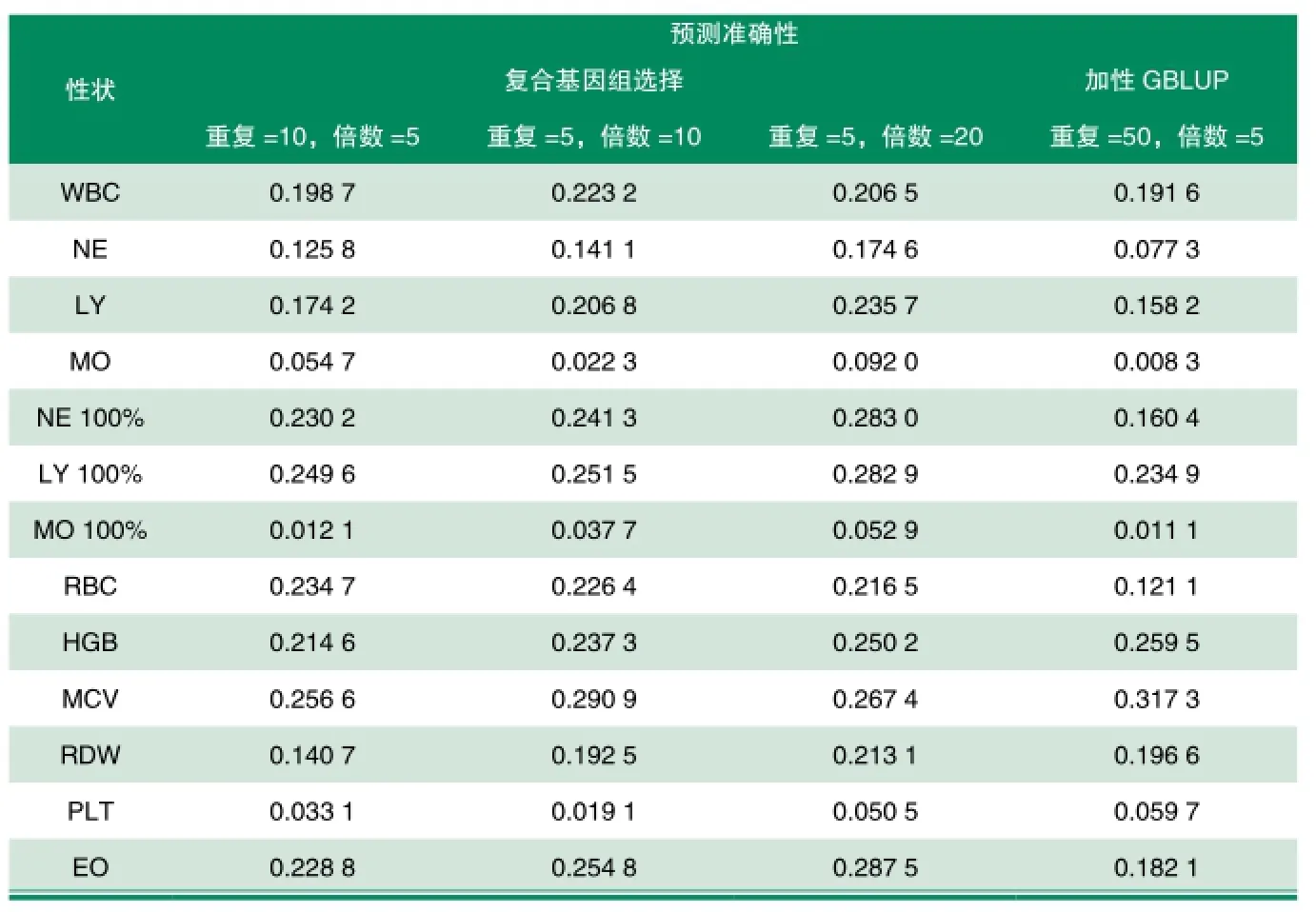
表2 各性状复合基因组预测结果
2 结果
2.1 基因型填充结果
基因组分型数据,我们使用R程序包synbreed的codeGeno命令将SNP芯片数据文件中缺失的SNP标记位点NA值进行填充,并根据需要转换数据形式,将字母基因型转换成数字基因型,统一格式后得到复合基因组选择的输入数据。表1为不同参数(nmiss)条件下基因组SNP数据的填充结果。从表1可以看出,随着缺失率增大,填充位点所占比例也跟着增加,在理论上这会使后续数据分析的准确性亦相应降低。所以,该研究选取nmiss=0.1时的填充结果用于复合基因组选择分析。
2.2 复合基因组选择结果
基于基因组遗传关系矩阵的GBLUP已被证明优于基于分子血缘矩阵的常规BLUP,GBLUP是基因组选择中最为广泛使用的方法之一。同时,在发展基因组选择的方法学研究中,GBLUP也为新方法提供了最为常用的参照。该研究用标准GBLUP作为对照。表2给出基于加性遗传关系矩阵的标准GBLUP预测和同时利用加性、显性、加性×加性、显性×显性、加性×显性遗传关系矩阵的复合基因组预测结果。除血小板(PLT)等3个性状外,其余所有性状的复合基因组预测的准确性均高于标准GBLUP预测法。研究结果表明,基于全部遗传组分的复合基因组预测的准确性,确实较基于加性遗传组分的预测准确性有明显提高。综合结果来看,无论哪种预测方法,单核细胞(MO)、单核细胞百分比(MO%)的预测准确性均为最低,这可能与MO、MO%的遗传力较低有关。此外,从表2的结果来看,在复合基因组预测的交叉验证参数上,交叉验证倍数对预测准确性有明显的影响,在一定程度上呈现出预测准确性随交叉验证倍数增加而提高的趋势。但这一趋势在不同性状上的表现不尽一致,如白细胞(WBC)、平均红细胞体积(MCV)的预测准确性最高的交叉验证倍数为10,交叉验证倍数过低和过高,其预测准确性均有所降低,而血小板(PLT)的表现趋势刚好与WBC和MCV性状相反。另外,红细胞(RBC)性状较为特殊,基因组选择结果表现出随着交叉验证倍数的提高,预测准确性逐渐下降的趋势。
3 讨论
该研究针对猪育种基础群规模通常有限的特点,提出了预测准确性更高的复合基因组选择方法。用猪血液免疫性状的实际分析发现,复合基因组预测的准确性高于基于单遗传组分的标准GBLUP法。该研究同时发现,复合基因组预测交叉验证参数的选择,可以影响性状的预测准确性。一般来说,重复数越大,分析结果越可靠。对于交叉验证倍数,我们发现没有绝对的标准,不同性状的最适宜交叉验证倍数并不完全一致,这跟性状的遗传结构有关。对于重复数和交叉验证倍数的确定,一方面要考虑计算成本,另一方面要考虑性状的遗传特性。在实际应用中,应综合计算机性能和性状的具体特性选择最适宜的验证参数。
另外,由于复合基因组预测涉及A、D、AA、DD和AD等全部遗传组分,模型设计矩阵的维数十分庞大,这给复合基因组的计算带来了巨大挑战。如果用贝叶斯或回归类方法,面对数量庞大的设计矩阵,在计算上很难实现。所以,当考虑纳入全部遗传组分的复合基因组预测时,基于混合线性模型的GBLUP是首选,这是因为GBLUP是利用全基因组标记构建的遗传关系矩阵,遗传关系矩阵的维数与样本含量直接相关,而与标记数目无关。假设不考虑二阶或高阶上位互作,只同时考虑A、D、AA、DD和AD遗传组分,无论基因组标记数目如何,实际纳入分析模型的遗传关系矩阵只有五个矩阵,GBLUP混合线性模型的计算量不会像贝叶斯或回归类方法那样随着分子标记数目增加而增加。所以,面对数量庞大的基因组分子标记,在可操作性上GBLUP混合线性模型是实现复合基因组预测的可行方法。
在理论上,复合基因组预测利用了全部遗传组分信息,其准确性较只利用单一遗传组分的预测准确性高。该研究实际结果也证明复合基因组预测的准确性确实高于传统的GBLUP预测法,利用复合基因组预测可以明显提高基因组选择的准确性。不过,复合基因组预测由于涉及加性遗传组分、显性遗传组分、加性×加性互作组分、显性×显性互作组分、加性×显性互作组分,属于多重遗传组分模型。多重遗传组分模型的计算复杂度明显高于基于加性遗传关系矩阵的单遗传组分GBLUP模型,当样本含量很大时,如奶牛基因组遗传评估[3],其计算负担偏大,可操作性仍不强。所以,该研究提出的复合基因组预测方法,主要适用于较小群体规模的基因组选择。
虽然该研究的实际分析结果支持复合基因组预测法优于标准GBLUP法,但该研究所使用的数据样本含量偏小,性状只限于血液免疫指标。该结论是否具有绝对的普遍性,仍然需要更多实际数据分析结果的支持。除了免疫性状以外,猪其他经济性状、以及其他遗传背景的猪群是否也具有相同的表现,仍需要进一步验证。综上所述,该研究提出了复合基因组选择(复合基因组预测)的概念,并用实际数据为复合基因组预测优于标准GBLUP法的结论提供了初步证据。
[1] HENDERSON C R.Best linear unbiased estimation and prediction under a selection model[J]. Biometrics,1975:423-447.
[2] M E U W I S S E N T,H A Y E S B J,GODDARD M E.Prediction of total genetic value using genomewide dense marker maps[J]. Genetics,2001,157(4):1819-1829.
[3] S C H A E F F E R L R.S t r a t e g y for applying genome wide selection in dairy cattle[J]. Journal of Animal Breeding and Genetics,2006,123(4):218-223.
[4] 李娅兰,梅盈洁,刘敬顺,等.基因组选择及其在猪育种中的应用[J].广东农业科学,2012,39(17): 106-109.
[5] 向安静.杜洛克×二花脸F2资源群体构建及其血液指标,免疫性状的测定和分析[D].武汉:华中农业大学,2010.
2016-08-03)
本研究受国家自然科学基金(31372302、31361140365)和湖北省公益性科技研究项目(2012DBA25001)资助
张巧霞,女,华中农业大学在读硕士
朱猛进,男(1974-),博士,副教授,主要致力于猪遗传改良的基因素材挖掘、基因组选择和GWAS等统计基因组学方法发展等,E-mail:zhumengjin@mail.hzau.edu.cn
