水稻端粒酶RNA候选序列的鉴定
2016-09-15朱莲莲林高强叶聪莹刘小川
朱莲莲,林高强,叶聪莹,黄 倩,刘小川
(浙江理工大学生命科学学院生物工程研究所,杭州 310018)
水稻端粒酶RNA候选序列的鉴定
朱莲莲,林高强,叶聪莹,黄倩,刘小川
(浙江理工大学生命科学学院生物工程研究所,杭州 310018)
端粒酶由端粒酶反转录蛋白和端粒酶RNA构成,端粒酶RNA中除模板序列保守外,其余区域的保守度极低,难以基于同源序列实施克隆。目前,植物中仅拟南芥端粒酶RNA得到了克隆。为了鉴定一条具水稻端粒酶RNA特征的候选序列,研究中进行了对模板序列单碱基T/A突变(5′-AAACCCTAA-3′)后的转基因愈伤组织端粒酶活性的比较,结果显示在缺乏dATP的端粒重复序列扩增法反应中,突变型的端粒酶活性明显高于未突变型;对体外延伸得到的端粒ssDNA进行测序分析,结果发现所测10条片段均含有突变特征序列;再对转基因植株进行端粒序列的检测,结果所测8个端粒序列中,有2个含有突变特征序列。因此,初步判断该候选序列为水稻端粒酶RNA基因。
水稻;端粒酶;端粒酶RNA;TRAP;定点突变
0 引 言
真核生物端粒酶是一种依赖于RNA的DNA聚合酶,是保证染色体末端结构稳定,维持细胞正常分裂的重要生物酶。它由端粒酶逆转录酶(Telomerase reverse transcriptase,TERT)和RNA亚基(Telomerase RNA,TER)组成,不管是体内还是体外环境[1-3],对于端粒酶活性来说TERT和TER都是必不可少的核心组件,其中TER中含有决定端粒重复序列的一段短模板序列[4]。端粒酶以染色体3′末端为前导引物,以其自身的TER为模板合成串联重复序列(TTTAGGG)n,TER模板区直接决定了延伸的端粒末端序列。线虫、酵母和多种脊椎动物的端粒酶TER已经得到了克隆,而在植物中仅报道了拟南芥端粒酶含有两个TER形式,分别为TER1和TER2,两者的模板序列均为5′-CTAAACCCTA-3′,其中TER1被认为是端粒重复序列的主要模板提供者[5]。分析显示TER中除了模板区域高度保守外,其长度及序列在这些物种之间都不具相似性[6]。
目前对水稻端粒酶序列的研究已有初步进展,马登旭通过水稻愈伤组织的端粒酶提取总RNA,接头连接后反转录得到cDNA,再设计多条特异性引物进行克隆,经过分析得到了一条具有水稻TER特征的候选序列[7];杨力媛等[8]建立了一套切实可行的水稻端粒酶RNA模板基因序列的鉴定体系。
通过对拟南芥,水稻,大麦,玉米等植物的研究发现,端粒酶主要分布在具有活跃细胞分裂和分化的分生组织中[9-11],而在已分化的组织中几乎检测不到端粒酶活性。植物细胞具有全能性[12],高度分化的成熟细胞可通过脱分化恢复分化能力,进而通过再分化成长为一个新植株,说明脱分化能重新激活端粒酶基因的表达。对烟草的研究发现,烟草叶片脱分化形成的愈伤组织中端粒酶活性比其外植体叶片有明显提高,而再分化后形成的再生植株叶片的端粒酶活性又下降到了原始植株水平[13],说明植物愈伤组织中有较高的端粒酶表达。本研究将正常的及模板区定点突变了的TER转导入水稻愈伤组织,以此为实验材料进行端粒酶活性比较等相关实验。
端粒酶活性的检测有多种方法,这些方法能应用于较高端粒酶活性的动物组织的检测,而植物组织,即便是分生组织或愈伤组织的酶活性都十分有限,目前还没有完全适用于植物端粒酶活性的高灵敏度检测技术。据报道,端粒酶重复序列扩增法(telomeric repeat amplification protocol,TRAP)相对灵敏度较高,其原理是首先合成一段长度约为18 nt的单链寡聚核苷酸作为前导引物,端粒酶结合前导引物末端的GTT并合成端粒重复序列,前导引物被延伸成为含有多个端粒重复序列的DNA单链,同时结合PCR扩增技术可提高检测端粒酶活性的灵敏度[14]。其中引物的设计也是关键的一步,故在本研究实验中采用TRAP法,通过设计特异的引物来进一步提高检测的灵敏度。
本研究将对马登旭发现的这条具有水稻TER特征的候选序列进行验证,实验采用杨力媛建立的鉴定体系,通过改变候选序列的模板区即可导致合成的端粒末端重复序列的变化,从而实现对候选序列的鉴定,为之后进一步研究端粒酶TER的结构和功能奠定基础。
1 材料与方法
1.1实验材料与仪器
主要实验材料有中花11号水稻(OryzasativaL.)种子、E.coliDH5α、pCambia1301、农杆菌LBA4404。
主要仪器有高压蒸汽灭菌锅、超净工作台、植物生长培养箱、Bio-Rad电泳仪、SynGene凝胶成像系统、NANODROP 2000分光光度计、Bio-Rad电击转化仪。
1.2方法
1.2.1水稻愈伤组织的诱导
利用水稻中花11的成熟胚为材料诱导愈伤组织[8-15]。
1.2.2端粒酶TER候选序列的定点突变及穿梭载体的构建
在已经得到的一条TER候选序列基因的基础上,比对水稻的全基因组,在目标序列区向上游延伸700 bp,向下游延伸500 bp,按照此序列设计一对扩增全长目的序列的引物24RNAPS1和24RNAPA1,再针对模板区设计一对定点突变引物22RNAPS2和27RNAPA2(表1)。采用三重PCR方法,将候选序列模板区的5′-AAACCCTAA-3′(Y)突变为5′-AAACCCAAA-3′(T),从而获得突变型序列。然后通过构建pCambia1301-T/Y穿梭质粒的方法[8], 将两组基因导入农杆菌LBA4404细胞,于—80℃冻存备用。
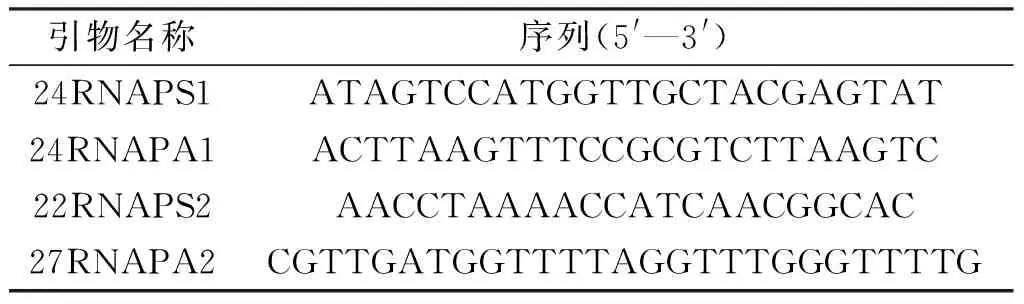
表1 获得端粒酶RNA候选序列的引物
1.2.3水稻转基因植株的培养
用分别含有穿梭载体pCambl1301-T/Y的农杆菌LBA4404侵染继代培养4次的水稻愈伤组织,并对侵染后的愈伤组织进行两次抗性筛选[8]。
分别挑取状态良好的未突变型(Contorl,C)和突变型(Mutant,M)愈伤组织5~6颗,移入装有分化培养基的培养皿中,25℃,14 h光照和10 h黑暗条件下培养分化成苗,20 d继代1次,待苗长至1 cm左右,移入装有生根培养基的组培瓶中壮苗。分别留下一部分愈伤组织用于基因组和端粒酶的提取。
待组培瓶中的转基因苗长至顶部时,及时开盖,并加入适量无菌蒸馏水,炼苗3 d,期间每天换水。然后洗去苗根部的琼脂,移栽到温室的土钵中。7 d后将苗移至室外实验田中。
1.2.4利用转基因愈伤组织鉴定水稻端粒酶TER候选序列
1.2.4.1转基因愈伤组织的鉴定
分别称取新鲜的未突变型和突变型愈伤组织各100 mg,用试剂盒(TaKaRaMiniBEST Universal Genomic DNA Extraction Kit Ver.5.0)提取全基因组。以基因组为PCR模板,根据载体pCambl1301内含的潮霉素基因序列以及插入水稻基因组的T-DNA序列设计上下游引物TUP和TDW(表2),进行PCR验证,并测序鉴定。

表2 鉴定抗性愈伤组织的引物
1.2.4.2端粒酶活性及酶促产物的检测
参照Fitzgerald MS等报道的方法提取水稻成熟胚愈伤组织的端粒酶[16]。对未突变型和突变型愈伤组织的端粒酶蛋白进行分组实验[17],分别为C组、M组及相应的对照组C-灭活组、M-灭活组,灭活组在酶促反应前加0.5μL 10 mg/mL RNaseA,冰上放置5 min,将端粒酶进行灭活。采用Kim建立的基于PCR基础上的TRAP法[18],结合陈波等[19]设计的前导引物CHEN03(表3)和不含dATP和dCTP的dNTPs,同时进行体外酶促反应。C组、M组在酶促反应后加0.5μL 10 mg/mL RNaseA,冰上放置5 min,将端粒酶进行灭活。酶促反应产物用氯仿抽提,醋酸钠、乙醇沉淀纯化的方法回收后,使用NANODROP 2000 spectrophotometer进行ssDNA的含量测定,并对各组进行比较分析。
通过对酶促产物ssDNA序列排列方式的推测,设计下游引物T3T-1(表3),以M组的酶促产物为模板,CHEN03和T3T-1为引物进行TD-PCR扩增。PCR产物用试剂盒进行纯化后装载与pMD18-T vector并转化大肠杆菌细胞,挑取单菌落后进行菌液PCR鉴定阳性克隆,引物为pMD18-T vector测序用的上下游引物M13-20和M13-48(表3)。并选择合适的克隆进行测序分析,以进一步确定TER候选序列的正确性。
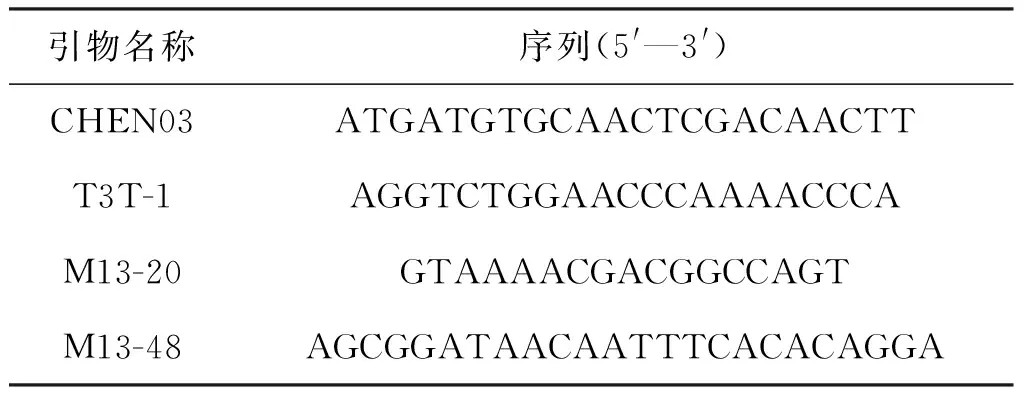
表3 酶促反应及克隆鉴定的引物序列
1.2.5转基因植株端粒的检测
1.2.5.1转基因植株的鉴定
分别称取鲜嫩叶片100 mg,用试剂盒提取5个样本的全基因组。按照转基因愈伤组织的PCR鉴定方法,使用相同的引物和程序对5个样本进行PCR扩增,使用突变型愈伤组织基因组作为对照,进行转基因植株的鉴定。
1.2.5.2染色体末端的检测
随机选取第2号染色体短臂设计上游引物02pF,下游引物为端粒重复序列P-R(表4)。分别以五个植株的基因组DNA为模板进行PCR反应。取一部分PCR产物进行回收,将回收后的产物装载到pMD18-T vector中用于测序检测,以确定样品端粒的组成。
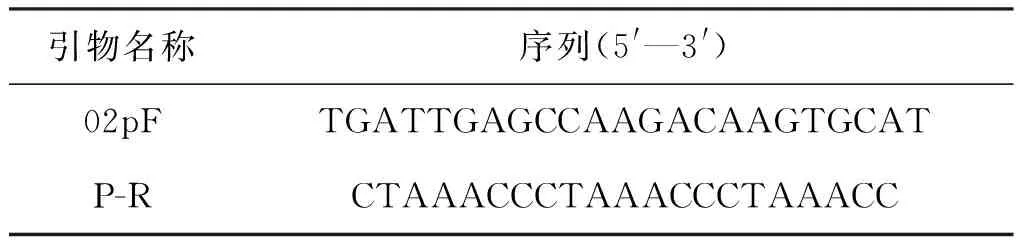
表4 扩增端粒的引物序列
2 实验结果
2.1水稻转基因植株的获取
将未突变型和突变型的抗性愈伤组织进行诱导分化,发现在相同的培养条件下,突变组出绿点率高于未突变组。取长有绿色颗粒状组织的愈伤组织进行继代培养后,突变组内近一半组织褐变死亡,但有两块愈伤组织上分化出苗(图1(a));未突变组大部分褐变死亡,未能分化成苗。重复诱导分化实验,出现相似的结果。待突变型转基因苗长至1cm左右时,将其转接到含有生根培养基的组培瓶中进行壮苗(图1(b)),再移栽入土栽培为转基因植株(图1(c))。

图1 转基因植株诱导培养的不同阶段
2.2转基因愈伤组织及植株的鉴定
分别提取未突变型和突变型两种愈伤组织,以及五个突变型转基因植株的基因组后,经PCR鉴定目标片段大小均在2500 bp左右,符合预期(图2(a)-(b)),结合测序结果证明转基因成功。

图2 转基因愈伤组织及植株的PCR鉴定结果注:M.Marker;c.未突变型愈伤组织基因组; m.突变型愈伤组织基因组;1~5. 转基因植株基因组。
2.3利用转基因愈伤组织鉴定水稻端粒酶TER候选序列
2.3.1端粒酶活性的比较
基于TRAP方法,在缺乏dATP和dCTP的体外酶促延伸反应中,未突变型端粒酶将受到抑制或停止反应,而突变型端粒酶则能顺利延伸前导引物,增加ssDNA含量。结果显示用100 μg未突变型和突变型的端粒酶经体外延伸后获得的ssDNA量(表5),未突变型的差值应为加入的前导引物,其与突变型组的差值为突变型端粒酶作用的结果,说明突变型愈伤组织的端粒酶,在缺乏dATP和dCTP的条件下能有效延伸前导引物,但相对于动物组织的端粒酶,它的酶活性仍不强。
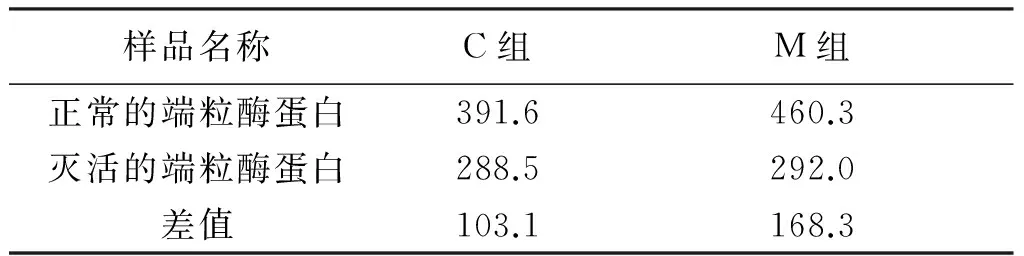
表5 未突变型(C)和突变型(M)端粒酶的
2.3.2酶促产物的检测
为解析延伸的ssDNA,对M组的酶促产物进行回收,并通过T-vector进行克隆。从获得的23个克隆(图3)中,选取了10个含所有不同分子量的克隆进行了测序分析,结果显示:10个克隆中都含有或多或少的GGGTTTT重复,与突变型端粒酶RNA模板序列5′-AAACCCAAA-3′相符(表6)。

图3 M组酶促产物的T克隆PCR鉴定结果 注:M.Marker;1~23.23个不同的克隆。

名称序列(5'—3')No1…ATGATGTGCAACTCGACAACTT(G)GGGTTTTGGGGTTTGGGTTTT…-3'No3…ATGATGTGCAACTCGACAACTT(G)GGGTTTTGGGTTTTGGGTTTT…-3'No5…ATGATGTGCAACTCGACAACTT(G)GGGTTTTGGGTTTT…-3'No6…ATGATGTGCAACTCGACAACTTGGGGTTT(G)GGGTTTTGGGGTCTTGGGTTTT…-3'No7…ATGATGTGCAACTCGACAACTTGGGTTTTGGGTTTT…-3'No10…ATGATGTGCAACTCGACAACTTT(G)GGGTTTT(G)GGGTTTTGGGTTTT…-3'No14…ATGATGTGCAACTCGACAACTT(GG)GGGTTTTGGGTTTT…-3'No17…ATGATGTGCAACTCGACAACTTGGGGTTTGGGGTTCTGGGTTTT…-3'No19…ATGATGTGCAACTCGACAACTTGGGTTTTGGGTTTT…-3'No23…ATGATGTGCAACTCGACAACTT(G)GGGTTTT(G)GGGTTTTGGGTTTT…-3'
2.4转基因植株端粒的检测
2.4.1单个端粒序列的扩增
以02pF和P-R作为上下游引物对获得的五株转基因水稻染色体端粒进行扩增并分析(图4)。每个泳道均出现了弥散条带,与预期情况相符。
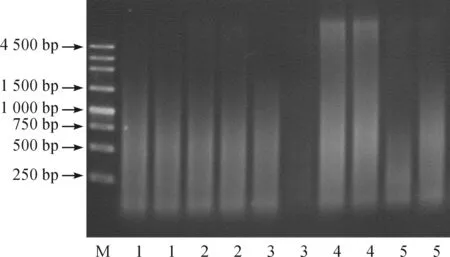
图4 单个端粒重复序列的PCR扩增结果 注:M.Marker;1~5.5个转基因水稻基因组。
2.4.2染色体端粒序列的检测
为解析转基因植株染色体末端的组成,对PCR扩增后的端粒重复序列进行回收,并通过T-vector进行克隆。结果获得了40个克隆(图5),选取8个含不同插入片段的克隆进行测序分析。显示8个克隆中均含有(AGGGTTT)n,与野生型端粒酶RNA模板序列5′-AAACCCTAA-3′相吻合;其中,2个克隆出现5次GGGTTTT序列,与突变型端粒酶RNA模板序列5′-AAACCCAAA-3′一致(表7)。
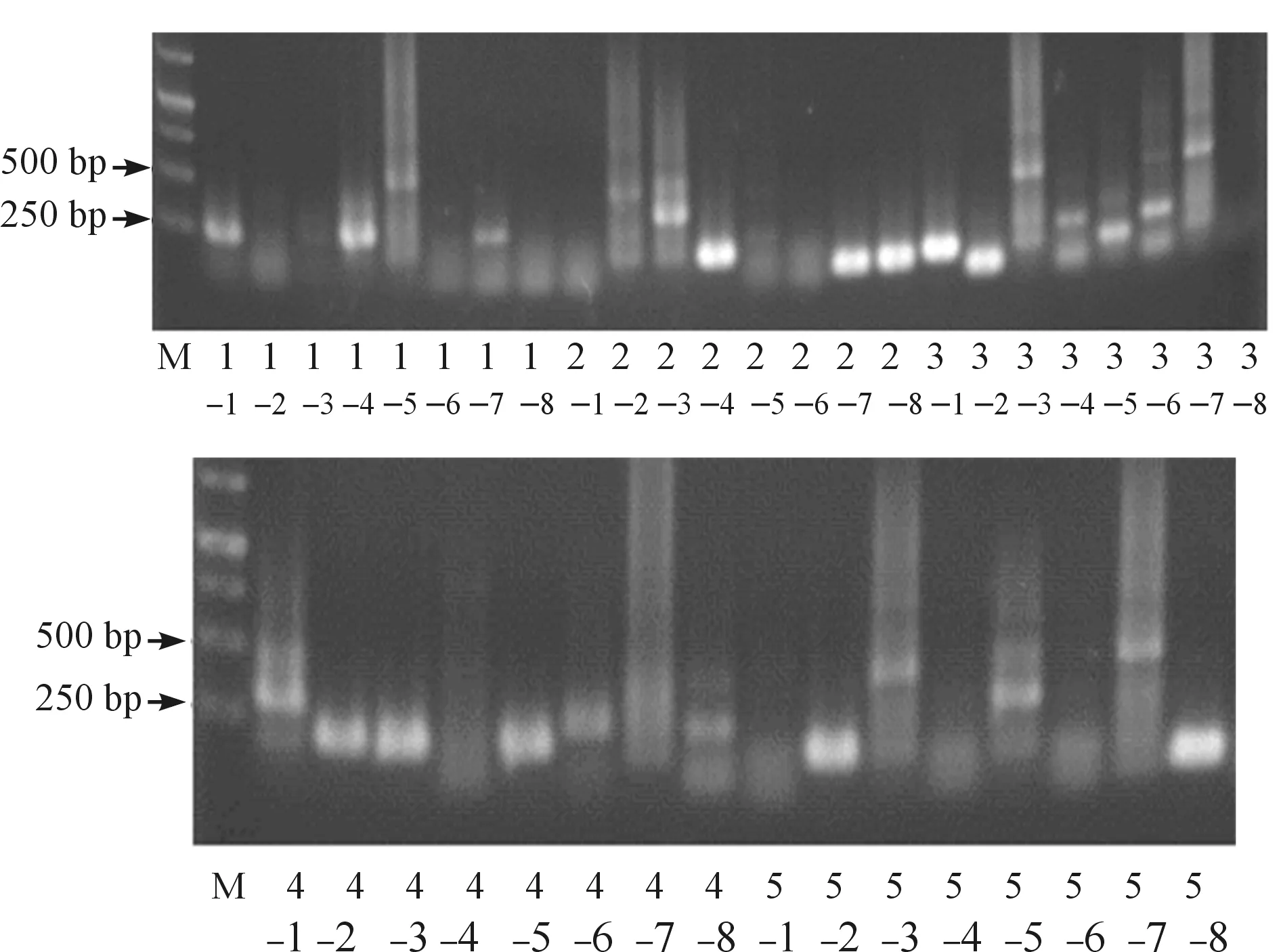
图5 染色体端粒序列T克隆的PCR鉴定结果注:M.Marker;1-1~5-8.以02pF和M13-48为引物获得的40个克隆。

名称序列(5'—3')No1-5…TGATTGAGCCAAGACAAGTGCATTGCTTCATTAAGGTTGGGGTT(AGGGTTT)25AGGGTTTT(AGGGTTT)8AGGGTT(AGGGTTT)3AGAGTTT(AGGGTTT)3AGGGTTTT(AGGGTTT)6AGGGTTTT(AGGGTTT)2-3'No2-2…TGATTGAGCCAAGACAAGTGCATTGCTTCGTTAAGGTTGGGGTT(AGGGTTT)5AGGGTT(AGGGTTT)20AGGGTT(AGGGTTT)3AGTGTTT(AGGGTTT)9-3'No2-3…TGATTGAGCCAAGACAAGTGCATTGCTTCATTAAGGTTGGGGTT(AGGGTTT)4AGGGTT(AGGGTTT)9AGGGTT(AGGGTTT)4AGTGTTT(AGGGTTT)9-3'No3-1…TGATTGAGCCAAGACAAGTGCATTGCTTCATTAAGGTTGGGGTT(AGGGTTT)2-3'No3-7…TGATTGAGCCAAGACAAGTGCATTGCTTCATTAAGGTTGGGGTT(AGGGTTT)3AGGGCTTAGGGTT(AGGGTTT)9AGGGTT(AGGGTTT)11-3'No4-1…TGATTGAGCCAAGACAAGTGCATTGCTTCATTAAGGTTGGGGTT(AGGGTTT)4AGGGTT(AGGGTTT)9AGGGTT(AGGGTTT)6-3'No5-3…TGATTGAGCCAAGACAAGTGCATTGCTTCATTAAGGTTGGGGTT(AGGGTTT)4AGGGTT(AGGGTTT)17AGGGTT(AGGGTTT)13-3'No5-7…TGATTGAGCCAAGACAAGTGCATTGCTTCATTAAGGTTGGGGTT(AGGGTTT)4AGGGTT(AGGGTTT)9AGGGTT(AGGGTTT)7AGGGTTTT(AGGGTTT)3AGGGTT(AGGGTTT)4GGGGTTT(AGGGTTT)2AGGGTTTT(AGGGTTT)11-3'
3 讨 论
在抗性愈伤组织的诱导分化过程中,愈伤组织的褐变死亡现象是导致低分化率的最大原因。植物组织培养中产生褐变的原因很多,可能是在多次转接过程中愈伤组织受到了多次切割,切口部位酚类物质被氧化的面积越来越大,导致了褐变程度的加深。在抗性筛选过程中使用了Cef来抑制农杆菌的生长,但Cef却易于引起伤口细胞的褐变死亡,也会抑制芽的分化[20]。在愈伤组织诱导过程中pH值的变化,也可能改变酚类与酚氧化酶的结合部位,进而影响酚类的氧化程度。在之后的实验中,可以深入探索防止植物组织培养褐变的措施,从而提高分化率。
在解析突变型愈伤组织端粒酶体外酶促反应的产物时,表6显示酶促产物中出现8次(G)GGGTTTT和1次(GG)GGGTTTT,可能是RNA模板的突变降低了端粒酶的保真性所导致。3次GGGGTTT序列的出现可能是由于突变型愈伤组织中内源的野生型端粒酶RNA模板造成的干扰所致。根据陈波等[19]的研究,端粒酶在延伸前导引物时倾向于合成GGTTTAG、TTAGGGT、TAGGGTT这三种类型的重复序列,且体外酶促条件下前导引物与TER模板区结合的位点一般是两个碱基,这意味着反应过程中TER模板可发生左右滑动,使得野生型端粒酶在不含dATP的情况下通过错配一个碱基G来完成端粒的延伸,但其效率应远远低于突变型端粒酶的催化作用。表6的结果可以较清晰地阐明体外延伸的部分为端粒重复序列,且出现了突变型目标重复序列,与拟南芥体外延伸试验结果基本一致[5]。
由于转基因植株体内既有内源的野生型端粒酶又有转入的突变型端粒酶,野生型端粒酶可能合成的端粒序列有AGGGTTT、TTAGGGT、TAGGGTT三种类型,而突变型端粒酶可能合成的端粒序列有GGGTTTT、TTTGGGT、TTGGGTT、TGGGTTT四种类型。在检测转基因植株染色体末端的组成时,表7的测序结果符合预期。其中7个克隆均出现了或多或少的AGGGTT序列,总计达14次,还出现了AGAGTTT、AGTGTTT、AGGGCTT、GGGGTTT这4个单一的序列,推测是突变型端粒酶RNA模板序列的存在降低了内源野生型端粒酶的保真性所导致的。从测序结果来看,突变型端粒酶合成的端粒序列GGGTTTT次数很少,出现这种情况可能有两方面的原因,其一,水稻有24条染色体,共48个染色体末端,这里只选择了其中一条染色体的一个端粒末端来进行实验,突变型端粒酶产生作用的概率相对较低;其二,体内端粒合成过程中,转基因植株的内源野生型端粒酶占优势,故合成的野生型端粒重复序列居多,推测突变型端粒酶只是间或插入野生型端粒酶作用的进程中,故合成的序列很少。
综合上述分析,研究认为马登旭克隆得到的这条候选序列为水稻端粒酶RNA序列。根据已报道的拟南芥端粒酶的情况,其拥有两个形式的TER[5],水稻也可能存在多个形式,而现克隆得到的这条序列也许只是其中一种,水稻端粒酶是否拥有另一个TER形式还有待进一步的研究。
[1] MITCHELL J R, COLLINS K. Human telomerase activation requires two independent interactions between telomerase RNA and telomerase reverse transcriptase[J]. Molecular Cell, 2000, 6(2): 361-371.
[2] BEATIIE T L, ZHOU W, ROBINSON M O, et al. Reconstitution of human telomerase activityinvitro[J]. Current Biology, 1998, 8(3): 177-180.
[3] WEINRICH S L, PRUZAN R, MA L, et al. Reconstitution of human telomerase with the template RNA component hTR and the catalytic protein subunit hTRT[J]. Nature Genetics, 1997, 17(4): 498-502.
[4] GREIDER C W, BLACKBURN E H. Identification of a specific telomere terminal transferase activity in Tetrahymena extracts[J]. Cell, 1985, 43(2): 405-413.
[5] CIFUENTES-ROJAS C, KANNAN K, LIN T, et al. Two RNA subunits and POT1a are components of Arabidopsis telomerase[J]. Proceedings of the National Academy of Sciences, 2011, 108(1): 73-78.
[6] FENG J, FUNK W D, WANG S S, et al. The RNA component of human telomerase[J]. Science, 1995, 269(5228): 1236-1241.
[7] 马登旭, 杨力媛, 马国兴, 等. 水稻端粒酶RNA候选序列的克隆及特征解析[J]. 浙江理工大学学报, 2013, 30(4): 579-584.
[8] 杨力媛, 郑洁, 马登旭, 等. 水稻端粒酶 RNA 模板基因序列鉴定体系的建立[J]. 浙江理工大学学报, 2013, 30(4): 590-594.
[9] HELLER K, KILIAN A, KLEINHOFS A, et al. Telomerase activity in plant extracts[J]. Molecular and General Genetics MGG, 1996, 252(3): 342-345.
[10] RIHA K, FAJKUS J, SIROKY J, et al. Developmental control of telomere lengths and telomerase activity in plants[J]. The Plant Cell Online, 1998, 10(10): 1691-1698.
[11] KILIAN A, HELLER K, KLEINHOFS A. Development patterns of telomerase activity in barley and maize[J]. Plant Molecular Biology, 1998, 37(4): 621-628.
[12] HABERLANDT G. Experiments on the culture of isolated plant cells[J]. The Botanical Review, 1969, 35(1): 68-88.

[14] LIU X C, LIANG Y H, Chen B. Quantitative analysis for telomerase activity in rice[J]. China Biotechnology, 2007, 27(5): 45-49.
[15] 陈惠, 赵原,种康. 一种改进的水稻成熟胚愈伤组织高效基因转化系统[J]. 植物学通报, 2008, 25(3):322-331.
[16] Fitzgerald M S, MCKNIGHT T D, SHIPPEN DE. Characterization and developmental patterns of telomerase expression in plants[J]. Proceedings of the National Academy of Sciences, 1996, 93(25): 14422-14427.
[17] 刘红彦, 马国兴, 杨力媛, 等. 西兰花端粒酶分离与活性测定探究[J].浙江理工大学学报, 2012, 29(4):604-608.
[18] KIM N W, PIATYSZEK M A, PROWSE K R, et al. Specific association of human telomerase activity with immortal cells and cancer[J]. Science, 1994, 266(5193): 2011-2015.
[19] 陈波, 梁江丽, 田晓平, 等. 水稻愈伤组织端粒酶催化特征检测[J]. 浙江理工大学学报, 2009, 26(4):597-601.
[20] 程振东, 卫志明, 许智宏. 根癌农杆菌对甘蓝型油菜的转化及转基因植株的再生[J]. 植物学报, 1994, 36(9): 657-63.
(责任编辑: 许惠儿)
Identification of Telomerase RNA Candidate Sequence in Rice (OryzaSativaL.)
ZHULianlian,LINGaoqiang,YECongying,HUANGQian,LIUXiaochuan
(Institute of Bioengineering, College of Life Science, Zhejiang Sci-Tech University, Hangzhou 310018, China)
Telomerase is composed of telomerase inverse transcription and telomerase RNA. Except template sequence which is conservative in telomerase RNA, conservation degree in olther zones is very low, so it is very hard to clone based on homologous sequence. Currently, only Arabidopsis thaliana RNA has been cloned in plants. To identify a candidate telomerase with the characteristic of telomerase RNA, the activity of the transgenic callus telomerase after site-directed mutagenesis T/A of the template sequence (5’-AAACCCTAA-3’) was compared. The result shows the activity of mutant telomerase is significantly higher than that of non-mutant telomerase in the absence of dATP during the TRAP approach. Furthermore, a sequencing result from the ssDNA extended in vitro by the transgenic telomerase shows that all of the 10 fragments contain mutant feature sequence. Then, telomere detection of transgenic plants was conducted. The results show that among 8 telomere sequences, 2 contain mutant feature sequence. Therefore, we can preliminarily judge the candidate sequence is telomerase RNA gene in rice.
rice; telomerase; telomerase RNA; TRAP; site-directed mutation
10.3969/j.issn.1673-3851.2016.01.019
2015-05-04
朱莲莲(1989- ),女,浙江绍兴人,硕士研究生,主要从事水稻端粒酶方面的研究。
刘小川,E-mail:xcliu@zstu.edu.cn
Q785
A
1673- 3851 (2016) 01- 0116- 06 引用页码: 010705
