表面增强拉曼光谱(SERS)技术对非标记蛋白质的研究进展
2016-07-12孔卫贺韩晓霞
陈 雷,孔卫贺,韩晓霞,赵 冰*
1. 吉林师范大学环境友好材料制备与应用教育部重点实验室,吉林 四平 136000
2. 吉林大学超分子结构与材料国家重点实验室,吉林 长春 130012
表面增强拉曼光谱(SERS)技术对非标记蛋白质的研究进展
陈 雷1, 2,孔卫贺1,韩晓霞2,赵 冰2*
1. 吉林师范大学环境友好材料制备与应用教育部重点实验室,吉林 四平 136000
2. 吉林大学超分子结构与材料国家重点实验室,吉林 长春 130012
基于表面增强拉曼光谱(SERS)技术在非标记蛋白质研究方面的最新进展。SERS是一个特殊的拉曼光谱现象,对于众多被吸附到粗糙金属表面上的拉曼活性分析物,可以提供增强拉曼信号(通常可以增强几个数量级)。SERS是一个灵敏的,选择性的,和通用的技术,并且可以实时、快速的对数据进行采集。因此,在基于仪器仪表技术和数据分析方法以及SERS在生物体系中的诸多优势,SERS经历了快速的发展阶段。重点介绍几个采用SERS技术对生物体系的代表性研究。某些SERS的生物应用发展比较成熟,并已经可以小范围临床应用,而有些还停留在发展的初始阶段(实验室研究阶段)。讨论了最近发展起来的几种基于SERS技术定量分析的方法, 选择不同SERS活性基底和技术(如生物分子在电极上,胶体纳米粒子,周期性图案结构和基于针尖拉曼技术)对蛋白质进行直接研究。此外,根据SERS指纹信息的变化可以用来研究蛋白质-蛋白质,蛋白质-配体间的相互作用。基于SERS技术对生物分子进行定性和/或定量分析方面显示出了相当大的优势。
表面增强拉曼光谱;蛋白质;非标记方法;定性分析;定量分析
1 表面增强拉曼光谱技术的一般概述
拉曼光谱和红外吸收光谱是通过分析分子振动信息实现对分子结构的解析,目前已经成为最广泛使用的分析技术。拉曼光谱可以提供解析分子结构的指纹信息。特别是,拉曼光谱在生物体系研究中具有诸多优势,它可以实现生物分子的无损分析和超高的检测灵敏度。并且拉曼光谱特别适用于水体系研究,因此拉曼光谱也被认为是生命体系研究。拉曼散射是光子与分子之间的非弹性碰撞[1-3]。当光从分子中被分散出来,散射光的一小部分(约1千万分之1个光子),通过激发被散射出来,因为散射光子与入射的光子具有不同的频率,因此光子与分子之间发生了能量交换而产生拉曼散射。SERS是表面敏感的分析技术,当分子吸附于粗糙的贵金属表面(例如金,银,和铜)时会产生拉曼信号增强的现象(如图1)。
根据实验中发现的不同现象,关于SERS的许多理论在早期阶段就被提出来了,并且有很多种解释。在众多理论中大多数研究人员都接受一个由两部分组成的理论。拉曼信号的增强,通常归因于两个不同的因素[4-12]:电磁(EM)和化学增强(电荷转移,CT)。在电磁增强方面,一个分析物被吸附到或接近粗糙金属表面时,由于金属表面能够在表面发生等离子体震荡现象,因此分析物可以受到电磁场的影响而被增强。在化学增强或电荷转移方面,首先,分析物要吸附在金属表面,通过电子转移产生增强的现象(金属-分子-金属)。因此,化学增强仅仅可能发生在吸附表面的分析物的第一层,而EM增强可能发生在第二层或其后表面层。有些增强的效能已经高达10纳米或离金属表面层更远的距离[4-5]。

图1 靶分子吸附于金属粒子基底的SERS示意图
2 表面增强拉曼光谱技术在蛋白质研究方面的优势和限制
蛋白质对于生命体来说是必要的,因为他们是细胞新陈代谢的主要组成部分。SERS是一项非常灵敏的技术,在高通量的生物分子检测方面已经被证实具有很大的潜力,特别是在蛋白质的检测方面。关于蛋白质的研究仅仅涉及到氨基酸的侧链,而一些研究小组从两个酰胺基团和侧链取得了SERS信号。目前,生物分子(非标记法)的内部结构信息和外在的SERS标签(标记法)是用于直接和间接检测生物分子的两种独立的方法。标记法需要适当的染料标签,该标签若在某一激发线下存在共振的现象,则可以提供附加的增强信号,因此它的检测灵敏度更高。而非标记的手段,得到的都是蛋白质内部氨基酸骨架的信息,该光谱的重现性差,蛋白质吸附位点的非重现性以及蛋白质吸附于金属表面的变性等问题,都会对非标记方法研究带来困难。但是非标记手段给出的信息更直接的分析蛋白质结构,对蛋白质的解析有很大帮助。
3 应用不同SERS活性材料用于蛋白质的直接分析
SERS活性基底对于SERS技术的生物分析与检测方面都起着重要的作用。由于SERS的发现,各种各样的SERS活性基底被发展起来并应用于生物检验。一个理想的SERS活性基底应该具有以下四个特点:(1)具有很高的拉曼增强效应;(2)具有均匀的表面,使得SERS信号重现性好;(3)提供良好的光谱稳定性;以及(4)能够从杂散SERS信号中区分出其本身。
蛋白质的直接的SERS分析可以给出蛋白质本身的结构信息,这种方法更可靠、更灵敏并且更具有选择性。随着新型SERS活性基底的发展,拉曼光谱仪器,以及样品前处理的方法的改进,使得该分析方法更具有权威性,并且被认为是具有前途的分析技术。
3.1 金属电极
基于电化学粗糙的金属电极的蛋白质研究中,通常利用循环伏安法,处理电极表面结构[13-14]。银或金电极表面可以通过自组装膜等覆盖,提高了基底表面的生物相容性(图2)[15-18]。
电极上的信号增强机制同样可以依靠电磁场和化学增强机制来解释。金属电极的表面粗糙度通常通过电化学粗糙化产生,其对SERS活性的改善具有非常重要的作用。基于SERS活性电极的蛋白质研究,主要围绕着细胞色素类蛋白质研究,通过外加电源,根据对SERS信号的分析,来对电荷在蛋白质内部转移过程进行研究。

图2 细胞细素c吸附于电化学粗糙的金或银电极表面示意图
3.2 胶体纳米粒子
目前,对蛋白质的检测最常用的SERS活性基底是金属胶体粒子(例如,金,银,及金属复合物纳米粒子)[19-21]。这些胶体粒子的制备方法简单,而表现出非常优异的应用价值。然而,随着银溶胶的使用而出现一些问题,是当分析物加入后,金属胶体粒子聚集严重,这使得胶体不稳定,经常导致SERS光谱的非重现性。因此,一些稳定剂例如聚(乙烯醇),聚(乙烯基吡咯烷酮),柠檬酸盐和十二烷基硫酸钠等分子被广泛的用来减少这种凝结问题,然而,这些稳定剂的使用可能会产生拉曼信号干扰[22-24]。在SERS活性基底的分析实验中,近段时间的研究中,已经结合了胶体悬浮液的优点和固体基底的稳定性的共同优势来对生物体系进行研究。
我们曾经开发的方法之一就是(图3)结合蛋白质印记和银溶胶染色技术,从而实现了基于SERS技术的非标记蛋白质的检测(例如牛血清白蛋白,血红素蛋白质,溶菌酶,和人血清白蛋白(HSA))[25]。用于蛋白质转移载体的硝酸纤维素膜是一个拥有诸多优点的很好的固定化膜,如蛋白结合的能力较强,高灵敏度,和易用性。在此,我们用银胶体染色和蛋白质印记技术结合的手段开发了一个新型简便的方法(无任何其它干扰试剂)拓展了蛋白质印记技术的检测方法。由于蛋白质和银纳米粒子之间的相互作用,蛋白质条带会被银溶胶染色,呈现出较清晰的条带。进而,利用SERS技术对条带进行分析,可以得到不同蛋白质的指纹信息,帮助我们分析蛋白质的吸附行为。因此,非标记的多目的蛋白质可以在没经处理的条件下,经蛋白质印迹转移到硝酸纤维素膜表面而被检测到。蛋白质印记SERS研究方法是结合了银溶胶染色技术,提高了待测物的检测灵敏度。利用该方法可以实现对血红素蛋白质的达到纳克水平的检测灵敏度。

图3 基于蛋白质印迹SERS对蛋白质检测示意图 血红素蛋白质和牛血清白蛋白SERS光谱
在另一项研究中(图4),银胶体纳米粒子[26]经酸化的硫酸钠作为聚集剂被用来诱导高的电磁场增强效应。这里,几种非标记蛋白质(如溶菌酶,核糖核酸酶B,抗生物素,过氧化氢酶,以及血红素蛋白质)在水溶液中被检测。展现出这种方法的诸多优点。

图4 硫酸盐诱导的金属纳米粒子聚集 用于非标记蛋白质检测示意图
Fig.4 Schematic of acidified sulfate as an aggregation agent for label-free protein detection in aqueous solution
此外,Ozaki教授团队的研究报道的,电解质和非标记蛋白质共同吸附在银胶体上用来分析蛋白质的吸附机制。由于蛋白质和电解质两种物质的特征峰都可以在SERS光谱上被观察到,表明蛋白质和NO-3共吸附在纳米银上。这些研究结果将有助于理解蛋白质在Ag胶体上的吸附机理,并对设计制备蛋白检测的SERS活性基底样品非常有用[27]。Weidinger和Hildebrandt研究团队报道了关于血红素蛋白质装饰在磁性与金属纳米粒子复合结构表面,用于捕获毒性物质(如NO2-, CN-和H2O2等)。当这些具有一定毒性物质吸附于血红素蛋白质表面时,会诱导血红素蛋白质结构的变化,并且可以利用SERS技术检测蛋白质结构变化过程。因此,通过这一特点,可以对血红素蛋白质和毒性物质的共检测[28]。
3.3 有序阵列结构
微纳米尺度有序阵列是一种模型结构,有助于我们理解SERS增强机制。这些阵列是通过电子束印刷技术,纳米压印光刻技术或纳米球光刻等技术制备而形成的,这使得高度重复性图案的制作能够具有各种几何形状和尺寸的结构[29-32]。SERS活性基底的周期模式被用来研究蛋白质的结构,Das等已经发表了蛋白质的结构分析(如:溶菌酶、核糖核酸酶B、牛血清白蛋白和血红素蛋白质),他们利用电镀和电子束刻蚀技术制备几种金纳米聚集体结构的直径在80和100纳米之间、间隙在10~30 nm区间(图5),提高基底的SERS活性以及SERS光谱重现性。
有研究者构筑一种检测模型,将蛋白质夹在Au/Ag三明治结构中, 作为基于SERS技术的检测对象。细胞色素c作为媒介用来构造Au/Ag三明治结构,由于金和银层形成共振腔,提供了较强的电磁场贡献而提高的基底的SERS活性。获得的光谱具有较高的灵敏度和分辨率, 这表明这种SERS活性基底在蛋白质检测中是一种强大的工具, 并在蛋白质芯片应用方面潜力巨大[33]。

图5 周期性有序金或银纳米阵列示意图(a); 血红素蛋白质SERS光谱(b)
Fig.5 (a) Schematic of periodic pattern Au/Ag substrate;(b) SERS spectrum of Mb
三维二氧化硅-银核壳SERS活性基底是通过利用自组装和种子催化结合的方法制备而成。该方法使我们能够精确地控制和重塑成片的银覆盖在硅纳米材料表面,可以制备出大片的连续的结构并提供较大的电磁场贡献。二氧化硅表面上聚集的银纳米粒子是通过无电沉积90 s得到的。这一方法制备出来的SERS活性基底允许非标记蛋白质直接吸附,得到的SERS光谱具有重现性好,灵敏度高的特点(图6)。
3.4 基于针尖拉曼光谱技术
针尖拉曼技术(TERS)被认为是最有前景研究化学增强机制的一项技术。作为一个有潜途的技术,TERS 被用来分析蛋白质的结构,尤其是细胞色素类蛋白质。TERS和SERS会得到不同的分子结构信息,这种差异更有助于研究分析物的结构。目前对于细胞色素类蛋白质可能有三种检测结果(图7)[34]。

图6 细胞色素c和溶菌酶的SERS光谱 插入图为细胞色素c和溶菌酶吸附于SERS活性基底示意图
The inset is the schematic illustrations of cyt c and lysozyme adsorbed on the SSCS substrate
第一,当制备AFM针尖距离样品表面有一定距离时,第二,当针尖贴近细胞色素类蛋白质的卟啉,这里面主要得到是卟啉的结构信息,而很难观测到氨基酸骨架信息,峰值强度相对于远场方法增强两倍。第三,当针尖贴近蛋白质氨基酸部分,得到的SERS信息主要来自于氨基酸骨架的信息,对分析蛋白质结构更加有利。目前,基于TERS技术的对其它类蛋白质,如血红素蛋白质和胰岛素都有报道[35-39]。
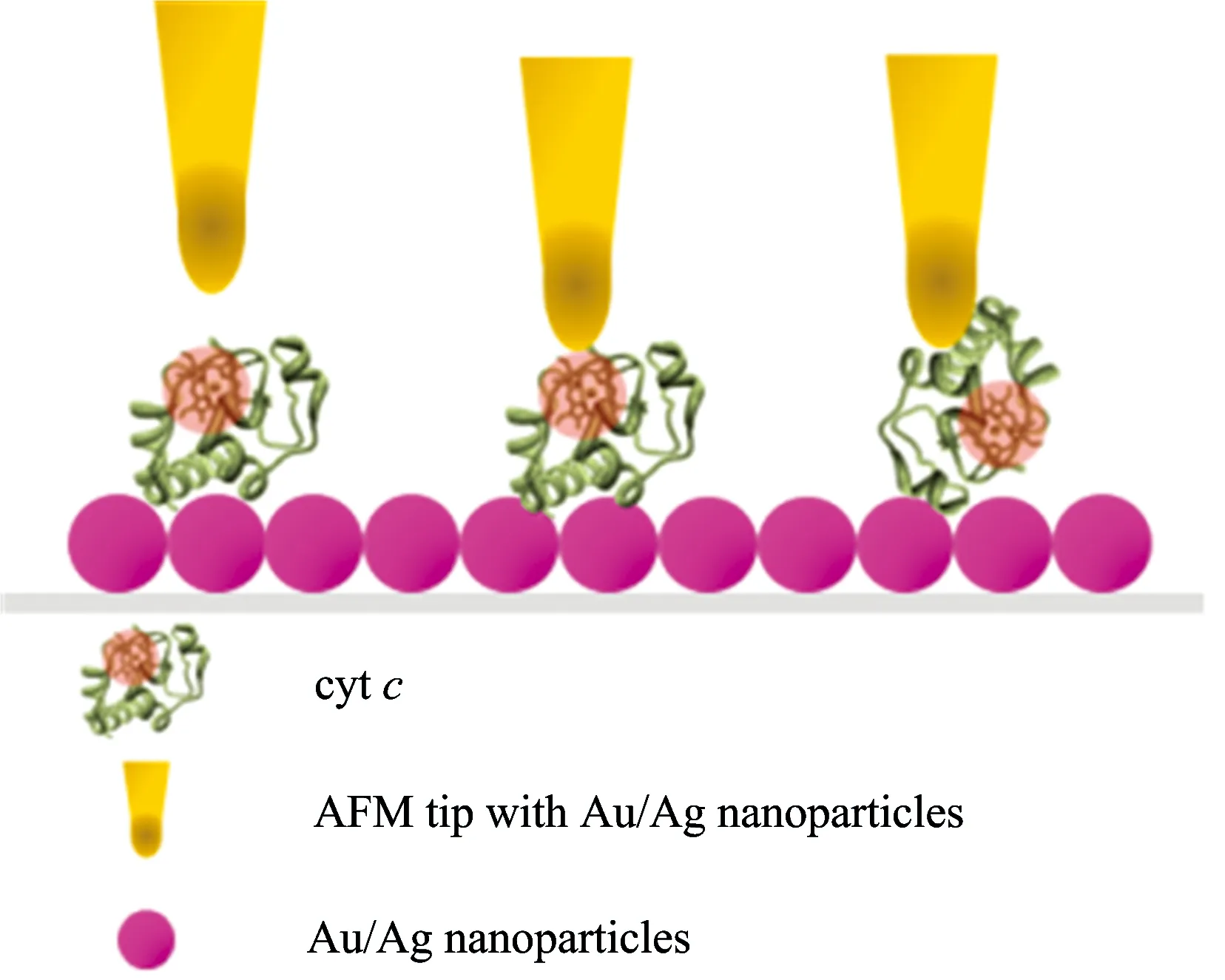
图7 金或银AFM探针检测细胞色素c的三种可能位置
4 展 望
概述了SERS基底技术及其在生物学应用趋势和发展,总结了近段时间不同类型SERS活性基底用于蛋白质组学分析中。SERS基底直接定量分析蛋白质:主要研究重点是蛋白质的SERS指纹信息变化,这可能会进一步被用于分析蛋白质的结构和功能之间的关系。SE(R)RS基底技术的进一步研究将会从疾病相关蛋白质研究目标出发。利用不同的SERS活性材料选择性的研究蛋白质及蛋白质间的相互作用。最重要的优势是, 它将提供快速和高灵敏度信息,为蛋白质结构以及蛋白质-蛋白质间的研究提供帮助,对于相关疾病的早期诊断具有一定的参考价值。
[1] Raman C V, Krishnan K S. Nature, 1928, 121: 501.
[2] Sasic S. Pharmaceutical Applications of Raman Spectroscopy, John Wiley & Sons, Inc., Hoboken, NJ, 2007.
[3] Siebert F, Hildebrandt P. Vibrational Spectroscopy in Life Science, WILEY-VCH Verlag GmbH & Co. KGaA, Weinheim, 2008.
[4] Aroca R. Surface-Enhanced Vibrational Spectroscopy, John Wiley & Sons, Ltd, Chichester, 2006.
[5] Kneipp K, Moskovits M, Kneipp H. Surface-Enhanced Raman Scattering-Physics and Applications, Springer, Berlin, Heidelberg, 2006.
[6] Bonham A J, Braun G, Pavel I, et al. J. Am. Chem. Soc., 2007, 129: 14572.
[7] Kleinman S L, Ringe E, Valley N, et al. J. Am. Chem. Soc., 2011, 133: 4115.
[8] Brus L. Acc. Chem. Res., 2008, 41: 1742.
[9] Qian X M, Zhou X, Nie S M. J. Am. Chem. Soc., 2008, 130: 14934.
[10] Camden J P, Dieringer J A, Zhao J, et al. Acc. Chem. Res., 2008, 41: 1653.
[11] Dasary S S R, Singh A K, Senapati D, et al. J. Am. Chem. Soc., 2009, 131, 13806.
[12] Willets K A, Stranahan S M, Weber M L. J. Phys. Chem. Lett., 2012, 3: 1286.
[13] Lin Z B, Tian J H, Xie B G, et al. J. Phys. Chem. C, 2009, 113(21): 9224.
[14] Wu D Y, Li J F, Ren B, et al. Chem. Soc. Rev., 2008, 37: 1025.
[15] Murgida D H, Hildebrandt P. J. Am. Chem. Soc., 2001, 123: 4062.
[16] Todorovic S, Pereira M M, Bandeiras T M, et al. J. Am. Chem. Soc., 2005, 127: 13561.
[17] Jin B, Wang G X, Millo D, et al. J. Phys. Chem. C, 2012, 116: 13038.
[18] Feng J J, Gernert U, Sezer M, et al. Nano Lett., 2009, 9: 298.
[19] Kneipp K, Kneipp H, Itzkan I, et al. Chem. Rev., 1999, 99: 2957.
[20] Wells S M, Retterer S D, Oran J M, et al. ACS Nano, 2009, 3: 3845.
[21] Pinkhasova P, Puccio B, Chou T, et al. Chem. Commun., 2012, 48, 9750.
[22] Durocher S, Rezaee A, Hamm C, et al. J. Am. Chem. Soc., 2009, 131: 2475.
[23] Johnson C J, Dujardin E, Davis S A, et al. J. Mater. Chem., 2002, 12, 1765.
[24] Li M D, Cui Y, Gao M X, et al. Anal. Chem., 2008, 80: 5118.
[25] Han X X, Jia H Y, Wang Y F, et al. Anal. Chem., 2008, 80: 2799.
[26] Han X X, Huang G G, Zhao B, et al. Anal. Chem., 2009, 81: 3329.
[27] Zhou Z, Yang I S, Huang G G, et al. Analyst, 2010, 135: 2372.
[28] Han X X, Schmidt A M, Marten G, et al. ACS Nano, 2013, 7: 3212.
[29] Mahajan S, Abdelsalam M, Suguwara Y, et al. Phys. Chem. Chem. Phys., 2007, 9: 104.
[30] Theiss J, Pavaskar P, Echternach P M, et al. Nano Lett., 2010, 10: 2749.
[31] Xue M, Zhang Z, Zhu N, et al. Langmuir, 2009, 4347.
[32] Wang C X, Ruan W D, Ji N, et al. J. Phys. Chem. C, 2010, 114: 2886.
[33] Ruan W D, Ji W, Xue X X, et al. J. Raman Spectrosc., 2011, 42: 1492.
[34] Yeo B S, Mädler S, Schmid T, et al. J. Phys. Chem. C, 2008, 112: 4867.
[35] Treffer R, Brhme R, Deckert-Gaudig T, et al. Biochem. Soc. Trans., 2012, 40: 609.
[36] Alexander K D, Schultz Z D. Anal. Chem., 2012, 84: 7408.
[37] Kurouski D, Deckert-Gaudig T, Deckert V, et al. J. Am. Chem. Soc., 2012, 134: 13323.
[38] Wood B R, Asghari-Khiavi M, Bailo E, et al. Nano Lett., 2012, 12: 1555.
[39] Sonntag M D, Klingsporn J M, Garibay L K, et al. J. Phys. Chem. C, 2012, 116: 478.
(Received Jun. 28, 2015; accepted Nov. 15, 2015)
*Corresponding author
Study of Proteins Based on Surface-Enhanced Raman Spectroscopy (SERS)
CHEN Lei1,2, KONG Wei-he1, HAN Xiao-xia2, ZHAO Bing2*
1. Key Laboratory of Preparation and Applications of Environmental Friendly Materials, Ministry of Education, Jilin Normal University, Siping 136000, China
2. State Key Laboratory of Supramolecular Structure and Materials, Jilin University, Changchun 130012, China
This article outlines the recent progress in surface enhanced Raman spectroscopy (SERS) based proteins applications. SERS is a specific Raman spectroscopic technique that provides enhanced Raman signals (several orders of magnitude greater than normal) for numerous Raman-active analyte molecules adsorbed onto rough metal surfaces. SERS is a sensitive, selective, and versatile technique that lends to fast data acquisition in actual time. Therefore, SERS has undergone rapid development because of its technical advantages in instrumentation, data analysis methods and its multiple biological applications. This article highlights several representative areas in proteins where SERS could be employed. Some of the proteins applications of SERS are more maturely developed, whereas others are in their initial stages of development (in laboratories). This article discusses the recent developments in SERS based quantitative analysis: directly with different substrates (e.g. biomolecules on electrodes, colloidal particles, and periodic pattern structure and tip based substrates). Furthermore, SERS based techniques are advantageous for obtaining valuable information on protein-protein, protein-ligand, and protein-drug recognitions via spectral differences among molecular bridges. SERS based techniques show considerable promise for qualitative and/or quantitative analyses of biological systems.
SERS;Proteins; Label-free method; Quantitative analysis; Qualitative analysis
2015-06-28,
2015-11-15
国家自然科学基金项目(21403082,21273091),吉林省科技发展计划项目(20150520015JH),吉林省教育厅项目(2016-217)和吉林师范大学博士科研启动项目(2014004)资助
陈 雷,1982年生,吉林师范大学环境友好材料制备与应用教育部重点实验室讲师 e-mail: chenlei@jlnu.edu.cn *通讯联系人 e-mail: zhaob@jlu.edu.cn
O657.3
A
10.3964/j.issn.1000-0593(2016)10-3087-05
猜你喜欢
杂志排行
光谱学与光谱分析的其它文章
- Gd靶激光等离子体光源离带辐射及其等离子体演化的研究
- Probing the Binding of Torasemide to Pepsin and Trypsin by Spectroscopic and Molecular Docking Methods
- Mn(Ⅱ)-5-Br-PADAP共沉淀-火焰原子吸收光谱法测定虾、贝样中的镉
- Near Infrared Spectroscopy Study on Nitrogen in Shortcut Nitrification and Denitrification Using Principal Component Analysis Combined with BP Neural Networks
- 内蒙古草原植被最大光能利用率取值优化研究
- 健康和糖尿病大鼠红细胞荧光光谱非线性程度差异
