大豆光周期调控开花与产量性状的研究进展
2016-05-28孔凡江赵晓晖刘宝辉
孔凡江,赵晓晖,刘宝辉
(中国科学院 东北地理与农业生态研究所 大豆分子设计育种重点实验室,黑龙江 哈尔滨 150081)
大豆光周期调控开花与产量性状的研究进展
孔凡江,赵晓晖,刘宝辉
(中国科学院 东北地理与农业生态研究所 大豆分子设计育种重点实验室,黑龙江 哈尔滨 150081)
摘要:大豆作为重要的经济作物,是人类植物油和植物蛋白的主要来源,但目前我国大豆产业面临严峻的挑战。大豆光周期开花不仅影响大豆的种植适应性,而且决定着大豆的产量。文章阐述了大豆特有的光周期开花调控途径PhyA-E1-FT,同时论述了光周期开花调控基因间的相互作用关系。大豆光周期反应中重要基因的克隆为理解开花期和生育期分子机理提供理论基础,同时对大豆分子育种和提高大豆产量具有重要意义。图1,参53。
关键词:大豆;光周期;生育期;开花期;产量
0引言
在植物生命循环过程中,开花是一个很重要的标志,它标志着植物由营养生长向生殖生长的重要转变,是在整合外界环境因子(如光、温度)和植物本身内源信号基础上的外在表现[1-2]。对开花起重要调控作用的是光周期[3- 4],植物能够随着日照长短的变化而改变的现象被称为光周期现象(Photoperiodism)[5]。大豆是典型的短日照作物,即在短日照条件下(小于12小时光照)比在长日照条件下(大于16小时光照)开花明显提前,而且韩天富证明了大豆的光周期反应不仅存在于开花前,而且在开花后的鼓粒期至成熟期都存在[6]。大豆光周期的研究不仅具有重要的理论意义,而且在农业上对指导大豆开花及产量提高具有重要的实践意义。文章综述了大豆主要生育期基因的研究现状及目前所揭示的大豆光周期调控途径,并分析了光周期对大豆产量性状的调控作用。
1大豆主要生育期基因的研究现状
大豆生育期(开花期和开花后期)特别是开花期是大豆光周期反应的重要指标,是重要的农艺性状,对大豆的产量、品质和适应性至关重要。根据经典遗传学的研究方法,对近等基因系进行比较,已发现了10个控制大豆开花期和成熟期基因位点,分别是E1、E2[7]、E3[8]、E4[9]、E5[10]、E6[11]、E7[12]、E8[13]、E9[14]和J[15],这些基因位点如果是显性,则在自然昼长的情况下表现为延迟花期与延长成熟期的作用,但是每个基因影响的程度却不一样[16]。在已知的这些E系列基因位点中,E1-E4和E9研究较为深入,均已被分子克隆。
大豆生育期基因E3和E4不但与大豆开花期和成熟期有关,同时也与光周期的敏感性有关。大豆植株在人工控制的不同红光∶远红光(R∶FR)量子比的光质量下会产生不同的反应[17]。通过用荧光灯以较高的R∶FR比延长至20小时,第一次证实了E3位点,其隐形纯合体在长日照条件下早花[8]。通过用白炽灯以低的R∶FR比延长自然光至20小时,又发现了E4位点[9],e4e4隐性等位基因并不能单独的对光周期产生不敏感性。因此,大豆植株在长日照条件下提前开花,必须携带隐性纯合基因e3[9,17-18]。因此,E3和E4双显性基因型延迟开花和成熟,对光周期反应敏感;而纯合双隐性基因型提早开花和成熟,对光周期反应不敏感。E3基因的效应大于E4,并且对E4具有上位性作用[18]。2009年Watanabe等利用图位克隆方法将E3基因克隆出来,E3基因是大豆光敏色素A基因的一个拷贝(GmphyA3)。此外,还发现E3位点在栽培品种中存在两种自然突变类型,一种是光敏色素第四个外显子缺失的Harosoy-e3类型,另一种是在第三个外显子上氨基酸置换的Misuzudaizu类型[19]。Liu等首次将E4基因克隆出来并证明其为大豆光敏色素A基因之一(GmphyA2),e4突变型是由于在GmphyA2基因第一个外显子上插入一个LTR型的反转录转座子SORE1,因此造成该基因失活。光敏色素A基因的另一个拷贝GmphyA1基因被定位在连锁群O,与连锁群I的E4位点区域是同源的。在连续的远红光下,纯合体e4等位基因的植株表现出黄化,也有部分非黄化的植株,表明e4单突变并不能促使GmphyA功能的完全丧失[20]。
大豆生育期基因E2为拟南芥GIGANTEA(GI)的同源基因GmGIa。Watanabe等成功克隆了E2基因,并发现大豆品种Misuzudaizu以及Harosoy近等基因系材料的DNA序列第10个外显子上有单碱基突变,恰好导致终止密码子产生,使E2基因的翻译提前终止,以至于功能发生了改变。在自然光照条件下e2基因型的开花期明显提前,并且e2突变体中的开花整合因子GmFT2a基因被上调表达。大豆E2及其近等基因系材料(e2)在高纬度43°N和中纬度36°N的开花时间相近,表明E2基因对大豆开花期的调控可能不依赖于光周期,以至于地理适应性更强[21]。E2基因对生育期贡献大,对光周期反应影响小,而且受外在环境影响较小。因此,E2基因在育种实践上具有广阔的应用前景。
大豆生育期基因E1对开花期及成熟期影响最大[7,22-25]。Xia等用Harosoy-E1(E1e2E3E4e5)与 Harosoy(e1e2E3E4e5)建立的群体,成功地实现了该基因的克隆。E1基因含有一个双边核定位信号和DNA结合位点,并含有一个B3结构域,其编码蛋白定位在细胞核中,而且E1是豆科植物特有的转录因子。在短日照条件下E1基因的表达受到明显抑制,而长日照条件下则表现出双峰的昼夜节律模式,表明E1基因受光周期调控,且长日照条件诱导E1的表达。然而,在e3/e4遗传背景下E1被长日照诱导表达的作用却被消除,表明E1基因受控于E3和E4。以早花品种Kariyutaka为受体材料进行大豆转基因,超表达E1的转基因株系与对照相比大大延迟开花,并且下调关键开花基因GmFT2a和GmFT5a的表达[26]。即便是在e3/e4遗传背景下,E1基因也表现出抑制开花的作用,尤其在远红光丰富的长日照条件及R∶FR量子比小于1的条件下[17]。最近Xu等对E1家族基因E1-like-a(E1La)和E1Lb在大豆光周期方面的分子基础进行研究,通过光暗转移试验(Transitionbetween light and dark phases)和暗中断试验(Night-break experiment)研究发现,E1家族基因仅仅在有光的条件下表达,而且前一天黄昏前的光照诱导对E1家族基因的表达起关键作用。进一步研究发现E1-like基因和E1在功能上是相同的,把E1家族基因沉默掉以后大豆植株表现出早花以及成熟期提前[27]。这些结果表明E1及其同家族基因在抑制大豆开花期和成熟期方面起关键作用。随着E1基因的成功克隆、同家族基因的功能分析及对其作用机制的深入研究,可进一步揭示大豆特有的光周期反应及开花调控机制。
在隐性纯合e1-nl背景下,利用两个回交杂交群体,Kong等发现了E9基因,显性纯合的E9E9基因控制早花,隐性纯合的e9e9基因控制晚花[14]。通过图位克隆的方法,Zhao等克隆了E9基因,证明E9由大豆GmFT2a基因编码,隐性e9基因由于在GmFT2a基因的第一个内含子插入了反转录转座子SORE1,导致GmFT2a基因的表达量降低,从而使其功能受到抑制,延迟大豆开花。进一步研究表明,反转录转座子SORE1插入GmFT2a基因内含子两侧DNA区域存在着甲基化现象,GmFT2a基因的内含子序列中可能存在该基因的顺式调控元件,这一区域的甲基化影响了GmFT2a基因的表达[28]。
2大豆光周期调控途径
随着分子生物学技术的不断进步和更新,拟南芥中对开花贡献起关键作用的基因已被鉴定和分离,对这些基因的研究,使人们对植物开花调控的分子机制得以不断深入。对模式植物拟南芥的光周期开花途径研究的较为透彻,首先通过光受体(Photoreceptors)感受光信号,然后传递此信号给生物节律钟(Circadian clock),由其产生生物节律(Circadian rhythm),但也并不是所有光受体调控的开花都是通过生物钟来实现的[29-30]。然后将节律输出给下游的节律调节基因(Circadian regulated genes)GI。GI的表达以及功能发挥还会受到光受体的直接调控。节律调节基因通过输出基因CO(CONSTANS)调控下游的开花时间基因(Flowering time genes),如成花途径整合因子(Floral integrators)FT基因,从而调控花分生组织识别基因(Floral meristem identity genes)的表达,最终控制拟南芥的开花时间。因此,拟南芥开花转变的光周期途径为GI-CO-FT模式。其中CO基因在光周期调控中起重要的调节作用,由Putterill采用图位克隆的方法分离得到,CO编码锌指蛋白转录因子[31],是植物生物钟与开花之间的桥梁。在韧皮部表达的CO能够激活开花植物FT的表达,而且有实验证明FT是CO蛋白的直接作用底物[32]。拟南芥FT蛋白转移至茎尖生长点,促使SOC1、LFY、AP1等基因表达,诱导植物开花[33]。然而,Jung等研究发现在拟南芥的光周期途径中GI除了调控CO基因表达以外,还调控miR172及其靶基因TOE1,即便是在缺失功能的co突变体中过表达miR172,无论长日照还是短日照条件植株都会提前开花,但最终也作用于FT基因[34]。因此,表明拟南芥miR172促进光周期调控的开花是不依赖于CO基因的一条独特途径,即GI-miR172-TOE1-FT模式。拟南芥miR172途径的发现是对光周期调控开花途径的一个完善,具有重要意义。
在大豆光周期调控途径中,目前已经揭示了包括生育期基因E1-E4在内的部分光周期调控大豆开花网络,见图1。Xu等在E3和E4基因型的大豆Harosoy近等基因系中分析E1及E1-like基因的表达,结果显示虽然在Harosoy-e3/e4材料中E1的表达模式与Harosoy(E3/E4)相同,但表达量却显著降低,甚至几乎不表达,充分证明E1及其同家族基因受到光受体基因E3和E4的调控,与Xia和Cao等的研究结果一致,Cao等还发现E3对E1影响比E4基因强烈[26-27,35]。通过大豆转基因以及病毒诱导基因沉默技术(Virusinducedgene silencing,VIGS)鉴定E1及其家族基因的功能,结果显示它们均是大豆开花抑制因子,强烈延迟大豆开花期及成熟期,但最终也是通过调控开花基因GmFT2a和GmFT5a实现对开花时间的控制[26-27]。GmFT2a和GmFT5a是大豆关键的开花整合因子,在光周期途径中协同调控开花[36],但对下游基因的调控又是不同的[37],表明二者在功能上产生了分化,冗余且协调的发挥作用。其同源基因GmFT4功能与之相反,为大豆开花抑制因子[38]。三个GmFT2a/5a/4基因均受到E1的调控。于是在大豆的光周期反应中形成了以生育期基因E1为核心的调控开花的主途径,即E3/E4-E1-GmFT。由于E1基因是豆科作物特有的转录因子,在拟南芥和水稻等物种中没有同源基因,所以E1基因的研究结果揭示了大豆基因组中独特的控制开花期与成熟期的调控途径。
随着对大豆光周期反应研究的深入,在后续的研究中发现光周期调控开花途径中的很多基因都与E1存在相互调节作用。拟南芥CO-like基因GmCOL1a过表达的大豆转基因株系在长日照条件和自然光照条件下表现出延迟开花,gmcol1b突变体与非转基因的对照相比为早花表型,表明大豆CO-like基因是大豆开花抑制因子,并且调控下游开花基因GmFT2a/5a和GmFT4的表达。当GmCOL1a过表达时下调大豆生育期基因E1和E2,而在E1和E2的近等基因系材料中GmCOL1a却被上调表达,所以极有可能在GmCOL1a、E1和E2基因之间存在反馈调节环,但仍需要进一步试验验证。同时,研究还发现E3和E4促进E2和GmCOL1a/1b的表达[35]。然而,CO基因在调控植物开花方面的功能是相对保守的,在很多物种中起到诱导开花的作用,如拟南芥、水稻[39]、牵牛花[40-41]及小麦[42]等,而大豆GmCOL1a和GmCOL1b的功能却与之相反,这可能与GmCOL1a/1b受到大豆特有转录因子E1的调控有关,也可能与大豆基因组的复杂性有关。根据Cao等的研究结果不难发现GmCOL1a/1b基因受到GI同源基因E2的调控,并且调控GmFT2a/5a的表达,所以大豆GmCOL1a/1b参与调控的光周期途径也是相对保守的,与模式植物拟南芥相同为GI(E2)-CO-FT模式;又因E1基因为豆科作物所特有,所以受E1调控的GmCOL1a/1b光周期途径又是独特的。
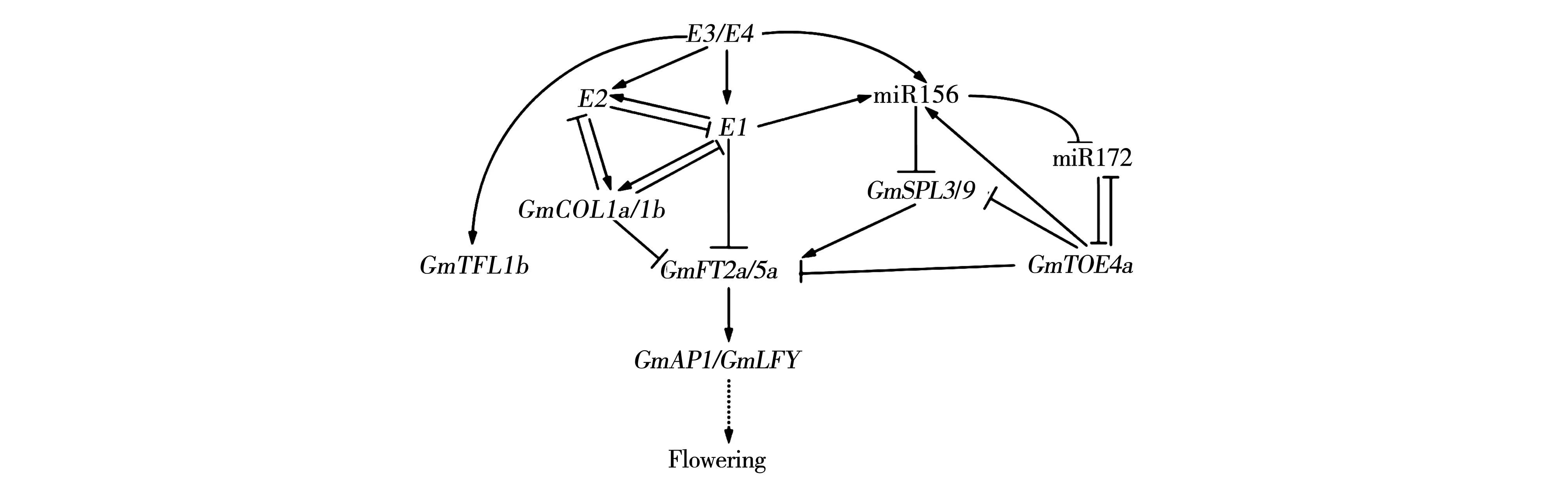
图1 大豆光周期调控开花网络模式图Fig.1 Proposed model for molecular network in the regulation of flowering time in soybean
植物miR172及其靶基因在生长发育过程中发挥调控作用,并且miR172作用于miR156的下游促进植物成年特征(Adult epidermal identity)的出现[43]。在光周期调控的大豆开花网络中miR156延迟开花,而且E1基因也参与到miR156/GmSPL通路中[44]。miR156通过其靶基因GmSPL3/9调控miR172的表达,miR172又通过靶基因GmTOE4a对miR156和GmSPL3/9进行反馈调控[45]。因此,miR156和miR172之间可能存在着一定的平衡关系,从而调控植物在适当的时间发育并完成发育阶段的时相转换,进而繁殖产生后代,以保证物种的延续。大豆miR172靶基因GmTOE4a过表达以后,转基因大豆株系延迟开花。研究发现GmTOE4a延迟从营养生长到生殖生长期的转换是通过抑制开花相关基因GmFT2a、GmFT5a、GmAP1、GmLFY和miR156的靶基因GmSPL3/9的表达,而促进GmFT4和miR156的表达实现的。同时研究发现miR172与其靶基因GmTOE4a之间存在着负反馈调控关系。进一步研究发现GmTOE4a参与的大豆光周期调控开花途径是在生育期基因E3和E4的作用下,依赖于GmCOL1a的表达,并且其转录水平不受GI同源基因E2的调控[45]。这是有别于模式植物拟南芥miR172途径的一种调控方式,再次表明大豆的光周期途径既有保守的一面,又有其独特性的一面。这可能与大豆经历了2次基因组复制成为具有复杂基因组的古四倍体物种有关。
大豆的光周期反应是一个极为复杂的过程,需要多基因间相互协调的发挥作用才能最终实现开花。目前所揭示的以E1为中心的开花网络仅仅处在转录水平,基因间的互作关系及分子机制有待于学者们深入研究,需要大量的工作来补充和完善。
3光周期对大豆产量性状的调控
大豆光周期反应不仅对开花诱导有显著影响,还对开花后的营养生长和生殖生长具有调控作用。韩天富利用原产自中国主要生态区、生育期不同的代表品种研究了大豆开花后的光周期反应问题,结果表明,不同成熟期的大豆品种开花后普遍存在着对光照长度的反应,这种反应属于典型的光周期现象;开花后的光周期反应不仅存在于大豆花荚期还存在于鼓粒期至近成熟期;并且提出光敏色素参与到控制开花后的光周期反应中[46-47]。大豆生育期基因E3和E4分别为大豆光敏色素A基因GmPhyA3和GmphyA2[19-20],二者不但调控大豆开花期,同时还决定开花后期的主茎节数、豆荚发育及茎的生长习性[48]。而大豆结荚习性、主茎节数、茎粗、株高、节间距、叶片大小和分枝数等是与产量相关的重要农艺性状,表明E3和E4基因不仅影响大豆开花期,对产量形成也具有重要作用。
大豆结荚习性又叫大豆茎生长习性,是指大豆开花和结荚的方式,是大豆的一个重要生态性状,光周期是影响大豆顶端花序发育的重要因素[46,48-49]。Bernard等依据花期长短、茎生长状态、茎粗细变化、节间数、叶大小变化及茎顶端结荚情况等综合性状的表现将大豆品种划分为有限结荚习性、亚有限结荚习性和无限结荚习性3种类型[50]。大豆在低纬度地区种植,表现出植株矮小、开花期及成熟期提前,产量降低。这可能与低纬度地区的短日照有关,影响大豆顶端花序的发育[47]。目前大豆结荚习性控制基因已克隆了Dt1[51]和Dt2[52],分别控制无限结荚习性和亚有限结荚习性。结荚习性是重要的株型性状之一,是联系大豆开花期与产量性状的桥梁。
E3和E4基因下游的GmTOE4a转基因大豆株系表现出明显的延迟开花、株高降低、节间缩短、叶片变小及主茎增粗等表型,该基因对大豆开花期和多个株型性状均有调控作用。同时,研究还发现GmTOE4a基因与调控生长发育时相转换的miR156及其靶基因GmSPL之间存在着反馈调控关系[45,53]。GmTOE4a属于AP2(APETALA2)家族基因,具有转录因子活性,对开花期和株型性状的调控作用表明该基因是参与大豆多个生长发育过程的多效基因,而且是适应环境压力和提高产量很有潜质的基因,在一定程度上能够为大豆理想株型的构建提供分子元件。但目前GmTOE4a基因对株型性状调控的分子机制还不清楚,有可能与大豆生育期基因E3和E4的调控有关系,也有可能该基因处在多个调控网络的交叉点,见图1。随着测序技术的不断更新和生物信息学的迅猛发展,对光周期调控网络中关键基因的克隆及分子机制的揭示对大豆分子设计育种和产量的提高具有现实的指导意义,处于大数据时代背景下的大豆光周期研究将会有突破性的进展。
参考文献:
[1]Kobayashi Y,Weigel D.Move on up,it′s time for change-mobile signals controlling photoperiod-dependent flowering[J].Genes and Development,2007,21(19):2371-2384.
[2]Mouradov A,Cremer F,Coupland G.Control of flowering time: Interacting pathways as a basis for diversity[J].The Plant Cell,2002,14(S):111-130.
[3]Cerdán P D,Chory J.Regulation of flowering time by light quality[J].Nature,2003,423(6942):881-885.
[4]Hayama R,Coupland G.Shedding light on the circadian clock and the photoperiodic control of flowering[J].Current Opinion in Plant Biology,2003,6(1):13-19.
[5]Garner W W,Allard H A.Effect of the relative length of day and night and other factors of the environment on growth and reproduction in plants[J].Agric Res,1920,18(2):553-606.
[6]韩天富.不同生态类型大豆品种开花后光周期反应的研究[D].哈尔滨:东北农业大学,1994.
[7]Bernard R L.Two major genes for time of flowering and maturity in soybeans[J].Crop Science,1971,11(2):242-244.
[8]Buzzell R I.Inheritance of a soybean flowering response to fluorescent-daylength conditions[J].Canadian Journal of Genetics and Cytology,1971,13(4):703-707.
[9]Buzzell R I,Voldeng H D.Inheritance of insensitivity to long daylength[J].Soybean Genet Newsl,1980,7:26-29.
[10]McBlain B A,Bernard R L.A new gene affecting the time of flowering and maturity in soybeans[J].Journal of Heredity,1987,78(3):160-162.
[11]Bonato E R,Vello N A.E6,a dominant gene conditioning early flowering and maturity in soybeans[J].Genetics and Molecular Biology Science,1999,22(2):229-232.
[12]Cober E R,Voldeng H D.Low R:FR light quality delays flowering of E7E7 soybean lines[J].Crop Science,2001,41:1823-1826.
[13]Cober E R,Molnar S J,Charette M,et al.A new locus for early maturity in soybean[J].Crop Science,2010,50(2):524-527.
[14]Kong F J,Nan H Y,Cao D,et al.A new dominant geneE9 conditions early flowering and maturity in soybean[J].Crop Science.2014,54:2529-2535.
[15]Ray J D,Hinson K,Mankono J E B,et al.Genetic-control of a long-juvenile trait in soybean[J].Crop Science,1995,35(4):1001-1006.
[16]Cober E R,Thnner J W,Voldeng H D.Genetic control of photoperiod response in early-maturing,near isogenic soybean lines[J].Crop Science,1996,36(3):601-605.
[17]Cober E R,Tanner J W,Voldeng H D.Soybean photoperiod-sensitivity loci respond differentially to light quality[J].Crop Science,1996,36(3):606-610.
[18]Saindon G,Voldeng H D,Beversdorf W D,et al.Genetic control of long day length response in soybean[J].Crop Science,1989,29(6):1436-1439.
[19]Watanabe S,Hideshima R,Xia Z J,et al.Map-based cloning of the gene associated with the soybean maturity locusE3[J].Genetics,2009,182(4):1251-1262.
[20]Liu B H,Kanazawa A,Matsumura H,et al.Genetic redundancy in soybean photoresponses associated with duplication of the Phytochrome A gene[J].Genetics,2008,180(2):995-1007.
[21]Watanabe S,Xia Z J,Hideshima R,et al.A map-based cloning strategy employing a residual heterozygous line reveals that theGIGANTEAgene is involved in soybean maturity and flowering[J].Genetics,2011,188(2):395-407.
[22]Abe J,Xu D H,Miyano A,et al.Photoperiod-insensitive Japanese soybean landraces differ at two maturity loci[J].Crop Science,2003,43(4):1300-1304.
[23]Stewart D W,Cober E R,Bernard R L.Modeling genetic effects on the photothermal response of soybean phenological development[J].Agronomy Journal,2003,95(1):65-70.
[24]Yamanaka N,Ninomiya S,Hoshi M,et al.An informative linkage map of soybean reveals QTLs for flowering time,leaflet morphology and regions of segregation distortion[J].DNA Research,2001,8(2):61-72.
[25]Yamanaka N,Watanabe S,Toda K,et al.Fine mapping of the FT1locus for soybean flowering time using a residual heterozygous line derived from a recombinant inbred line[J].Theoretical and Applied Genetics,2005,110(4):634-639.
[26]Xia Z J,Watanabe S,Yamada T,et al.Positional cloning and characterization reveal the molecular basis for soybean maturity locusE1 that regulates photoperiodic flowering[J].Proceedings of the National Academy of Sciences,2012,109(32):E2155-E2164.
[27]Xu M,Yamagishi N,Zhao C,et al.The soybean-specific maturity geneE1 family of floral repressors controls niht-break responses through down-regulation ofFLOWERINGLOCUSTorthologs[J].Plant Physiology,2015,168(4):1735-1746.
[28]Zhao C,Takeshima R,Zhu J,et al.A recessive allele for delayed flowering atthe soybean maturity locusE9 is a leakyallele ofFT2a,aFLOWERINGLOCUSTortholog[J].BMC Plant Biology,2016,16:20.
[29]Millar A J,Straume M,Chory J,et al.The regulation of circadian period by phototransduction pathways inArabidopsis[J].Science,1995,267(5201):1163-1166.
[30]Shirley B W,Kubasek W L,Storz G,et al.Analysis ofArabidopsismutants deficient in flavonoid biosynthesis[J].The Plant Journal,1995,8(5):659-671.
[31]Putterill J,Robson F,Lee K,et al.TheCONSTANSgene of Arabidopsis promotes flowering and encodes a protein showing similarities to zinc finger transcription factors[J].Cell,1995,80(6):847-857.
[32]Suárez-López P,Wheatley K,Robson F,et al.CONSTANSmediates between the circadian clock and the control of flowering inArabidopsis[J].Nature,2001,410:1116-1120.
[33]Huang T,Böhlenius H,Eriksson S,et al.The mRNA of the Arabidopsis gene FT moves from leaf to shoot apex and induces flowering[J].Science,2005,309(5741):1694-1696.
[34]Jung J H,Seo Y H,Seo P J,et al.TheGIGANTEA-regulated microRNA172 mediates photoperiodic flowering independent ofCONSTANSinArabidopsis[J].Plant Cell,2007,19(9):2736-2748.
[35]Cao D,Li Y,Lu S J,et al.GmCOL1a and GmCOL1b function as flowering repressors in soybean under long-day conditions[J].Plant and Cell Physiology,2015,56(12):2409-2422.
[36]Kong F J,Liu B H,Xia Z J,et al.Two coordinately regulated homologs of FLOWERING LOCUS T are involved in the control of photoperiodic flowering in soybean[J].Plant Physiology,2010,154:1220-1231.
[37]Nan H Y,Cao D,Zhang D Y,et al.GmFT2a and GmFT5a redundantly and differentially regulate flowering through interaction with and upregulation of the bZIP transcription factor GmFDL19 in soybean[J].PLoS One,2014,9(5):e97669.
[38]Zhai H,Lü S X,Liang S,et al.GmFT4,a homolog ofFLOWERINGLOCUST,is positively regulated byE1 and Functions as a flowering repressor in soybean[J].PLoS One,2014,9 (2): e89030.
[39]Yano M,Katayose Y,Ashikari M,et al.Hd1,a major photoperiod sensitivity quantitative trait locus in rice,is closely related to the Arabidopsis flowering time geneCONSTANS[J].The Plant Cell,2000,12:2473-2483.
[40]Liu J Y,Yu J P,McIntosh L,et al.Isolation of aCONSTANSortholog fromPharbitisniland its role in flowering[J].Plant physiology,2001,125: 1821-1830.
[41]Hayama R,Agashe B,Luley E,et al.A circadian rhythm set by dusk determines the expression ofFThomologs and the short-day photoperiodic flowering response inPharbitis[J].The Plant Cell,2007,19(10): 2988-3000.
[42]Nemoto Y,Kisaka M,Fuse T,et al.Characterization and functional analysis of three wheat genes with homology to theCONSTANSflowering time gene in transgenic rice[J].The Plant Journal,2003,36(1): 82-93.
[43]Zhu Q H,Helliwell C.Regulation of flowering time and floral patterning by miR172[J].Journal of Experimental Botany,2011,62(2) :487-495.
[44]Cao D,Li Y,Wang J L,et al.GmmiR156b overexpression delays flowering time in soybean[J].Plant Molecular Biology,2015,89(4): 353-363.
[45]Zhao X H,Cao D,Huang Z J,et al.Dual functions of GmTOE4a in the regulation of photoperiod-mediated flowering and plant morphology in soybean[J].Plant Molecular Biology,2015,88: 343-355.
[46]韩天富,王金陵.大豆开花后光周期反应的研究.植物学报[J],1995,37(11): 863-869.
[47]Han T F,Wu C X,Tong Z X,et al.Post flowering photoperiod regulates vegetative growth and reproductive development of soybean[J].Environmental and Experimental Botany,2006,55(1-2):120-129.
[48]Xu M L,Xu Z H,Liu B H,et al.Genetic variation in four maturity genes affects photoperiod insensitivity and PHYA-regulated post-flowering responses of soybean[J].BMC Plant Biology,2013,13:91.
[49]Jiang Y,Wu C X,Zhang L X,et al.Long-day effects on the terminal inflorescence development of a photoperiod-sensitive soybean [Glycinemax(L.) Merr.]variety [J].Plant Science,2011,180(3): 504-510.
[50]Bernard R.Two genes affecting stem termination in soybeans [J].Crop Science,1972,12:235-239.
[51]Liu B H,Watanabe S,Uchiyama T,et al.The soybean stemgrowth habit geneDt1 is an ortholog of ArabidopsisTERMINALFLOWER1[J].Plant Physiology,2010,153(1):198-210.
[52]Ping J,Liu Y,Sun L,et al.Dt2 is a gain-of-function MADS-domain factor gene that specifies semideterminacy in soybean[J].The Plant Cell,2014,26(7):2831-2842.
[53]赵晓晖.miR172及其靶基因在大豆光周期调控开花中的功能研究[D].哈尔滨: 中国科学院东北地理与农业生态研究所,2015.
Research Progresses on Photoperiodic Flowering and Yield Traits in Soybean(GlycinemaxMerr.)
KONG Fanjiang,ZHAO Xiaohui,LIU Baohui
(KeyLaboratoryofSoybeanMolecularDesignBreeding,NortheastInstituteofGeographyandAgroecology,CAS,Harbin
150081,China)
Abstract:Soybean as an important industrial crop,which is a predominant plant source for human vegetable oil and protein.But now our country′s soybean industry faces severe challenges.Soybean photoperiod regulated flowering not only influences soybean adaptation but also affects soybean grain yield.In this review,we demonstrated that soybean contains a specific photoperiod regulated flowering pathway PhyA-E1-FT.We also elucidated the genetically regulation relationship between identified flowering genes in soybean.The clone of key genes in soybean photoperiod response will serve as a theoretical basis for flowering and maturity molecular mechanism,and will be of great importance in soybean molecular breeding and yield improvement.
Key words:soybean; photoperiod; maturity; flowering time; yield
中图分类号:S565.1
文献标识码:A
作者简介:第一作者及通讯孔凡江(1972-),男,黑龙江哈尔滨人,研究员,博士生导师,长期从事大豆光周期调控开花研究.
基金项目:国家自然科学基金(31430065,31371643,31571686).
收稿日期:2016-03-15.
文章编号:2095-2961(2016)02 -0065 -07
doi:10.11689/j.issn.2095-2961.2016.02.001
