Leu1p参与调节V-ATP酶的活性
2016-04-26高堂杰张震宇
高堂杰,张震宇
(江南大学糖化学与生物技术教育部重点实验室,江苏无锡,214122)
Leu1p参与调节V-ATP酶的活性
高堂杰,张震宇
(江南大学糖化学与生物技术教育部重点实验室,江苏无锡,214122)
摘要:RAVE(regulator of the H+-ATPase of the vacuolar and endosomal membrane)由Rav1p、Rav2p和Skp1p3个亚基组成,是V-ATP酶活性调节机制中一个关键的多亚基复合物调节因子; Leu1p能够与RAVE相互作用。通过融合PCR、同源重组分别构建了酿酒酵母BY4742的Rav1、Rav2、Leu1和Vma2缺陷型菌株。进一步研究重组菌株对pH及CaCl2的耐受能力发现:Leu1缺陷型菌株(3-异丙基苹果酸异构酶(Leu1p)缺失)的生长状况与Rav1、Rav2 缺陷型菌株的生长状况基本一致,表明RAVE对V-ATP酶的活性调节极有可能需要通过与Leu1p结合才能实现。笔者所在实验室为进一步研究RAVE与Leu1p的相互关系及其对V-ATP酶的活性调节提供了重要依据。
关键词:V-ATP 酶;RAVE;Leu1p;同源重组;融合PCR
V-ATP酶(vacuolar ATPase)又称液泡ATP酶,广泛存在于生物的细胞膜和细胞器膜中。它能够通过水解ATP产生的能量来转移质子,从而调节细胞内外的pH,使细胞处于一个相对稳定的pH环境[1];同时,V-ATP酶担负着调节细胞内部分离子浓度的功能,如Ca2+、Zn2+等[2-3]。V-ATP酶的质子泵效应与生物体内的许多生化反应及代谢活动息息相关,同时也与一系列疾病(如癌症、艾滋等)的产生与治疗有关[4-6]。目前,医学人员已越来越多地以V-ATP酶为目标来研发靶向性药物。V-ATP酶由V1和V0两部分组成。V1位于细胞质当中,其主要功能是水解ATP;V0镶嵌于细胞膜当中,负责转移质子。单独的V1、V0是没有活性的,只有当二者结合形成完整的V-ATP酶,才能发挥各自的功能,共同起到调节细胞内环境稳定的作用[7-9]。V-ATP酶的活性调节受诸多因素的影响,其调节机制目前尚未研究透彻,但文献[10-11]的研究表明,RAVE(regulator of the H+-ATPase of the vacuolar and endosomal membrane)在V-ATP酶的活性调节当中具有重要作用。RAVE由Ravlp、Rav2p和Skplp 3个亚基组成。当细胞内环境发生变化时,RAVE可以通过与V1的结合或分离来调节V1与V0的分离与结合,从而起到调节V-ATP酶活性的作用,RAVE的缺失会导致V-ATP酶的形成严重受阻,且形成的V-ATP酶活性也严重受到影响。
目前,关于RAVE研究最多的是Skplp,且已经得到其晶体结构[11]。但是对Ravlp、Rav2p的研究还相对较少。笔者所在实验室致力于RAVE复合物的研究,目前通过亲和纯化及质谱鉴定,确定了一系列能与RAVE产生相互作用的蛋白,其中3-异丙基苹果酸异构酶(Leu1p)与RAVE具有相互作用关系的发现尚属首次[12]。Leu1p在亮氨酸合成途径第一步反应中将α-异丙基苹果酸异构为β-异丙基苹果酸,为亮氨酸的合成提供前体[13]。为了进一步研究Leu1p与RAVE及V-ATP酶之间的关系,本实验通过构建重组菌株来研究Leu1p是否影响V-ATP酶的功能活性。
1材料与方法
1.1材料
1.1.1菌株与质粒
大肠杆菌JM109、质粒pRS425(带有Leu2基因序列)及pUCm-T载体,保存于笔者所在实验室。用到的酵母菌株如表1所示。

表1 本实验用到的酵母菌株
1.1.2试剂
PfuDNA聚合酶、Taq DNA聚合酶、dNTP混合物、十二烷基磺酸钠(SDS)、Tris、溴化乙锭(EB)、异丙基-β-D-硫代半乳糖苷(IPTG)、琼脂糖M、小量质粒提取试剂盒、DNA凝胶回收试剂盒、PCR产物纯化试剂盒、氨苄青霉素、卡那霉素(G418 硫酸盐)、1 kb DNA Marker,BBI公司;限制性内切酶BamH I、NdeI、XhoI,1 kb DNA Ladder Marker,λ-Hind III digest DNA Marker,TaKaRa公司;T4 DNA ligase,Fermentas公司;Lyticase酵母破壁酶,Sigma-Aldrich公司;其余试剂均购自国药集团化学试剂有限公司。
1.2方法
1.2.1酿酒酵母基因组DNA提取
挑取酿酒酵母BY4742Flag-R2单菌落于YPD液体培养基中,30 ℃、200 r/min过夜培养18~20 h。参照文献[14]的方法提取基因组DNA。
1.2.2目的基因的融合与克隆
酿酒酵母BY4742是Leu2缺陷型菌株,通过融合PCR(图1)在Rav1、Rav2和Vma2片段中插入Leu2基因的同时敲除原基因的部分片段,得到与基因组DNA具有同源互补序列的重组基因片段:R1F-R1LEU2-R1D、R2F-R2LEU2-R2D、V2F-V2LEU2-V2D(其中R1F、R2F和V2F分别为Rav1、Rav2和Vma2的上游同源序列;R1D、R2D和V2D分别为Rav1、Rav2和Vma2的下游同源序列)。融合后的目的片段转化酵母BY4742,经同源重组整合到其基因组当中,通过SD-Leu培养基筛选到重组菌株。
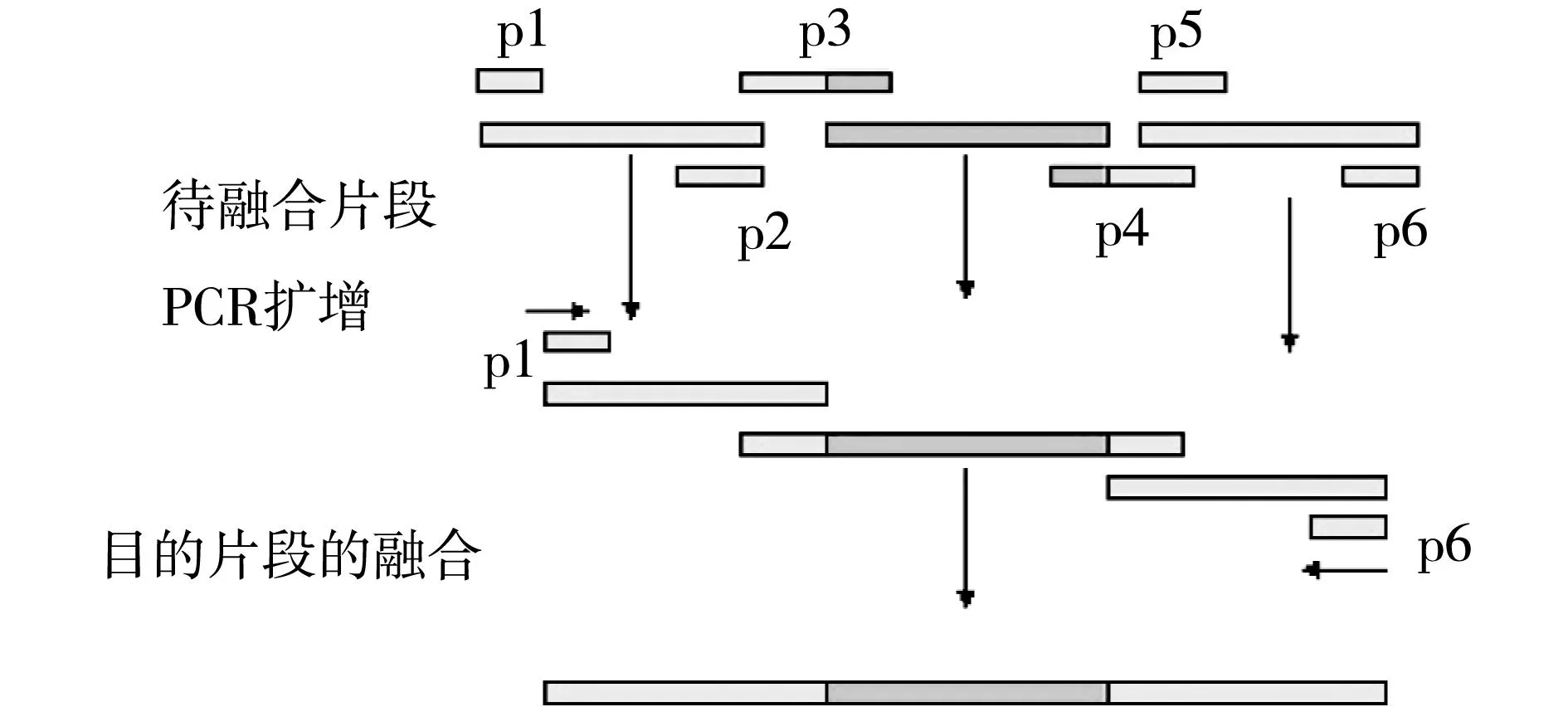
图1 融合PCR示意图Fig.1 Schematic diagram of fusion PCR
由于Leu1p与Leu2p同为亮氨酸合成途径当中的酶,因此在构建Leu1缺陷型菌株时不能以SD-Leu培养基进行筛选,故选取KanMX6作为抗性标记,以含G418的YPD固体培养基进行筛选。通过融合PCR及酶切连接等方法,在Leu1基因片段当中插入Leu2及KanMX6基因,同时敲除Leu1的部分核苷酸序列,得到与基因组DNA具有同源互补序列的重组片段:LEU1F-LEU2-KanMX6-LEU1D(其中LEU1F、LEU1D分别为Leu1的上游同源序列及下游同源序列)。重组片段转化酵母BY4742经同源重组整合到基因组当中,通过选择培养基进行筛选得到重组菌株。
以酿酒酵母BY4742F-R2基因组为模板,通过PCR扩增R1F、R1D、R2F、R2D、V2F、V2D、LEU1F、LEU1D及KanMX6,PCR引物对分别为R1p1/R1p2、R1p5/R1p6、R2p1/R2p2、R2p5/R2p6、Vp1/Vp2、Vp5/Vp6、Lp1/Lp2、Lp5/Lp6、Lp7/Lp8(表2)。PCR反应程序:94 ℃预变性4 min;然后98 ℃变性1 min,58 ℃退火30 s,72 ℃延伸1 min,30个循环;最后72 ℃延伸10 min。扩增产物通过PCR产物纯化试剂盒纯化回收。
以质粒pRS425为模板,R1p3/R1p4、R2p3/R2p4、Vp3/Vp4、Lp3/Lp4为引物,分别扩增Leu2。PCR反应程序:94 ℃预变性4 min;然后98 ℃变性1 min,58 ℃退火30 s,72 ℃延伸1 min 20 s,30个循环;最后72 ℃延伸10 min。扩增产物通过PCR产物纯化试剂盒纯化回收。
以上述PCR产物为模板,R1p1/R1p6、R2p1/R2p6、Vp1/Vp6为引物,先采用KOD Plus Neo通过融合PCR分别扩增R1F-LEU2-R1D、R2F-LEU2-R2D及V2F-V2LEU2-V2D;然后通过Taq DNA Polymerase在3′末端加A。PCR反应程序:94 ℃预变性2 min;98 ℃变性30 s,58 ℃退火30 s,68 ℃延伸1 min10 s,30个循环。最后加入1 μL Taq DNA Polymerase,72 ℃延伸30 min。扩增产物经琼脂糖凝胶电泳割胶后,用胶回收试剂盒回收。
纯化后的融合PCR产物,经T4 DNA Ligase作用连接到pUCm-T载体,通过蓝白斑进行筛选。挑选白色单菌落培养后,提取质粒,经双酶切及PCR验证重组载体,并选取二者验证皆正确的重组载体进行测序验证。
LEU1F-LEU2-KanMX6-LEU1D由于片段较长,很难通过融合PCR进行连接,故先通过融合PCR分别连接LEU1F/LEU2、KanMX6/LEU2D。然后通过酶切连接的方式依次将二者连接到载体pET28a上,再通过PCR扩增,得到全长的目的片段。
吕凌子极具耐心地听她发完牢骚,说,发票复印件我会催物业公司的陈主任明天给送过去,这会1402号业主也不知道在不在家,我马上去看看,要是在的话,麻烦你再派人来一趟。

表2 所使用的核苷酸序
注:1、2、3分别为NdeI、BamH I、XhoI酶切位点。
1.2.3酿酒酵母BY4742感受态细胞的制备
酿酒酵母BY4742感受态细胞的制备参照文献[12]。
1.2.4目的基因的转化及其筛选
参照文献[15]的方法将目的基因分别转化酿酒酵母BY4742,重组菌株通过SD-Leu培养基或含200 μg/mL G418的YPD培养基进行筛选。
挑取上述转化后长出的单菌落于10 mL SD-Leu培养基中,过夜培养18~20 h,提取基因组DNA,以其为模板进行PCR验证。
1.2.5酿酒酵母BY4742重组菌不同pH培养基中生长差异的研究
分别挑取酿酒酵母BY4742及各重组菌株单菌落接种于50 mL的YPD液体培养基中过夜培养16~18 h,用分光光度计测其吸光度OD600,取少量菌液稀释至相同OD值,按1%的接种量接种至100 mL的YPD液体培养基中(3个平行样),于30 ℃、200 r/min摇床培养,每隔2 h取样测其OD600,直至36 h,并测培养结束时的菌液pH,绘制生长曲线。
1.2.6重组菌株对CaCl2的耐受性研究
分别挑取酿酒酵母BY4742及各重组酿酒酵母单菌落接种于50 mL的YPD液体培养基中过夜培养16~18 h,用分光光度计测其OD600,取少量菌液稀释至相同OD后,按梯度1、10-1和10-2取相同量的菌液分别涂布于pH为5.5、7.5含100 mmol/L CaCl2的YPD固体培养基上(各3个平行),同时接种相同菌液至不含CaCl2的培养基作为对照,30 ℃恒温培养3~5 d。
2结果与讨论
2.1通过融合PCR获取重组基因
按照1.2.2节的方法分别扩增各待融合的DNA片段。各片段的理论大小如表3所示;PCR产物的电泳检测如图2所示。

表3 各基因片段的理论大小
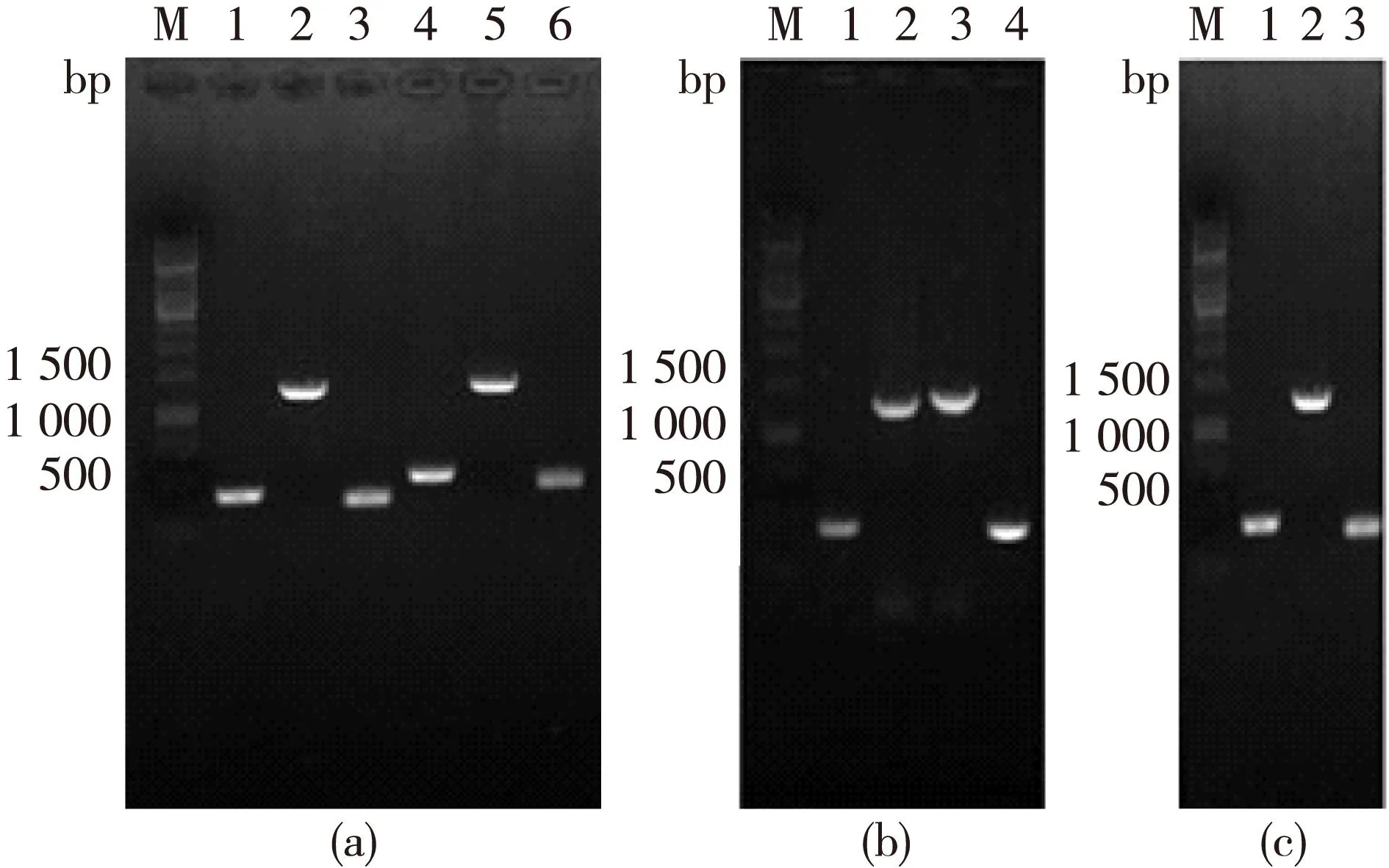
M均为1kb DNA Marker;(a):1~6分别为R2F、R2LEU2、R2D、R1F、R1LEU2、R1D;(b):1~4分别为LEU1F、LEU2、KanMX6、LEU1D;(c):1~3分别为V2F、LEU2、V2D图2 各待融合DNA片段PCR产物Fig.2 PCR products of the DNA fragment to be fused
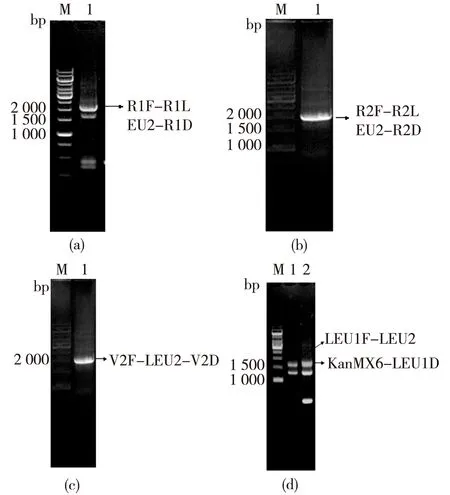
M均为1 kb DNA Marker;(a)1—R1F、R1LEU,2—R1D融合PCR产物R1F-R1LEU2-R1D;(b)1—R2F、R2LEU2、R2D融合PCR产物R2F-R2LEU2-R2D;(c)1—V2F、 LEU2、V2D融合PCR产物V2F-V2LEU2-V2D;(d)1、2分别为LEU1F与LEU2、KanMX6与LEU1D的融合PCR产物LEU1F-LEU2及KanMX6-LEU1D 图3 经融合PCR所得到的目的基因Fig.3 The target gene obtained by fusinon PCR
经融合PCR所得到的目的基因经纯化后连接到克隆载体进行克隆。其中R1F-R1LEU2-R1D、R2F-R2LEU2-R2D及V2F-V2LEU2-V2D经TA克隆连接到pUCm-T载体上,分别命名为pUCm-T-R1、pUCm-T-R2、pUCm-T-V2;LEU1F-LEU2及KanMX6-LEU1D,通过酶切连接的方式依次连接到pET 28a载体上,命名重组载体为pET28a-LEU1,从而可以通过PCR得到完整的目的片段:LEU1F-LEU2-KanMX6-LEU1D。连接好的重组载体分别转化大肠杆菌JM109感受态进行克隆。重组克隆载体经双酶切、PCR及测序验证均正确。
2.2目的基因转化酵母BY4742及其验证
以上述构建的重组载体为模板,通过PCR分别扩增各目的片段,经割胶回收纯化后,按1.2.4节的方法转化酵母BY4742 感受态细胞,筛选出目的菌株,提取基因组DNA进行PCR验证,同时以酵母BY4742的基因组DNA为模板,进行PCR对照,验证结果分别如图4所示。
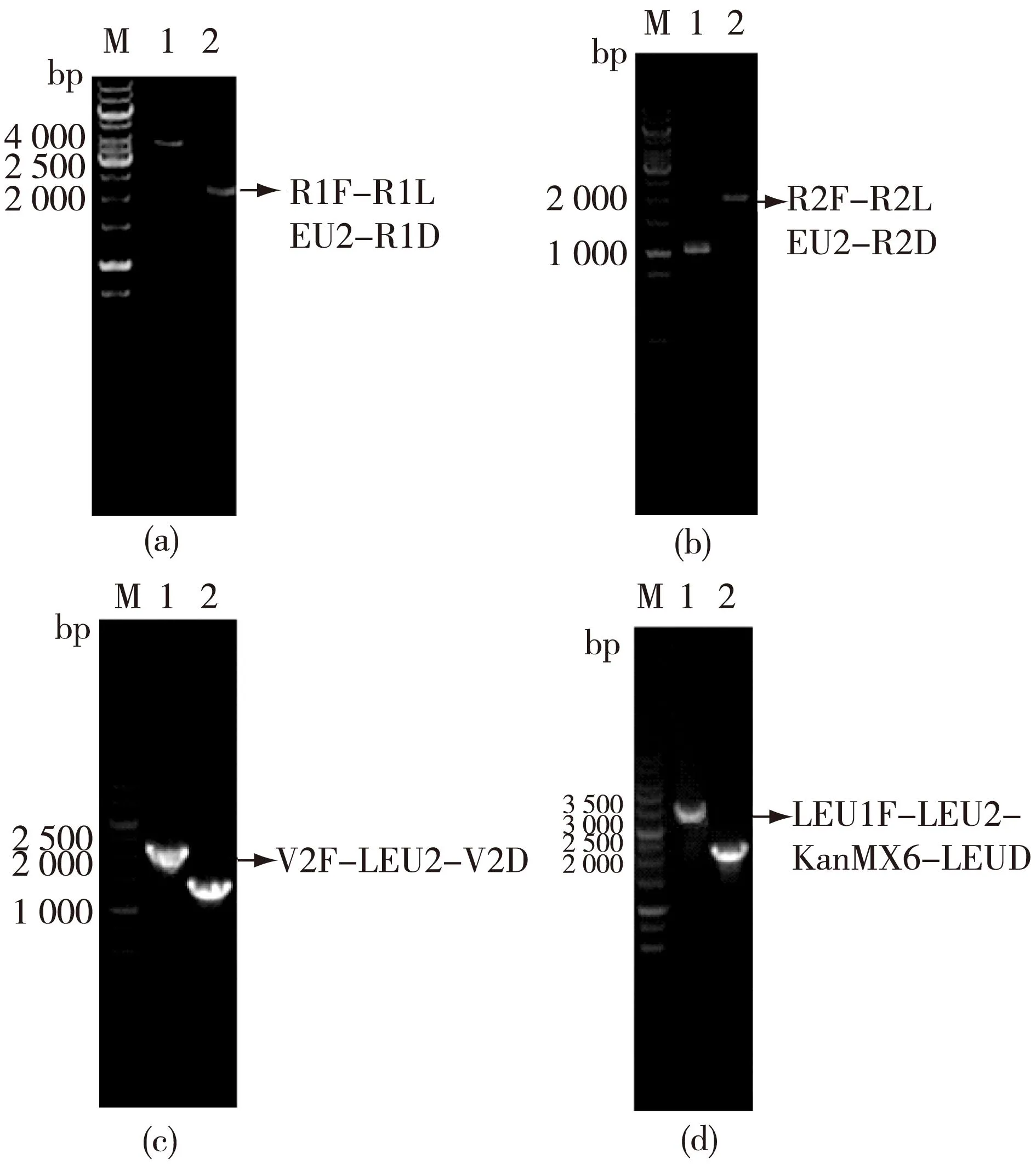
M均为1 kb DNA Marker;箭头所指条带分别为以各筛选到的重组菌株基因组为模板进行PCR所得片段;非箭头所指条带为采用相对应的引物并以BY4742基因组DNA为模板进行PCR所得片段图4 各重组菌株PCR验证Fig.4 PCR results of recombinant strain
2.3重组酵母菌株在不同pH条件下的生长状况
图5为重组酵母菌株在pH 5.5条件下的生长曲线。由图5可知:在pH为5.5的YPD液体培养基中,原始菌株及4株缺陷型重组菌(rav1Δ、rav2Δ、vma2Δ和leu1Δ)的生长曲线基本一致,在10 h左右进入对数生长期,15 h左右进入稳定期,且菌密度也基本一致。这是因为酿酒酵母的最适生长pH为5.5左右,所以五者的生长趋势基本一致。

图5 酵母BY4742及其各重组菌在pH为5.5的 YPD液体培养基中的生长曲线 Fig.5 Growth curves of the yeast BY4742 and the recombinant strain cultured in YPD mediu(pH=5.5)
图6为重组酵母菌株在pH 7.5条件下的生长曲线。由图6可知:在pH为7.5的培养基中,原始菌株及4株缺陷型重组菌(rav1Δ、rav2Δ、vma2Δ 和leu1Δ)均在13 h左右开始进入对数生长期,但是原菌BY4742生长迅速,较快达到稳定期,而缺陷型重组菌生长缓慢,直到25 h左右才达到稳定生长期,且菌密度相较于原始菌要低,尤以Vma2缺陷型菌株生长最为缓慢,其稳定期的菌密度最低,OD600只达到3.8左右。
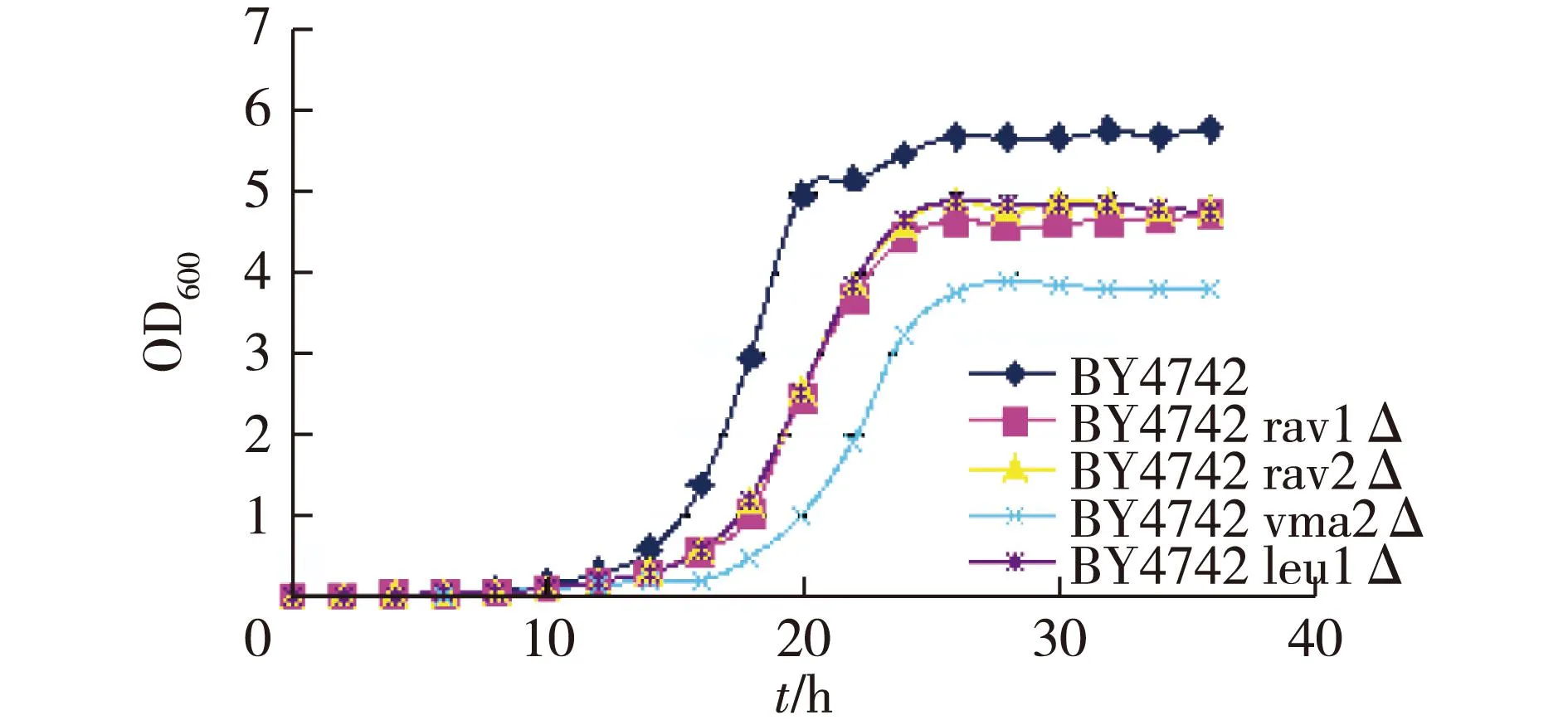
图6 酵母BY4742及其各重组菌在pH为7.5的 YPD液体培养基中的生长曲线Fig.6 Growth curves of the yeast BY4742 and the recombinant strain cultured in YPD mediu(pH=7.5)
同时,在培养结束时测得的pH显示,在pH 5.5的培养基中,最终的pH变化不大,均在5.3左右;在pH 7.5的培养基中,原始菌株及4株缺陷型重组菌(rav1Δ、rav2Δ、vma2Δ和leu1Δ)的pH分别为5.6、6.4、6.2、6.9和6.5。由此可见,Rav1、Rav2、Vma2、Leu1的敲除使得酵母菌对pH的调节受到了严重阻碍,从而影响其生长。同时,菌株生长及培养基pH的结果显示Leu1的敲除所产生的影响与Rav1、Rav2的敲除所产生的影响基本一致,而与Vma2的敲除有较大差别,这从一定程度上说明Leu1p通过与RAVE结合,参与对V-ATP酶的活性调节,因为Vma2的缺失导致V-ATP酶完全不能形成;而RAVE的缺失则只是使其组装严重受阻,且活性下降。
2.4重组酵母菌株对CaCl2的耐受性

图7 各重组菌株对CaCl2及pH的耐受性Fig.7 Tolerance of recombinant strains to CaCl2 and pH
按照1.2.6节的方法将各菌液分别涂布于含100 mmol/L CaCl2、pH 5.5及7.5的YPD培养基上,30 ℃恒温培养3~5 d,其结果如图7所示。由图7可知:在含100 mmol/L CaCl2、pH为5.5的YPD固体培养基中,酿酒酵母BY4742 的Rav1、Rav2、Vma2及Leu1的缺陷型菌株的生长受到了极大抑制,相同菌量的涂布,其菌落数目要远远小于酿酒酵母BY4742,而四者之中,又以Vma2缺陷型菌株的生长状况最差,这可能是由于缺少Vma2p,导致V-ATP酶的V1部分无法形成,不能组装成为完整的V-ATP酶,从而失去了对CaCl2及pH的调控作用。由图2可知,Leu1缺陷型菌株的生长状况与Rav1、Rav2 缺陷型菌株的生长状况基本一致,这从一个方面说明,Leu1p通过与RAVE结合,共同调节V-ATP酶的分离与集装,从而起到调控其活性的作用,不过Leu1p也有可能通过其他途径影响菌株对CaCl2及pH的耐受性,有待进一步的实验验证。V-ATP酶的活性调节机制相当复杂。普遍认为RAVE复合物在其调节机制中扮演重要角色,但具体的调节机制尚不清楚。RAVE复合物主要由Rav1p、Rav2p和Skp1p构成,与V-ATP酶的众多亚基具有相互作用关系;同时由于Skplp是组成SCF复合物的一个关键骨架蛋白,还能与其他众多蛋白产生作用关系,故其调节机制可能还要受到其他蛋白的影响。
3结论
Leu1p是笔者所在实验室最新发现的能与RAVE相互结合的蛋白。笔者通过融合PCR及同源重组的方法分别构建了酿酒酵母BY4742的Rav1、Rav2、Vma2和Leu1的缺陷型菌株,研究Leu1p与RAVE之间的相互功能关系。上述结论表明Leu1p对酿酒酵母BY4742的pH及CaCl2耐受性产生极大影响,且通过与重组菌株(rav1Δ、rav2Δ、vma2Δ)的对照实验发现,RAVE对V-ATP酶活性的调节极有可能需要通过与Leu1p结合才能得以实现。
V-ATP酶在生物体内的重要作用正越来越多地受到人们的关注,随着研究的深入,解开V-ATP酶的活性调节机制,使其服务于医疗卫生已是新的方向和热点。本研究的发现为进一步研究RAVE与Leu1p的相互关系及其对V-ATP酶的活性调节提供了重要依据。
参考文献:
[1]FORGAC M.Vacuolar ATPase:rotary proton pumps in physiology and patbophysiology[J].Nat Rev Mol Cell Biol,2007,8(11):917-929.
[2]LIN C W,TSAI F J,WAN L,et al.Binding interaction of SARS coronavirus 3CL(pro)protease with vacuolar H+-ATPase G1 subunit[J].FEBS Lett,2005,579(27):6089-6094.
[3]OTERO-REY E M,SOMOZA-MARTIN M,BARROS-ANGUEIRA F,et al.Intracellular pH regulation in oral squamous cell carcinoma is mediated by increased V-ATPase activity via over expression of the ATP6VIC1 gene[J].Oral Oncol,2008,44(2):193-199.
[4]GRAHAM L A,FLANNERY A R,STEVENS T H.Structure and assembly of the yeast V-ATPase[J].J Bioenerg Biomembr,2003,35(4):301-312.
[5]INOUE T,WILKENS S,FORGAC M.Subunit structure,function,and arrangement in the yeast and coated vesicle V-ATPase[J].J Bioenerg Biomembr,2003,35(4):291-299.
[6]JEFFERIES K C,CIPRIANO D J,Forgac M.Function,structure and regulation of the vacuolar(H)-ATPases[J].Arch Biochem Biophys,2008,476(1):33-42.
[7]DAWSON K,TOONE W M,JONES N,et al.Loss of regulators of vacuolar ATPase function and ceramide synthesis results in multidrug sensitivity inSchizosaccharomycespombe[J].Eukaryot Cell,2008,7(6):926-937.
[8]CIPRIANO D J,WANG Y,BOND S,et al.Structure and regulation of the vacuolar ATPases[J].Biochim Biophys Acta,2008,1777(7/8):599-604.
[9]ZHANG Z,ZHENG Y,MAZON H,et al.Structure of the yeast vacuolar ATPase[J].J Biol Chem,2008,283(51):35983-35985.
[10]SMARDON A M,KANE P M.RAVE is essential for the efficient assembly of the C subunit with the vacuolar H+-ATPase[J].J Biol Chem,2007,282(36):26185-26194.
[11]SEOL J H,SHEVCHENKO A,SHEVCHENKO A,et al.Skp1 forms multiple protein complexes,including RAVE,a regulator of V-ATPase assembly[J].Nature Cell Biol,2001,3(4):384-391.
[12]顾春银,张震宇.亲和纯化酿酒酵母RAVE复合物[EB/OL].[2013-12-29].[2013-01-16].http://www.paper.edu.cn/html/releasepaper/2013/01/732/.
[13]KOHLHAW G B.Leucine biosynthesis in fungi:entering metabolism through the back door[J].Microbiol Mol Biol Rev,2003,67(1):1-15.
[14]CRYER D R,ECCLESHALL R,MARMUR J.Isolation of yeast DNA[J].Methods Cell Biol,1975,12:39-44.
[15]ITO H,FUKUDA Y,MURATA K,et al.Transformation of intact yeast cells treated with alkali cations[J].J Bacteriol,1983,153(1):163-168.
(责任编辑荀志金)
Leu1p involved in regulating V-ATPase activity
GAO Tangjie,ZHANG Zhenyu
(Key Laboratory of Carbohydrate Chemistry & Biotechnology of the Ministry of Education,Jiangnan University,Wuxi 214122,China)
Abstract:RAVE(regulator of the H+-ATPase of the vacuolar and endosomal membrane)complex containing Rav1p,Rav2p and Skp1p,is a key regulatory factor of V-ATPase activity regulating mechanism. Latest news showed Leu1p capable of interacting with RAVE. This study,we constructed 4 Saccharomyces cerevisiae BY4742 recombinant strains that were lack of Rav1,Rav2,Vma2 and Leu1 respectively by fusion PCR and homologous recombination. RAVE for V-ATPase activity regulation is likely to be achieved by conjunction with Leu1p. Our study provided an important basis for studying the relationship between RAVE and Leu1p,also the study of regulation mechanism of the V-ATPase.
Keywords:V-ATPase;RAVE;Leu1p;homologous recombination;fusion PCR
中图分类号:Q936
文献标志码:A
文章编号:1672-3678(2016)02-0051-07
作者简介:高堂杰(1987—),男,湖南浏阳人,研究方向:生物化学、发酵工程,;张震宇(联系人),教授,E-mail:zhangzy@jiangnan.edu.cn
基金项目:国家自然科学基金(30800018、30970058)
收稿日期:2014-01-02
doi:10.3969/j.issn.1672-3678.2016.02.010
