滚环扩增技术最新研究动态及展望
2016-04-21孙亚军
孙亚军, 王 亮, 蔡 俊
湖北工业大学工程技术学院, 发酵工程教育部重点实验室, 武汉 430068
滚环扩增技术最新研究动态及展望
孙亚军,王亮,蔡俊*
湖北工业大学工程技术学院, 发酵工程教育部重点实验室, 武汉 430068
摘要:滚环扩增技术(rolling circle amplification,RCA)的建立模拟了自然界中环状病原生物DNA通过滚环模型方式自我复制的原理,经长期科学研究和实践应用,取得了诸多突破性成果。对最近几年在滚环扩增技术研究领域的最新动态进行了较全面的总结,其中包括了网状RCA、锁式探针RCA、目标成环RCA和跨越式RCA,也对滚环扩增中存在的问题进行了探讨,重点介绍了该技术在基础研究、实际检测、医疗诊断及纳米材料等方面的应用,最后对核酸等温扩增技术产业化的发展前景进行了展望。
关键词:RCA;纳米材料;转基因;锁式探针
建立于20世纪90年代中期的滚环扩增技术(rolling circle amplification,RCA),模拟了自然界中环状病原生物DNA通过滚环模型方式进行自我复制的原理,在研究初期就得到了世界范围内科研人员的高度关注。经过不断研究和改善,现已成为了包括DNA链置换扩增(SDA)、环介导的等温扩增(LAMP)和依赖解旋酶的恒温基因扩增(HDA)等核酸恒温扩增技术中的重要一员。与传统的核酸扩增技术相比,RCA技术摆脱了对精良仪器的依赖和进行反复热变性等缺点的局限,且有较高的灵敏度和特异性,反应时间也相对缩短,使得以此项技术为基础的核酸扩增检测技术能更有效的投入到普遍和成规模的实际应用中。通过对目前国内外在滚环扩增技术中的研究及应用调查发现,相较国外,我国的RCA应用研究还是有些不足,因此本文对最近几年国内外在滚环扩增技术研究领域的最新动态进行了较全面的总结,对滚环中存在的问题进行了探讨,介绍了该技术在基础研究、实际检测、医疗诊断及纳米材料等方面的应用[1,2],以期为我国科学研究中RCA技术的广泛应用提供参考。
1RCA技术
最早对RCA技术的研究包括了线性RCA(即单引物RCA,又叫LRCA)、指数RCA(即双引物RCA,也被称为超分支滚环扩增,HRCA)、多引物RCA和免疫RCA等[3,4]。RCA技术操作的基本原理是在DNA聚合酶作用下,引物沿着环状模板链延伸和扩增,产物为与单环模板序列完全相同的线性链,但长度却扩大了数千倍[5]。随着RCA技术的发展,研究者在这几种比较基础的RCA技术上不断改善、创新,开发了一批更加高效和实用的RCA技术,其中就包括了如网状RCA、锁式探针RCA、目标成环RCA和跨越式RCA在内的一系列RCA技术。
1.1网状RCA
在LRCA和HRCA的基础上建立的网状RCA(net rolling circle amplification,NRCA)是引入了切刻内切酶的一种更高效率的核酸扩增技术,切刻内切酶可识别双链中特定的核苷酸序列,并只切割双链中的一条单链。其在LRCA阶段的产物上设计了一个切刻内切酶的酶切位点,切割下的单链与环状DNA模板互补并充当引物,从而继续诱发LRCA和HRCA反应,使其在LRCA和HRCA基础上实现了进一步的扩增,扩增产物的原子力显微表征显示为结构较为单一的网状结构。如果把LRCA产物视为树干,那么HRCA就是具有众多枝桠的一棵完整大树,NRCA则是发展为由无数棵大树组成的茂密森林(图1)[6]。与LRCA和HRCA相比,该技术一方面没有牺牲原有技术在操作性、使用成本和扩增所需时间等方面的优势,另一方面其在原有技术基础上进一步实现了信号放大,从而为更低丰度核酸样本的分析检测提供了良好的技术条件[6]。
1.2锁式探针RCA
经特异性生成环状DNA,HRCA和LRCA都可用于线性DNA的扩增,即只有目标DNA存在的条件下,另一条线状DNA才可成环,且只有成环的线状DNA才可以进行复制和扩增,为了进一步保证此反应的特异性,可采用双切口滚环扩增[7]。线状探针DNA的长度只有几十个碱基,两端能与目标DNA一段连续的序列相结合,在探针两端序列之间留一段缺口,通过在缺口处设计既能发挥探针功能又能行使引物作用的一段序列,且当另一部分探针和设计的引物序列都和模板完全互补配对后,两个切口才能被连接酶特异性连接成环,也只有这种由两部分探针形成的环状模板才能在两条引物作用下扩增。这个方法类似于将线状DNA分子特异性地锁定在目标DNA上,可以很好的应用在单核苷酸特异性基因诊断的研究中[8]。
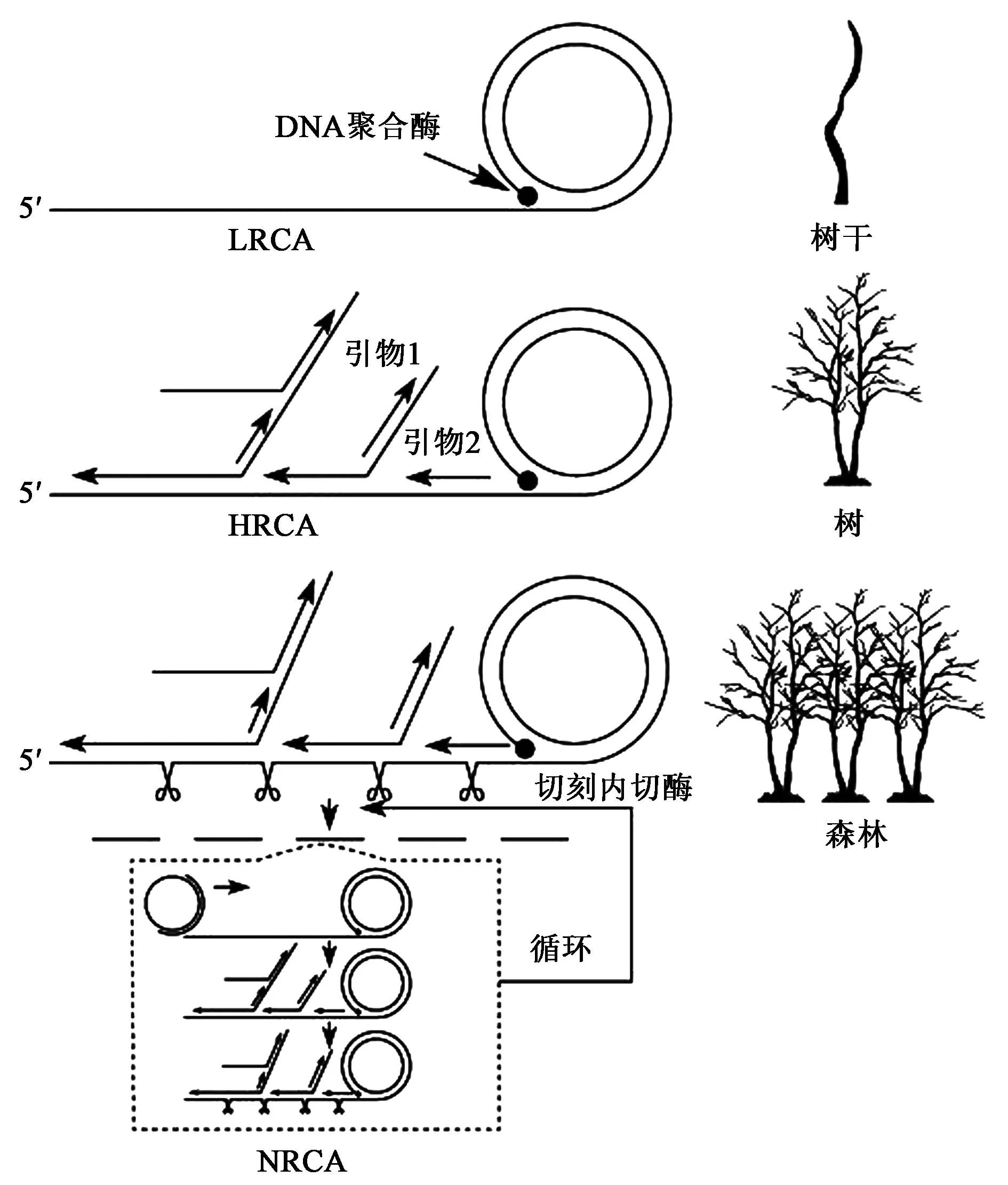
图1 LRCA、HRCA和NRCA对比示意图[6]Fig.1 The LRCA、HRCA and NRCA contrasts in diagram[6].
1.3目标成环RCA
目标成环RCA(target circle RCA,TC-RCA)克服了锁式探针RCA的单链探针不稳定、探针与模板容易错配、扩增产物并非目标序列而是互补探针自身等缺点。用限制性内切酶酶切样品,产生具有9nt粘性末端的序列,其次,设计与目标序列粘性末端完全互补配对的双链接头,经连接酶的作用,同目标序列连接成环,然后再利用具有链置换活性的DNA聚合酶,在两条引物的作用下引发HRCA,最后,扩增产物再经过限制性内切酶酶切,得到扩增后的目的片段。TC-RCA能在无背景信号的条件下扩增目标DNA片段,同时接头能稳定结合于固相磁珠表面而不影响扩增,因此能在芯片上实现目标成环等温扩增。在此基础上,利用高特异性的DNA连接酶、生物素修饰接头、链霉亲和素磁珠以及磁性分离等手段,构建了磁珠辅助的TC-RCA技术。该项技术能够在等温条件下从大量背景核酸中检测出目标序列,从根本上解决了检测特异性和灵敏度之间的矛盾关系,在检测食品中的有害微生物方面十分适用[9]。
1.4跨越式RCA
通过具有链置换活性及热稳定性的Bst DNA聚合酶引入RCA反应所提出的跨越式滚环等温扩增(stride RCA,SRCA),对RCA反应中所产生的背景信号机理提供了线索,其原理和过程与早期指数RCA和多引物RCA类似,但由于Bst DNA聚合酶具有在聚合过程中不受DNA缺口影响的特殊活性,当核酸合成过程中遇到复制模板缺空后可跨越并继续合成延伸,因此,SRCA能够以非闭合环形DNA为模板启动扩增反应。Bst DNA聚合酶最开始以上游引物为模板合成其互补链形成双链DNA后,该酶发挥核苷酸转移酶活性在其引物的3′末端沿5′→3′方向随机掺入脱氧核糖核苷酸聚合形成寡聚核苷酸序列,即DNA的合成反应跨越了互补链与下游引物间的缺口,以下游引物为模板继续互补配对;然后Bst DNA聚合酶继续将脱氧核糖核苷酸随机添加到下游互补序列的3′末端后形成寡聚核苷酸序列,继续催化聚合反应合成互补新链,完成一轮合成;最后,通过其链置换酶活力替换上一轮引物,往复进行滚环扩增,从而形成重复扩增产物。通过该特性设计的非闭合模板,巧妙设计引物,避免了引物之间的扩增,该技术能很好的驱动RCA反应,为基于RCA技术的检测方法及开发提供了新的理论依据和借鉴意义,也对开发等温扩增技术检测微生物有一定帮助[10]。
2RCA技术的应用
RCA技术在基础研究、实际检测、医疗诊断及纳米材料等方面都有很好的应用,结合细胞原位检测、单核苷酸多态性(SNPs)、蛋白质分析、免疫芯片和全基因组的扩增等研究都能发挥其巨大的优势[11~14],而且弥补了相应技术中存在的某些不足,例如在细胞原位检测中避免了传统原位PCR技术扩增产物会扩散出细胞的问题,且恒温扩增特性保证了组织形态学特征的完整性[15];在单核苷酸多态性检测中可以在合适的反应环境和探针设计条件下同步检测多组等位基因[16],甚至还实现了对极低丰度基因组DNA样本的检测;近来研究显示RCA技术对miRNA的检测也非常灵敏和高效[17,18]。当前RCA技术越来越多的应用到更多的领域,充分证明了其重要的价值。
2.1蛋白质分析免疫芯片检测
对于一些疾病的检测诊断和定向对药物药靶的筛选已通过免疫芯片技术得到了部分优化,但对于进行高通量及高灵敏性的检测是传统信号扩增检测力所不及的,基于RCA技术的免疫芯片检测不仅克服了这一缺陷,而且使芯片靶点间的空间独立性和抗原抗体结合体的整体性得到了保证[19,20],基于两者技术的交叉结合对于实现如数百个目标分子同时检测这样的成规模生产大有裨益,还可以将微阵列技术与免疫RCA结合以实现多元高通量并行分析蛋白质。
Miller等[21]制成能检测前列腺癌患者及健康人血清中蛋白的186中抗体芯片,对患者与健康人中表达差异蛋白成功进行了筛选。Zhou等[22]用不同标记素双色RCA芯片对血清蛋白质进行了相对水平的检测,验证了其信号强度相对于间接和直接标记检测法大幅提升,比诸如HLISA法等方法的检测过程中具有更高的重复性与准确性。近年来,免疫RCA技术与纳米材料相结合还可用于蛋白质的高灵敏度甚至超高灵敏度检测[23]。Ou等[24]通过DNA封装脂质体发展的免疫RCA多功能分析平台对于超灵敏检测蛋白质十分实用。Cheng等[25]也提出了一种高灵敏性的级联信号放大蛋白质分子检测策略,该策略融合了RCA、功能化量子点、生物素-亲核素系统和电化学溶出伏安检测技术。
2.2RCA全基因组扩增
对于分子生物学的研究,核酸的扩增无疑是最重要的手段之一,在对较大的环状DNA模板的研究中,例如噬菌体DNA、叶绿素DNA和质粒等[26],采取指数RCA技术对模板的扩增能达到均一和全面的效果[27]。RCA技术也拓展应用到了全基因组的扩增(WGA),有研究报道phi29 DNA聚合酶和核酸酶抗性引物在30℃温育6 h条件下,应用RCA技术能够从少至10个拷贝的基因组中扩增DNA至20~30 μg, 产物的平均长度大于10 kb。因此,采用RCA技术可以实现利用全血样品扩增人类基因组DNA的目标[28]。
Wang等[29]开发了一种全新的限制性环化RCA (RCA-RCA),这是一种改进的全基因组扩增技术,可针对于出现了明显降解和交联的DNA样品。此方法用限制性内切酶将DNA降解成短片段后环化成DNA环状单链,最后进行HRCA。Niel等[30]利用RCA可以由多条引物引发的特点,从人血浆样品中对细环病毒(Torquetenovirus)的全基因组进行扩增,由于四条引物同时使用,使得所有成环扩增子扩增几率更加均等,每个片段都可以扩增到30 kb以上,样品均一,覆盖率在90%以上。
2.3转基因检测及食品安全
转基因植物及其产品正不断涌入人们的生活,引起了世界范围对于食品安全性的广泛关注,因此,对于转基因植物检测技术的需求显得迫切重要。RCA技术用于植物转基因成分的检测相比传统的PCR方法检测和ELISA等方法具有更加方便、高效的优点。陶震等[31]通过效仿复合式PCR原理采用复合式HRCA方法成功的对转基因烟草进行了检测,并取得了理想的结果。通过食物中提取转基因模板,锁式探针再与其特异性杂交成环,之后进行RCA反应扩增[32],能够应用于进出口食品中转基因成分的快速而高效检测中。食品中卵清蛋白的检测也可基于抗体抗原结合的特异性,利用RCA技术来进行扩增[33]。还有研究者把食物中重金属铅的检测结合RCA,利用Aptamer特异性结合Pb2+后,判断目标物的存在与否[34、35]。张健[36]利用RCA技术对食源性致病微生物进行了特异性检测,例如副溶血性弧菌、单核增生李斯特氏菌等,RCA检测体系还可以研制成试剂盒以更方便的进行快速、高灵敏性及准确性的检测[37]。
2.4RCA在纳米技术中的应用
2.4.1DNA纳米结构合成RCA可用于合成特定的DNA纳米结构,在体外模拟复杂的DNA复制。建立人工DNA纳米材料已成为研究热点,而RCA重复串联产物序列是能够通过编码其环状模板来实施精确调控的[38],因此,基于这点特性可与人工合成DNA纳米结构技术相结合。现已成功把RCA技术用于与端粒酶六聚体核苷酸序列互补的一个环状模板复制,来人工合成及制备人端粒酶,对于人工合成端粒酶在活体研究中的意义重大。Lin等[39]利用RCA技术成功复制出平行互换DNA分子(paranemic crossover DNA, PX DNA),建立了人工DNA纳米结构,表明RCA能够作为复制其他复杂DNA纳米结构的有效工具,为将来核酸药物的发展提供了方法。
利用具有链置换作用及高持续合成力的Phi 29 DNA聚合酶在RCA进程中不会受到模板自身二级结构约束的性质,如双链、发卡、结头等这些在功能化核酸和DNA纳米技术中的关键部分二级结构可通过RCA技术合成。研究者们把基于RCA扩增的放大合成用于DNA四通接头结构上,提出了用于大尺寸树枝状DNA接头制备的新策略。在对交联度较高的复杂DNA纳米结构RCA反应放大后,对胞内病毒DNA克隆提供了一个新的可能途径。Nangreave等[40]通过RCA实现了经单链DNA分子构建的DNA四面体-三维纳米结构的放大合成。
2.4.2构建周期性纳米组装结构因RCA反应所具有的在扩增反应中通过聚合酶聚合核苷酸结合到引物末端而不断延伸扩增的特性,通过简便的环状序列长度的调节,就能精确地实现对组装单元间距的调节与控制,所以对于一维周期性纳米组装结构的构建,RCA产物所特有的重复串联结构就理想的成为其模板。
Deng等[41]和Beyer等[42]以重复串联核酸序列为模板,通过对有互补序列金纳米颗粒的杂交修饰,成功构建了有序的周期性金纳米颗粒组装结构。Zhao等[43]利用巯基修饰RCA过程中的引物,修饰成功的引物与金纳米颗粒进行组装后进行RCA反应,扩增得到的重复串联单链DNA作为模板就可用于DNA修饰的小尺寸纳米颗粒的组装,这是基于RCA技术构建的一种新型的三维周期性纳米组装支架。Cheglakov等[44]和Winner等[45]以RCA产物分别作为支架及拉链状结构成功构建了三维DNA纳米结构和可控尺寸的DNA纳米管,还通过RCA反应对于核酸适体的放大特性构建了周期性的蛋白质和纳米颗粒的复合纳米结构。当延着RCA产物长链连续性组装的四边形或六边形基本单元所合成的二维片层达到一定尺度后就会发生卷曲变性,这种变性导致了具有一定尺寸的DNA纳米管的形成。Hamblin等[46]也将RCA反应的重复串联DNA大片段作骨架很好的运用在了三棱柱DNA纳米结构的构建上,经一系列构建的产物深度分析后发现这种结构对于核酸酶的降解抵抗力和结构稳定性大幅提升,还可有效的被癌细胞摄取。
3RCA技术存在的问题和解决策略
3.1锁式探针连接效率
核酸链结构、离子强度和杂交温度等都是可制约及干扰核酸分子杂交的因素,在RCA反应中的锁状探针的杂交及其连接的效率也是一个关键性的制约因素,只有确保了探针分子的杂交及高效的连接才会有高灵敏度和特异性的检测效果[47]。王星宇[9]设计的目标成环RCA(TC-RCA)提高了探针与模板配对的正确率和稳定性。Szemes等[48]的研究发现,热循环法对于核酸分子的杂交有很高的效率,通过此项技术辅助锁状探针的杂交,保证了高效率的杂交后对马铃薯晚疫病原的成功分析。除此之外,不同的连接酶所要求的连接条件及连接效率有着较大的差异,包括锁式探针的小二级结构等也影响着反应的进程。根据相关报道,NaCl 浓度干扰着T4 DNA连接酶的保真性,当浓度处于50~150 mmol/L之间时,此酶的保真性会随着NaCl浓度的升高而不断增加。因此,必须要按照不同的实验设计来调控合适的连接条件及连接酶种类的选择,这样才能提高连接酶的连接效率及保真性,从而加强检测的灵敏性。
3.2背景信号干扰
RCA扩增反应的背景信号干扰问题一直以来都是严重干扰检测效率的一个棘手问题,背景信号的来源广泛,既有连接反应中非特异性连接经过多个循环扩增后产生的较强信号,又有未经成环的锁式探针等的干扰[49],因此,如何解决背景信号的干扰显得至关重要。孟兆祥等[10]利用热稳定的Bst DNA聚合酶驱动跨越式滚环扩增反应为RCA反应中的背景信号干扰问题提供了线索,对于背景信号干扰的降低或消除,目前有几种主要方法:①通过无模板的阴性对照实验来确定背景信号的来源,经不同的荧光染料在RCA反应与芯片分析中作标记后进行双重荧光芯片分析,能够极大限度的提升对背景信号和阳性信号区分的准确性;②筛选及弃除非特异性模板和未成环的锁式探针,可通过利用固体支持物上固定的生物素和抗生物素蛋白捕获探针和纯化探针来进行特异选择,但这也增大了RCA技术的繁琐性及成本;③对未结合探针的模板和线性探针采用核酸外切酶降解,以克服未成环探针的背景信号对信号检测的干扰。
3.3拓扑结构的制约
研究显示当DNA单链分子通过互补配对与锁状探针杂交后会导致与拓扑结构相类似的区域结构形成,一旦产生这种结构就会严重干扰聚合酶对锁状探针的扩增[50],所以基于锁状探针的RCA扩增分子检测手段的可行性饱受研究学者们的质疑。通过Beyer等[51]研究设计的一种特殊的锁式探针(earring probe)在与DNA分子退火杂交后,可自发形成特定的拓扑结构,再通过具有特殊链置换功能的Bst DNA聚合酶扩增杂交后的环状锁式探针,从而获得RCA产物,研究结果表明即使不对模板进行变性处理,RCA反应也会正常发生,从而很好的解决了所质疑的问题。解决了拓扑结构对扩增反应的制约,为RCA技术在癌基因的检测、基因分型、单核苷酸多态性等广泛的核酸检测领域夯实了理论基础。
4展望
生物技术产业作为21世纪最有潜力的高新技术产业,是继信息产业后的又一个新兴经济技术增长点,在社会和经济可持续发展中具有战略性和先导性。RCA作为生物技术中的一名重要成员,因其具有独特的等温扩增条件,克服了PCR反应需要反复温度变化的循环过程,其优势也使得它对仪器的要求大大简化,反应时间大大缩短,检测灵敏度高,检测特异性强,同时也易于同其他技术联合,实现高通量与自动化检测,因此具有巨大的商业价值和广阔的市场潜力。但同时国内的RCA发展也面临诸如研究资金来源单一、创新性不足、成果转化较慢等一系列问题。我国的科研人员必须抓住挑战和机遇,才能促使我国核酸等温扩增技术的产业化得到进一步发展。
参考文献
[1]Ali M M, Li F, Zhang Z,etal.. Rolling circle amplification: a versatile tool for chemical biology, materials science and medicine[J]. Chem. Soc. Rev., 2014, 43(10):3324-3341.
[2]Long Y, Zhou X, Xing D. An isothermal and sensitive nucleic acids assay by target sequence recycled rolling circle amplification[J]. Biosens. Bioelectron.,2013,46:102-107.
[3]Jin G, Wang C, Yang L,etal.. Hyperbranched rolling circle amplification based electrochemiluminescence aptasensor for ultrasensitive detection of thrombin[J]. Biosens. Bioelectron. 2015,63:166-171.
[4]Chen J, Tong P, Lin Y,etal.. Highly sensitive fluorescent sensor for mercury based on hyperbranched rolling circle amplification[J]. Analyst, 2015, 140(3): 907-911.
[5]Zou B J, Ma Y J, Wu H P,etal.. Signal amplification by rolling circle amplification on universal flaps yielded from target-specific invasive reaction[J]. Analyst,2012,137(3):729-734.
[6]张慧慧.新型滚换扩增技术的研究[D].上海:上海大学,硕士学位论文,2014.
[7]王勤涛. 双切口滚环扩增检测方法及原理初探[D].上海:上海海洋大学,硕士学位论文,2013.
[8]Song W, Zhang Q, Sun W. Ultrasensitive detection of nucleic acids by template enhanced hybridization followed by rolling circle amplification and catalytic hairpin assembly[J].Chem. Commun.,2015,51(12):2392-2395.
[9]王星宇.基于靶序列环化的新型滚换扩增方法的建立及其应用[D].青岛:中国海洋大学,博士学位论文,2014.
[10]孟兆祥,张 伟,檀建新,等.一种DNA扩增的新技术:利用热稳定的Bst DNA聚合酶驱动跨越式滚环等温扩增反应[J].中国生物化学与分子生物学报,2013,29(9):892-898.
[11]Ge J, Zhang L L, Liu S J,etal.. A highly sensitive target-primed rolling circle amplification (tprca) method for fluorescent in situ hybridization detection of microrna in tumor cells[J]. Anal. Chem.,2014,86(3):1808-1815.
[12]Chen H, Wu S, Dong F,etal.. A novel chemiluminescence immunoassay for highly sensitive and specific detection of protein using rolling circle amplification and the multiplex binding system[J]. Sens. Actuat. B: Chem.,2015,221:328-333.
[13]Li Y, Zeng Y, Ji X,etal.. Cascade signal amplification for sensitive detection of cancer cell based on self-assembly of DNA scaffold and rolling circle amplification[J]. Sens. Actuat. B: Chem.,2012,171-172:361-366.
[14]Xu W, Xie X, Li D,etal.. Ultrasensitive colorimetric DNA detection using a combination of rolling circle amplification and nicking endonuclease-assisted nanoparticle amplification (NEANA)[J]. Small,2012,8(12):1846-1850.
[15]Larsson C, Grundberg I, Soderberg O,etal.. In situ detection and genotyping of individual mRNA molecules[J].Nat. Methods,2010,7(5):395-397.
[16]Zhu X, Shen Y, Cao J,etal.. Detection of microRNA SNPs with ultrahigh specificity by using reduced graphene oxide-assisted rolling circle amplification[J].Chem. Commun.,2015, 51:10002-10005.
[17]Deng R, Tang L, Tian Q, etal.. Toehold-initiated rolling circle amplification for visualizing individual microRNAs in Situ in single cells[J].Angewandte Chem. Int. Edit.,2014, 53(9):2389-2393.
[18]Liu H, Li L, Duan L,etal.. High specific and ultrasensitive isothermal detection of microrna by padlock probe-based exponential rolling circle amplification[J].Anal. Chem.,2013,85(16): 7941-7947.
[19]Göransson J, Ke R, Nong R Y,etal.. Rapid identification of bio-molecules applied for detection of biosecurity agents using rolling circle amplification[J].PLoS ONE,2012,7(2):e31068.
[20]Lu L, Liu B, Zhao Z,etal.. Ultrasensitive electrochemical immunosensor for HE4 based on rolling circle amplification[J].Biosens. Bioelectron.,2012,33(1):216-221.
[21]Miller J C, zhou H, Kwekel J,etal.. Antibody microarray profiling of human prostate cancer sera:antibody screening and identification of potential biomarkers[J]. Proteomics, 2003,3(1):56-63.
[22]Zhou H, Bouwman K, Schotanus M,etal.. Two-color,rolling circle amplification on antibody microarrays for sensitive,multiplexed serunrprotein measurements[J].Genome Biol.,2004,5(4): 28.
[23]He P, Liu L, Qiao W,etal.. Ultrasensitive detection of thrombin using surface plasmon resonance and quartz crystal microbalance sensors by aptamer-based rolling circle amplification and nanoparticle signal enhancement[J]. Chem. Commun., 2014, 50(12): 1481-1484.
[24]Ou L J, Liu S J, Chu X,etal.. DNA encapsulating liposome based rolling circle amplification immunoassay as a versatile platform for ultrasensitive detection of protein[J].Anal. Chem., 2009,81(23):9664-9673.
[25]Cheng W, Yan F, Ding L,etal.. Cascade signal amplification strategy for subattomolar protein detection by rolling circle amplification and quantum dots tagging[J].Anal. Chem.,2010, 82(8):3337-3342.
[26]Brasino M, Cha J N. Isothermal rolling circle amplification of virus genomes for rapid antigen detection and typing[J].Analyst,2015,140:5138-5144.
[27]Bi S, Zhao T, Luo B,etal.. Hybridization chain reaction-based branched rolling circle amplification for chemiluminescence detection of DNA methylation[J].Chem. Commun.,2013,49(61): 6906-6908.
[28]Rockett R, Barraclough K A, Isbel N M,etal.. Specific rolling circle amplification of low-copy human polyomaviruses BKV, HPyV6, HPyV7, TSPyV, and STLPyV[J]. J. Virol. Methods, 2015,215:17-21.
[29]Wang G, Maher E, Brennan C,etal.. DNA amplification method tolerant to sample degradation [J]. Genome Res.,2010,14:2357-366.
[30]Niel C, Diniz-Mendes L, Devalle S. Rolling-circle amplification of Torque teno virus (TTV) complete genomes from human and swine sera and identification of a novel swine TTV genogroup[J].J. General Virol,,2005,86(5):1343-1347.
[31]陶 震,蔡新锋,颜志强,等.HRCA技术在转基因植物检测中的的应用[J]. 生物工程学报,2003,19(3):294-299.
[32]Pang S, Qureshi F, Shanahan D. Investigation of the use of rolling circle amplification for the detection of GM food [J]. Eur. Food Res. Technol.,2007,225(1):59-66.
[33]Kobori T, Matsumoto A, Takahashi H,etal.. Rolling Circle Amplification for signal enhancement in ovalbumin detection[J]. Anal. Sci.,2009,25(12):1381-1383.
[34]Jacobi Z E, Li L, Liu J W. Visual detection of lead(II)using a label-free DNA-based sensor and its immobilization within a monolithic hydrogel[J]. Analyst,2012,137(3):704-709.
[35]Tang S, Tong P, Li H,etal.. Ultrasensitive electrochemical detection of Pb2+based on rolling circle amplification and quantum dotstagging[J].Biosens. Bioelectron.,2013,42:608-611.
[36]张 健.利用滚环扩增技术检测食源性致病微生物的研究与应用[D].青岛:中国海洋大学,硕士学位论文,2013.
[37]郝振明,赵 鑫,吴孝槐.超分支滚换扩增结合试纸法检测食品中多种转基因组分[J].食品科学,2010,31(6):263-266.
[38]Hamblin G D, Carneiro K M M, Fakhoury J F,etal.. Rolling circle amplification-templated DNA nanotubes show increased stability and cell penetration ability[J]. J. Am. Chem. Soc.,2012,134(6):2888-2891.
[39]Lin C, Wang X, Liu Y,etal.. Rolling circle enzymatic replication of a complex multi-crossover DNA nanostructure[J]. J. Am. Chem. Soc., 2007,129(46):14475-14481.
[40]Nangreave J, Lin C X, Liu Y,etal.. A replicable tetrahedral nanostructure self-assembled from a single DNA strand[J]. Am. Chem. Soc., 2009,131(36):13093-13098.
[41]Deng Z, Ribbe A E, Mao C,etal.. DNA-encoded self-assembly of gold nanoparticles into one-dimensional arrays[J]. Angew. Chem., 2005,117(23):3648-3651.
[42]Beyer S, Nickels P, Simmel F C. Periodic DNA nanotemplates synthesized by rolling circle amplification[J].Nano. Lett., 2005,5(4):719-722.
[43]Zhao W, Kandadai S A, Brook M A,etal.. DNA polymerization on gold nanoparticles through rolling circle amplification:Towards novel scaffolds for three-dimensional periodic Nnanoassemblies[J]. Angew. Chem. Int. Edit., 2006, 45(15):2409-2413.
[44]Cheglakov Z, Wilner O I, Winner I,etal.. Increasing the complexity of periodic protein nanostructures by the rolling-circle-amplified synthesis of aptamers[J]. Angew. Chem. Int. Edit.,2008,47(1):126-130.
[45]Wilner O I, Orbach R, Henning A,etal.. Self-assembly of DNA nanotubes with controllable diameters[J]. Nat. Commun.,2011,2:540.
[46]Hamblin G D, Fakhoury J F, Sleiman H F,etal.. Rolling circle amplification-templated DNA nanotubes show increased stability and cell penetration ability[J]. J. Am. Chem. Soc.,2012,134(6):2888-2891.
[47]Yoshimura T, Suzuki T, Mineki S,etal.. Controlled microwave heating accelerates rolling circle amplification[J].PloS ONE,2015,10(9):e0136532.
[48]Szemes M, Bonants P, Weeixh M,etal.. Diagnostic application of padlock probes-multiplex detection of plant pathogens using universal microarrays[J]. Nucl. Acids Res., 2005,33(8):701-704.
[49]Clausson C M, Arngården L, Ishaq O,etal.. Compaction of rolling circle amplification products increases signal integrity and signal-to-noise ratio[J]. Sci. Rep.,2015,5:12317.
[50]Beyer S, Nickels P, Simmel F C. Periodic DNA nanotemplates synthesized by rolling circle amplification[J].Nano. Lett.,2005,5(4):719-722.
[51]Kuhn H, Demidov V V, Frank-Kamenetskii M D. Topological links betweenduplex DNA and a circular DNA single strand[J]. Angew. Chem. Int. Edit.,1999,38(10):1446-1449.
Progress and Prospects of Rolling Circle Amplification
SUN Ya-jun, WANG Liang, CAI Jun*
KeyLaboratoryofFermentationEngineering,MinistryofEducation,EngineeringandTechnologyCollege,HubeiUniversityofTechnology,Wuhan430068,China
Abstract:The establishment of rolling circle amplification simulates the principle of self-replication of the ring-shaped pathogen DNA through the rolling circle model in nature, experiencing long-term scientific research and practical application, many breakthroughs have been achieved. This paper made a comprehensive summary on the latest research in recent years on rolling circle amplification(RCA), including net RCA, padlock probe RCA, target circle RCA and stride RCA. The paper also discussed the problems in the process of rolling circle amplification. We mainly introduced the application of RCA in basic research, the practical detection, medical diagnosis and Nano-materials. At last, the expectation towards the development of isothermal amplification of nucleic acid was prospected.
Key words:RCA; nanomaterials; transgene; padlock probe
DOI:10.3969/j.issn.2095-2341.2016.02.09
作者简介:孙亚军,本科生,研究方向为生物工程。E-mail:sunyajun43@163.com。*通信作者:蔡俊,教授,博士生导师,主要从事微生物发酵工程研究。E-mail:caijun@mail.hbut.edu.cn
收稿日期:2015-11-11; 接受日期:2015-11-26
