基于互信息研究的乳腺癌与阿尔茨海默症的免疫系统发病机理探寻
2016-02-16牟晓阳
刘 芳 孔 薇* 牟晓阳
1(上海海事大学信息工程学院,上海 201306)2(美国罗文大学生物化学系,美国 新泽西 08028)
基于互信息研究的乳腺癌与阿尔茨海默症的免疫系统发病机理探寻
刘 芳1孔 薇1*牟晓阳2
1(上海海事大学信息工程学院,上海 201306)2(美国罗文大学生物化学系,美国 新泽西 08028)
近年来,越来越多的流行病学研究显示多种癌症与阿尔茨海默症(AD)呈现负相关,但分子生物学机制尚不明确。从基因信号传导及调控网络构建方面研究这种负相关,将对两种疾病致病机理的探寻起重要作用。选取乳腺癌(BC)与AD进行对比研究。考虑到传统特征基因提取方法注重单个基因在不同样本中的表达差异而忽视基因之间的关联性,从基因之间的关联性出发,利用互信息(MI)首先提取两种疾病中共有的差异表达基因作为特征基因。在此基础上,鉴于网络成分分析(NCA)约束条件较强、运行时间过长等局限性,采用快速网络成分分析算法(FastNCA),推演出特征基因中转录因子的表达活性及其对靶基因的调控强度,并分别构建两种疾病的转录调控网络。实验结果表明,转录因子POLR2E、RFC5、THOC4、FBXO22、KPNA1、MYST3、PTBP1等在两种疾病中表达活性及调控作用相反,如转录因子RFC5的表达活性从健康到BC患病过程中由0.269降低至0.077,而从健康到AD患病过程中则由-0.430升高至0.307。通过分子生物学分析可知,它们所影响的调控关系及生物过程对BC及AD的致病起着关键作用,对两种疾病之间呈现负相关性机制的探寻具有重要意义。
乳腺癌;阿尔茨海默症;互信息;快速网络成分分析;机体免疫
引言
近年来,越来越多的流行病学统计显示癌症与老年痴呆症特别是阿尔茨海默症(Alzheimer′s disease, AD)之间呈现负相关即癌症患者患AD的可能性较其他人大幅降低,反之亦然[1]。为了更清楚地了解癌症和AD之间负相关关系,很多学者就癌症和AD分别利用基因表达数据等进行了多方面的研究,研究结果显示,与癌症和AD相关的基因、通路等或许共同参与了一些与细胞增殖和凋亡有关的基本的生物学过程,但在两类疾病中却起着相反的作用[2]。已证实的Pin1通过扩大致癌基因信号通路促进恶性肿瘤的发生,Pin1表达降低会有损tau蛋白功能及淀粉样前体蛋白加工最终导致神经元纤维缠结及神经元退变从而促进AD的发生[2]。Wnt信号通路上调会阻止神经元退化,保证其活性更容易形成癌症,相反,抑制Wnt通路能使神经元死亡或消失促使AD的发生,在Wnt通路中关键分子的表现、基因组的多态性和扰动性决定其发展为癌症或是AD[2]。然而目前对于两种疾病在免疫过程中为什么会呈现负相关及其呈现负相关的根本原因还知之甚少,因在AD与女性雌激素及内分泌相关的癌症中这种负相关性更加明显[3]。为此本课题选择乳腺癌(breast cancer, BC)与AD进行对比研究,通过提取与免疫系统紧密相关的显著特征基因,构建转录调控网络对它们的负相关关系及其致病机理进行探寻。
传统的基因提取算法往往只注重单个基因在两类样本中的表达差异而忽略了基因相互之间行为的复杂性,而熵和互信息(mutual information, MI) 则能抓住模式上的相似性,能有效处理基因间复杂关系。对于任意两条基因若互信息大说明二者在同一样本中共表达程度高,反之则弱。运用互信息算法[4]在基因表达数据中挖掘差异表达基因,就是搜索那些在健康样本与患病样本中互信息值相差很大的基因作为特征基因。另一方面,基因表达数据无法体现基因转录调控信息,考虑到快速网络成分分析(fast-network component analysis, FastNCA)算法[5-6]摒除了传统网络成分分析(network component analysis,NCA)算法不稳定、运行时间长等缺点,本研究将FastNCA算法用于共有转录因子,分别推演出两种疾病患病前后转录因子活性变化及其对靶基因调控强度的变化,并构建调控网络,结合分子生物学研究探讨BC和AD在免疫反应下负相关的原理,为它们致病机理的深入研究提供了有益的依据。
1 算法
1.1 互信息算法
在信息论中,随机变量出现的期望值或自信息的均值用熵来衡量,两个系统间的统计相关或一个系统能提供给另一个系统的信息量的多少是用互信息来衡量的。为了能有效抓住基因之间复杂的关联性,有效提取出复杂疾病的致病基因[7],采用互信息算法提取特征基因。设基因变量X=[x1,x2,…,xs]是一个基因表达模式,S表示基因表达数据中的样本的数量,基因变量X的熵即为该基因表达模式所包含的信息量,即
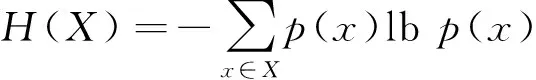
(1)
联合熵表示一对基因变量X和Y的不确定的度量,即
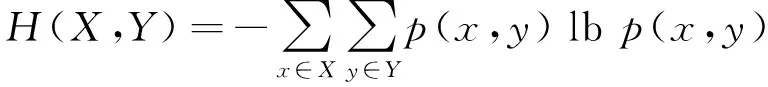
(2)
互信息表示对于两个随机变量X和Y,其中一个变量能提供给另一个变量的信息量,即
(3)
综上,可以得到基因变量X,Y之间的互信息,有
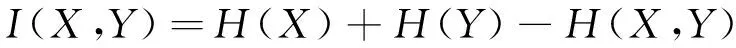
(4)
根据式(4)分析两个基因变量间的关联性,若I(X,Y)较大则表明基因变量X,Y之间相关性较大,可以认为它们在生物学上存在着较强的关联性,反之,若I(X,Y)为0或较小,则表明基因变量X,Y不相互包含任何信息,即在生物学上不存在关联性。
1.2 基于互信息算法的特征基因提取
由于基因变量的连续性,因此在计算基因变量的熵或互信息时,首先应对其离散化,本研究借助直方图方法[9]将基因表达数据全部离散化,分别求出各个基因的熵及基因之间的联合熵,最后根据式(4)计算基因变量之间的互信息。
基因表达数据微阵列E=(E(i,j))NS,N表示基因的数量,S表示数据样本数。E(i,j)表示第i条基因在第j个样本下的表达水平值,对于只包含健康对照样本和患病样本的情况,可将E分为健康对照样本Ec和患病样本Ea两部分,利用式(4)分别计算得出Ec和Ea中基因之间的互信息Ic和Ia,其中I为N×N的矩阵。I(i,j)是指第i条基因和第j条基因在两类样本中的互信息值,两条基因之间的互信息值越大,则说明二者在该样本中关联程度较高,反之关联程度较弱,基于互信息的性质,研究中最终选定了两类状态的基因做为特征基因:第1类是将在健康对照样本中互信息值较小的基因称其与其他基因失联,若该基因在患病样本中的互信息值较高则称其与其他基因关联。则认为此类基因在疾病产生过程中从失联状态变化为较高关联状态势必对疾病的产生具有重要的作用,因此将上述基因作为从失联到关联状态的特征基因;第2类相反,从关联到失联状态的基因,也将其选为特征基因。以第2类状态为例,求取从关联到失联状态的特征基因的提取方法。
选取两个阈值Tc和Ta其中Tc>Ta,并对Ic和Ia进行如下具体算法步骤:
(5)
(6)
(7)
(8)
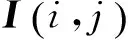
为了将所有基因表达数据值映射到[-1,1]区间,首先对数据进行了归一化,最后对BC的17个健康对照样本、45个无转移样本及AD的13个健康对照样本、10个患病样本分别通过式(1)计算每条基因的信息熵,并将信息熵应用于基因表达数据中,若某基因对应的信息熵值越大,则表明该基因在相应样本中含有的信息量越大,对分类贡献率越高。
1.3 快速网络成分分析算法
由于基因表达数据不能直接体现基因间转录调控信息,两种疾病所提取特征基因无法比较分析其转录调控功能和方向的异同,本研究将目前生物学上已知的TF-TG(transcription factor-target gene)调控关系作为先验知识,基于NCA构建转录调控网络。
NCA是一种用来分析转录网络基因表达数据的算法[8],其实质是根据基因表达数据和转录因子-靶基因调控关系的连通性网络,进而推演出TF活性及其对TG的调控强度,可表示为
(9)
式中,矩阵ENM表示N个基因在M个样本中的基因表达数据,矩阵CNL表示L个转录因子对N个靶基因的调控关系,矩阵PLM表示L个转录因子在M个样本下的表达活性。
由式(9)不难看出,要将基因表达矩阵E分解为调控矩阵C和活性矩阵P的解不是唯一的,需要分别给C和P矩阵添加更多的约束条件才能满足唯一解的需求,如矩阵C必须是列满秩矩阵P必须是行满秩,且矩阵C的每一列必须至少有L-1个0,当满足了上面的约束条件后,分解E矩阵最优解可以通过以下目标函数来求解,即
(10)

FastNCA算法包括3个主要步骤。
步骤1:对矩阵E利用奇异值分解,求得其秩为L的EYM逼近,即
(11)
式中,UL=W。
步骤2:估计调控矩阵C。对于i=1,2,…,M,将矩阵W进行重排,则调控矩阵C的第i列可以表示为
(12)
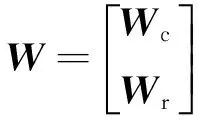
步骤3:估计转录因子活性矩阵P=C+EL。
此处规定L=M,从而获得更好的性能,与传统利用最小二乘法的NCA算法相比,利用奇异值分解的FastNCA算法,在算法的运行速度上明显比NCA有较大的提高,而且计算复杂度低,具有很高的稳定性。
1.4 实验数据
所使用的基因表达数据取自美国国家生物技术信息中心(National Center for Biotechnology Information, NCBI)基因表达数据库。乳腺癌数据使用的是GSE42568数据集,其121个样本包括17个健康对照样本、45个无转移患病样本(BCno)及59个腋下淋巴转移样本(BCmeta)。所使用的是17个健康对照样本和45个无转移患病样本(BCno)。阿尔茨海默症数据使用的是GSE5281数据集,其包含了161组不同大脑皮层组织样本的基因表达数据,所使用的是海马区(hippocampus,HIP)基因表达数据,它包括13个健康对照样本和10个患病样本。数据集GSE42568 和GSE5281中每个样本都含有54 675个探针数据。转录因子对靶基因的调控关系选用的是提供了人类4 105个转录因子对1 974个靶基因的调控关系的ITFP(integrated transcription factor platform, http://itfp.biosino.org/itfp/)平台。
2 结果
2.1 互信息提取特征基因
最终通过计算分别对上述4类样本挑选出满足上述条件的5 000条基因,以BC为例,对BC 分别计算在正常样本和患病样本5 000条基因的互信息值可得两互信息矩阵Ic和Ia,利用互信息提取特征基因的关键在于阈值Tc和Ta的选取,阈值选取不得当会造成得到的特征基因数过多或过少,都将直接影响后续的FastNCA算法构建调控网络。为了使结果具有生物学意义,阈值的选取可根据图中孤立点的比例或者图的密度来选择,关于孤立点及图的密度定义可参考文献[4]。具体阈值取值及得到的特征基因数部分实验数据如表1所示,经过多次实验得出,对BC而言从关联到失联较为理想的阈值为Tc=2.32和Ta=1.9,将BC、AD各自两类样本得到的两个互信息矩阵Ic和Ia分别进行式(5)~(8)处理。最终对于BC和AD分别得到两种状态对称矩阵I1和I2共4个互信息实对称矩阵。分别对上述4个实对称互信息矩阵行求和,并以和值所在行为基准进行降序排列,其中和值越大则表示该条基因在对应的样本中与许多基因相关联,反之和值为0的基因则表示该基因在对应样本中并不与其他基因有关联,依据上述步骤最终挖掘出BC两种状态下的基因分别为769条和923条、共计1 572条BC特征基因,同理对AD两种状态提取共计1 476条特征基因,对BC和AD两组实验得到的差异表达基因取交集,最后得到175条在两组数据集中都差异表达的显著基因。
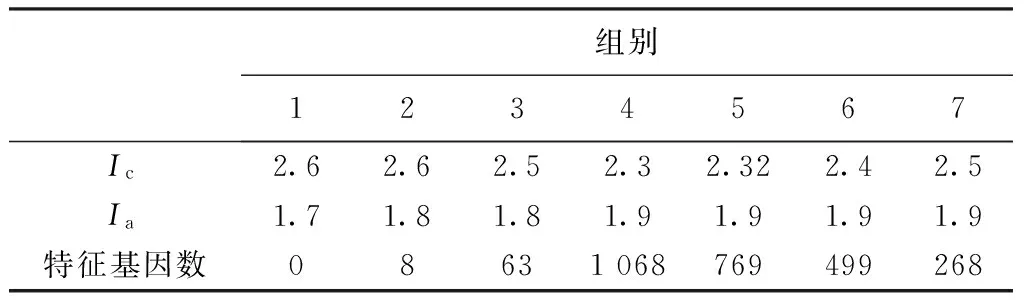
表1 阈值选取与特征基因提取Tab.1 Threshold selection and extracting feature gene
利用DAVID 方法(http://david.abcc.ncifcrf.gov/home.jsp),对上述175条差异表达显著的基因进行分析,得到的GO功能注释[10]的生物过程(BP)、细胞组分(CC)、分子功能(MF)和KEGG通路[11]分别如表2~5所示,这175个特征基因在BC和AD中表达差异显著。从表格中可以看出,上述表达差异显著的基因主要参与的生物过程有磷酸代谢过程、细胞凋亡、程序性细胞死亡等,其细胞组分主要是细胞器官腔、细胞骨架等,其分子功能主要是核苷酸结合、核糖核苷酸结合、蛋白质激酶活性及金属离子结合等。其中,细胞凋亡、程序性细胞死亡等是免疫反应的主要组成部分,它与许多疾病尤其是癌症和AD的发生发展密切相关。

表2 175个表达差异显著基因的GO功能注释(BP)Tab.2 175 significantly differentially expressed genes in GO functional annotation (BP)

表3 175个表达差异显著基因的GO功能注释(CC)Tab.3 175 significantly differentially expressed genes in GO functional annotation (CC)
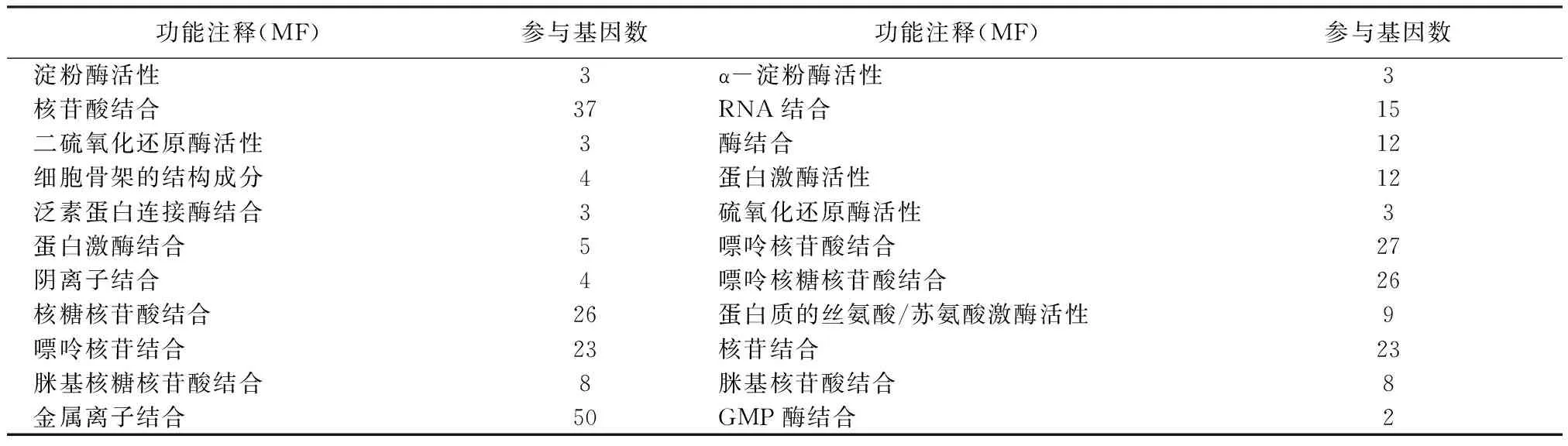
表4 175个表达差异显著基因的GO功能注释(MF)Tab.4 175 significantly differentially expressed genes in GO functional annotation (MF)
表5 175个表达差异显著基因参与的KEGG通路
Tab.5 175 significantly differentially expressed genes involved in the KEGG pathway
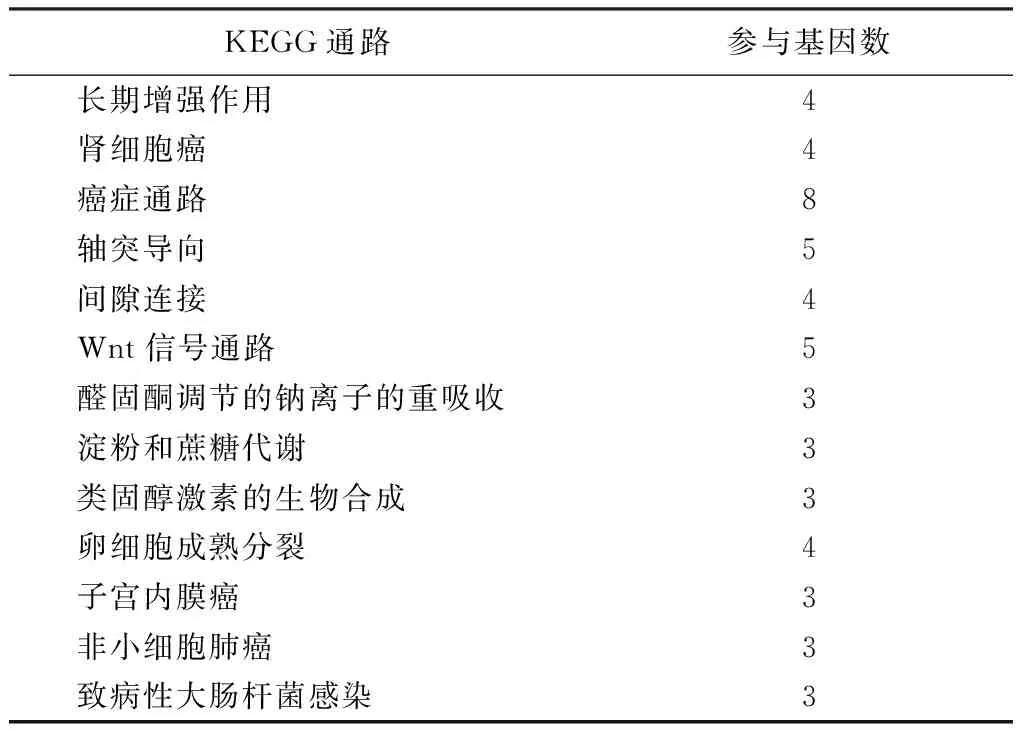
KEGG通路参与基因数长期增强作用4肾细胞癌4癌症通路8轴突导向5间隙连接4Wnt信号通路5醛固酮调节的钠离子的重吸收3淀粉和蔗糖代谢3类固醇激素的生物合成3卵细胞成熟分裂4子宫内膜癌3非小细胞肺癌3致病性大肠杆菌感染3
2.2 基于FastNCA构建转录调控网络
为了得到两种疾病中差异共表达基因中起主要作用的转录因子及其对靶基因的调控强度信息,首先将175个差异表达基因作为靶基因与转录库进行匹配,得到了520个TFs及其调控的87个TGs共1 004条调控关系。选取调控基因个数较多(≥6)的14个TFs及其调控的34个靶基因。为了能更形象具体地观察转录因子对靶基因的调控强度及其活性,现将FastNCA得到的结果利用Cytoscape软件(http://www.cytoscape.org/)分别构建BC、AD健康对照样本与其各自患病样本下的转录调控网络图。为了便于观察分析,上述两幅图中的基因都位于相同的位置,分别如图1、2所示。
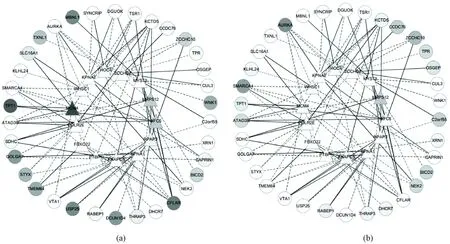
图1 BC样本转录调控网络图。(a)健康对照样本;(b)患病样本Fig.1 BC samples transcriptional regulatory network diagram.(a)Healthy control samples;(b)Diseased samples
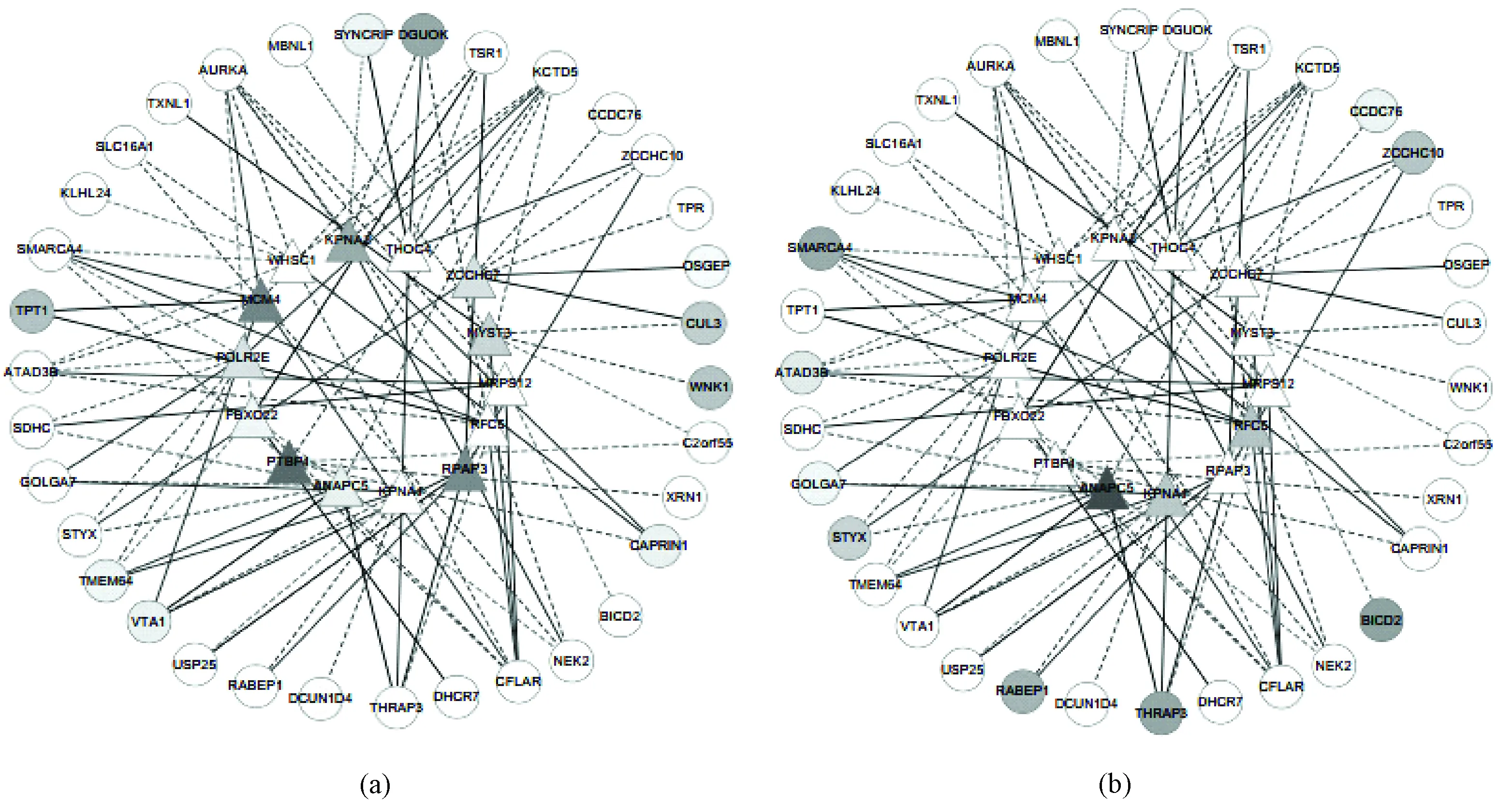
图2 AD样本转录调控网络图。 (a) 健康对照样本;(b)患病样本Fig.2 AD samples transcriptional regulatory network diagram.(a) Healthy control samples;(b) Diseased samples
在两幅图中,圆形节点代表靶基因,三角形节点代表转录因子。节点的白色和黑色分别表示表达水平或活性的上调和下调,颜色的深浅代表了表达水平或活性的高低,连线的实线和虚线分别表示转录因子对靶基因的调控强度的正负。从构建的转录调控网络图,可见转录因子和靶基因之间存在一对多及多对一的情况,充分印证了所选取互信息算法提取差异共表达基因的必要性。比较图1(a)、(b),可以明显看出在BC患病前后转录因子表达活性升高的有WHSC1、ANAPC5、POLR2E、THOC4、PTBP1、MRPS12、MYST3、FBXO22,转录因子表达活性降低的有MCM4、RFC5、RPAP3、KPNA1、KPNA2、ZCCHC7。同理,比较图2(a)、(b),AD患病前后的转录调控网络图中转录因子表达活性升高的有WHSC1、ANAPC5、RFC5、KPNA1、MRPS12,转录因子表达活性降低的有MCM4、RPAP3、 KPNA2、ZCCHC7、POLR2E、THOC4、FBXO22、MYST3、PTBP1。
将BC和AD共用的14个转录因子相对于各自的健康参照样本的转录活性变化趋势进行构图,具体如图3所示,图中横轴的1、2分别代表健康对照样本、患病样本。纵坐标代表转录因子活性的高低,其中圆形端点线段代表了BC的转录因子活性变化趋势,三角形端点的线段代表AD的转录因子活性变化趋势。
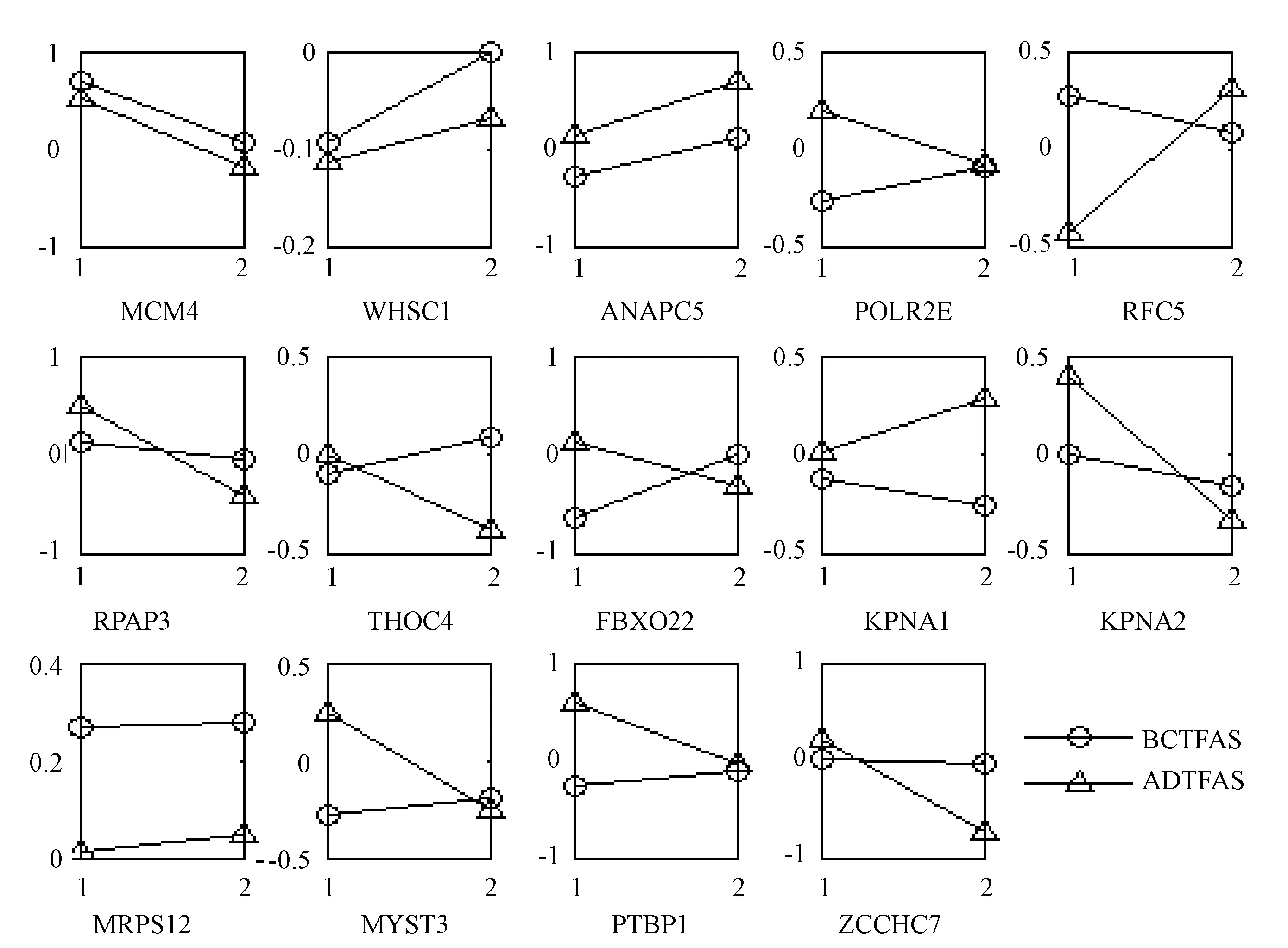
图3 BC与AD转录因子表达活性对比图(各子图中横坐标“1”和“2”分别表示健康对照样本和患病样本,纵坐标表示对应转录因子的表达活性,两者均为无量纲量)Fig.3 BC and AD transcription factor expression activity comparison chart(The ‘1’and ‘2’ in the abscissas of each sub-figure represents healthy and AD/BC sample respectively, and the ordinate denotes the activities of the corresponding TFs.Both of them are dimensionless variables)
从图3中可以清楚地看到,BC与AD转录因子表达活性相反的有POLR2E、RFC5、THOC4、FBXO22、KPNA1、MYST3、PTBP1,其余的7个转录因子的表达活性是一致的。
3 讨论
癌症与AD之间呈现负相关关系已在流行病学统计中越来越明显,但目前对于其发生的根本原因仍知之甚少。利用生物信息学方法进行两种疾病特征基因选择和信号传导通路分析是目前有针对性的有效分析方法之一。本课题选用互信息这一更能体现基因相关性的算法提取BC和AD共有的175条差异表达基因,利用GO数据库对提取的特征基因所参与的生物学过程及通路进行分类分析发现其参与的主要生物学过程有磷酸代谢过程、蛋白质氨基的磷酸化、调控细胞死亡、雌激素代谢过程、氧化还原及程序性细胞死亡,即在两种病中都包含的常见的生物学过程但作用效果却不同,如在细胞代谢过程中对BC而言补偿的代谢效应是糖酵解的上调即瓦尔堡效应,而对AD而言则是氧化磷酸化的上调与瓦尔堡效应相反[12]。
本课题提取出了癌症通路涉及的基因有APC、AR、CUL2、HGF、PRKCA、TPR、KRAS、CRK,轴突导向通路涉及的基因有EPHA4、CHP、EFNB2、RGS3、KRAS、类固醇激素的生物合成通路涉及的基因有AKR1C1、COMT、HSD17B1,参与Wnt通路的基因有APC、CHP、CSNK1A1、CSNK2A2、PRKCA,文献[2]已经证实Wnt信号通路上调会阻止神经元退化,保证其活性更容易形成癌症,相反,抑制Wnt通路能使神经元死亡或消失促使AD的发生。由此可见更深入的探寻BC和AD致病基因相关通路是研究两者呈现负相关根本原因的重要任务之一。
在信使RNA合成方面,本课题对所提取的转录因子POLR2E患病前后表达活性值进行计算可知,其在乳腺癌中的表达活性升高0.169而在AD中的活性降低0.27。目前已有研究表明POLR2E主要编码RNA聚合酶Ⅱ[13],RNA聚合酶Ⅱ被认为在负责信使RNA合成中作用最大,故POLR2E表达活性上调会间接导致mRNA合成量增多从而加速细胞的增殖。由图1、2可知转录因子RFC5调控靶基因SMARCA4且其表达活性从健康到BC患病过程中由0.269降低至0.077,而从健康到AD患病过程中则由-0.43升高至0.307。SMARCA4属于SWI / SNF的成员,已有研究表明SWI / SNF表达紊乱会导致BC和AD的发生[14-15]。THOC4基因的表达也对胚胎细胞起重要的作用且受ZFP206的调控[16]。Zhang 等的研究发现,ZFP206调控胚胎干细胞的增殖和分化,并在胚胎细胞增殖过程中过表达,这刚好符合图3中THOC4在BC和AD中的表达活性情况,即THOC4表达活性升高促进细胞增殖促使乳腺癌的形成同时降低了AD患病概率。在炎症反应方面,FBXO22、SKP1、GogB相互作用具有抗炎作用,GogB升高会抑制NF-κB,降低则会增加炎症反应。而许多研究均已证实炎症在AD的发病过程中扮演着重要的角色[17]。研究显示BIG3通过封锁KPNA1与PHB2(抗增殖蛋白)的链接区域达到抑制PHB2表达的效果,与此同时PHB2又和雌激素受体之间在细胞核中存在相互作用[18]。文献[3]则表明雌激素可以下调炎症基因的表达同时会增加乳腺癌的风险。研究表明p53功能性突变上调MYST53会促进肿瘤的生长。Sheikh等的研究表明,MOZ(MYST3, KAT6A)通过INK4A-ARF通路能够抑制衰老[19]。这正好与图3中MYST3作为BC和AD共有的转录因子表达活性相符,即在BC中表达活性升高了0.079同时在AD中表达活性降低了0.502。PTBP1与乳腺肿瘤的形成有关,并对肿瘤细胞的生长和保持改变属性是必不可少的,PTBP1表达水平的下降会导致PKM1表达的上调,同时PKM1会进一步促进氧化磷酸化并减少肿瘤的形成[20]。而Demetrius等的研究表明,在AD中存在氧化磷酸化的上调迹象[12]。由图3不难看出,该转录因子在BC中的表达活性上升而在AD中表达活性降低。综上所述本课题所提取的与BC和AD相关基因、转录因子及涉及的通路共同参与了细胞增殖和凋亡有关的基本的生物过程,但在两种疾病中却起着相反作用,这为生物学实验及有关BC和AD负相关致病机理的分析提供了坚实的依据和基础。
4 结论
鉴于BC和AD发病机制复杂且都与免疫系统有关,以及相关研究显示二者呈负相关,为了更进一步了解二者呈负相关的原因,本研究首先对BC和AD基因表达数据分别利用特征基因提取算法获取二者的特征基因并取其共有的特征基因。为了避免传统特征基因提取方法只注重单个基因在不同样本中的表达差异而忽视了基因之间的关联性,研究中选用互信息算法分别提取BC和AD的致病特征基因,然后通过FastNCA算法构建TF-TG转录调控网络,最后运用Cytoscape构图直观呈现TF对TG的调控强度及表达活性变化。在最后得到的在两种疾病中表达活性及调控作用呈相反关系的转录因子,包括POLR2E、RFC5、THOC4、FBXO22、KPNA1、MYST3、PTBP1等。通过分子生物学分析可知,它们所影响的调控关系及生物过程对BC及AD的致病起着关键作用。通过对调控网络的分析,也发现了很多BC和AD致病的共有基因,对两种疾病之间呈现负相关机制的探寻具有重要意义。
[1] Catalá-López F, Crespo-Facorro B, Vieta E, et al. Alzheimer′s disease and cancer: current epidemiological evidence for a mutual protection[J]. Neuroepidemiology, 2014, 42(2): 121-122.
[2] Driver JA. Inverse association between cancer and neurodegenerative disease: review of the epidemiologic and biological evidence[J]. Biogerontology, 2014, 15(6): 547-557.
[3] Realmuto S, Cinturino A, Arnao V, et al. Tumor diagnosis preceding Alzheimer’s disease onset: is there a link between cancer and Alzheimer’s disease?[J]. Journal of Alzheimer's Disease, 2012, 31(1): 177-182.
[4] 张焕萍,王惠南,卢光明,等. 基于互信息的差异共表达致病基因挖掘方法[J]. 东南大学学报: 自然科学版, 2009, 39(1): 151-155.
[5] Chang C, Ding Z, Hung YS, et al. Fast network component analysis for gene regulation networks[C]// 2007 IEEE Workshop on Machine Learning for Signal Processing. Thessaloniki:IEEE, 2007: 21-26.
[6] Chang C, Ding Z, Hung YS, et al. Fast network component analysis (FastNCA) for gene regulatory network reconstruction from microarray data[J]. Bioinformatics, 2008, 24(11): 1349-1358.
[7] 孙啸,陆祖宏,谢建明. 生物信息学基础[M]. 北京:清华大学出版社,2005.
[8] Liao JC, Boscolo R, Yang YL, et al. Network component analysis: reconstruction of regulatory signals in biological systems[J]. Proceedings of the National Academy of Sciences, 2003, 100(26): 15522-15527.
[9] Steuer R, Kurths J, Daub CO, et al. The mutual information: detecting and evaluating dependencies between variables[J]. Bioinformatics, 2002, 18(Suppl 2): S231-S240.
[10] Gene Ontology Consortium. The Gene Ontology (GO) database and informatics resource[J]. Nucleic Acids Research, 2004, 32(Suppl 1): D258-D261.
[11] Kanehisa M, Goto S. KEGG: Kyoto encyclopedia of genes and genomes[J]. Nucleic Acids Research, 2000, 28(1): 27-30.
[12] Demetrius LA, Simon DK. The inverse association of cancer and Alzheimer's: A bioenergetic mechanism[J]. Journal of the Royal Society Interface, 2013, 10(82): 20130006.
[13] Ye X, Xiao P, Hu X, et al. Crystallization and preliminary X-ray analysis of the RPB5 subunit of human RNA polymerase II[J]. Acta Crystallographica Section F: Structural Biology and Crystallization Communications, 2011, 67(11): 1391-1393.
[14] Reisman D, Glaros S, Thompson EA. The SWI/SNF complex and cancer[J]. Oncogene, 2009, 28(14): 1653-1668.
[15] Schirer Y, Malishkevich A, Ophir Y, et al. Novel marker for the onset of frontotemporal dementia: early increase in activity-dependent neuroprotective protein (ADNP) in the face of Tau mutation[J]. PloS ONE, 2014, 9(1): e87383.
[16] Zhang W, Walker E, Tamplin OJ, et al. Zfp206 regulates ES cell gene expression and differentiation[J]. Nucleic Acids Research, 2006, 34(17): 4780-4790.
[17] Pilar AVC, Reid-Yu SA, Cooper CA, et al. Active modification of host inflammation by Salmonella[J]. Gut microbes, 2013, 4(2): 140-145.
[18] Kim NH, Yoshimaru T, Chen YA, et al. BIG3 inhibits the estrogen-dependent nuclear translocation of PHB2 via multiple karyopherin-alpha proteins in breast cancer Cells[J]. PLoS ONE, 2015, 10(6):e0127707.
[19] Sheikh BN, Phipson B, El-Saafin F, et al. MOZ (MYST3, KAT6A) inhibits senescence via the INK4A-ARF pathway[J]. Oncogene, 2015,34(47): 5807-5820.
[20] He X, Arslan AD, Ho TT, et al. Involvement of polypyrimidine tract-binding protein (PTBP1) in maintaining breast cancer cell growth and malignant properties[J]. Oncogenesis, 2014, 3(1): e84.
Exploration of Pathogenesis in Immune System of Breast Cancer and Alzheimer′s Disease Based on Mutual Information
Liu Fang1Kong Wei1*Mou Xiaoyang2
1(InformationEngineeringCollege,ShanghaiMaritimeUniversity,Shanghai201306,China)2(DepartmentofChemistryandBiochemistry,RowanUniversity,NJ08028,USA)
In recent years, a growing number of epidemiological studies have shown that many kinds of cancer and Alzheimer′s disease have an inverse association, but the molecular biological mechanism remains unclear. Researching the inverse association from gene signal transduction and regulatory networks will play an important role in exploring the pathogenesis of both diseases. Breast cancer (BC) and AD were selected to be analyzed. Taking account of that the traditional genes extraction algorithms focused on a single gene expressed differently in different samples and ignored the links among the correlation genes, mutual information (MI) was utilized to extract the differentially expressed genes in the two diseases basing on the correlation among genes using as feature genes. In this paper, considering the limitation of network component analysis (NCA),such as the strong constraint conditions and the long running time, fast-network component analysis (FastNCA), improved by NCA,was brought up to get the activity of transcription factors among feature genes and TF′s regulate strength of target genes, and construct two diseases transcriptional regulatory networks, respectively. Experimental results showed that the activities and the regulate and control strength of TFs were totally opposite in the two diseases, for example POLR2E, RFC5, THOC4, FBXO22, KPNA1, MYST3 and PTBP1, for example, transcription factors RFC5 activities in BC decreased from 0.269 to 0.077, and in AD increased by -0.430 to 0.307. According to the experiment and analysis of molecular biology, the regulate relationship and the biological process influence from these TFs play a vital role in BC and AD.
breast cancer; Alzheimer′s disease; mutual information; fast network component analysis; immunity
10.3969/j.issn.0258-8021. 2016. 03.006
2015-10-28, 录用日期:2016-03-21
国家自然科学基金(61271466);上海市教委科研创新项目(15ZZ079)
Q343.1
A
0258-8021(2016) 03-0292-09
*通信作者(Corresponding author), E-mail:weikong@shmtu.edu.cn
