调控因子SlyA和RfaH在E.coli K4合成果糖软骨素中的生理作用
2015-11-11杨爱华张震宇刘立明
杨爱华,刘 佳,张震宇,刘立明
(1.江南大学 食品科学与技术国家重点实验室,江苏 无锡 214122;2.江南大学 工业生物技术教育部重点实验室,江苏 无锡 214122)
调控因子SlyA和RfaH在E.coli K4合成果糖软骨素中的生理作用
杨爱华1,2,刘 佳1,2,张震宇1,刘立明1,2
(1.江南大学 食品科学与技术国家重点实验室,江苏 无锡 214122;2.江南大学 工业生物技术教育部重点实验室,江苏 无锡 214122)
利用基因工程手段过量表达E.coli K4荚膜多糖(K4CPS)合成途径中相关代谢调控因子SlyA和RfaH,以构建高效合成K4CPS的菌株。结果发现:slyA和rfaH基因的单独过量表达和共表达重组菌的K4CPS的产量较对照菌分别提高了81.8%、46.5%和153.8%;甘油比消耗速率较对照菌降低了12.5%、18.8%和31.3%,而乙酸含量则较对照菌降低了98.6%、39.7%和91.6%。因此,调控蛋白SlyA和RfaH的过量表达能够大幅度提高E.coli K4的K4CPS合成,并增强菌体对甘油的利用率,而乙酸合成明显降低。而与单独表达策略相比,共表达策略能够更加有效地增强菌体合成K4CPS的能力。
硫酸软骨素;E.coli K4;K4CPS;转录调控蛋白
硫酸软骨素(chondroitin sulfate,CS)是重要的糖胺聚糖产品之一,目前主要被药用于骨关节炎症状的治疗,此外还具有抗寄生虫和病毒感染等功能,在再生医学以及抗肿瘤药物的开发等方面潜在的应用价值同样受到广泛关注[1-2]。CS还可用作膳食补充剂和保湿剂[3]。
多种细菌可以合成类硫酸软骨素的荚膜多糖,这使基于微生物发酵法生产CS成为可能。果糖软骨素是Escherichia coli O5∶K4∶H4荚膜中的糖胺聚糖,其结构式为(GlcUA(Fru)-1,3-GalNAc-1,4)(GlcUA代表葡萄糖醛酸,GalNAc代表乙酰半乳糖胺,Fru代表果糖),具有与 CS类似的多糖骨架[4-5]。目前,已利用E.coli K4发酵与化学修饰相结合的方法已成功合成了包括CS-A和CS-C的多种CS[6]。
K4CPS属于大肠杆菌Group 2荚膜类型,由包含3个功能区(region 1、regioin 2和region 3)的基因簇控制合成[7]。其中,region 1为单独转录单元,由位于region 1上游的σ70启动子(PR1)控制转录[8]。region 3和region 2共同形成一个转录单元,由启动子PR3控制转录[9]。从PR3方向的转录过程需要反终止因子RfaH的参与[10]。该蛋白与PR3下游非翻译区(UTR)713 bp的ops序列结合,从而保证从启动子PR3起始,经过region 2的转录过程正常进行[11]。基因rfaH的突变或ops序列的缺失,将导致荚膜合成能力的丧失,而rfaH的过量表达能有效促进PR3方向的转录[9,12]。基因簇的转录表达调控系统复杂而严密,除反终止因子RfaH以外,多种调控蛋白也参与其中,以调控蛋白SlyA最为重要。SlyA的表达受到温度的控制,与蛋白H-NS共同参与调控基因簇在37℃正常转录,而20℃的转录抑制。但SlyA的表达并不依赖于H-NS蛋白。20℃时,SlyA的表达量较低,主要为H-NS蛋白结合在启动子区域,抑制了基因簇的转录。在37℃条件下,SlyA的表达量上升,与H-NS形成SlyA-DNAH-NS复合物激活从PR1和PR3的转录[7,11,13-14]。可见,调控蛋白SlyA对荚膜多糖合成基因簇具有明显的正调控作用。荚膜多糖合成基因簇中包括编码单糖合成及转运、多糖的聚合、多糖跨膜转运等功能蛋白的多个基因,K4CPS的合成需要以上基因的协同表达。
因此,本研究中,基于荚膜多糖合成基因簇的转录调控因子SlyA和RfaH对基因簇转录调控的重要作用,克隆slyA和rfaH基因,构建2株单表达重组菌E.coli pTH-slyA、E.coli pTH-rfaH,以研究SlyA和RfaH对K4CPS合成的影响,并构建共表达重组菌E.coli pTHslyA-rfaH,增强特别是从启动子PR3方向的基因簇的转录水平,以构建高效的K4CPS合成菌株。
1 材料与方法
1.1 菌株与载体
PMD 19-T-simple,购于TaKaRa公司;pTrcHis A载体,购于Invitrogen公司;大肠杆菌JM109保存于笔者所在实验室,大肠杆菌K4(Escherichia coli O5∶K4∶H4),购于American Type Culture Collection,保藏编号为ATCC23502。
1.2 主要试剂和仪器
限制性内切酶BamH I、Kpn I、Hind III、T4 DNA连接酶、Ex Taq DNA聚合酶购于TaKaRa公司;质粒提取试剂盒、胶回收试剂盒、异丙基硫代-β-D-半乳糖苷(IPTG)购于上海生工生物工程公司;细菌基因组提取试剂盒购于北京天根生化科技有限公司;PCR仪、全自动凝胶成像系统购于美国Bio-Rad公司;UltiMate 3000型高效液相色谱仪,美国Dionex公司。
1.3 培养基
菌株构建过程中所用培养基和种子培养基均为LB培养基。
发酵培养基组成[15](g/L):甘油20,大豆蛋白胨1,K2HPO49.7,KH2PO42,(NH4)2SO43,柠檬酸三钠0.5,MgCl20.1。
1.4 实验方法
1.4.1 引物的设计和slyA和rfaH基因的扩增
根据Genbank中收录的 E.coli K4的 slyA和rfaH基因序列,设计特异扩增引物,如表1所示。引物合成由上海翰宇生物科技有限公司完成。将E.coli K4用LB培养基在37℃、200r/min培养过夜,收集菌体细胞,提取其基因组。以该基因组DNA为模板,使用slyA up和slyA down1扩增出用于单表达的slyA片段,使用slyA up和slyA down2扩增出用于共表达的slyA片段,使用rfaH up1和rfaH dowm扩增出用于单表达的rfaH片段,使用rfaH up2和rfaH down扩增出用于共表达的rfaH基因片段。PCR产物经过胶回收后,与pMD19-T simple在16℃下连接过夜。链接产物转化至E.coli JM109感受态细胞。筛选阳性转化子,提取质粒,经酶切验证,得到阳性质粒T-slyA1、T-slyA2、T-rfaH1和T-rfaH2,送至上海生工生物工程有限公司测序。

表1 本研究中所使用的引物Table 1 PCR primers used in this study
1.4.2 重组质粒和工程菌的构建
用限制性内切酶BamH I和Hind III分别对表达载体pTrcHis A和质粒T-slyA1进行双酶切,胶回收pTrcHis A线性质粒和目标基因,将表达载体和目标基因片段相连,得到单表达重组质粒pTH-slyA。用限制性内切酶BamH I和Hind III分别对表达载体pTrcHis A和质粒T-rfaH1进行双酶切,胶回收pTrcHis A线性质粒和目标基因,将表达载体和目标基因片段相连,得到单表达重组质粒pTH-rfaH。
用限制性内切酶BamH I和Kpn I分别对表达载体pTrcHis A和质粒T-slyA2进行双酶切,胶回收pTrcHis A线性质粒和目标基因片段。将表达载体和目标基因片段相连,得到重组质粒pTH-slyA。该质粒经Kpn I和Hind III酶切后胶回收。胶回收的片段与T-rfaH2的Kpn I和Hind III酶切片段相连,得到共表达重组质粒pTH-slyA-rfaH。共表达载体同其单表达载体一样,均由Trc启动子启动,且每个基因带有独立和相同的核糖体结合位点。将上述构建的表达载体pTH-slyA、pTH-rfaH和pTH-slyA-rfaH转入E.coli K4感受态细胞,在加有氨苄的LB培养基上培养过夜,挑取阳性转化子,酶切及PCR验证。
1.4.3 重组大肠杆菌的诱导表达及鉴定
将选定的重组大肠杆菌接入含有氨苄抗生素的LB液体培养基中,37℃、200r/min振荡培养过夜。种子液按10%的接种量接至含有氨苄抗生素的发酵培养基中,培养至600nm处的吸光值(OD600)达到0.6时,加入IPTG诱导剂至终浓度0.4 mmol/L,37℃诱导培养。细胞总蛋白和胞内组分经十二烷基硫酸钠-聚丙烯酰氨胶凝电泳(SDS-PAGE)检测。
1.4.4 K4CPS含量检测
发酵液8 000r/min离心5min,收集上清液,上清液用3倍体积的无水乙醇沉淀多糖,12 000r/min离心10min,所得沉淀用于K4CPS含量的测定。K4CPS含量测定用硫酸咔唑法[16]。
1.4.5 甘油和乙酸浓度的测定
用高效液相色谱(HPLC)仪检测发酵液上清中甘油和乙酸含量。色谱柱为Aminex HPX-87H色谱柱(美国Bio-Rad公司);流动相为5 mmol/L H2SO4,流速0.6mL/min;RID检测器。
2 结果与讨论
2.1 重组菌的构建和表达
以E.coli K4基因组为模板,利用PCR扩增出目的片段slyA和rfaH并连接至pMD19-T simple vector。获得重组质粒T-slyA和T-rfaH,且基因的测序结果表明,slyA和rfaH的基因序列与NCBI登记序列完全相同,长度分别为441和489 bp。按照1.4.2节中的方法构建重组表达质粒 pTH-slyA、pTH-rfaH和pTH-slyA-rfaH,经BamH I和Hind III双酶切验证,酶切片段大小与理论值相符,证明重组质粒构建成功,质粒的构建和验证结果如图1所示。
将质粒pTH-slyA、pTH-rfaH和pTH-slyA-rfaH分别导入E.coli K4中得到重组菌E.coli pTH-slyA、E.coli pTH-rfaH和E.coli pTH-slyA-rfaH。经IPTG诱导表达,菌体破碎后的离心上清进行SDSPAGE分析,结果如图2所示。由图2可知:重组菌E.coli pTH-slyA、E.coli pTH-rfaH和E.coli pTH-slyA-rfaH均有明显的目的蛋白表达条带,且蛋白条带的大小均与预期相符(蛋白SlyA和RfaH大小分别约1.617×104和1.793×104)。表明基因slyA和rfaH均在E.coli K4中得到了过表达。
2.2 调控因子SlyA和RfaH过量表达对菌体生长的影响
摇瓶发酵比较各重组菌与出发菌E.coli K4的生长情况,结果如图3所示。由图3可知:重组菌E.coli pTH-slyA和E.coli pTH-slyA-rfaH延滞期明显延长,分别在3、6 h进入对数生长期。而E.coli K4几乎没有延滞期,在8 h时便可达到稳定期。重组菌在对数生长期的最大比生长速率(0.42、0.32 h-1)也分别低于E.coli K4的69.1%和76.5%。虽然不同菌株细胞比生长速率各不相同,但至发酵结束时,3株菌E.coli K4、E.coli pTH-slyA和E.coli pTH-slyA-rfaH的OD600基本一致。而E.coli pTH-rfaH尽管初始生长状态良好,与E.coli K4同时进入稳定期,但它的最大菌体密度仅有(10.98±0.16)g/L,分别比E.coli K4、E.coli pTH-slyA和E.coli pTH-slyA-rfaH降低了18.0%、10.0%和18.1%。因此,调控蛋白SlyA的过表达明显抑制了菌体的生长,而RfaH的过量表达对菌体的比生长速率影响较小。
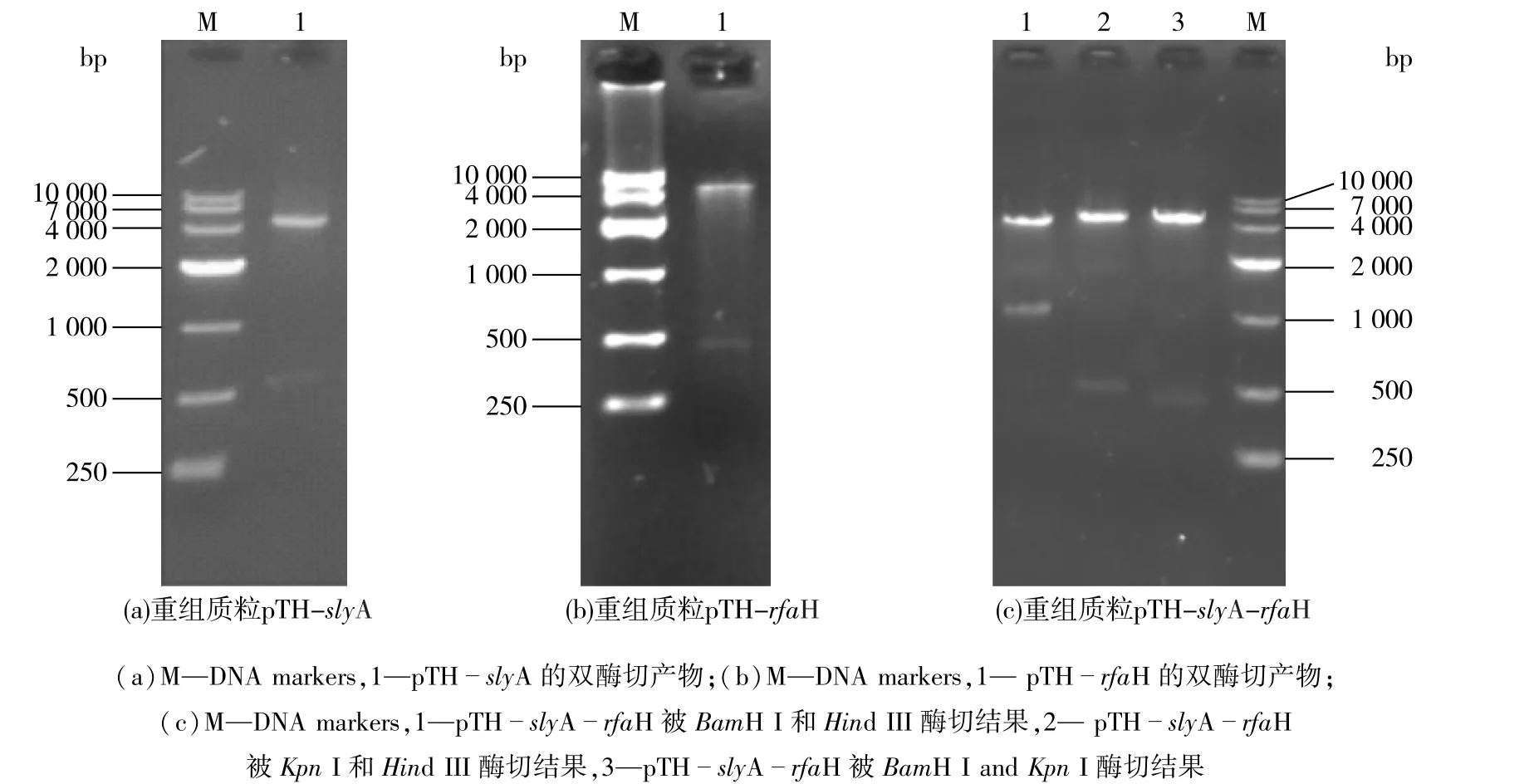
图1 表达载体的构建与验证Fig.1 Construction and confirmation of recombinant plasmid

图2 重组菌蛋白诱导表达SDS-PAGE分析结果Fig.2 SDS-PAGE analysis of expression proteins in three recombinant stains
2.3 重组菌的甘油消耗和乙酸合成分析
图4为菌株的甘油消耗和乙酸合成分析。从图4中可以看出:4株菌消耗甘油的规律基本一致,在菌体的对数生长期,甘油快速消耗。但至发酵结束,E.coli K4培养基中的甘油质量浓度为(3.12±0.35)g/L,而重组菌 E.coli pTH-rfaH、E.coli pTH-slyA和E.coli pTH-slyA-rfaH仅有(1.29±0.1)、(0.40±0.01)和(0.31±0.03)g/L,比E.coli K4甘油消耗得更为彻底,细胞产生乙酸的含量也有较大变化。重组菌在代谢过程中产生的乙酸量明显比E.coli K4降低。乙酸是E.coli K4发酵过程中产生的主要有机酸副产物(乙酸含量至少占总有机酸含量的80%)[17]。一般而言,乙酸在E.coli有氧发酵条件下的产生与2种因素有关[18]:①培养基内溶解氧浓度较低;②底物摄取与转化成生物量及产物之间不平衡,使得乙酰-CoA由三羧酸循环(TCA)转向乙酸合成。在培养环境一致的条件下,重组菌 E.coli pTH-rfaH、E.coli pTH-slyA和E.coli pTH-slyA-rfaH平均甘油比消耗速率(0.14、0.13和 0.11 h-1)比E.coli K4(0.16 h-1)分别将低了12.5%、18.8%和31.3%。较低的甘油比消耗速率,不利于乙酸的合成。乙酸含量比对照菌降低了 98.6%、39.7%和91.6%。另一方面,培养基内较低的乙酸含量也使得甘油可以持续被菌体消耗。

图3 重组菌与出发菌的细胞生长情况比较Fig.3 Comparision of cell growth(in term of OD600)between E.coli K4 and recombinant stains
2.4 重组菌合成K4CPS性能的分析
对重组菌合成K4CPS性能进行分析,结果如图5所示。从图5可以看出:重组菌产K4CPS量较出发菌均有提高。单独过量表达slyA与rfaH基因分别使K4CPS积累量达到(1 000±44)和(806±58)mg/L,较E.coli K4的(550±34)mg/L提高了81.8%和46.5%;而共表达slyA和rfaH基因的重组菌K4CPS的产量高达(1 396±55)mg/L,较出发菌株提高了153.8%。K4CPS的平均比合成速率分别为6.42、6.30和8.86 h-1,比E.coli K4(5.10 h-1)提高了25.9%、23.5%和73.7%。且K4CPS对单位菌体的得率由100 mg/g分别提高至 199、178和 253 mg/g,提高幅度高达99.0%、78.0%和 153.0%,表明调控因子 SlyA和RfaH的过量表达能够大幅度增强菌体的K4CPS合成能力。另外,与 E.coli K4相比,重组菌 E.coli pTH-slyA、E.coli pTH-rfaH、E.coli pTH-slyA-rfaH的K4CPS对甘油的得率也分别提高了54.5%、30.3%和115.1%。结合2.3节中重组菌消耗了更多的甘油而产生很少量的乙酸,说明调控蛋白SlyA和RfaH的过表达有效激活了K4CPS合成路径,减少了副产物的产生,使甘油更多地转化为产物。

图4 重组菌与出发菌甘油消耗及乙酸产生情况比较Fig.4 Comparision of glycerol consumed and acetate acid production in E.coli K4 and recombinant strains

图5 基因slyA和rfaH的过量表达对K4CPS积累情况的影响Fig.5 Effects of overexpression of slyA and rfaH genes on K4CPS accumulation
将重组菌和出发菌实验数据进行整理,得到的发酵过程参数如表2所示。表2说明调控因子基因的过量表达有效地提高了K4CPS的积累量,且共表达策略能够更加有效地激活荚膜多糖合成基因簇的表达,从而将碳代谢流转入荚膜多糖合成途径。

表2 对照菌株与工程菌株发酵参数比较Table 2 Comparion of fermentation parameters between wild type and recominant stains
3 结论
基因slyA不仅能够加强K4CPS的合成能力,将K4CPS由(550±34)mg/L增加至(1 000±44)mg/L,提高了81.8%,还能一定程度上增强菌体对甘油的利用能力。反终止因子RfaH的过量表达则使K4CPS产量提高了46.5%。针对与单独表达slyA和rfaH基因明显促进K4CPS积累的实验结果,采用共表达策略使编码SlyA和RfaH调控蛋白的基因共同由Trc启动子启动,构建一个串联表达系统。结果发现,slyA和rfaH的共表达,能够大幅度提高K4CPS的产量,较E.coli K4和单表达slyA、rfaH的菌株分别提高了153.8%、81.8%和46.5%。本研究的实验结果表明,荚膜多糖合成基因簇的转录调控因子SlyA和RfaH的过量表达可明显促进K4CPS的合成。
[1]Volpi N.Therapeutic applications of glycosaminoglycans[J].Curr Med Chem,2006,13(15):1799-1810.
[2]何兆雄,金艳,张天民.硫酸软骨素的结构及其在骨关节炎治疗中的应用[J].中国药学杂志,2012,47(5):387-391.
[3]吴秋林,刘立明,陈坚.发酵法生产硫酸软骨素的研究进展[J].生物工程学报,2012,28(11):1281-1293.
[4]Manzoni M,Bergomi S,Molinari F,et al.Production and purification of an extracellularly produced K4 polysaccharide from Escherichia coli[J].Biotechnol Lett,1996,18(4):383-386.
[5]Rodriguez M L,Jann B,Jann K.Structure and serological characteristics of the capsular K4 antigen of Escherichia coli O5:K4:H4,a fructose-containing polysaccharide with a chondroitin backbone[J].Eur J Biochem,1988,177(1):117-124.
[6]Bedini E,de Castro C,de Rosa M,et al.A microbiologicalchemical strategy to produce chondroitin sulfate A,C[J].Angew Chem Int Ed Eng,2011,50(27):6160-6163.
[7]Whitfield C. Biosynthesis and assembly of capsular polysaccharides in Escherichia coli[J].Ann Rev Biochem,2006,75:39-68.
[8]Simpson D A,Hammarton T C,Roberts I S.Transcriptional organization and regulation of expression of region 1 of the Escherichia coli K5 capsule gene cluster[J].J Bacteriol,1996,178(22):6466-6474.
[9]Stevens M P,Clarke B R,Roberts I S.Regulation of the Escherichia coliK5 capsule gene clusterby transcription antitermination[J].Mol Microbiol,1997,24(5):1001-1012.
[10]Stevens M P,Hanfling P,Jann B,et al.Regulation of Escherichia coli K5 capsular polysaccharide expression:evidence for involvement of RfaH in the expression of group II capsules[J]. FEMS Microbiol Lett,1994,124(1):93-98.
[11]Xue P,Corbett D,Goldrick M,et al.Regulation of expression of the region 3 promoter of the Escherichia coli K5 capsule gene cluster involves H-NS,SlyA,and a large 5'untranslated region[J].J Bacteriol,2009,191(6):1838-1846.
[12]Cimini D,de Rosa M,Carlino E,et al.Homologous overexpression of rfaH in E.coli K4 improves the production of chondroitin-like capsular polysaccharide[J].Microb Cell Fact,2013,12:1-12.
[13]Corbett D,Bennett H J,Askar H,et al.SlyA and H-NS regulate transcription of the Escherichia coli K5 capsule gene cluster,and expression of slyA in Escherichia coli is temperature-dependent,positively autoregulated,and independent of H-NS[J].J Biol Chem,2007,282(46):33326-33335.
[14]Rowe S,Hodson N,Griffiths G,et al.Regulation of the Escherichia coli K5 capsule gene cluster:evidence for the roles of H-NS,BipA,and integration host factor in regulation of group 2 capsule gene clusters in pathogenic E.coli[J].J Bacteriol,2000,182(10):2741-2745.
[15]Wu Q,Yang A,Zou W,et al.Transcriptional engineering of Escherichia coli K4 for fructosylated chondroitin production[J]. Biotechnol Prog,2013,29(5):1140-1149.
[16]Bitter T,Muir H M.A modified uronic acid carbazole reaction[J]. Anal Biochem,1962,4(4):330-334.
[17]Cimini D,Restaino O F,Catapano A,et al.Production of capsular polysaccharide from Escherichia coli K4 for biotechnological applications[J].Appl Microbiol Biotechnol,2010,85(6):1779-1787.
[18]de Mey M,de Maeseneire S,Soetaert W,et al.Minimizing acetate formation in E.coli fermentations[J].J Ind Microbiol Biotechnol,2007,34(11):689-700.
(责任编辑 周晓薇)
Effects of overexpression of transcriptional regulators SlyA and RfaH on fructosylated chondroitin production in E.coli K4
YANG Aihua1,2,LIU Jia1,2,ZHANG Zhenyu1,LIU Liming1,2
(1.State Key Laboratory of Food Science and Technology,Jiangnan University,Wuxi 214122,China;2.Key Laboratory of Industrial Biotechnology of the Ministry of Education,Jiangnan University,Wuxi 214122,China)
To enhance K4CPS production of E.coli K4,the transcriptional factor SlyA and RfaH were over expressed in E.coli K4 to create E.coli pTH-slyA,E.coli pTH-rfaH and co-expression strain E.coli pTH-slyA-rfaH.The over-expression of SlyA and RfaH significantly enhanced K4CPS production.The K4CPS concentration for E.coli pTH-slyA,E.coli pTH-rfaH and E.coli pTH-slyA-rfaH was increased by 81.8%、46.5%and 153.8%,compared with that of E.coli K4,respectively.The glycerol consumption rate of recombinant strain decreased by 12.5%、18.8%and 31.3%than that of E.coli K4.Moreover,the concentration of acetic acid decreased by 98.6%、39.7%and 91.6%compared with that of E.coli K4.The overexpression of slyA and rfaH could significantly enhanced K4CPS production.
chondroitin sulfate;E.coli K4;K4CPS;transcriptional regulator
TQ929.2
A
1672-3678(2015)05-0014-06
10.3969/j.issn.1672-3678.2015.05.003
2014-03-13
江苏省科技支撑计划(BE2010617);中组部青年拔尖人才支撑计划;江苏省杰出青年基金(BK2010002)
杨爱华(1988—),女,山东聊城人,硕士研究生,研究方向:生物工程;刘立明(联系人),教授,mingll@jiangnan.edu.cn;张震宇(联系人),教授,zhangzy@jiangnan.edu.cn
